Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.21 no.3 Lima July/set. 2004
TRABAJOS ORIGINALES
Variabilidad genética de Aedes aegypti en algunas áreas del Perú usando Single Stranded Conformational Polymorphism (SSCP)
Nélida Leiva G 1; Omar Cáceres R 2
1 Escuela de Biología, Facultad de Ciencias Naturales y Matemáticas, Universidad Nacional Federico Villarreal. Lima, Perú.
2 Laboratorio de Biotecnología y Biología Molecular, Centro Nacional de Salud Pública. Instituto Nacional de Salud. Lima, Perú.
RESUMEN
Aedes aegypti es el vector responsable de la transmisión del virus del dengue, su distribución geográfica se ha ampliado rápidamente debido principalmente a la intervención de los seres humanos. Objetivo: Analizar la variabilidad genética de este mosquito mediante la comparación del Segundo Espaciador Transcrito Interno (ITS 2) perteneciente al ADN ribosomal (rADN). Materiales y Métodos: Se analizaron muestras de ocho localidades (Jaén, Tingo María, Iquitos, Lambayeque, el distrito de El Rimac, Sullana y Zarumilla) y uno de la provincia de Huaquillas (Ecuador). El análisis de la variabilidad se determinó usando la técnica conocida como SSCP (Single Stranded Conformation Polymorphism). Resultados: El estudio muestra que existe variabilidad genética entre las poblaciones analizadas, principalmente entre las muestras localizadas en la costa del Perú (Zarumilla, El Rímac, Sullana) y Huaquillas y las muestras del nororiente (Tingo María, Iquitos, Jaén y Lambayeque) Conclusión: Se determinaron dos variantes genéticas entre las poblaciones de Aedes aegypti: Costeña y Nororiental, que probablemente provienen de dos ancestros diferentes y cuyo ancestro común sufrió de aislamiento por distancia. Se observó que no existe relación entre las distancias genéticas y las distancias geográficas indicando que la migración de estas poblaciones es el resultado de la intervención de los seres humanos que diseminan al vector y no por la migración activa del mosquito. Se plantea el papel de la Cordillera de los Andes en la migración y separación de las poblaciones de Aedes.
Palabras clave: Aedes/genética; Variación (Genética); Polimorfismo Conformacional Retorcido-Simple(SSCP); Perú. (fuente: BIREME).
ABSTRACT
Aedes aegypti is the responsible vector for transmission of dengue fever virus, and its geographical distribution has been widely broadened, mainly because of human intervention. Objectives: To analyze the mosquito genetic variability comparing the Second Internal Trascribed Spacer (ITS-2) from the ribosomal DNA (rDNA). Materials and Methods: Samples from seven Peruvian sites (Jaen, Tingo Maria, Iquitos, Lambayeque, Rimac district in Lima, Sullana, and Zarumilla) and one site from Ecuador (Huaquillas Province) were assessed. Variability analysis was determined using the SSCP (Single-Stranded Conformational Polymorphism) technique. Results: The study shows that there are genetic variability between the analyzed populations, mainly between the samples from the Peruvian northern and central coast (Zarumilla, Sullana, and Rímac) as well as in the Ecuador sample (Huaquillas) and the samples from the northeastern region in Peru (Tingo Maria, Iquitos, Jaen, and Lambayeque). Conclusions: Two genetic variants were determined for Aedes aegypti populations: Coastal and northeastern, which probably come from different lineages, and whose common ancestor became isolated because of the distance. It was observed that there is no relationship between genetic distances and geographical distances, indicating that the migration of the mosquito populations is a consequence of human intervention disseminating the vector and not because of active mosquito migration. We propose that there is a role for the Andes Mountains with respect to Aedes populations migration and separation.
Key words: Aedes/genetic, Variation (Genetics), ITS 2, Single-Stranded Conformational Polymorphism (SSCP),Peru. (source: BIREME).
INTRODUCCIÓN
El dengue es una de las enfermedades virales más importantes en cuanto a la morbilidad y mortalidad que afecta a miles de millones de personas en más de 100 países del mundo, es endémica en la mayoría de las regiones tropicales y subtropicales 1. En los últimos 50 años la incidencia ha aumentado 30 veces, se ha estimado que en el mundo existen 2500 millones de personas que viven en zonas de riesgo, se calcula que cada año se infectan entre 50 y 100 millones de personas por dengue, de los cuales entre 250 mil y 500 mil casos son dengue hemorrágico (DH) con por lo menos 22 000 de muertes, afectando sobre todo a niños menores de 15 años 1,2. Se estima que en los siguientes 10 años, el ambiente y la sociedad determinarán que el riesgo de transmisión del dengue continúe expandiéndose, y otro billón de personas estará en riesgo de adquirir la enfermedad, como producto de la intensificación del proceso de urbanización y de los cambios en el clima global y local 3,4.
Debido a que hasta hoy no se encuentran disponibles ni una vacuna y ningún tratamiento específico contra esta enfermedad, la prevención del dengue depende enteramente del control de su vector Aedes aegypti, el cual transmite el virus a través del ciclo mosquito-hombre- mosquito 1,5,6.
Aedes aegypti es considerado como uno de los vectores más eficientes en la transmisión del dengue debido a que es altamente antropofílico, vive y se reproduce dentro de las casas 1,5. Este mosquito ha expandido rápidamente su distribución geográfica debido a la falta de atención a los programas de control y erradicación, al rápido crecimiento y urbanización de las poblaciones humanas que originan el aumento de las vías y medios de transporte 1,5.
En nuestro país, Ae. aegypti fue detectado por primera vez en el año 1852, este mosquito habría ingresado por la frontera norte desde la región de Guayaquil (Ecuador), extendiéndose progresivamente a lo largo de la costa, incluyendo a Lima 7. En 1934, se determinó que 191 localidades de 11 departamentos estaban infestadas con Ae. aegypti iniciándose en 1938, las primeras campañas de control antiaédica 8. En 1948 se implementó el programa de erradicación y en 1958 se anunció la erradicación de Ae. aegypti como resultado de la intensa campaña de control ejecutada por el Ministerio de Salud con el apoyo de la Organización Panamericana de la Salud (OPS)7.
Sin embargo, una reinfestación de Ae. aegypti fue detectado en Loreto en 1984, de donde se expandió hacia San Martín y Huánuco. En los siguientes años su presencia se reportó en la región oriental, la selva central y la región norte del país 7. En el año 2000 Ae. aegypti ya había infestado 111 distritos del país (repartidos en 12 departamentos) encontrándose incluso en Lambayeque, La Libertad, Ancash y Lima 8.
En julio de 2001 Sevilla A et al.7 reportaron la presencia de Ae. aegypti en la ciudad de Lima (distrito del Rímac), señalaron además que hasta esa fecha este mosquito había sido colectado en al menos 15 de los 24 departamentos del país. Por último, en el año 2002, se notificó su presencia en los distritos de Ate Vitarte y Puente Piedra (Lima)9, convirtiéndose en un riesgo potencial para la salud pública.
La diseminación del dengue puede ser directamente atribuida a Ae. aegypti el cual se ha distribuido ampliamente en todo el mundo. Según Aitken T et al.10 la divergencia genética de las especies con frecuencia está asociada a la ampliación geográfica de ellos, dando origen a la aparición de poblaciones alopátricas genéticamente divergentes. Asimismo, estos autores señalan que en el caso de este mosquito, la divergencia genética podría afectar o incluso mejorar su capacidad de transmitir el virus dengue.
La capacidad vectorial puede expresarse de manera diferente entre cepas de mosquitos o entre especies cercanamente relacionadas, ya que se sabe que dicha capacidad se determina genéticamente en poblaciones diferentes. Esta característica hace que Ae. aegypti sea una especie compleja en el sentido de que presenta variaciones morfológicas, fisiológicas y de conducta mucho mayores que la mayoría de otros insectos 11.
Una forma de analizar la variación genética es mediante la comparación de las secuencias del ADN ribosomal (rADN), y más específicamente las regiones que corresponden a los Espaciadores Transcritos Internos o Internal Transcribed Spacer (ITS por sus siglas en inglés), los cuales son adecuados para realizar estudios filogenéticos debido al relativo grado de conservación entre los componentes de un rADN en distintas poblaciones del vector 12-14.
Entre las herramientas moleculares actualmente disponibles se tiene el Single Stranded Conformational Polymorphism (SSCP), método que es ampliamente usado en el análisis de mutaciones, estructuras genéticas y genética de poblaciones debido a su extrema sensibilidad y poder de resolución 15-22. Esta técnica se basa en la movilidad electroforética diferencial de moléculas de ADN de cadena simple 16, es decir cualquier mutación que afecte las interacciones dentro de una secuencia de nucleótidos va a generar estructuras secundarias diferentes que alterarán su movilidad durante la electroforesis. Así, moléculas diferentes (distinta secuencia de nucleótidos) van a tener diferentes movilidades electroforéticas, mientras que moléculas idénticas (misma secuencia de nucleótidos) tendrán movilidades idénticas 17.
Por tanto, el objetivo del presente estudio fue determinar las variantes genéticas de Ae. aegypti que están circulando en algunas localidades del país a través de la comparación del Segundo Espaciador Transcrito Interno (ITS 2) haciendo uso del SSCP.
MATERIALES Y MÉTODOS
MATERIAL BIOLÓGICO
Se analizaron siete mosquitos Ae. aegypti por cada una de las zonas donde se realizó la colecta de este vector, dichas zonas fueron: Sullana (Piura), Zarumilla (Tumbes), Iquitos (Loreto), Lambayeque, Jaén (Cajamarca), Tingo María (Huánuco), San Juan de Amancaes del distrito de El Rímac (Lima) y la provincia de Huaquillas (Ecuador). Las muestras se conservaron en alcohol al 70%, luego fueron enviadas desde los laboratorios referenciales de dichos departamentos hasta los laboratorios centrales de entomología y biología molecular del Instituto Nacional de Salud. Para la identificación taxonómica de la especie los entomólogos siguieron las claves dicotómicas de Savage y Smith 23, Forattini 24 y Carpenter y La Casse 25.
SET DE OLIGONUCLEOTIDOS
Los oligonucleotidos o primers usados para la amplificación del ITS 2 fueron reportados por Wesson et al.14 y fueron sintetizados, a solicitud nuestra, por Integrated DNA Technologies, Inc. (USA). La secuencia de estos oligonucleotidos se detalla a continuación:
CP – P1A 3- TGGATCCTGTGAACTGCAGGACACAT
CP – P1B 5- GTGTCGACATGCTTAAATTTAGGGGGT
AISLAMIENTO DE ADN DE MOSQUITOS
El aislamiento del ADN se llevó a cabo usando el Kit Genomic Prep TM Cells and Tissue DNA Isolation Kit (Amersham Biosciences) siguiendo las instrucciones del fabricante. Brevemente, cada mosquito fue triturado individualmente en 150 µL de PBS 1X (pH 7) usando un microhomogeneizador descartable estéril, luego se agregó 200 µL de la solución de lisis helada al cual se le agregó 3 µL de proteinasa K (20 mg/mL) y se dejó incubando a 55 ºC en baño maría durante 12 horas. Luego se le agregó 3 µL de la solución de RNasa A (10 mg/mL) y se incubó a 37 ºC durante 30 minutos. A la solución se le agregó 200 µL de la solución precipitante de proteínas mezclándose por agitación luego los tubos fueron centrifugados a 14000 x g por 5 minutos. El ADN fue precipitado con 600 µL de isopropanol absoluto y lavado con 600 µL de etanol al 70%. Finalmente el ADN precipitado fue resuspendido con 50 µL de la solución de hidratación y se dejó a 4 ºC durante 12 h. Finalmente, la muestra se calentó a 65 ºC durante 1 hora para ayudar a que el ADN se disperse y se conservó a 4 ºC.
La concentración del ADN fue cuantificada usando un espectrofotómetro UV/Visible (Spectronic 21D) a 260 y 280 nm. Se asumió que para el ADN de doble hebra 1 D.O 260 es igual a 50 mg/mL de ADN. La pureza del ADN extraído se obtiene de la relación de D.O 260 nm/280 nm el cual debe ser mayor de 1,7 26.
AMPLIFICACIÓN DEL ITS2 POR LA REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
La optimización de los diversos parámetros necesarios para la estandarización del PCR fueron realizadas según lo descrito 27. La concentración final de la reacción de PCR fue de 1X para el buffer, 200 µM para la mezcla de nucleótidos, 0,28 µM para cada uno de los primers, 2,25 mM de MgCl 2, 20 ng de ADN y 1U de Taq Gold® ADN polimerasa (Applied Biosystems) para 25 mL de volumen final de reacción. El PCR fue realizado en un termociclador 2400 (Applied Biosystems) donde los ciclos de amplificación fueron: 1 ciclo de denaturación inicial a 95 ºC x 10 minutos; 30 ciclos de 95 ºC x 30 segundos; 60,8 ºC x 1 minuto y 72 ºC x 30 segundos y una extensión final de 72 ºC x 10 minutos.
Cinco microlitros de la reacción de PCR fueron colocados en un gel de agarosa al 1,5% en buffer TAE 1X junto con 250 ng de un marcador de peso molecular (100 pb DNA Ladder, Promega). El gel fue sometido a electroforesis horizontal a 100 V por 40 minutos, posteriormente fue teñido con una solución de 0,5 µg/mL de bromuro de etidio por 10 minutos, los productos de amplificación fueron primero visualizados en un transiluminador de luz UV (UVP) y luego fotografiados usando una cámara Polaroid® y películas Polapan 665 PN IS0 80/20º (Polaroid).
PURIFICACIÓN DEL PRODUCTO DE PCR DE ITS 2
Los productos de PCR fueron purificados a partir de gel de agarosa de bajo punto de fusión (LMP) (Promega), 25 µL del producto de PCR fue corrido en dicho gel a 70 V durante 60 minutos. Las bandas de ADN correspondientes al ITS 2 se extrajeron usando laminillas cubreobjetos estériles cortando las porciones de agarosa que contenían la banda amplificada, los cuales fueron colocados un tubos de 1,5 mL. El ADN fue liberado del gel utilizando el Kit QIAquick Gel Extraction (Qiagen) siguiendo el procedimiento descrito por el fabricante. Los productos de amplificación correspondientes al ITS 2 se almacenaron a –20 ºC y se usaron para el SSCP.
SSCP (SINGLE STRANDED CONFORMATIONAL POLYMORPHISM)
Los productos purificados del ITS 2 fueron sometidos a una electroforesis vertical en gel de poliacrilamida no denaturante 0,5X (Gel MDE, BioWhittaker Molecular Bioproducts). El gel se preparó mezclando 1,25 mL de Gel MDE 2X, 0,6 mL de buffer TBE 5X, 3,15 mL de agua, 7 µL de TEMED y 70 µL de APS al 10% y se dejó polimerizar por 30 minutos a temperatura ambiente. Se tomó 5 µL del producto purificado de cada localidad y se mezcló a razón de 1:1 con una solución denaturante (sucrosa 50%, NaOH 0,015 N, azul de bromofenol 0,25% y formamida 30%), las muestras fueron calentadas a 95 ºC durante 5 minutos e inmediatamente después fueron colocados en hielo. Esta solución denaturada fue colocada en el gel MDE ya preparado y sometido a electroforesis con buffer TBE 0,6X a 150 V por 2 h.
El gel fue teñido con nitrato de plata con algunas modificaciones 28. El gel fue fijado con 100 mL de ácido acético glacial al 10% durante 30 minutos luego lavado con agua destilada por tres veces durante 2 minutos cada vez. Después de eliminar el agua el gel se secó por 10 segundos, luego del cual se colocó en 100 mL de la solución de tinción (AgNO3 0,1% w/v y 150 µL de formaldehído 37%) durante 30 minutos. Posteriormente el gel fue lavado con agua destilada por 5 segundos luego del cual fue colocado en la solución de revelado (Na2CO3 3%, 150 µL de formaldehído 37% y 400 µL de tiosulfato de sodio pentahidratado 10 mg/mL), hasta la aparición de las bandas, en ese momento se agregó 100 mL de la solución de fijación fría hasta que el burbujeo generado se detuvo. Finalmente el gel fue colocado sobre papel filtro 3 MM y se secó en un secador de geles 583 Gel Dryer (BioRad) durante 30 minutos a 80 ºC.
DETERMINACIÓN DE LAS DISTANCIAS DE CORRIDA (RF)
Dado que los resultados del SSCP se expresan en índices de similitud o diferencia de las distancias alcanzadas por las cadenas simples de ADN durante la corrida electroforética, el procedimiento para obtener dicho índice fue el siguiente: Para cada mosquito se calculó de manera separada las distancias de corrida (Rf) alcanzadas por cada una de las dos bandas de cadena simple de cada ITS 2 amplificado (Rf1 y Rf2 ), luego se obtuvo un Rf promedio (Rfp ) para los 7 mosquitos de cada localidad analizada. El análisis estadístico de los resultados se realizó usando la prueba F de Fisher y la comparación de medias se hizo con el método de Tukey 29.
Finalmente, se determinó el porcentaje de similitud de los Rf promedio entre los departamentos analizados, es decir si siete mosquitos de un departamento obtenían el mismo Rf promedio que otros siete mosquitos de otro departamento, estos tenían 100% de similitud. Con estos resultados se construyó una matriz de similitud que sirvió para la construcción de un árbol filogenético utilizando el método de evolución mínima usando el software Mega Ver. 2,130, debido a que se estaba analizando la variación genética de un mismo locus dentro una misma especie 31.
MATRIZ DE DISTANCIAS GEOGRÁFICAS
Para la construcción de esta matriz, se tomaron en cuenta las distancias entre cada uno de los departamentos donde las muestras fueron colectadas, dichos datos fueron proporcionados por el Instituto Geográfico Nacional (IGN), y al igual que la matriz de similitud del SSCP, también se construyó un árbol de distancias entre los departamentos para relacionarlos.
RESULTADOS
AMPLIFICACIÓN Y PURIFICACIÓN DEL ITS 2
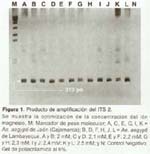
El tamaño del producto obtenido fue de aproximadamente 350 pb (Figura 1), la purificación se hizo necesaria debido a que fue difícil obtener una sola banda pues a pesar de que los primers usados habían sido diseñados a partir de las regiones conservadas del 18S y 28S para los extremos 3 y 5 respectivamente éstos no se unieron específicamente a la región deseada del ADN. Hay que mencionar que los primers usados (CP-P1A y CP-P1B) fueron diseñados para amplificar el ITS 2 de Ae. aegypti formosus 14 mientras que la subespecie circulante en nuestro país es Ae. aegypti aegypti. Una vez finalizada esta etapa se tuvieron en total, las regiones amplificadas y purificadas de 56 mosquitos.
DETERMINACIÓN DEL POLIMORFISMO DE Ae.aegypti MEDIANTE SSCP
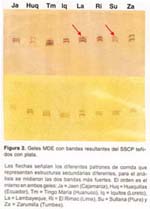
Para el cálculo de los Rf se tomaron en cuenta las dos bandas más fuertes, dejándose de lado aquellas bandas débiles que también se encontraron en la corrida (Figura 2). Los Rf obtenidos estuvieron dentro del rango de 1,5 a 2,8 cm siendo el promedio total igual a 2,17 cm (Tabla 1). El análisis estadístico no reveló diferencias significativas entre los Rfp de las muestras de una localidad con respecto al Rfp de otra (Fc = 0,171, Ft = 2,21; a = 0,05). Asimismo, el número de Tukey calculado fue de 0,695, y ninguno de los valores obtenidos en la comparación de los Rfp fueron estadísticamente significativos (p > 0,05).
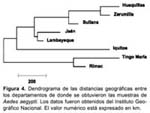
Por otro lado, la construcción de la matriz de similitud y diferencia con los Rf obtenidos (Tabla 2) dieron como resultado un árbol filogenético (construido bajo el modelo de evolución mínima) donde se encuentran dos clusters o variantes principales (Figura 3). La primera variante agrupa a Tingo María, Iquitos, Lambayeque y Jaén; y la segunda a Huaquillas (Ecuador), Zarumilla, al distrito de El Rímac y Sullana. Esta matriz arrojó una distancia promedio total entre las variantes igual a 71,4%.
Al analizar los perfiles de corrida dentro de las poblaciones (siete mosquitos por departamento) no se encontró ninguna diferencia, pues todas tenían las mismas distancias.
ANÁLISIS DE DISTANCIAS GEOGRÁFICAS
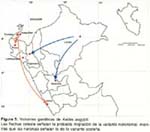
La relación geográfica entre las localidades estudiadas está representada en la Figura 4. Este dendograma muestra dos ramas principales, la primera de ellas a su vez está dividida en otras dos subramas, la primera agrupa a Huaquillas (Ecuador) y Zarumilla; Sullana, Jaén y Lambayeque en tres diferentes clusters mientras que la segunda subrama sólo tiene a Iquitos. Finalmente, la segunda rama principal sostiene a las localidades de Tingo Maria y El Rímac (Lima). Nótese que el árbol generado agrupa a las localidades del norte en un cluster y a las del centro en otro cluster.
DISCUSIÓN
ESPECIFICIDAD DEL PCR
Considerando el hecho de que los primers no fueran específicos para la especie y teniendo en cuenta que el genoma de un mosquito de Ae. aegypti cuenta con un promedio de 1200 copias de cistrones de rDNA ribosomal 32, fue muy difícil obtener una banda específica perteneciente al ITS 2. La aparición de bandas adicionales o inespecíficas (Figura 1) podrían ser resultado de la variación intragenómica de esta región, lo cual provoca que un genoma simple contenga parálogos divergentes 33, y en el caso específico de Ae. aegypti tal variación intragenómica origina una variación intraespecífica que alcanza el 1,17%14. Además, la aparición de dichas inespecificidades puede también ser explicadas por la aparición de problemas de unión entre los primers y los sitios de alineamiento 20. Es por esta razón que no quedó otra alternativa que purificar, a partir de gel de agarosa, el producto del PCR que posteriormente fue empleado para desarrollar los análisis del SSCP, ya que esta es una técnica que sobretodo exige pureza del producto 16.
SSCP
En algunas corridas se apreció la aparición de bandas adicionales a las dos esperadas (cada una correspondiente a cada cadena simple) (Figura 2). Las verdaderas causas de estas observaciones aún permanecen sin definir. Sin embargo, una hipótesis es que la cadena simple de ADN adopta una de un número reducido de conformaciones 21 los cuales producirían un número limitado de cambios de movilidad asociados a substituciones de bases simples. Una banda bastante marcada o condensada podría pertenecer a una conformación única predominante o a un grupo de conformaciones que se interconvierten rápidamente, mientras que la interconversión más lenta puede ser la explicación de la aparición de bandas dispersas. Sólo dos bandas a partir de un segmento implicaría que sólo existen dos conformaciones o dos grupos de conformaciones que no se interconvierten entre sí 21.
VARIABILIDAD GENÉTICA Y DISPERSIÓN DE Ae.aegypti
Según el análisis del SSCP, las poblaciones de Ae. aegypti estudiadas pueden ser divididas en dos grandes grupos o clusters o variantes genéticas principales el primero incluye a Iquitos, Jaén, Lambayeque y Tingo María; y el segundo incluye a Huaquillas (Ecuador), Zarumilla, Sullana y El Rímac (Lima) (Figura 3) notándose claramente dos poblaciones, una Costeña y otra Nororiental. Según De Sousa et al.34, el conocimiento del origen geográfico de los mosquitos introducidos puede ser muy útil para identificar los patrones potenciales o actuales del flujo de genes de los vectores que con el tiempo generarán polimorfismo genético. Por lo tanto, si se toma en cuenta el proceso de invasión de Ae. aegypti al Perú, primero en 1852 por Ecuador y luego en 1984 por Iquitos, puede asumirse que este vector provendría de dos ancestros diferentes cuyo ancestro común cambió a través del tiempo y estuvo sujeto a «aislamiento por distancia» al tomar dos rutas distintas (divergencia alopátrica) cuando ingreso al Perú, haciendo que ambas poblaciones (costa norte y nororiente) sean diferentes. Nuestros resultados demostraron que las divergencias genéticas eran independientes de las distancias geográficas (compárese las figuras 3 y 4), tal como fue determinado en varios estudios recientes donde se analizó la distancia intraespecífica entre poblaciones de Ae. aegypti, y no se halló ninguna correlación entre las distancias genéticas y geográficas 34-36.
Es sabido que el flujo de genes entre poblaciones disminuye con el aumento de las distancias geográficas, lo cual es conocido como aislamiento por distancia. Para el caso de Aedes aun no se ha determinado la distancia exacta del aislamiento genético, pero un estudio en Puerto Rico dice que a 100 km el flujo de genes sigue siendo continuo 11. En el caso de las poblaciones peruanas de Ae. aegypti, éstas están separadas por la Cordillera de los Andes, la cual es una barrera geográfica que no puede ser cruzada por los mosquitos, por eso las poblaciones del nororiente y la costa norte son diferentes.
El flujo de las poblaciones de Aedes definitivamente está relacionado con la migración del hombre, sus relaciones comerciales y el transporte. Esto se ve reflejado en Zarumilla, Sullana y la provincia ecuatoriana de Huaquillas, donde estas tres localidades comparten la misma población de mosquitos.
La población de Ae. aegypti de Tingo María, está más relacionada con l a población de Iquitos, lo que indicaría que la migración va de norte a sur; lo mismo sucede con la población presente en El Rímac, la cual está relacionada con Sullana, y a su vez está con la de Zarumilla (Figura 5). Sin embargo, no es posible hacer mayores presunciones con respecto a la migración de la población de El Rímac porque se careció de muestras pertenecientes a departamentos intermedios entre Lima y Piura a excepción de Lambayeque.
Con respecto del medioambiente, la diferenciación genética también puede atribuirse a la heterogeneidad del hábitat. Las poblaciones que viven en hábitats aislados o extremos pueden alterar su composición genética 36. Por esta razón las poblaciones pertenecientes a las áreas secas como la costa también son diferentes de aquellas poblaciones de áreas húmedas como Iquitos y Tingo María. Además, probable-164 mente ésta sea la razón por la cual las poblaciones de Aedes presentes en Lambayeque y Jaén, geográficamente muy cercanas, estén más relacionadas con la población nororiental, pues estos departamentos no comparten las mismas características climáticas que Piura o Tumbes donde el clima es cálido y muy seco, sino que más bien tienen un clima semicálido y húmedo para el caso de Lambayeque y templado húmedo para el caso de Jaén. Estudios posteriores donde se realizaron análisis de secuencias nucleotídicas han ubicado a la población de Lambayeque dentro de la variante costera (dato no mostrado que será publicado posteriormente).
En cuanto a la migración de Ae. aegypti, no se ha podido comprobar que el mosquito está migrando de norte a sur, pero la distribución de las poblaciones y la historia de infestación en nuestro país, indican que la dirección seguida por este mosquito es en esa dirección, dividida por la Cordillera de los Andes, originando la existencia de las variantes Costeña y Nororiental de Ae. aegypti en el Perú.
La migración y dispersión de este vector es principalmente producto de las migraciones humanas, transacciones comerciales y medios de transporte (migración pasiva)37, más que debido a la migración activa del mosquito. Además, como no existe relación entre las distancias genéticas de las poblaciones de Aedes estudiadas y las distancias geográficas de las localidades analizadas, entonces es muy probable que las barreras geográficas desempeñen un papel importante sobre el flujo de poblaciones y en nuestro país la Cordillera de los Andes estaría separando las dos variantes determinadas en este estudio.
No podemos descartar que los huevos o larvas pueden ser llevados por el hombre de un lugar a otro (antropocoria) y mezclarse con las poblaciones nativas, en nuestros estudios no hemos encontrado Aedes de la variante costeña en la región oriental y viceversa.
En este estudio el SSCP ha probado ser un método útil, barato, sencillo y rápido para la búsqueda y determinación de variantes genéticas de Ae. aegypti o cualquier otro insecto con cierta precisión, sin embargo como los datos del SSCP son obtenidos midiendo los Rf de las bandas, los valores muchas veces están sujetos a la interpretación subjetiva que dependen de la experiencia del investigador, así en nuestro estudio SSCP no fue capaz de discriminar fehacientemente las poblaciones de Aedes analizando las bandas generadas en los geles, debido a que al aplicar el análisis estadístico no hubo diferencias significativas entre las localidades (Ft > Fc), sin embargo cuando se aplico el análisis filogenético utilizando mínima evolución recién se logró determinar a las variantes de las poblaciones de Aedes. Una explicación posible es que el SSCP no tiene el suficiente poder discriminativo en poblaciones cuya distancia filogenética es muy corta o está en proceso de divergencia genética tal como esta sucediendo con las poblaciones de Aedes estudiadas por lo que es útil además utilizar otro tipo de análisis que apoyen al SSCP; pero en poblaciones cuya filogenia está bien establecida el método tiene un gran poder de discriminación.
Es la primera vez que se hace este tipo de estudio de polimorfismo genético de este vector en el Perú y debido a la importancia que representa para la salud pública era necesario conocer y tener un panorama de cómo son las poblaciones de Ae. aegypti en el país. Nuestros resultados nos llevaron a iniciar otros estudios más precisos utilizando marcadores genéticos más finos que abarquen todo el genoma con el fin de determinar con mayor precisión la variabilidad genética y las rutas de migración de este vector.
AGRADECIMIENTOS
Los autores agradecen al entomólogo Pablo Villaseca de la División de Entomología del Instituto Nacional de Salud por la identificación de los mosquitos analizados en este estudio al igual que al entomólogo Abraham Cáceres por proveernos de Aedes del distrito de El Rímac (Lima). Agradecemos también a los LRR de Tumbes, Piura II, Lambayeque, Cajamarca, Huánuco y Loreto por la captura y envío de los mosquitos usados en este estudio.
REFERENCIAS BIBLIOGRÁFICAS
1. Guzmán M, Kouri G. Dengue: an update. Lancet Infect Dis 2002; 2(1): 33-42. [ Links ]
2. World Health Organization. DengueNet – WHO´s internet- based system for the global surveillance of dengue fever and dengue haemorrhagic fever. Weekly Epidemiological Record. 2002; 77(36): 300-04. [ Links ]
3. World Health Organization. Scientific working group on dengue. Meeting Report, Geneva, Switzerland, 3-5 april 2000. Geneva: WHO; 2000. [ Links ]
4. Pinheiro FP, Chuit R. Emergence of dengue hemorrhagic fever in the Americas. Infect Med 1998; 15(4): 244-51. [ Links ]
5. Monath T P. Dengue: the risk to developed and developing countries. Proc Natl Acad Sci USA 1994; 91(7): 2395- 400. [ Links ]
6. Ravel S, Monteny N, Velasco Olmos D, Escalante J, Cuny G. A preliminary study of the population genetics of Aedes aegypti (Diptera: Culicidae) from Mexico using microsatellite and AFLP markers. Acta Trop 2001; 78(3): 241-50. [ Links ]
7. Andrade C, Cáceres A, Vaquerizo A, Ibáñez-Bernal S, Cachay L. Reappearance of Ae. aegypti (Díptera: Culicidae) in Lima, Peru. Mem Inst Oswaldo Cruz 2001; 96(5): 657-58. [ Links ]
8. Espejo LR. Prevalencia de criaderos urbanos e índices de infestación para Aedes aegypti y Culex pipiens en los distritos de Independencia y Comas. Lima: Ministerio de Salud, Dirección de Salud III Lima Norte AIS-SBS-COMAS; 2000. [ Links ]
9. Perú, Oficina General de Epidemiología, Ministerio de Salud. Situación del dengue en el departamento de Lima y en la provincia constitucional del Callao. Boletín Epidemiológico Semanal 2002; 11(39): 1-9. [ Links ]
10. Aitken T, Downes W, Shope R. Aedes aegypti strain fitness for yellow fever virus transmission. Am J Trop Med Hyg 1977, 26(5 Pt1): 985-89. [ Links ]
11. Tabachnick WJ, Powell J R. A world-wide survey of genetic variation in the yellow fever mosquito, Aedes aegypti. Genet Res 1979; 34(3): 215-29. [ Links ]
12. Fritz GN, Conn J, Cockburn A, Seawright J. Sequence analysis of the ribosomal DNA internal transcribed spacer 2 from populations of Anopheles nuneztovari (Diptera: Culicidae). Mol Biol Evol 1994; 11(3): 406-16. [ Links ]
13. Schlötterer C, Hauser M, von Haeseler A, Tautz D. Comparative evolutionary analysis of rDNA ITS regions in Drosophila. Mol Biol Evol 1994; 11(3): 513-22. [ Links ]
14. Wesson D, Porter C, Collins F. Sequence and secondary structure comparisons of ITS rDNA in mosquitoes (Diptera: Culicidae). Mol Phylogenet Evol 1992; 1(4): 253-69. [ Links ]
15. Hayashi K. PCR-SSCP: a simple and sensitive method for detection of mutations in the genomic DNA. PCR Methods Appl 1991; 1(1): 34-38. [ Links ]
16. Orita M, Iwahana H, Kanazawa H, Hayashi K, Sekiya T. Detection of polymorphisms of human DNA by gel electrophoresis as single-strand conformation polymorphisms. Proc Natl Acad Sci USA 1989; 86(8): 2766- 70. [ Links ]
17. Gening L, Klincheva S, Gusev A, Suroroy A, Potapov V. SSCP screening of individual aptamers. Biotechniques 2001; 31(4): 828-34. [ Links ]
18. Soto D, Sukumar S. Improved detection of mutations in the p53 gene in human tumors as single-stranded conformation polymorphs and double-stranded heteroduplex DNA. PCR Methods Appl 1992; 2(1): 96-98. [ Links ]
19. Fulton R, Salasek M, DuTeau N, Black WC 4 th. SSCP analysis of cDNA markers provides a dense linkage map of Aedes aegypti genome. Genetics 2001; 58(2): 715-26. [ Links ]
20. Kumeda Y, Asao T. Single-Strand Conformation Polymorphism Analysis of PCR-amplified ribosomal DNA Internal Transcribed Spacers to differentiate species of Aspergillus Section Flavi. Appl Environ Microbiol 1996 62(8): 2947-52. [ Links ]
21. Liu Q, Scaringe W, Sommer S. Discrete mobility of single-stranded DNA in non-denaturing gel electrophoresis. Nucleic Acids Res 2000; 28(4): 940-43. [ Links ]
22. Burt A, Carter DA, Koenig GL, White T, Taylor J.Molecular markers reveal cryptic sex in the human pathogen Coccidioides immitis. Proc Natl Acad Sci USA 1996; 93(2): 770-73. [ Links ]
23. Savage HM, Smith GC. Aedes albopictus y Aedes aegypti en las Américas: implicaciones para la transmisión de arbovirus e identificación de hembras adultasdañadas. Bol Oficina Sanit Panam 1995; 118 (6):473-78. [ Links ]
24. Forattini OP. Entomología Médica. Ed. Universidad de São Paulo. São Paulo: 1965. p. 416. [ Links ]
25. Carpenter SJ, LaCasse WJ. Mosquitoes of North America (North of Mexico). Berkeley: University of California Press; 1955. p. 360. [ Links ]
26. Sambrook J, Fritsch EF, Maniatis T. Molecular cloning: A laboratory manual. 2 nd Ed. New York: Cold Spring Harbor Laboratory Press 1989; Vol III. p. E5. [ Links ]
27. Saiki R, Gelfand D, Stoffel S, Scharff S, Higuchi R, Horn G, et al. Primer-directed enzymatic amplification of DNA with thermostable DNA polymerase. Science 1988; 239(4839): 487-91. [ Links ]
28. Bassam BJ, Caetano-Anolles G, Gresshoff PM. Fast and sensitive silver staining of DNA in polyacrylamide gels. Anal Biochem 1991; 196(1): 80-83. [ Links ]
29. Montgomery DC. Diseño y análisis de experimentos. Grupo Editorial Iberoamérica. Ciudad de México: 1991. p. 393-408. [ Links ]
30. Kumar S, Tamura K, Jakobsen IB, Nei M. MEGA2: Molecular Evolutionary Genetics Analysis software. Bioinformatics 2001; 17(12): 1244-45. [ Links ]
31. Rzhetsky A, Nei M. A simple method for estimating and testing minimum evolution trees. Mol Biol Evol 1992, 9(5): 945-67. [ Links ]
32. Park YJ, Fallon AM. Mosquito ribosomal RNA genes: Characterization of gene structure and evidence for changes in copy number during development. Insect Biochem 1990; 20(1): 1-11. [ Links ]
33. Harris DJ, Crandall KA. Intragenomic variation within ITS1 and ITS2 of freshwater crayfishes (Decapoda: Cambaridae): implications for phylogenetic and microsatellites studies. Mol Biol Evol 2000; 17(2): 284-91. [ Links ]
34. De Sousa G, Blanco A, Gardenal C. Genetic relationship among Aedes aegypti (Diptera: Culicidae) populations from Argentina using Random Amplified Polymorphic DNA polymerase chain reaction markers. J Med Entomol 2001; 38(3): 371-75. [ Links ]
35. Failloux AB, Darius H, Pasteur N. Genetic differentiation of Aedes aegypti, the vector of dengue virus in French Polynesia. J Am Mosq Control Assoc 1995; 11(4): 457-62. [ Links ]
36. Gorrochotegui-Escalante N, Munoz ML, Fernández-Salas I, Beaty BJ and Black WC 4 th. Genetic isolation by distance among Aedes aegypti populations along the northeastern coast of Mexico. Am J Trop Med Hyg 2000; 62(2): 200-09. [ Links ]
37. Nelson MJ. Aedes aegypti, biología y ecología. Washington DC: Organización Panamericana de la Salud; 1986. [ Links ]
Correspondencia:
Omar Cáceres Rey.
Laboratorio de Biotecnología y Biología Molecular, Centro Nacional de Salud Pública, Instituto Nacional de Salud.
Dirección: Cápac Yupanqui 1400. Lima 11, Perú.
Teléfono/Fax: (511) 4719920 anexo 129
Correo electrónico: ocaceres@ins.gob.pe