Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.22 n.1 Lima Ene./mar. 2005
TRABAJOS ORIGINALES
Perfiles genéticos (IS6110) y patrones de resistencia en aislamientos de M. tuberculosis de pacientes con tuberculosis pulmonar. Lima Sur, Perú* Luis Capcha A1; Martha Urbina B1; Lucy Vásquez C2; Luis Asencios S2; Neyda Quispe T2; Elena Leo H2; Christian Baldeviano V3; Amparo Zavaleta P4.
1 Laboratorio del Hospital María Auxiliadora. Lima, Perú.
2 Laboratorio de Referencia Nacional de Mycobacterias, Instituto Nacional de Salud. Lima, Perú.
3 Laboratorio de Biología Molecular, Instituto Nacional de Salud. Lima, Perú.
4 Departamento de Biología Molecular, Facultad de Farmacia y Bioquímica, Universidad Nacional Mayor de San Marcos. Lima, Perú.
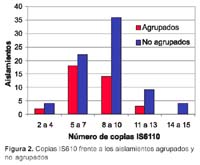
* Esta investigación contó con el apoyo técnico - financiero del Proyecto "Enfrentando las amenazas de las enfermedades infecciosas emergentes y reemergentes" Vigía (MINSA / USAID), en el marco del III CONCURSO PARA PROYECTOS DE INVESTIGACIÓN EN ENFERMEDADES INFECCIOSAS EMERGENTES Y REEMERGENTES – AÑO 2002. RESUMEN Objetivos: Conocer los perfiles genéticos de M. tuberculosis y determinar el patrón de resistencia a drogas en una población de sujetos infectados provenientes del sur de Lima mediante el marcador genético IS6110 (RFLP-IS6110). Materiales y Métodos: Entre octubre de 2002 y abril de 2003 se incluyeron pacientes mayores de 15 años con tuberculosis (TB) pulmonar frotis positivo procedentes de servicios de salud del distrito Villa María del Triunfo y del Hospital María Auxiliadora. Se realizó la prueba de sensibilidad a las cuatro drogas de primera línea rifampicina (RIF), isoniacida (INH), estreptomicina (SM) y etambutol (EMB) por el método de proporciones, y la genotipificación mediante el método estándar de RFLP-IS6110. Se recolectó información de los casos de los registros del establecimiento e historias clínicas. Resultados: De 118 aislamientos de M. tuberculosis se identificaron 97 perfiles genéticos variando entre 2 a 15 bandas por perfil. El 29,7% de los aislamientos dio origen a 14 grupos o clusters genéticos mientras que el resto mostró patrones variables de bandas. De otro lado, los perfiles de resistencia revelaron que cerca de 33% de los sujetos participantes nunca tratados presentaron resistencia a drogas y 58% de los tratados con anterioridad. La multidrogoresistencia fue de 8,42% y 36% en los nunca y anteriormente tratados respectivamente. Palabras clave: Tuberculosis; M. tuberculosis / genotipificación; Resistencia a drogas; Códigos genéticos; ABSTRACT Objectives: To know the genetic profile of M. tuberculosis, and to determine its drug resistance pattern in a population consisting in infected subjects from southern Lima using the IS610 genetic marker (RFLP-IS6110). Materials and Methods: Between october 2002 and april 2003 patients older than 15 years of age diagnosed with pulmonary tuberculosis (TB) by a positive sputum smear were included. Patients were recruited from health facilities in Villa Maria del Triunfo district and Maria Auxiliadora Hospital. Susceptibility testing for all first-line drugs was performed: rifampin (RIF), isoniazid (INH), streptomycin (SM), and ethambutol (EMB) using the proportion method, and genotyping was performed using the standard RFLP-IS6110 method. Information was collected from case registries in health facilities, as well as from clinical records. Results: Out of 118 M. tuberculosis isolates, 97 genetic profiles were identified, with between 2 to 15 bands per profile. 29.7% of isolates originated 14 genetic groups or clusters, while the others showed variable patterns in their bands. On te other hand, resistance profiles revealed that nearly 33% of participating subjects who never received any previous therapy had drug resistance, as well as 58% of those previously treated. Multidrug resistance was present in 8.42% and in 36% of those never before treated and those previously treated, respectively. Key words: Tuberculosis; M. tuberculosis/genotipification; Drug resístanse; Genetic code; Polymorphism, restriction fragment length; Molecular Epidemiology; Peru (source: DeCS BIREME). INTRODUCCIÓN A pesar de los esfuerzos mundiales para combatir la tuberculosis (TB), la enfermedad permanece como el mayor problema de salud pública en países industrializados y en países en vías de desarrollo como el Perú1,2. La enfermedad activa producida por Mycobacterium tuberculosis, se desarrolla después de una infección adquirida recientemente (primaria) o de la reactivación de una infección adquirida en el pasado (latente)3. El síndrome de inmunodeficiencia adquirida (SIDA) es el mayor factor de riesgo tanto para la reactivación de la infección de M. tuberculosis latente, como de enfermedad primaria en los países europeos y en Estados Unidos de América4,5. En los países en vías de desarrollo parece ser que el principal factor de riesgo son las condiciones económicas y sociales6,7. También se ha determinado que el diagnóstico tardío de la tuberculosis esta asociado con la mayor morbilidad y mortalidad debido al incremento de la carga vacilar en el individuo, los factores asociados con el diagnóstico tardío son: automedicación, percepción de un tiempo de espera prolongado, percepción de un costo elevado y desconocimiento de la existencia de programas de control de tuberculosis8. Otra de las grandes dificultades para el control de la TB es la emergencia de cepas multidrogoresistentes (MDR)9. Entre los factores claves para el control de la TB están la rápida detección y una adecuada terapia para detener una futura transmisión10. En el Perú, esta enfermedad presenta altas tasas de incidencia, en el año 2000 se notificó el diagnóstico de 39 918 casos de TB en todas sus formas, 58% de los cuáles fueron informados en Lima y Callao (incidencia anual de 133,60 x 100 000 habitantes). En el caso de la zona sur de Lima (donde se realiza el presente estudio), los datos epidemiológicos notificados por la Dirección de Salud de Lima Sur señalan que la incidencia de tuberculosis pulmonar frotis positivo (TBP-FP) es 125,8 x 100 000 habitantes; y los niveles de resistencia primaria y adquirida en el Perú son de 17,8 y 23,5%, respectivamente (1999). Además, existen algunas zonas de alta incidencia de tuberculosis en los distritos de San Juan de Miraflores y Villa María del Triunfo llamados "bolsones tuberculosos"11. Con el fin de entender mejor la dinámica de la transmisión y la patogénesis de la TB se han desarrollado en los últimos años diversas técnicas moleculares que han permitido caracterizar genéticamente aislamientos de M. tuberculosis. Uno de estos métodos es el análisis del polimorfismo de longitud de los fragmentos de restricción usando como sonda la secuencia de inserción IS6110 (RFLP-IS6110, por sus iniciales en inglés), convirtiéndose en una poderosa herramienta de la epidemiología molecular en diferentes países12-16. En nuestro país, los estudios que han usado esta herramienta molecular son escasos, uno de ellos, realizado en la provincia del Callao, encontró genotipos En ese sentido, el objetivo del presente estudio fue conocer los perfiles genéticos (frecuencia y agrupamientos) de M. tuberculosis en una zona del sur de Lima (distrito de Villa María del Triunfo) mediante el marcador genético IS6110 (RFLP-IS6110) así como determinar el patrón de resistencia a drogas en esta población. MATERIALES Y MÉTODOS DESCRIPCIÓN DEL ÁREA DEL ESTUDIO Villa María del Triunfo es un distrito ubicado en el cono sur de la ciudad de Lima, capital del Perú. Con una extensión de 70,57 km2, su área urbana ocupa sólo 21 km2, siendo el resto de su territorio zonas áridas. Este distrito se divide en 6 zonas (José Carlos Mariátegui, Cercado-capital del distrito-, Inca Pachacútec, Nueva Esperanza, Tablada de Lurín y Villa Poeta José Gálvez Barrenechea). Tiene una población de 330 348 habitantes, de los cuales 51% son mujeres y más de 60 % son jóvenes. Presenta un crecimiento caracterizado por una constante migración de población procedente principalmente de las provincias andinas del interior del país, y además tiene al 30% de su población en situación de extrema pobreza. POBLACIÓN Y MUESTRAS CLÍNICAS INCLUIDAS Entre el 1 de octubre de 2002 y el 30 de abril de 2003, se incluyeron a pacientes mayores de 15 años con diagnóstico de TB pulmonar mediante baciloscopía positiva, procedentes de diferentes servicios de salud del distrito Villa María del Triunfo y del Hospital María Auxiliadora (hospital nacional de referencia de la zona sur de Lima). Los casos incluidos fueron los "nunca tratados" y los "anteriormente tratados", las muestras clínicas de esputo fueron transportadas al laboratorio del Hospital María Auxiliadora, acompañada de la ficha clínico-epidemiológica estándar correctamente llenada (que incluye datos de filiación: edad, sexo, procedencia, antecedentes de tratamiento). Cuando fue necesario completar los datos de los pacientes, se revisó el libro de registros de baciloscopías y cultivos o las fichas de tratamiento del centro de salud, y en el caso de los pacientes del hospital se acudió a su historia clínica. Además, en el caso de los pacientes "agrupados", cuyas cepas de M. tuberculosis forman parte de un mismo cluster, se indagó por su relación epidemiológica (antecedentes de contacto con un paciente con TB, antecedentes de TB clínica, antecedentes de hospitalización previa en el hospital o en algún otro hospital, y antecedentes de tratamiento ambulatorio). CULTIVO Y PRUEBA DE SENSIBILIDAD A DROGAS Las muestras fueron recibidas para la realización de cultivo de M. tuberculosis, de acuerdo con los procedimientos estándares 18. De estos, sólo las muestras aisladas fueron transportadas al Laboratorio Nacional de Referencia de Mycobacterias (LNRM), Centro Nacional de Salud Pública, Instituto Nacional de Salud (INS) donde inicialmente se realizó el reaislamiento bacteriano y la prueba de susceptibilidad a drogas de primera línea. Estas muestras fueron las que finalmente se incluyeron para el estudio molecular. La prueba de sensibilidad a drogas de primera línea (rifampicina, RIF; isoniacida, INH; estreptomicina, SM y etambutol, EMB) fue realizada de acuerdo con el método convencional de las proporciones de Canetti, Rist y Grosset, en su variante económica 19. Adicionalmente, se consideraron algunos criterios básicos para sospechar de la presencia de mycobacterias no tuberculosas, como: observación de características de morfología y tamaño de colonias, presencia de ramificación, velocidad de crecimiento, coloración y temperatura de incubación 18. CARACTERIZACIÓN GENÉTICA MEDIANTE EL La caracterización genética de los aislamientos fue realizado en la División de Biología Molecular del INS. La cepa de referencia M. tuberculosis 14323, proporcionada por el LNRM, fue incluida en cada corrida de electroforesis como un control para el análisis de caracterización genética por el método de RFLP-IS6110. El método empleado consistió en la restricción enzimática o RFLP (de las siglas en inglés: Restriction Fragment Lenght Polimorphism) e hibridación de ADN con una sonda correspondiente a un fragmento de la secuencia de inserción IS6110. Esta secuencia de inserción está presente en número (0 a 20 copias) y posición variable en distintas cepas de M. tuberculosis, y esta forma genera un polimorfismo de bandas (perfil genético RFLP-IS6110). El procedimiento se realizó siguiendo el protocolo estándar propuesto por van Embden 20, y van Soolingen 21. Extracción de ADN genómico. Para ello, una asada de cultivos jóvenes (25 a 35 días) fue resuspendida en 400 µL buffer TE (0.01 M Tris-HCl, 0,001 M EDTA [pH 8]) e inactivadas a 85 °C durante 20 minutos. Luego, se adicionó 50 µL de lisozima, 10 mg/mL (SIGMA) y se incubó a 37 °C durante 2 horas, para posteriormente adicionar 70 µL SDS 10% (SIGMA) y 5 µL de proteinasa K, 10 mg/mL(SIGMA) y se incubó a 65 °C por 15 minutos. Finalmente, 100 µL de NaCl 5M y CTAB-NaCl fueron adicionados para incubarlo a 65 °C por 15 minutos. El ADN fue extraído mediante cloroformo: alcohol isoamílico (24:1), precipitado mediante isopropanol absoluto y resuspendido en buffer TE. Restricción enzimática y transferencia de ácidos Hibridación con la sonda 245pb-IS6110 y obtención de los perfiles genéticos RFLP-IS6110. La hibridación de ácidos nucleicos fue llevada a cabo empleando el kit ECL (Amersham Pharmacia, Biotech) de acuerdo con las instrucciones del fabricante. Para ello, se preparó un volumen de buffer de hibridación y se agregó la sonda marcada. La sonda empleada fue un producto de amplificación de 245 pb de la secuencia de inserción IS6110 previamente amplificado por PCR usando los primers INS1 (5'-CGTGAGGGCATCGAGGTGGC-3') e INS2 (5'-GCGTAGGCGTCGGTGACAAA-3') y purificados mediante electroforesis en gel de bajo punto de fusión (SIGMA). La sonda fue marcada con la enzima peroxidasa siguiendo el procedimiento del kit ECL. Luego de una hibridación de 12 horas, la membrana fue lavada con un buffer conteniendo urea (Urea 7M, SDS %, SSC 0,5), y expuesto a un film (Hipefilm ECL, Amersham Pharmacia, Biotech). Los patrones de restricción fueron visualizados exponiendo la película de fotografía con soluciones de revelado y de fijación. ANÁLISIS DE LOS PERFILES GENÉTICOS RFLP-IS6110 Para comparar patrones genéticos que proceden de diferentes geles, la cepa de referencia M. Tuberculosis 14323 fue usada como control, e incluida en el primer y último carril. Los perfiles genéticos generados en base al número y posición de la secuencia de inserción IS6110 en el genoma de los diferentes aislamientos de M. tuberculosis fueron analizado empleando el programa GelCompar II versión 4.0 (Window 98, Applied Maths, Kortrijk, Bélgica). Los perfiles genéticos generados a partir de diferentes geles fueron introducidos en una base de datos, aquellos geles cuyos patrones genéticos de la cepa de referencia 14323 no coincidieron en un 100% fueron repetidos. Luego de eliminar aislamientos repetidos de un mismo paciente, se definieron cepas que provienen de una misma población clonal como aislamientos "agrupados" si ellos compartieron patrones idénticos o con sólo una banda de diferencia (como se sabe las secuencias de inserción son elementos genéticos móviles, por lo que entre cepas de una misma cadena de transmisión [cluster] puede ocurrir un cambio de 1,5%, además estos mínimos cambios no afectan la interpretación de los resultados 23 ).Y "no agrupados" si sus patrones genéticos fueron totalmente diferentes entre sí. Para estudiar la relación genética de las cepas se elaboró también un árbol de similaridad basado en el algoritmo UPGMA y el coeficiente de Dice. Todos los patrones fueron inspeccionados visualmente para detectar posibles errores, eliminando y volviendo a repetir los ensayos cuyos patrones IS6110 de la cepas 14323 no coincidieron en 100%. RESULTADOS PERFIL DE SUSCEPTIBILIDAD De 144 pacientes reclutados, 128 fueron seleccionados por cumplir los criterios de inclusión para la prueba de susceptibilidad. Los pacientes tuvieron una edad media de 24,52 ± 11,80 años, y 76 (59,37%) fueron de sexo masculino. Los pacientes "nunca tratados" fueron 95 (74,21%) y los "antes tratados" fueron 33 (25,78%). El análisis de perfil de sensibilidad a drogas de las cepas estudiadas, mostró de forma global que 50 (39,0%) presentaron resistencia de una a más drogas, con un nivel de resistencia primaria y adquirida de 32,63 y 57,57%, respectivamente. En el grupo de los pacientes "nunca tratados", 64 (67,37%) presentaron sensibilidad a todas las drogas de primera línea, 31 (32,63%) mostraron resistencia de una a más drogas. En el grupo de los "antes tratados", 14 (42,43%) fueron sensibles a todas las drogas, mientras que 19 (57,57%) fueron resistentes de una a más drogas. Se encontraron 20 cepas (54,78%) con el patrón de multidrogorresistencia (MDR), de los cuales 8,42% pertenecían a los "nunca tratados" y 36,36% a los "antes tratados" (Tabla 1). PERFILES GENÉTICOS MEDIANTE RFLP De los 144 pacientes reclutados se excluyeron 19 pacientes, debido a que pese a repetidos ensayos de extracción de ADN de sus respectivas cepas de M. tuberculosis que se practicó, no fue posible recuperar sus ADN genómicos en óptimas condiciones para el análisis de RFLP; finalmente se realizó el RFLP-IS6110 a 125 cepas de M. tuberculosis; sin embargo, siete perfiles genéticos con menos de cuatro bandas IS6110 si bien están incluidas en el dendograma fueron posteriormente excluidas del análisis de agrupamiento porque el análisis del RFLP no tiene un buen poder discriminativo en cepas menores de 5 bandas, quedando como población final 118 cepas (Figura 1). El número de copias IS6110 varió desde 2 hasta 15 bandas y la mayoría estuvo entre 5 a 11 bandas (Tabla 2). Además el número de copias de IS6110 se presentó con menor frecuencia en las poblaciones clonales que en las no clonales (Tabla 1, Figura 2). En total, 97 perfiles genéticos RFLP-IS6110 diferentes fueron obtenidos de 118 aislamientos; 35(29,7%) perfiles genéticos se agruparon en 14 clusters y 83 perfiles RFLP-IS6110 tuvieron ocurrencia única. De los 14 cluster encontrados la mayoría agrupó pacientes que presentaron perfiles genéticos desde 5 a 11 bandas con 100% de similitud, los cuales no presentaron link epidemiológico que determine una transmisión activa de la tuberculosis, a excepción del cluster VIII que agrupó a dos pacientes con un perfil genético de 10 bandas, cuyo antecedente epidemiológico fue que eran hermanos y compartían la misma vivienda, demostrándose en este cluster la transmisión activa de la TB. Además, el cluster XII presentó 10 bandas y sus pacientes agrupados (02) vivían en la misma localidad (Daniel Alcides Carrión) y presentaron similar patrón de resistencia a isoniazida y rifampicina; asimismo, el cluster XIII presentó perfiles genéticos con nueve bandas de similitud y el mismo patrón de resistencia a una droga (estreptomicina). DISCUSIÓN La epidemiología molecular usando el RFLP-IS6110 es una herramienta valiosa para estudiar la transmisión de la TB 24,25. En el presente estudio se encontró una gran diversidad de patrones genéticos diferentes (97 patrones), de las cuales 35 se agruparon en 14 cluster, aunque un cluster de tres cepas y que presentó menos de cuatro bandas no fue considerado en el análisis de transmisión. Probablemente a estas cepas se les debería hacer una subtipificación con otro método molecular, tales como el spoligotyping, VNTRs o DRE-PCR para confirmar su pertenencia a un cluster. De nuestros hallazgos, en el cluster VIII se encontraron genotipos relacionados (dos pacientes hermanos y que vivían en la misma vivienda), lo que indicaría una transmisión activa de la enfermedad y una relación clonal con el agente etiológico que causó la enfermedad en ambos casos. En el resto de clusters, dado que no se pudo establecer alguna relación epidemiológica de transmisión activa, se tratarían de casos de reactivación endógena, que probablemente hayan tenido diversas fuentes de contagio en el pasado. Sin embargo, cabe resaltar que no en todos los pacientes se pudo realizar una indagación epidemiológica que nos pudiera indicar un mayor indicio del encontrado. Este método mediante RFLP-IS6110 se presenta como una excelente herramienta para definir transmisión reciente y reactivación de TB 12,13,16,17. Las investigaciones de numerosos brotes han demostrado que muestras de M. tuberculosis con algún link epidemiológico presentan similares patrones genéticos, mientras que muestras no relacionadas tienen patrones diferentes 22. En países de alta prevalencia de TB como el nuestro, emplear este método será de gran valor, dado que permitirá identificar grupos de alto riesgo de transmisión, la identificación temprana de casos, y una mejor elección de los tratamientos antituberculosos. Como se aprecia, la zona escogida para el estudio (Villa El Salvador) presenta altos niveles de resistencia y multidrogoresistencia, y metodologías como la empleada, ligada a un fuerte componente epidemiológico y de antecedentes de tratamiento de los casos, nos ayudaría a seleccionar alternativas de tratamiento, con la consiguiente reducción de la transmisión de la enfermedad. Si bien se encontró un alto porcentaje de TB-MDR, nuestro estudio no reveló una transmisión activa de MDR en la zona. Se sugiere continuar con este tipo de investigaciones de epidemiología molecular, que nos permita caracterizar la diversidad clonal de las cepas provenientes de diferentes zonas del país, así como definir grupos de perfiles genéticos asociados a transmisión. AGRADECIMIENTOS Agradecemos a los médicos, enfermeras, tecnólogos médicos, técnicos y auxiliares de los diferentes centros de salud del distrito de Villa María del Triunfo que colaboraron desinteresadamente en el estudio. A los biólogos del Laboratorio de Biología Molecular por sus consejos y experiencia en el transcurso del trabajo. Al Dr. Leonid Lecca por su gran aporte en la finalización del trabajo. REFERENCIAS BIBLIOGRÁFICAS 1. Perú, Ministerio de Salud. Tuberculosis en el Perú. Informe 1999. Lima: MINSA / DGSP; 2000. [ Links ] 2. Raviglione MC, Snider DE Jr, Kochi A. Global epidemiology of tuberculosis: morbidity and mortality of a worldwide epidemic. JAMA 1995; 273(3): 220-26. [ Links ] 3. Chaves F, Dronda F, Alonso-Sanz M, Noriega AR. Evidence of exogenous reinfection and mixed infection with more than one strain of Mycobacterium tuberculosis among spanish HIV-infected inmates. AIDS 1999; 13(5): 615-20. [ Links ] 4. Barnes P, Bloch AB, Davidson PT, Snider DE Jr. Tuberculosis in patients with human immunodeficiency virus infection. N Engl J Med 1991; 324(23): 1644-50. [ Links ] 5. Coronado VG, Beck-Sague CM, Hutton MD, Davis BJ, Nicholas P, Villarreal C, et al. Transmission of multidrug-resistant Mycobacterium tuberculosis among persons with the human immunodeficiency virus infection in an urban hospital: epidemiologic and restriction fragment length polymorphism analysis. J Infect Dis 1993; 168(4): 1052-55. [ Links ] 6. Garcia-Garcia ML, Jimenez-Corona ME, Ponce-de-Leon A, Jimenez-Corona A, Palacios-Martinez M, Balandrano-Campos S, et al. Mycobacterium tuberculosis drug resistance in a suburban community in southern Mexico. Int J Tuberc Lung Dis 2000; 4(12 Suppl 2): S168-70. [ Links ] 7. Harrow EM, Rangel JM, Arriega JM, Cohen I, Regil Ruiz MI, DeRiemer K, et al. Epidemiology and clinical consequences of drug-resistant tuberculosis in a Guatemalan hospital. Chest 1998; 113(6): 1452-58. [ Links ] 8. Muñoz D, Rios G, Villalva C, Muñoz S. Factores asociados al diagnóstico tardío de pacientes con tuberculosis pulmonar en Lima Este. Peru. Rev Peru Med Exp Salud Publica 2004; 21(1): 18-22. [ Links ] 9. Taylor JP, Bergmire-Sweat D, Suarez L. Epidemiology of drug-resistant tuberculosis in Texas. Am J Epidemiol 1999; 149(4): 359-65. [ Links ] 10. Farmer P, Kim JY. Community based approaches to the control of multidrug resistant tuberculosis: introducing "DOTS-plus". BMJ 1998; 317(7159): 671-74. [ Links ] 11. Perú, Ministerio de Salud. Tuberculosis en el Perú. Informe 2000. Lima: MINSA / DGSP; 2001. [ Links ] 12. Alland D, Kalkut GE, Moss AR, McAdam RA, Hahn JA, Bosworth W, et al. Transmission of tuberculosis in New York City. An analysis by DNA fingerprinting and conventional epidemiologic methods. N Engl J Med 1994; 330(24): 1710-16. [ Links ] 13. Diel R, Schneider S, Meywald-Walter K, Ruf CM, Rusch-Gerdes S, Niemann S. Epidemiology of tuberculosis in Hamburg, Germany: long-term population-based analysis applying classical and molecular epidemiological techniques. J Clin Microbiol 2002; 40(2): 532-39. [ Links ] 14. Ellis BA, Crawford JT, Braden CR, McNabb SJ, Moore M, Kammerer S. Molecular epidemiology of tuberculosis in a sentinel surveillance population. Emerg Infect Dis 2002; 8(11): 1197-209. [ Links ] 15. Gutierrez MC, Vincent V, Aubert D, Bizet J, Gaillot O, Lebrun L, et al. Molecular fingerprinting of Mycobacterium tuberculosis and risk factors for tuberculosis transmission in Paris, France, and surrounding area. J Clin Microbiol; 36(2): 486-92. [ Links ] 16. Small PM, Hopewell PC, Singh SP, Paz A, Parsonnet J, Ruston DC, et al. The epidemiology of tuberculosis in San Francisco. A population-based study using conventional and molecular methods. N Engl J Med 1994; 330(24): 1703-9. [ Links ] 17. Baldeviano C, Quispe N, Bonilla C, Gastiaburu D, Pro J, Llanos-Zavalaga L. Perfiles genéticos (RFLP-IS6110) y resistencia a drogas en aislamientos de M. tuberculosis de pacientes internados en un hospital referencial del Callao, Perú. Rev Peru Med Exp Salud Publica 2003; 20(2): 72-7. [ Links ] 18. Instituto Nacional de Salud. El laboratorio de salud pública frente a la emergencia de la tuberculosis resistente. Lima: INS; 2001. Documento Técnico Nº3. [ Links ] 19. Canetti G, Rist N, Grosset J. [Measurement of sensitivity of the tuberculous bacillus to antibacillary drugs by the method of proportions. Methodology, resistance criteria, results and interpretation.]. Rev Tuberc Pneumol (Paris) 1963; 27: 217-72. [ Links ] 20. van Embden JD, Cave MD, Crawford JT, Dale JW, Eisenach KD, Gicquel B, et al. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J Clin Microbiol 1993; 31(2): 406-9. [ Links ] 21. van Soolingen D, de Haas P, Kremer K. Restriction fragment length polymorphism (RFLP) typing of mycobacteria. Bilthoven, The Netherlands: National Intitute of Public Health and The Environment; 2002. p. 52. [ Links ] 22. Daley CL, Small PM, Schecter GF. An outbreak of tuberculosis with accelerated progresión among persons infected with the human immunodeficiency virus: an analysis using restriction-fragment-length polymorphisms. N Eng J Med 1992; 326(4): 231-35. [ Links ] 23. Niemann S, Rusch-Gerdes S, Richter E. Stability of IS6110 restriction fragment polymorphism patterns of Mycobacterium tuberculosis strains in actual chains of transmission. J Clin Microbiol 2000; 38(1): 152-57. [ Links ] 24. Fletcher HA. Molecular epidemiology of tuberculosis: recent developments and applications. Curr Opin Pulm Med 2001; 7(3): 154-59. [ Links ] 25. Narayanan S. Molecular epidemiology of tuberculosis. Indian J Med Res 2004; 120(4): 233-47. [ Links ] Correspondencia:
Luis Capcha A.
Dirección: Av. Miguel Iglesias s/n San Juan de Miraflores, Lima.
Teléfono: (511) 4665545.
Correo electrónico: luiscapcha@hotmail.com