Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.22 n.3 Lima jul./sep 2005
TRABAJOS ORIGINALES
Tipificación molecular del virus dengue 3 durante el brote epidémico de dengue clásico en Lima, Perú, 2005
Molecular typing of dengue 3 virus during the classic dengue outbreak in Lima-Peru, 2005
Enrique Mamani Z1; María García M1; Victoria Gutiérrez P1; César Cabezas S1; Eva Harris2
1 Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
2 Division of Infectious Diseases, School of Public Health, University of California. Berkeley, CA, United States.
RESUMEN
Objetivos: Identificar mediante trascripción reversa-reacción en cadena de la polimerasa (RT-PCR) y sitios específicos de restricción - reacción en cadena de la polimerasa (RSS-PCR) al agente causal del brote epidémico presentado en el distrito de Comas, Lima en abril del año 2005. Materiales y métodos: veinte muestras de suero colectadas durante el brote de dengue fueron procesados por RT-PCR para determinar el serotipo, esta técnica se realizó en un solo paso. Luego se aplicó la técnica RSS-PCR para la identificación del genotipo circulante y se corroboraron los resultados posteriormente con aislamiento viral y secuenciamiento. Resultados: El análisis del RTPCR del ARN extraído de las muestras presentó un producto amplificado de 290pb que corresponden al dengue serotipo 3 (DEN 3). El análisis de los productos de RSS-PCR del ARN extraído a partir de aislamientos de DEN 3 correspondió al patrón C, incluido en el genotipo III. Los aislamientos de los virus dengue 3 en líneas celulares C6/36, tipificadas por IFI y el secuenciamiento genético confirmaron los resultados obtenidos por las pruebas previamente descritas. Conclusión: Durante el brote epidémico de dengue clásico en Lima, circuló el genotipo III del virus DEN 3.
Palabras clave: Brote de dengue clásico; virus dengue 3; tipificación molecular; RT-PCR; RSS-PCR; Perú (fuente: DeCS BIREME).
ABSTRACT
Objectives: To identify the causative agent of the outbreak that occurred in Comas, Lima in April 2005 using reverse transcription-polymerase chain reaction (RT-PCR) and specific restriction site-PCR (RSS-PCR). Materials and methods: 20 serum samples collected during the dengue outbreak were assessed using RT-PCR for identifying serotypes, this technique was performed in a single step. Later, the RSS-PCR method was used to identify the circulating serotypes, and the results were corroborated by viral isolation and sequencing. Results: RT-PCR of viral RNA taken from the samples showed a 290-pb amplified product corresponding to dengue virus serotype 3 (DEN 3). RSS-PCR product analysis of RNA from DEN 3 isolates corresponded to pattern C, included in genotype III. Dengue 3 virus isolates in C6/36 cell lines identified using indirect immunofluorescence and gene sequencing confirmed the results obtained using the aforementioned tests. Conclusion: During the classic dengue fever outbreak in Lima, DEN 3 genotype III circulated.
Key words: Outbreak of classic dengue; dengue 3 virus; molecular typing; RT-PCR; RSS-PCR; Peru (source: DeCS BIREME).
INTRODUCIÓN
El dengue es la enfermedad metaxénica viral más importante en salud pública, causada por alguno de los cuatro serotipos (DEN-1, 2, 3 y 4) del virus dengue, un virus ARN de cadena positiva de la familia Flaviviridae, el cual produce un espectro de enfermedad que va desde una fiebre por dengue hasta el dengue hemorrágico / síndrome de choque (shock) por dengue, esta última una infección grave con anormalidad vascular y hemostática que puede llevar a la muerte1.
La vigilancia serológica de dengue se basa principalmente en la detección de anticuerpos específicos IgM, mientras que la detección de serotipos circulantes se realiza tradicionalmente por el aislamiento viral y su identificación.
Desde hace unos años se comenzó a aplicar la prueba de trascripción reversa- reacción en cadena de la polimerasa (RT-PCR), que permite la identificación de los serotipos en muestras de suero y sobrenadantes de células infectadas con muestras clínicas2-5. El RTPCR es útil para obtener información rápida de los serotipos circulantes de dengue; sin embargo, es muy importante aislar el virus para confirmar su identidad y realizar estudios más detallados.
Cada serotipo del virus está subdividido en subtipos basados en la diversidad genética del gen de envoltura (E)6. Estos subtipos son estudiados para un mejor entendimiento del origen y de la evolución de las cepas de virus y la correlación existente entre subtipos y grado de severidad de la enfermedad presentes en los brotes epidémicos.
Para determinar los subtipos, tradicionalmente, se realiza el secuenciamiento para generar el árbol filogenético mediante métodos laboriosos y sofisticados7,8. Recientemente se desarrolló un nuevo método para la identificación de los subtipos basado en un simple PCR denominado Sitios Específicos de Restricción - Reacción en Cadena de la Polimerasa (RSS-PCR)9.
Desde los últimos 100 años no se había documentado la presencia de casos autóctonos de dengue en Lima; sin embargo, la presencia del Aedes aegypti fue notificado por primera vez en marzo del año 2000 en cinco localidades del Lima10; para abril del año 2004 ya se registraba 44 localidades infestadas con el vector, en el cual estaba incluido el distrito de Comas11; aunado a la presencia de casos importados de dengue12 el riesgo de que se presente un brote en Lima era alto.
Por ello se estableció un sistema de vigilancia vectorial y de febriles de las zonas en riesgo, con el fin de detectar oportunamente los primeros casos de dengue y realizar acciones de control. En este contexto es que el 14 de abril de 2005 se notificaron en la zona de Comas, casos de febriles con sintomatología compatible con dengue; se realiza la búsqueda activa de casos de febriles en la comunidad, y se encontraron 75 pacientes a los cuales se les tomó muestras de suero para el diagnóstico de la causa del brote13.
El objetivo del estudio fue identificar rápidamente mediante técnicas moleculares RT-PCR de un solo paso y el RSS-PCR, al agente causal del brote epidémico de dengue en Comas, Lima ocurrido en abril de 2005.
MATERIALES Y MÉTODOS
MUESTRAS DE SUERO
De los 75 primeros casos del día 14 de abril, se seleccionó en forma randomizada un grupo de 20 muestras con un tiempo de enfermedad menor de cinco días al momento de la toma de muestra. Se procedió a realizar el RT-PCR, simultáneamente, las muestras fueron inoculadas en cultivo celular C6/36 para el aislamiento viral y analizados por RSS-PCR para la determinación de los subtipos genéticos.
EXTRACCIÓN DEL ARN VIRAL
El ARN fue extraído a partir de las muestras de suero utilizando: QIAamp viral RNA Mini Kit cat. 52906 (QIAGEN, Inc., Valencia, CA) de acuerdo con el protocolo establecido por el fabricante.
TRANSCRIPCIÓN REVERSA Y AMPLIFICACIÓN POR RT-PCR
Se usó un procedimiento descrito por Harris et al., esta técnica de multiplex RT-PCR fue realizada en un solo paso para la transcripción reversa y la amplificación del ARN viral usando para ello un set de cinco primers2.
REACCIÓN EN CADENA DE LA POLIMERASA – SITIOS ESPECÍFICOS DE RESTRICCIÓN (RSS-PCR) PARA DEN 3.
Para la subtipificación de DEN 3 por RSS-PCR se siguió los procedimientos descritos por Harris et al9. Este sistema de tipificación rápido consiste de una trascripción reversa y una amplificación de ADNc usando cuatro primers que corresponden a regiones que obtenidas mediante el uso de enzimas de restricción del gen de envoltura (E). Este método es de fácil ejecución, rápido, requiere equipo de laboratorio mínimo y se usa reactivos ampliamente distribuidos.
AISLAMIENTO VIRAL E IDENTIFICACIÓN POR INMUNOFLUORESCENCIA
Se inoculó 50µL del suero de los pacientes en línea celular C6/36 Aedes albopictus, incubadas a 33 ºC por diez días; se usó medio mínimo esencial EMEM( Gibco-BRL) con sales de Earles, L-glutamina, aminoácidos no esenciales, 0,11% bicarbonato de sodio, 105 U/mL de penicilina, 75 U/mL de estreptomicina y suero bovino fetal al 2%. Se procedió a realizar la inmunofluorescencia usando un anticuerpo policlonal grupo B, anticuerpos monoclonales para DEN 1, DEN 2, DEN 3 y DEN 4.
RESULTADOS
APLICACIÓN DEL RT-PCR PARA IDENTIFICACIÓN DEL SEROTIPO
Después de seis horas de entregadas las muestras de suero, el análisis del producto de RT-PCR por electroforesis en gel de agarosa al 1,5% del ARN ex-traído de las muestras presentó un producto amplificado de 290pb que corresponden al DEN 3 (Figura 1).
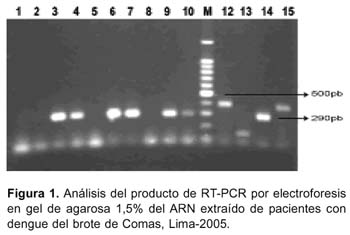
APLICACIÓN DE RSS-PCR PARA IDENTIFICACIÓN DE GENOTIPO
El análisis de los productos de RSS-PCR por electroforesis en gel de agarosa 1,5% del ARN extraído a partir de aislamientos de DEN 3 correspondieron al patrón C, incluido en el genotipo III o genotipo asiático, además se encontró una homología de 98,5% con la secuencia del DEN3 aislado en Nicaragua en 1996 (Figura 2).
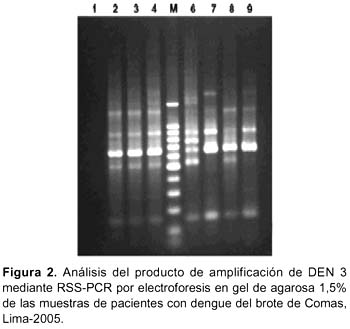
AISLAMIENTO VIRAL E IDENTIFICACIÓN POR INMUNOFLUORESCENCIA
Las muestras inoculadas en línea celular C6/36 presentaron efecto sincitial entre los siete a diez días después de la inoculación, la tipificación por IFI aplicando anticuerpos monoclonales dio como resultado DEN 3, confirmando los resultados obtenidos por las pruebas antes descritas.
DISCUSIÓN
El primer brote de dengue epidémico documentado en la localidad de Comas en Lima fue causado por el subtipo III o genotipo asiático del virus DEN 3, diagnosticado con las técnicas moleculares de RT-PCR a las seis horas de recibir la noticia de la presencia de febriles en la zona y subtipificado por RSS-PCR, posteriormente confirmado por el aislamiento viral e inmunofluorescencia (IFI).
El diagnóstico etiológico de dengue se realiza por el aislamiento viral en línea celular y la identificación de los serotipos por IF, procedimiento que requiere entre 7-15 días para obtener el resultado, además del alto costo, aplicación de técnicas laboriosas, obtención, almacenamiento y transporte de la muestra como factores que garantizan el éxito del diagnóstico. Este procedimiento se viene realizando en nuestro laboratorio desde 1990 fecha en el que se dio el primer brote de DEN 1 en la zona tropical de Loreto. Actualmente sólo se trabaja 10% de las muestras que vienen para este diagnóstico, debido principalmente al alto costo que representa su ejecución.
Alternativamente, la técnica del RT-PCR que aplicamos para definir el brote de Comas en un tiempo corto, previamente descrito 9 , está siendo usada en otros países como un método de vigilancia sólo en el inicio de brotes2-5 . Otros laboratorios han logrado optimizar este procedimiento de tal forma que lo vienen usando como un diagnóstico de rutina, previamente validado contra el aislamiento que es la prueba de oro 14 , lo cual permite simplificar el tiempo de respuesta y reducir los costos del diagnóstico. Esta es una opción importante, teniendo en cuenta que 90% de las muestras de suero de pacientes febriles con cuadro probable de dengue se encuentran dentro de los primeros cinco días de iniciado la enfermedad y que el aislamiento viral representa un alto costo y tiempo de respuesta.
La determinación del genotipo de los virus circulantes de dengue es una información valiosa cuando se presenta un brote, es sabido que algunos genotipos asiáticos están asociados con cuadros graves de dengue 15,16 .
El sistema de tipificación RSS-PCR 9 , es un procedimiento de fácil ejecución similar a un método de PCR y mucho más accesible que un análisis de secuencia y filogenia, por lo que se constituye en una importante herramienta en epidemiología molecular para laboratorios que no cuentan con sistemas de secuenciamiento.
Los resultados demostraron la circulación del subtipo III del virus DEN 3, este subtipo es miembro del subtipo Sri Lanka, de origen asiático y ha sido asociado con dengue hemorrágico. Por los antecedentes de brotes anteriores de DEN 3 en áreas endémicas de Perú como la región oriental y la costa norte 17,18 , hasta el momento sólo se ha observado la presencia del genotipo III y este ha estado asociado con casos clínicos de dengue hemorrágico, lo observado en el brote de Comas a la fecha son cuadros clínicos de dengue clásico.
El diagnóstico rápido y oportuno permitió que las autoridades del sector salud actuaran inmediatamente para realizar las medidas de control del brote.
AGRADECIMIENTOS
Al Sustanaible Sciences Institute por su colaboración con los reactivos y entrenamiento para la ejecución del estudio, en particular a la Dra. María Elena Peñaranda.
REFERENCIAS BIBLIOGRÁFICAS
1. Gubler DJ. Dengue and dengue hemorrhagic fever. Clin Microbiol Rev 1998; 11(3): 480-96. [ Links ]
2. Harris E, Roberts TG, Smith L, Selle J, Kramer LD, Valle S, et al. Typing of dengue viruses in clinical specimens and mosquitoes by single-tube multiplex reverse transcriptase PCR. J Clin Microbiol 1998; 36(9): 2634-39. [ Links ]
3. Henchal E, Polo S, Vorndam V, Yaemsiri C, Innis B, Hoke C. Sensitivity and specificity of a universal primer set for the rapid diagnosis of dengue virus infections by polymerase chain reaction and nucleic acid hybridization. Am J Trop Med Hyg 1991; 45(4): 418-28. [ Links ]
4. Lanciotti R, Calisher C, Gubler D, Chang G, Vorndam AV. Rapid detection and typing of dengue viruses from clinical samples by using reverse transcriptasepolymerase chain reaction. J Clin Microbiol 1992; 30(3): 545-51. [ Links ]
5. Seah CL, Chow V, Tan HC, Chan YC. Rapid, single-step RT-PCR typing of dengue viruses using five NS3 gene primers. J Virol Methods 1995; 51(2-3): 193-200. [ Links ]
6. Fields BN, Knipe DM eds. Virology. 4 th Ed. Philadelphia: Lippincott Williams & Wilkins; 2001. [ Links ]
7. Chungue E, Deubel V, Cassar O, Laille M, Martín PM. Molecular epidemiology of dengue 3 viruses and genetic relatedness among dengue 3 strains isolated from patients with mild or severe form of dengue fever in French Polynesia. J Gen Virol 1993; 74(Pt 12): 2765-70. [ Links ]
8. Lanciotti RS, Lewis JG, Gubler DJ, Trent DW. Molecular evolution and epidemiology of dengue-3 viruses. J Gen Virol 1994; 75(Pt 1): 65-75. [ Links ]
9. Harris E, Sandoval E, Johnson M, Xet-Mull AM, Riley LW. Rapid subtyping of dengue viruses by restriction site-specific (RSS)-PCR. Virology 1999; 253(1): 86-95. [ Links ]
10. Andrade CS, Caceres AG, Vaquerizo A, Ibañez-Bernal S, Cachay LS. Reappearance of Aedes aegypti (Diptera: Culicidae) in Lima, Perú. Mem Inst Oswaldo Cruz 2001; 96(5): 657-58. [ Links ]
11. Dirección General de Salud Ambiental. Vigilancia y control entomológico del Aedes aegypti. Lima ciudad. Lima: Ministerio de Salud / DIGESA; 2005. Informativo entomológico Nº 001-05/DESB/DIGESA [ Links ]
12. Legua P. Dengue importado en Lima: Riesgo para viajeros. Rev Farmacol Terapeut 1995; 4 (1-2): 67-68 [ Links ]
13. Cabezas C. Reemergencia del dengue en Lima. Crónica de una enfermedad anunciada. Rev Peru Med Exp Salud Publica 2005; 22(2): 159-60. [ Links ]
14. Balmaseda A, Sandoval E, Pérez L, Gutiérrez CM, Harris E. Application of molecular typing techniques in the 199 dengue epidemic in Nicaragua. Am J Trop Med Hyg 1999; 61(6): 893-97. [ Links ]
15. Lanciotti RS, Lewis JG, Gubler DJ, Trent DW. Molecular evolution and epidemiology of dengue-3 viruses. J Gen Virol 1994; 75(Pt 1): 65-75. [ Links ]
16. Rico-Hesse R, Harrison LM, Salas RA, Tovar D, Nisalak A, Ramos C, et al. Origins of dengue type 2 viruses associated with increased pathogenicity in the Americas. Virology 1997; 230(2): 244-51. [ Links ]
17. Cáceres O, Mamani E, Iwasaki R, Gutierrez V, Garcia M, Cobos M. Secuenciamiento genético del virus dengue 3 circulante en Ucayali-Perú, en el año 2004. Bol Inst Nac Salud (Perú) 2005; 11(1): 18-20. [ Links ]
18. Montoya Y, Holechek S, Cáceres O, Palacios A, Burans J, Guevara C, et al. Circulation of dengue viruses in North-Western Peru, 2000-2001. Dengue Bull WHO 2002; 27: 52-62. [ Links ]
Correspondencia: Blgo. Enrique Mamani Zapana. Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima Perú.
Dirección: Capac Yupanqui 1400, Lima 11.
Teléfono: (511) 2516151 anexo 542
Correo electrónico: emamani@ins.gob.pe














