Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.23 n.4 Lima oct./dic. 2006
COMUNICACIÓN CORTA
Amplificación del gen hsp18 para la detección de Mycobacterium leprae
hsp18 gene amplification for detecting Mycobacterium leprae
Róger Calderón E1; Carmen Luna C1
1 Laboratorio de Biotecnología y Biología Molecular, Instituto Nacional de Salud. Lima, Perú.
RESUMEN
Mediante PCR se amplificó un fragmento de 360 pb del gen hsp18, que codifica al antígeno proteico de 18kDa de M. leprae, a partir de una biopsia de un paciente con diagnóstico baciloscópico, histopatológico y clínico de lepra. Además, se evaluaron tejidos embebidos en parafina (fijados en formol) y ADN de otras micobacterias para determinar la especificidad, sensibilidad y confiabilidad del método. El ensayo de PCR amplificó clara y satisfactoriamente ADN de M. leprae procedente de la biopsia del paciente, pero fue incapaz de amplificar usando ADN purificado a partir de tejidos embebidos en parafina. No se observaron productos de amplificación al utilizar ADN genómico de varias micobacterias tales como M. tuberculosis, M. bovis, M. fortuitum, M. gordonae, M. kansasii, M. scrofulaceum, M. avium entre otras, así como de otras bacterias. Este sistema puede considerarse como una alternativa para la identificación de pacientes infectados con M. leprae orientando el esfuerzo para determinar la prevalencia oculta de la enfermedad mediante la identificación de casos asintomáticos con capacidad de transmisión de bacilos.
Palabras clave: Lepra / diagnóstico; Reacción en cadena de la polimerasa; Mycobacterium leprae, Proteínas del shock térmico.
ABSTRACT
A 360-kb fragment of the hsp-18 gene that codes for the 18-kDa-protein antigen of Mycobacterium leprae was amplified using a polymerase chain reaction (PCR). The fragment was obtained from a biopsy taken from a patient with a microscopic, histopathological, and clinical diagnosis of leprosy. Additional paraffin-embedded tissue samples (formalin-fixed), as well as DNA samples from other mycobacteria were also assessed, in order to determine specificity, sensitivity, and reliability of this method. PCR clearly and satisfactorily amplified M. leprae DNA from the patient's biopsy, but it was not capable of performing any amplification using purified DNA from paraffin-embedded tissue samples. No amplification products were observed when using genomic DNA from some other mycobacterial species, such as M. tuberculosis, M. bovis, M. fortuitum, M. gordonae, M. kansasii, M. scrofulaceum, and M. avium; as well as using DNA from other bacteria. This system can be used as an alternative for identifying M. leprae-infected patients, orienting efforts for detecting the hidden prevalence of the disease by identifying asymptomatic cases capable of bacilli transmission.
Key words: Leprosy / diagnosis; Polymerase chain reaction; Mycobacterium leprae, Heat-shock proteins.
INTRODUCCIÓN
La lepra es una enfermedad que continúa siendo un problema en la salud pública de los países en desarrollo, a pesar de la reducción de los casos registrados por la OMS. En el Perú, la realidad epidemiológica de la enfermedad aún es desconocida, aunque se reconocen zonas endémicas como Ucayali1. En el año 1998 se han notificado en el continente americano 119 279 casos y registrado en nuestro país 262 casos; los cuales fueron incluidos en su totalidad en los programas de tratamiento2.
Dado de que este microorganismo no es recuperable a partir de cultivo in vitro, el diagnóstico de la lepra es realizado por medio del examen clínico, apoyado por la presencia de M. leprae en las baciloscopías practicadas en la mucosa nasal, lóbulo de la oreja y lesión cutánea, en la demostración histopatológica de bacilos ácidoalcohol resistentes infiltrados en histiocitos, linfocitos; acompañados de la invasión de nervios cutáneos3-5. En los pacientes que se encuentran en un estado intermedio entre los tipos tuberculoide y lepromatoso se presentan dificultades diagnósticas en términos de sensibilidad y especificidad, dado que las características en las biopsias parecen ser inconsistentes con el número de organismos observados o cuando existe respuesta granulomatosa a pesar de que hay muy pocos o ningún organismo6. Así se evidencia la necesidad de mejorar las metodologías de detección de lepra en nuestro país, para que contribuyan con el bienestar en las zonas de alto riesgo e incidencia de esta enfermedad.
Algunos países se encuentran en un período en el que se puede pensar en la erradicación de la enfermedad, pero es muy importante conocer las características epidemiológicas ubicando las zonas geográficas en donde aun se reportan casos de lepra1. Es así como se pueden fortalecer o regenerar los programas de vigilancia y principalmente reasignar recursos para tal fin, tratando de detectar tempranamente los casos y eliminar las fuentes de transmisión mediante poliquimioterapias7.
El objetivo del presente trabajo preliminar fue implementar un sistema de amplificación mediante PCR para que pueda aplicarse en muestras clínicas de diverso origen y así establecer una eficiente alternativa de diagnóstico temprano y oportuno en casos intermedios y asintomáticos que pueden ser fuentes de infección y con ello se pueda contribuir con la salud pública de nuestro país.
EL ESTUDIO
MUESTRAS BIOLÓGICAS, BIOPSIAS Y CEPAS
La biopsia fue obtenida de un paciente multibacilar adulto que se encontraba en el quinto mes de tratamiento que acudió al Instituto de Investigación en Medicina Tropical "Alexander von Humboldt" del Hospital Nacional Cayetano Heredia en Lima. La fracción de la biopsia de 25 mg aproximadamente fue almacenada a -70 °C.
La División de Patología del Instituto Nacional de Salud proporcionó cuatro muestras de tejidos embebidos en parafina y con diagnóstico clínico e histopatológico positivo para lepra lepromatosa y una muestra con diagnóstico histopatológico negativo.
Las cepas de Mycobacterium tuberculosis H37Rv (ATCC 27294), M. bovis BCG, M. avium, M. gordonae, M. scrofulaceum, M kansasii, M. fortuitum fueron obtenidas del Laboratorio de Micobacterias del Instituto Nacional de Salud. Los aislamientos de Klebsiella pneumoniae, Staphylococcus aureus, Brucella sp. Pseudomonas aeruginosa, Neisseria meningitidis, Escherichia coli, fueron obtenidas del Laboratorio de IRAS e IIH del Instituto Nacional de Salud.
El ADN genómico de las cepas micobacterianas fue obtenido usando el protocolo de extracción con tiocianato de guanidina8. Los análisis electroforéticos fueron realizados en geles de agarosa al 1,5% y de poliacrilamida al 10% y revelados por tinción con bromuro de etidio (1%) y evaluados mediante exposición a luz UV o tinción con sales de plata.
PREPARACIÓN DE ADN DE M. leprae A PARTIR DE TEJIDOS
En un tubo de microcentrífuga de 1,5mL con 300ìL de buffer STE (0,1M Tris-HCl pH 8; 0,15M NaCl; 0,01M EDTA pH 8) se colocaron 25 mg del nódulo cutáneo. Se realizó una lisis inicial mediante ciclos de congelamiento (-70 °C) y descongelamiento a 95 °C. Luego se adicionaron 500 ìg de proteinasa K y fue incubado durante seis horas, realizando una agitación usando vórtex cada 30 min. Posteriormente se trató con 300 ìL un volumen similar de solución de lisis (5M tiocianato de guanidina, 0,1 M EDTA pH 8 y Tween 0,5%) agitando suave y totalmente el contenido. Luego se adicionaron 300ìL de etanol absoluto y se mezcló totalmente. Finalmente el ADN fue purificado usando columnas de purificación del QIAamp® DNA Mini kit (Qiagen, Hilden, Alemania) y se almacenaron a -20 °C hasta su utilización.
En el caso de los tejidos embebidos en parafina, se retiraron 25 mg de cada muestra y fueron lavados con 1,2 mL de xileno durante diez minutos y luego centrifugados a 14000 rpm durante cinco minutos. Posteriormente fueron lavados dos veces con etanol absoluto, el cual fue retirado por pipeteo y evaporación final a 37 °C durante 15 minutos. Luego se agregaron 300 ìL de buffer STE y se continuó con el procedimiento anteriormente descrito.
REACCIÓN DE AMPLIFICACIÓN DEL GEN DE LA PROTEÍNA DE 18KDa DE M. leprae
Un fragmento del gen de la proteína de shock térmico de 18KDa (hsp18) de M. leprae fue amplificada mediante un PCR9 usando oligonucleótidos sintéticos 18K1-F y 18K3-R (IDT DNA Technologies) cuyas secuencias fueron descritas por Williams et al10. La mezcla de reacción del PCR para un volumen final de 100 ìL estuvo constituída de la siguiente forma: buffer PCR II 1X; 200ìM de desoxinucleótidos fosfato; 1,5 mM de MgCl2; 0,25 ìM del oligonucleótido 18K1-F y 18K3-R y 1 U de enzima Taq DNA polimerasa (Applied Biosystem, SF California USA). Las temperaturas usadas para la amplificación fueron: 95 °C por cinco minutos, cuarenta ciclos de 95 °C por 30 segundos, 63 °C por 30 segundos y 72 °C por 90 segundos. Posteriormente una extensión final de 72 °C por diez minutos.
DETERMINACIÓN DE LA SECUENCIA DEL GEN hsp18 DE M. leprae
El producto de PCR fue purificado a partir de un gel de agarosa de bajo punto de fusión mediante el uso del kit Wizard® PCR Preps DNA Purification System (Promega Corp. Winsconsin, UK). Luego fue clonado en el vector pGEM®-T Easy (Promega) y mediante electroporación se realizó la transformación de la cepa E. coli XL1 Blue. Las bacterias transformantes fueron seleccionadas en medio de cultivo agar LB con 100 µg/mL de ampicilina, así como IPTG y X-Gal según lo especificado por Sambrook et al11. Luego, cada colonia recombinante fue crecida en medio de cultivo caldo LB con 100 µg/mL de ampicilina a 37 °C durante toda la noche. Los plásmidos recombinantes fueron extraídos siguiendo protocolos previamente descritos11 y purificados mediante el kit Wizard® Clean Up DNA Purification System (Promega).
Finalmente el plásmido recombinante fue secuenciado mediante el Thermo Sequenase* Cy5.5 Dye Terminator Sequencing Kit (Amersham Pharmacia Biotech, Nueva York, USA).
HALLAZGOS
Una total disgregación fue obtenida de la biopsia fresca del paciente multibacilar, usando el protocolo descrito.
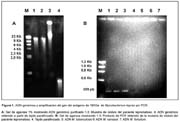
Así, la obtención del ADN a partir de la digestión del homogeneizado celular de la biopsia fue exitosa obteniendo una gran cantidad de ADN genómico (>1 mg), de elevado peso molecular, aunque principalmente perteneció al paciente (Figura 1).
Por otra parte el procedimiento de extracción de ADN a partir de los tejidos embebidos en parafina no fue satisfactorio observándose fragmentos de ADN menores a 300 pb (Figura 1).
El ensayo de PCR proporcionó un claro fragmento de amplificación de 358 pb. El ensayo de PCR resultó positivo sólo para el ADN de la biopsia del nódulo de piel del paciente. Sin embargo, no se determinó amplificación alguna al usar ADN genómico de las muestras de tejidos parafinizados, los cuales eran baciloscópicamente e histológicamente positivas para M. leprae.
Al comparar con las secuencias del mismo gen hsp18 pertenecientes a otras bacterias no se observó complementariedad alguna de los oligonucleotidos 18K1-F y 18K3-R. Al ensayar el PCR utilizando ADN genómico de M. tuberculosis, M. bovis, M. avium, M. gordonae, M. scrofulaceum, M kansasii, M. fortuitum y otras bacterias tampoco se generaron productos de amplificación.
La secuencia referencial completa del gen hsp18, de la proteína de 18KDa (GeneBank M19058.1) es mostrada en la Figura 2. Asimismo, se indican las regiones complementarias a los oligonucleótidos 18K1-F y 18K3-R para la obtención de la secuencia parcial de 358 pb. La secuencia obtenida mediante secuenciamiento del producto de PCR hsp-01 fue:

Mediante un análisis de secuencia, se reveló que de hsp-01 corresponde al extremo C-terminal del gen hsp18 de M. leprae (AL583923, M19058, M22587), observando una similitud >99% (Figura 3). El número de acceso GeneBank de la secuencia nucleotídica hsp-01 obtenida es AY608693.
DISCUSIÓN
El procedimiento de extracción de ADN a partir de la biopsia de piel del paciente resultó completamente ventajoso, dada la amplia capacidad de purificación y aplicabilidad en muestras de cualquier origen; suficiente para los motivos de amplificación de ADN de M. leprae. Sin embargo, la principal causa por la que los tejidos parafinizados no proporcionaron producto de amplificación fue el uso de formaldehído inapropiadamente tamponado (pH ácido) en la metodología de fijación tisular lo cual contribuyó directamente con la total destrucción del ADN.
El sistema de amplificación mostró una elevada especificidad, debido a que no mostró reacción cruzada con ADN de otras micobacterias, entre ellas M. tuberculosis. La secuencia hsp-01 amplificada mostró >99% de similitud con otras secuencias de hsp18 reportadas en el GenBank.
Es concluyente que este ensayo de amplificación mediante PCR descrito, tiene una gran especificidad en la detección de M. leprae a partir de muestras para una calidad y cantidad relativa de ADN bacteriano. Sin embargo, debe ser evaluado para determinar su verdadera aplicabilidad en una eficaz identificación de pacientes infectados con M. leprae. Es también importante señalar que la aplicación del PCR puede indicarnos el éxito o fracaso terapéutico en aquellos pacientes que se encuentren recibiendo fármacos antilepromatosos, debido a que el ADN de M. leprae va siendo degradado o fragmentado hasta llegar a niveles no amplificables12 producto de la quimioterapia. En el caso de pacientes infectados con bacilos resistentes, la reacción de PCR se mantendría positiva13,14.
Por todo ello, este ensayo podría considerarse como una herramienta de diagnóstico alternativo que permita la identificación de pacientes intermedios-asintomáticos, infectados con bacilos de Mycobacterium leprae. Estos pacientes son fuente importante de contaminación y transmisión de la enfermedad, ya que pueden contaminar silenciosamente a poblaciones susceptibles y generar la aparición de casos nuevos. Potencialmente se podrán dirigir oportunos esquemas terapéuticos a los pacientes identificados que finalmente contribuirán con el control y prevención de la transmisión de este microorganismo, para posteriormente consolidarse como una herramienta que ayude con la erradicación de la enfermedad en nuestro país.
REFERENCIAS BIBLIOGRÁFICAS
1. Burstein Z. Revisión histórica del control de la lepra en el Perú. Rev Med Exp 2001; 18(1-2): 40-44. [ Links ]
2. World Health Organization. Action programme for the elimination of leprosy. Status Report 1998. Geneva: WHO; 1999. [ Links ]
3. Ridley DS. The histopathological spectrum of the mycobacterioses. In: Ratledge C, Stanford J. The biology of mycobacteria. London: Academic Press; 1982. [ Links ]
4. Lopez-Antuñano F. Diagnóstico y tratamiento de la lepra. Salud Publica Mex 1998; 40(1): 66-75. [ Links ]
5. Ustianowsky AP, Lockwood D. Leprosy: current diagnostic and treatment approaches. Curr Opin Infect Dis 2003; 16(5): 421-27. [ Links ]
6. Scollard DM, Adams LB, Gillis TP, Krahenbuhl RW, Truman RW, Williams DL. The continuing challenges of leprosy. Clin Microbiol Rev 2006; 19(2): 338-81. [ Links ]
7. González Ochoa E, Abreu A. Vigilancia de la lepra en situaciones de baja prevalencia. Rev Panam Salud Pública 2001, 9(2): 94-101. [ Links ]
8. Popovic T, Bopp C, Olsvik Ø, Kiehbaluch J. Ribotyping in molecular epidemiology. In: Persing DH, Smith TF, Tenover FC, White TJ (ed.), Diagnostic molecular microbiology: principles and applications. Washington DC: American Society for Microbiology; 1993. p. 573-89. [ Links ]
9. Scollard D, Gillis TP, Williams DL. Polymerase chain reaction for the detection and identification of Mycobacterium leprae in patients in the United States. Am J Clin Pathol 1998. 109(5): 642-46. [ Links ]
10. Williams D, Gillis TP, Booth RJ, Looker D, Watson JD. The use of a specific DNA probe and Polymerase chain reaction for the detection of Mycobacterium leprae. J Infect Dis 1990; 162(1): 193-200. [ Links ]
11. Sambrook J, Fritsch EF, Maniatis T. Molecular cloning: a laboratory manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press 1989. [ Links ]
12. De Wit MY, Faber W, Krieg SR, Douglas JT, Lucas SB, Montreewasuwat N, et al. Application of a polymerase chain reaction for the detection of Mycobacterium leprae in skin tissues. J Clin Microbiol 1991; 29(5): 906-10. [ Links ]
13. Williams DL, Gillis TP, Fiallo P, Job CK, Gelber RH, Hill C, et al. Detection of Mycobacterium leprae and the potential for monitoring antileprosy drug therapy directly from skin biopsies by PCR. Mol Cell Probes 1992; 6(5): 401-10. [ Links ]
14. Donoghue H, Holton J, Spiegelman M. PCR primers that can detect low levels of Mycobacterium leprae DNA. J Med Microbiol 2001; 50(2): 177-82. [ Links ]
Correspondencia:
Blgo. Roger Calderón Espinoza. Laboratorio de Biotecnología y Biología Molecular. Instituto Nacional de Salud. Lima, Perú.
Dirección: Av. Defensores del Morro 2268. Chorrillos, Perú
Teléfono: (511) 251-6151
Correo electrónico: rivance@yahoo.com