Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.24 n.1 Lima ene./mar. 2007
ARTÍCULO ORIGINAL
Detección sensible y específica de Mycobacterium tuberculosis a partir de muestras clínicas, mediante la amplificación de un elemento repetitivo de la familia REP13E12
Sensitive and specific detection of Mycobacterium tuberculosis from clinical samples by amplification of a repeated sequence of the REP13E12 family
Christian Baldeviano V1a ; Carmen Luna C1a ; Tatiana Cáceres N2a ; Roger Calderón E1a
1 Laboratorio de Biotecnología y Biología Molecular, Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
2 Laboratorio de Micobacterias, Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
a Biólogo.
RESUMEN
Objetivos: Diseñar e implementar un método de PCR para la detección sensible y específica de M. tuberculosis, orientado al oportuno diagnóstico de la tuberculosis mediante su adecuada aplicación en muestras clínicas. Materiales y métodos: Se diseñaron oligonucleótidos específicos para la amplificación de un fragmento de 318pb luego de una alineación múltiple de secuencias REP13E12 de M. tuberculosis. Se realizó la estandarización del método de PCR y se evaluó la sensibilidad y especificidad diagnóstica utilizando muestras clínicas con diagnóstico baciloscópico y cultivo. Resultados: El REP13E12-PCR detectó hasta 100 fg de ADN genómico, fue específico para la detección del complejo M. tuberculosis y capaz de distinguirlos de micobacterias atípicas. Esta evaluación preliminar proporcionó 100% de sensibilidad y especificidad en muestras clínicas de pacientes con diagnóstico de TB. Conclusiones: La amplificación de REP13E12 mediante PCR es una alternativa para el diagnóstico rápido de pacientes con TB, especialmente en casos cuyo diagnóstico pueda ser dificultoso o poco claro mediante el uso de los métodos convencionales.
Palabras clave: Tuberculosis / diagnóstico; Mycobacterium tuberculosis; PCR (fuente: DeCS BIREME).
ABSTRACT
Objectives: To design and implement of PCR method for sensitivity and specific detection of M. tuberculosis oriented to opportune and correctly diagnosis of tuberculosis by the application on clinical specimens. Material an methods: Specific oligonucleotides for the amplification of a fragment of 318pb after a multiple alignment of sequences REP13E12 were designed. The PCR method was standarize and also the analytical and diagnostic sensibility and specifity was evaluate; using clinical samples with smear or culture diagnosis positive and negative. Results: The REP13E12-PCR detected up to 100 fg of genomic DNA. It was specific for the detection of complex M. tuberculosis and able to distinguish them of non-tuberculous mycobacteria. This preliminary evaluation provided 100% of sensitivity and specificity in sputum samples of patients with TB diagnosis. Conclusions: The REP13E12 amplification by PCR is an alternative method for the rapid diagnosis of patients with TB, especially for cases which difficult or unclear diagnosis by the gold standard technique.
Key words: Tuberculosis / diagnosis; Mycobacterium tuberculosis; PCR (source: DeCS BIREME).
INTRODUCCIÓN
La tuberculosis (TB) continúa siendo una de las principales causas de muerte en adultos, ocasionado por un único agente infeccioso. Se estima que anualmente existen entre siete a ocho millones de nuevos casos y entre dos a tres millones de muertes en todo el mundo1. El Perú comparte con Brasil la mayor carga de tuberculosis en la región de las Américas y desde la década de 1990 vive en una creciente endemia de TB multidrogorresistente (TB MDR), la forma más peligrosa de esta enfermedad 2.
En nuestro país, durante el año 2005, se ha presentado una tasa de incidencia de 109,7×100 000 hab. (30 226 casos nuevos), de los cuales 38,8% de pacientes con tuberculosis no se ha podido detectar mediante baciloscopía3, entre los que estarían los casos extrapulmonares, paucibacilares, entre otros.
Por ello, la detección rápida y oportuna de estos casos es de vital importancia en el control de TB; sin embargo, no existe hasta el momento un método de laboratorio para diagnosticar TB que sea rápido, sencillo y efectivo, aunque existe un considerable esfuerzo en la investigación de ésta área4.
La baciloscopía es una herramienta útil para el rápido tamizaje y la detección de casos, ya que es un método simple, barato y fácil de realizar, pero su sensibilidad es baja (aprox. 10 000 bacilos/mL de muestra5). El cultivo en medio Lowenstein-Jensen (LJ) que es usado como diagnóstico confirmatorio, es más sensible que la baciloscopía (10 a 100 bacilos/mL); pero puede demorar más de tres semanas debido a la biología del bacilo5.
Por otro lado, se ha desarrollo una variedad de métodos para detectar el crecimiento bacteriano de manera temprana, entre los más importantes se encuentran el sistema BACTEC 460 (Becton Dickinson Co, MD), el sistema BACTEC MGIT 960 (BD Biosciences, MD), cultivo Myco-ESP (Difco Laboratories, MC), entre otros. Estos nuevos métodos tienen sensibilidad y especificidad equivalente al cultivo, pero pueden proporcionar un resultado entre cuatro días a tres semanas6.
El avance en el campo de la genética molecular ha permitido el desarrollo de nuevas técnicas basada en la detección del material genético. Se han elaborado varios métodos moleculares basados en la técnica de la reacción en cadena de la polimerasa (PCR) los cuales han mejorado los métodos existentes7. La utilidad clínica de los métodos de amplificación genética ha sido bastante estudiada7-10, aunque en muestras paucibacilares no se encuentra completamente demostrada11.
Existe una gran cantidad de genes blancos; entre ellos, la secuencia de inserción IS6110, la proteína de 38 kDa, 32 kDa, la secuencia mtp-409-10,12-14. Recientemente, se han reportado PCR basados en las secuencias recA/ pps1, y el gen tipo histona hupB115-16. Sin embargo, aún son poco estudiados los sistemas de PCR que puedan detectar muestras paucibacilares.
En este artículo reportamos un nuevo blanco de amplificación basado en la secuencia repetitiva REP165 perteneciente a la familia de elementos repetitivos REP13E12 recientemente descrito17,18. Se diseñaron un par de primers para amplificar una región de la secuencia REP165, se estandarizaron las condiciones para el PCR, determinándose la sensibilidad y especificidad analítica. Adicionalmente, se evaluaron preliminarmente un grupo de muestras de esputo frotis positivo así como frotis y cultivo negativo. Esta técnica presenta enorme potencial en la detección en muestras paucibacilares.
MATERIALES Y MÉTODOS
OBTENCIÓN DE MUESTRAS
Se evaluaron dos grupos de muestras de esputo, las del grupo A (N= 45) de pacientes positivos a TB con dos o más baciloscopías positivas y al menos dos cultivos positivos, obtenidas como parte de un estudio que se llevó a cabo en el Instituto Nacional de Salud de Lima entre los meses de abril y mayo del 2004; las muestras del grupo B (N= 35) fueron recolectadas de pacientes sintomáticos respiratorios con un diagnóstico diferente a TB (al menos dos baciloscopías negativas y cultivo por LJ negativos a TB) en el Laboratorio de Micobacterias del Hospital Nacional Cayetano Heredia.
Todas las muestras fueron descontaminadas empleando la solución de NALC-NaOH y el ADN genómico fue obtenido mediante el uso del protocolo de extracción con tiocianato de guanidina19, precipitación con etanol y resuspendidos en buffer TE.
ALINEACIÓN MÚLTIPLE DE SECUENCIAS REP13E12 Y DISEÑO DE OLIGONUCLEÓTIDOS
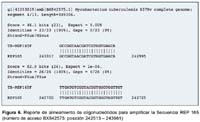
Con el objetivo de implementar un sistema de PCR para detectar de manera sensible y específica la presencia de M. tuberculosis a partir de muestras clínicas se diseñaron un par de oligonucleótidos TB-REP165F y TBREP165R (Tabla 1) alineando las secuencias repetitivas de dos secuencias de la familia REP13E12, empleando la secuencia REP165 de M. tuberculosis H37Rv, el cual se encuentra publicada en el Gen Bank (Número de acceso es BX842575).
Los oligonucleótidos fueron diseñados con la ayuda del software PrimerSelect 4.05 (DNASTAR Inc. Madison, USA) y posteriormente fueron sintetizados por Integrated DNA Technologies (IDT Inc. Coralville, ID, USA).
CONDICIONES DE LA REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
La reacción de amplificación fue estandarizada con una concentración final óptima de MgCl2 de 1,5mM, 0,2 mM de mezcla de dNTPs; 1,2 uM de cada primer y 1U de enzima Taq ADN polimerasa. Las condiciones empleadas fueron: 95 °C por 10 min, 40 ciclos de 95 °C durante 1 min, 64 °C por 1 min y 72 °C por 2 min, con una extensión final de 72 °C por 7 min.
Se estandarizaron las condiciones de la reacción de PCR en un volumen final de 25 µL. Se determinó la temperatura óptima de hibridación (60 a 65 °C) y la concentración óptima de los oligonucleótidos (entre 0,2 a 1uM), la concentración óptima final de MgCl2, entre 1mM y 3mM. Todos los ensayos fueron realizados empleando 1 unidad (U) de enzima Ampli Taq Gold DNA Polimerase (Applied Biosystem, USA) por tubo de reacción, usando el termociclador Amplitron II (Thermolyne)
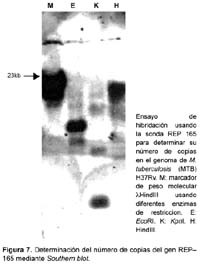
Finalmente, se determinó el número de copias del gen REP165 perteneciente a las secuencias repetitivas de la familia REP13E12 presentes en el genoma de M. tuberculosis H37Rv. Para ello 10 ìg de ADN genómico fueron digeridos con 50 U de las enzimas KpnI, EcoRI y Hind III. Los fragmentos fueron separados por electroforesis en agarosa al 1%, transferidos a una membrana de nylon (N+) y fijados por acción de luz ultravioleta. La membrana fue incubada con buffer de hibridación (Amersham Pharmacia Biotech) empleando como sonda el fragmento amplificado y el marcaje se realizó empleando el Direct Labelling Nucleic Acid and detection system (Amersham Pharmacia Biotech) registrando las señales mediante autoradiografía en film.
EVALUACIÓN DE LA SENSIBILIDAD Y ESPECIFICIDAD ANALÍTICA
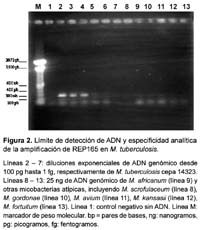
La sensibilidad analítica de la prueba de PCR fue evaluada mediante la determinación del límite de detección o mínima cantidad de ADN requerida, lo cual fue realizado utilizando diluciones exponenciales (desde 10 ng hasta 1 fg) de ADN genómico extraído18 de un cultivo joven (20-30 días) de M. tuberculosis en medio sólido LJ. Luego los productos de PCR fueron sometidos a una electroforesis en gel de agarosa al 1,5% y visualizados mediante tinción con bromuro de etidio al 1%. Para evaluar la especificidad analítica se incluyeron en el estudio ADN genómico de micobacterias no tuberculosas: Mycobacterium kansasii, M. bovis, M. gordonae, M. avium, M. fortuitum y M. scrofulaceum obtenidas del Laboratorio de Referencia Nacional de Micobacterias del Instituto Nacional de Salud, así como ADN de M. leprae obtenido a partir de una biopsia de un paciente con confirmación diagnóstica del Hospital Nacional Cayetano Heredia en Lima.
RESULTADOS
AMPLIFICACIÓN POR PCR DE LAS SECUENCIAS REP165
La alineación múltiple de las secuencias de la familia REP13E12 mostró un alto grado de conservación nucleotídica; especialmente entre REP165 (Rv1148c) y REP09F9 (Rv1945); así como en REP336 (Rv1587c) y REP251 (Rv0094c). Además, existe 100% de identidad en las secuencias REP165 y REP09F9 en distintas cepas de M. tuberculosis, tales como Erdman, H37Rv, CDC1551 y YP; y una divergencia con secuencias homólogas a REP en M. leprae, M. bovis, entre otras. La secuencia y características de los oligonucleótidos TB-REP165F y TB-REP165R, diseñados en el presente estudio (Tabla 1), amplifican una región interna de 318 pb de la secuencia repetitiva REP165 y REP09F9.
Además, los resultados del alineamiento sugieren que los oligonucleótidos diseñados también hibridizan parcialmente con las secuencias REP336 y REP251, rindiendo un producto de tamaño idéntico.
Por otro lado, mediante una búsqueda en el programa BLAST del servidor NCBI (http://www.ncbi.nlm.nih.gov/BLAST/) los oligonucleótidos hibridizan con secuencias de M. tuberculosis, M. bovis y no hibridizan con ADN de otras micobacterias tales como M. leprae, M. kansasii, M. avium.
SENSIBILIDAD Y ESPECIFICIDAD ANALÍTICA DE LA AMPLIFICACIÓN DE REP165
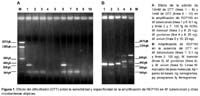
La sensibilidad de la reacción fue evaluada mediante diluciones exponenciales de ADN genómico. La sensibilidad y especificidad mejoró notablemente con la adición de 1mM de dithiotheitol (DTT), detectándose una única banda de 318 pb. Se logró detectar hasta 100 fg de ADN, equivalente a 20 microorganismos20 (Figuras 1 y 2). No se detectó producto en M. kansasii, M. gordonae, M. avium, M. fortuitum, ni M. scrofulaceum.
EVALUACIÓN DE LA AMPLIFICACIÓN DE REP165 EN MUESTRAS CLÍNICAS
En todas las muestras de esputo de los pacientes del grupo A se distinguió una banda de 318 pb, rindiendo un resultado positivo para la prueba, lo cual corresponde a un 100% de sensibilidad. Asimismo, en ninguno de los pacientes del grupo B se detectó una banda, siendo negativa la prueba, correspondiendo a una especificidad de 100%.
DISCUSIÓN
En las últimas décadas el desarrollo y la aplicación de las técnicas moleculares han iniciado una revolución en el diagnóstico y seguimiento de las enfermedades infecciosas. Dichas técnicas han logrado mejorar la sensibilidad, especificidad, y acortar el tiempo de diagnóstico de muchas enfermedades, incluyendo TB. Las secuencias de inserción IS6110 han sido ampliamente usadas en ensayos de PCR para el diagnóstico de la tuberculosis8-10,13. Sin embargo, se han reportado resultados falsos negativos debido a que se han aislado cepas que carecen de la secuencia IS6110 21. Además, resultados falsos positivos también han sido descritos en diversos estudios. Esto probablemente sea debido a la existencia de secuencias homólogas similares a IS6110 en otras bacterias, incluyendo micobacterias no tuberculosas, tal como se encuentra descrito22.
Otro ensayo basado en PCR para amplificar una región del gen mtp40 fue descrito por Del Portillo14. Este método es especie-específico para M. tuberculosis, y puede discriminar M. bovis de M. tuberculosis. No obstante, se ha reportado que mtp40 no se encuentra presente en todas las cepas de M. tuberculosis23 limitando así su aplicación en el diagnóstico de TB.
Con la disponibilidad de la secuencia genómica completa de la cepa de referencia Mycobacterium tuberculosis H37Rv18, es posible estudiar nuevas secuencias que podrían ser posibles blancos de amplificación para diagnósticos más sensibles y útiles por ejemplo para aplicarlos en casos paucibacilares. Especialmente convenientes son las secuencias repetitivas, las cuales se presentan en múltiples copias en el genoma y su detección mediante PCR presenta frecuentemente mayor sensibilidad comparada con aquellos métodos basados en blancos de secuencia única. Recientemente se ha descrito cerca de 56 loci con homología a secuencias repetitivas, entre ellas se identificó una nueva familia de secuencias repetitivas llamada REP13E12 17-18.
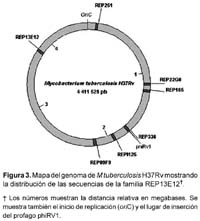
La familia REP13E12 está compuesta por un grupo de secuencias repetitivas similares a transposones, y se encuentran localizadas a lo largo del genoma de M. tuberculosis (Figura 3). Existen siete copias de ellas, las cuales presentan cierto grado de conservación de su secuencia nucleotídica. Las secuencias REP13E12 están presentes únicamente en miembros del complejo M. tuberculosis y ausentes en el genoma de otras micobacterias17,18. La alineación múltiple de las secuencias repetitivas REP13E12 sugiere que los oligonucleótidos teóricamente hibridizan tanto con las secuencias REP165 y REP09F9; como con REP336 y REP251. Aunque es necesario llevar a cabo más experimentos para demostrar la amplificación simultánea de dichas secuencias en el ensayo de PCR, en el presente trabajo fueron observadas bandas únicas en los resultados de amplificaciones. Adicionalmente, gracias al perfil de restricción enzimática e información nucleotídica completa (Figuras 4 y 5) del elemento repetitivo amplificado, la figura 6 muestra que el gen REP165 se presenta en copia única en el genoma de M. tuberculosis H37Rv, aunque al observar algunas hibridaciones inespecíficas podrían representar a las secuencias REP09F9, REP336 y REP251, debido a las condiciones de moderada especificidad en el Southern blot.
Por otro lado, este método de amplificación detectó hasta 100 fg, lo cual es similar a sistemas de PCR anteriormente descritos para secuencias repetitivas16,20,24. La amplificación de REP165 resultó negativa para las diferentes especies de micobacterias evaluadas que no pertenecen al complejo M. tuberculosis. A pesar que la prevalencia de micobacterias no tuberculosas es baja en el Perú, el ensayo de PCR podría ser capaz de discriminar entre infección con M. tuberculosis e infección con otras micobacterias atípicas. Luego de evaluar muestras de ADN de M. bovis se considera que la amplificación de REP165 mediante este método no discrimine entre las especies M. tuberculosis y M. bovis. Lee et al.25, reportaron secuencias repetitivas de 453 bp presentes en M. tuberculosis y M. bovis y ausentes en M. kansasii, M. smegmatis, M. simiae, M. fortuitum, M. scrofulaceum, M. intracellulare, M. avium, and M. haemophilum, los cuales fueron posteriormente identificadas como REP13E12 25. La presencia de las secuencias REP13E12 en 40 aislamientos diferentes de M tuberculosis, así como en múltiples cepas de referencia tales como H37Rv y H37Ra; Erdman, Canetti25, sugieren que estas secuencias podrían ser genéticamente estables.
Al evaluar en muestras de esputo proveniente de pacientes diagnóstico de TB positivo y negativo tanto por cultivo como por baciloscopía, encontramos 100% de sensibilidad y 100% de especificidad. Sin embargo, no fueron analizadas muestras de pacientes con TB baciloscopía negativa y cultivo positivo, ni TB con baciloscopía y cultivo negativo, por lo que son necesarios futuros estudios con un adecuado número y variado tipo de muestras clínicas, para determinar la utilidad de aplicar este método en el diagnóstico de la tuberculosis.
En resumen, se reporta un nuevo blanco de amplificación para la detección específica de miembros del complejo M. tuberculosis, el cual presenta elevada sensibilidad in vitro en esta evaluación preliminar con muestras de esputo. Esta prueba de PCR puede ser aplicable en casos de difícil diagnóstico, tales como TB paucibacilar, debiendo ser evaluado posteriormente.
REFERENCIAS BIBLIOGRÁFICAS
1. Raviglione MC. The TB epidemic from 1992 to 2002. Tuberculosis (Edinb) 2003; 83(1-3): 4-14. [ Links ]
2. Perú, Ministerio de Salud. Norma técnica de salud para el control de la tuberculosis. Lima: Estrategia Sanitaria Nacional de Prevención y Control de la Tuberculosis / MINSA; 2006.
3. Perú, Ministerio de Salud. Construyendo alianzas estratégicas para detener la tuberculosis: la experiencia peruana. Lima: MINSA; 2006.
4. Perkins MD. New diagnostic tools for tuberculosis. Int J Tuberc Lung Dis 2000; 4(12 Suppl 2): S182-88.
5. Laszlo A. Tuberculosis: 7. Laboratory aspects of diagnosis. CMAJ 1999; 160(12): 1725-29.
6. American Thoracic Society. Diagnosis standards and classification of tuberculosis in adults and children. Am J Respir Crit Care Med 2000; 161(4 pt 1): 1376-95.
7. Ieven M, Goossens H. Relevance of nucleic acid amplification techniques for diagnosis of respiratory tract infections in the clinical laboratory. Clin Microbiol Rev 1997; 10(2): 242-56.
8. Bennedsen J, Thomsen VO, Pfyffer Ge, Funke G, Feldmann K, Beneke A, et al. Utility of PCR in diagnosing pulmonary tuberculosis. J Clin Microbiol 1996; 34(6): 1407- 11.
9. Cohen RA, Muzaffar S, Schwartz D, Bashir S, Luke S, McGartland LP, et al. Diagnosis of pulmonary tuberculosis using PCR assays on sputum collected within 24 hours of hospital admission. Am J Respir Crit Care Med 1998; 157(1): 156-61.
10. Cheng VC, Yam WC, Hung If, Woo PC, Lau SK, Tang BS, et al. Clinical evaluation of the polymerase chain reaction for the rapid diagnosis of tuberculosis. J Clin Pathol 2004; 57(3): 281-85.
11. Sarmiento OL, Weigle KA, Alexander J, Weber DJ, Miller WC. Assessment by meta-analysis of PCR for diagnosis of smear-negative pulmonary tuberculosis. J Clin Microbiol 2003; 41(7): 3233-40.
12. Soini H, Shurnik M, Liippo K, Tala E, Viljanen MK. Detection and identification of mycobacteria by amplification of a segment of the gene coding for the 32- kilodalton protein. J Clin Microbiol 1992; 30(8): 2025-28.
13. Pierre C, Lecossier D, Boussougant Y, Bocart D, Joly V, Yeni P, et al. Use of a reamplification protocol improves sensitivity detection of Mycobacterium tuberculosis in clinical samples by amplification of DNA. J Clin Microbiol 1991; 29(4): 712-17.
14. Herrera EA, Segovia M. Evaluation of mtp40 genomic fragment amplification for specific detection of Mycobacterium tuberculosis in clinical specimens. J Clin Microbiol 1996; 34(15): 1108-13.
15. Prabhakar S, Mishra A, Singhal A, Katoch VM, Thakral SS, Tyagi JS, et al. Use of the hupB gene encoding a histone-like protein of Mycobacterium tuberculosis as a target for detection and differentiation of M. tuberculosis and M. bovis. J Clin Microbiol 2004; 42(6): 2724-32.
16. Saves I, Lewis LA, Westrelin F, Warren R, Daffe M, Masson JM. Specificities and functions of the recA and pps1 intein genes of Mycobacterium tuberculosis and application for diagnosis of tuberculosis. J Clin Microbiol 2002; 40(3): 943-50.
17. Gordon SV, heym B, Parkhill J, Barrell B, Cole ST. New insertion sequences and a novel repeated sequence in the genome of Mycobacterium tuberculosis H37Rv. Microbiology 1999; 145(Pt 4): 881-92.
18. Cole ST, Brosch R, Parkhill J, Garnier T, Churcher C, harris D, et al. Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence. Nature 1998 ; 393(6685): 537-44.
19. Popovic T, Bopp C, Olsvik O, Kiehbaluch J. Ribotyping in molecular epidemiology, p. 573. In Persing DH, Smith TH, Tenover FC, White TJ (ed.). Diagnostic molecular microbiology: principles and applications Washington, D.C: American Society for Microbiology; 1993.
20. Drake WP, Pei Z, Pride DT, Collins RD, Cover TL, Blaser MJ. Molecular analysis of sarcoidosis tissues for Mycobacterium species DNA. Emerg Infect Dis 2002; 8(11):1334-41.
21. Lazraq R, el Baghdadi J, Guesdon JL, Benslimane A. Evaluation of IS6110 as amplification target for direct tuberculosis diagnosis. Pathol Biol (Paris) 1999; 47(8): 790-96.
22. Kent L, Mchugh TD, Billington O, Dale JW, Gillespie SH. Demonstration of homology between IS6110 of Mycobacterium tuberculosis and DNAs of other Mycobacterium spp.? J Clin Microbiol 1995; 33(9): 2290- 93.
23. Weil A, Plikaytis BB, Butler WR, Woodley CL, Shinnick TM. The mtp40 gene is not present in all strains of Mycobacterium tuberculosis. J Clin Microbiol 1996; 34(9): 2309-11.
24. Magdalena J, Vachee A, Supply P, Locht C. Identification of a new DNA region specific for members of Mycobacterium tuberculosis complex. J Clin Microbiol 1998; 36(4): 937-43.
25. Lee TY, Lee TJ, Belisle JT, Brennan PJ, Kim SK. A novel repeat sequence specific to Mycobacterium tuberculosis complex and its implications. Tuber Lung Dis 1997; 78(1): 13-19.
Correspondencia:
Blgo. Róger I. Calderón Espinoza.
Departamento de Biología Molecular, Laboratorio Clínico Blufstein. Lima, Perú.
Dirección: Av. Jorge Basadre 1133, San Isidro. Lima, Perú.
Teléfono: (511) 222-0550 anexo 121
Correo electrónico: rivance@yahoo.com