Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.25 no.1 Lima Jan./mar 2008
ARTÍCULOS ORIGINALES
Características clínicas y moleculares de un brote de influenza en dos bases militares, Tumbes- Perú, 2007
Clinical and molecular characteristics of influenza outbreak in two military bases in Tumbes, Peru 2007
Tatiana Saldarriaga1,a, V. Alberto Laguna-Torres1,a,d , Juan Arrasco2,a, Luis Guillén3,b, Juan Águila3,b, Patricia Aguilar1,d, Merly Sovero1,c, Juan Pérez1,e, Jane Ríos1,c, María Gamero1,c, Rommell González4,a, Tadeusz Kochel1,d
1 US Naval Medical Research Center Detachment (NMRCD). Lima, Perú.
2 Oficina de Epidemiología, Dirección Regional de Salud Tumbes. Tumbes, Perú.
3 1era Brigada de Infantería Ejército del Perú. Tumbes, Perú.
4 Centro de Salud Zarumilla, Ministerio de Salud. Tumbes, Perú.
a Médico; b Enfermero Militar; c Bióloga; d Doctor en Ciencias; e Ingeniero analista / programador
RESUMEN
Objetivo. Describir un brote de Influenza en dos bases militares en Tumbes, Perú. Material y métodos. Se utilizó la definición de caso de síndrome gripal del Ministerio de Salud en casos con menos de cinco días de inicio de síntomas. Se tomó hisopado nasal para la prueba rápida de Influenza (PRI) e hisopado faríngeo para aislamiento viral en tres líneas celulares (MDCK, VERO, LLCMK2). Para la genotipificación de Influenza B se usó un secuenciamiento parcial de la región de hemaglutinina de 898 pares de bases teniendo como base la cepa de la vacuna del 2007 (B/Florida/4/2006) y para el tipo A se analizó 958 pares de bases teniendo como referencia la cepa vacunal del 2007 (A/Brisbane/10/2007 (H3N2). Resultados. Se tomó 124 hisopados, 20 de la base militar (BM) # 1 y 104 de la BM # 2. La PRI identificó a 41 casos positivos: Flu B: 27, Flu A: 02, Flu: 12, con 62% de sensibilidad y 100% de especificidad. Finalmente, 66 casos fueron confirmados por cultivo celular, 54 Flu B y 12 Flu A. Los aislamientos de Tumbes fueron genéticamente similares con la cepa B/Texas/4/ 2006 (n=34) y A/Texas/91/2007 (H3N2)/ (n=7). Los síntomas más comunes fueron fiebre, cefalea, malestar general, tos, dolor de garganta, mialgias y rinorrea. Las medidas de control consistieron en el aislamiento de los casos identificados y el uso de mascarillas. Conclusiones. Se confirmó 66 casos de Influenza por cultivo celular. Los aislamientos tuvieron la mayor similitud genética con las cepas denominadas B/Texas/4/2006 y A/Texas/91/2007 (H3N2).
Palabras clave: Influenza humana; Brote; Personal militar; Perú (fuente: DeCS BIREME).
ABSTRACT
Objective. To describe an Influenza outbreak in two military bases in Tumbes, department located in northern Peru. Material and methods. In patients within 5 days of symptoms the Ministry of Health case definition of Influenza-like illness was used. Nasal swabs were taken for Rapid Influenza Test (RIT) and throat swab for viral isolation, three cell lines (MDCK, VERO, LLCMK2) were used. For Influenza B genotypification we made a partial sequencing of hemaglutinine region of 898 pair bases, having 2007 vaccine strain B/Florida/4/2006 as a model. For Influenza A genotypification we analysed 958 pair bases, having 2007 vaccine strain A/Brisbane/10/2007 (H3N2) as a model. Results. We took 124 samples, 20 from military base (MB) # 1 and 104 from MB # 2. RIT promptly identified 41 positive cases: Flu B: 27, Flu A: 02, Flu: 12, with 62% of sensibility and specificity 100%. Finally, 66 cases were confirmed by virus isolation, 54 Flu B and 12 Flu A. Genotypification showed that isolates were genetically similar to Influenza B/Texas/4/2006 (n=34) and A/Texas/91/2007 (H3N2) (n=7). Most common symptoms found were fever, headache, malaise, cough, sore throat, myalgias and rinorrheae. Control strategies consisted to identify probable cases and to isolate them in a special room and to wear facemasks. Conclusions. We confirmed 66 cases by cellular culture. Isolates were genetically similar to Influenza B/Texas/4/2006 and A/Texas/91/2007 (H3N2).
Key words: Human Influenza; Outbreak, Militar personnel; Peru (source: DeCS BIREME).
INTRODUCCIÓN
El virus de la Influenza causa una infección respiratoria aguda altamente contagiosa que puede autolimitarse o causar graves epidemias o pandemias de gran impacto para la salud pública (1). El virus Influenza es muy infeccioso y fácilmente transmisible por la tos, estornudo, pañuelos o ropa contaminada. Normalmente, las partículas se depositan en el epitelio respiratorio y son barridas por los cilios de la membrana mucosa; aunque muchas veces estas micropartículas llegan directamente al tejido alveolar, burlando el mecanismo primario de defensa. Infectan rápidamente, se adhieren a las células epiteliales del huésped y las penetran por endocitosis (2).
Aun cuando es autolimitada, esta infección es capaz de alterar el normal desenvolvimiento de las actividades productivas al ocasionar incapacidad temporal para realizar actividades cotidianas. Asimismo, pueden aparecer complicaciones cardíacas, neurológicas y otros problemas graves como miositis (3) y síndrome del choque tóxico (4). Una peculiaridad de este virus es la interacción que tiene con bacterias patógenas que producen infecciones del parénquima pulmonar como el Streptococcus pneumoniae (5,6). Es con esta bacteria con quien tiene un sinergismo letal en el que la neuraminidasa cumple una importante función (7).
Los virus de la Influenza son genéticamente inestables y fácilmente pueden eludir la respuesta inmune del huésped mediante cambios en sus genes, que se traduce en modificaciones en los antígenos de superficie. Cambios menores llamados deriva antigénica (antigenic drift) suceden frecuentemente y resultan en la aparición de nuevas cepas en un mismo subtipo. Cambios mayores llamados salto antigénico (antigenic shift) (8) que resultan en la aparición de un nuevo virus con nuevas proteínas de superficie, hemaglutinina (H) y neuraminidasa (N), muy distinto a los que circularon previamente, este salto antigénico generalmente se produce por el intercambio genético entre diferentes virus (humanos y animales). Esta capacidad de adaptación que poseen los virus de la Influenza para eludir las defensas del huésped explicaría los ciclos endémicos, epidémicos y pandémicos que se suceden en el tiempo (9).
Las pandemias del siglo XX, se han caracterizado por su alta letalidad. La primera de ellas, llamada La Gripe Española (1918-1919), al parecer llegó al Perú recién en marzo de 1919. La siguiente pandemia en 1957, se inició al norte de China y rápidamente se diseminó a todo el mundo, llegando a Perú en julio de ese mismo año. Durante esta pandemia en el Perú, se notificaron 1081 defunciones (letalidad de 0,8%). Finalmente en 1968, denominada como La Gripe Asiática, se extendió rápidamente por todo el mundo, con alta morbilidad pero menor letalidad que las anteriores (10).
En la actualidad, el virus de la gripe aviar se está esparciendo cada vez más. Desde 2003, ha infectado no sólo a aves, sino también a seres humanos. Las cifras de La Organización Mundial de la Salud (OMS), muestran, según el último reporte del 25 de octubre del 2007, que un total de 332 personas han contraído la enfermedad, 204 de los cuales han fallecido, vale decir el 61% de la población infectada (11). Desde fines del 2003 hasta el 29 de mayo del 2007, son 59 los países con focos de Influenza aviar H5N1 en el mundo, siendo los más afectados China, Egipto, Indonesia, Tailandia y Vietnam (12).
Considerando que ya han ocurrido algunas mutaciones del virus, la gripe aviar representa una amenaza real para América Latina y el Caribe. La progresión de la enfermedad puede ser muy rápida. Se estima que se puede propagar en menos de 24 horas de un continente a otro considerando la gran cantidad de viajes internacionales.
Si ocurriese una pandemia de gripe aviar, la enfermedad puede llegar a cualquier país a través de un simple vuelo aéreo. A esto se suma la presencia de una variante de H5N1 encontrada en aves en Canadá que permitiría que este virus circule en América Latina y también el reporte de epizootias por Influenza aviar subtipo H9 (baja patogenicidad) en Colombia (13) y H7N3 (alta patogenicidad) en Chile (14), lo que indica que la región tiene riesgo de Influenza aviar por existir rutas migratorias permanentes de aves y por que éstas constituyen reservorio natural del virus H5N1. En el Perú, no existen estudios que indiquen que la Influenza tenga un comportamiento estacional a semejanza de otros países tropicales, son escasos los estudios peruanos publicados sobre Influenza (15-17). El Instituto Nacional de Salud (www.ins.gob.pe) reporta el aislamiento de virus Influenza en todo el año, con incrementos durante el invierno y a finales del año. En los últimos años, la Dirección General de Epidemiología ha descrito brotes epidémicos de Influenza en algunas comunidades indígenas amazónicas, en especial en aquellas de reciente contacto o en instituciones con poblaciones cautivas como centros penitenciarios (12).
En el Perú existe una vigilancia epidemiológica de Influenza y otros virus respiratorios basada en la directiva N.º 057-2005-MINSA-DGE, que define el síndrome gripal (18) y que incluye a los establecimientos del Ministerio de Salud (MINSA), el Naval Medical Research Center Detachment (NMRCD) participa en este sistema desde mayo del 2006. En el departamento de Tumbes se ha implementado la vigilancia centinela de Influenza en los Centros de Salud de Zarumilla y Pampagrande, basándose en los lineamientos del Plan Nacional de Preparación y Respuesta Frente a una Potencial Pandemia de Influenza (10) y durante los últimos años se han notificado casos de Influenza. En estos establecimientos de salud, durante el año 2007, hasta la semana epidemiológica 24 se ha confirmado 156 casos de Influenza A y 53 de B, siendo el serotipo A H1N1 el predominante. Los genotipos del virus de Influenza B que con mayor frecuencia se han encontrado son los genéticamente similares al tipo Hong Kong, Shanghai y Arizona.
En la semana epidemiológica 15 del 2007 (abril) se notificó a la Oficina de Epidemiología de Tumbes, casos de síndrome gripal procedentes de dos cuarteles de la guarnición militar de Tumbes, Perú (cuartel #1 y cuartel #2). El objetivo del presente trabajo es describir los genotipos circulantes y las características clínico epidemiológicas de un brote de Influenza A y B ocurrido en personal militar en el departamento de Tumbes, Perú.
MATERIALES Y MÉTODOS
Para la investigación de este brote se utilizó la definición de caso de síndrome gripal del MINSA identificando como caso a todo aquel que presentaba temperatura mayor o igual de 38 ºC de inicio brusco y con tos o dolor de garganta que podía o no estar acompañada de síntomas sistémicos como mialgias, postración, cefalea o malestar general (18). Personal del Centro de Salud de Zarumilla y de Epidemiología de la Dirección de Salud (DISA) de Tumbes, brindó apoyo técnico para ejecutar las medidas de tratamiento y control de los mencionados casos.
Se realizó entrevistas y exámenes médicos dirigidos, en cada paciente, consignándose los datos en la ficha de vigilancia de virus respiratorios del NMRCD y del MINSA. La ficha del MINSA fue entregada al personal encargado de su análisis.
Todos los pacientes con síntomas sistémicos que no podían continuar con sus actividades diarias fueron internados en la enfermería de la base militar. Se les administró antipiréticos y adecuada hidratación.
El aislamiento viral fue la prueba confirmatoria utilizada por el NMRCD. Con este fin, se estableció que se obtendría muestra para laboratorio a todos los casos de síndrome gripal que presentaban menos de cinco días de iniciado los síntomas. Se obtuvo dos tipos de muestra para diagnóstico, un hisopado nasal para la ejecución de prueba rápida (QUICKVUE Influenza A+B Test°) y un hisopado de secreción faríngea con el fin de realizar, específicamente, aislamiento viral. Los hisopados se obtuvieron con hisopos de dacrón.
La prueba rápida se procesaba in situ al colocar el hisopo con secreción nasal en un tubo con una solución de extracción de antígenos víricos, que ante la presencia de virus de Influenza A o B, deja expuestas las nucleoproteínas víricas. Luego se introducía la tira de prueba y si la muestra tenía antígenos daba una reacción de color entre los dos y diez minutos. Si era positivo a Influenza aparecía una línea de color rojo (línea de prueba) y una azul de control del procedimiento, mientras que si era negativo aparecía únicamente una línea azul. Inicialmente fue utilizada una versión de este kit que no discriminaba el tipo de Influenza, solamente si era positivo o negativo. Cuando el color rojo aparecía por encima de la línea de control, se calificaba como Influenza A y cuando por debajo como Influenza B. El resultado de la prueba rápida se brindaba al paciente en el momento de la evaluación.
La muestra de secreción faríngea fue colocada y almacenada en tubos con medio de transporte los cuales se congelaban a una temperatura de –70 °C. Las muestras fueron enviadas en cajas con hielo seco hacia el NMRCS en Lima, Perú. En Lima se sembró tres líneas celulares: para el aislamiento del virus de Influenza se utilizó células Madin-Darby de riñón canino (MDCK por sus siglas en inglés), así como Células de riñón de mono verde (VERO) y células LLCMK2, para identificar virus respiratorio sincitial (VRS), adenovirus, coxsackie, herpes virus y parainfluenza tipos 1, 2 y 3. Después de los diez días se procedió a la identificación viral, por medio de la prueba de inmunofluorescencia directa (IFD) para todos los virus antes descritos menos para el enterovirus en el cual se realizaba a la inmunofluorescencia indirecta (IFI).
El RNA se aisló del sobrenadante de células MDCK para el cual se utilizó el QIAamp Viral RNA kit (QIAGEN°, Valencia, CA) siguiendo los parámetros dados por la casa productora. Con el RNA obtenido se desarrolló la trascripción reversa y posteriormente la reacción en cadena de la polimerasa (RT-PCR) en un solo paso utilizando cebadores de la región de la hemaglutinina para identificar la Influenza A (subtipo H3 y H1) e Influenza B con el SuperScript III One-Step RT-PCR System kit (Invitrogen°, San Diego, CA). El RT-PCR fue desarrollado utilizando primers específicos (19) para 35-43. Brote de Influenza en militares cada subtipo de Influenza A, H1 (H1F-6 y H1R-1193), H3 (H3F-7 y R-1184) y para Influenza B (BHAF-36 y BHAR-1140) recomendados por Air Force Institute for Operational Health (AFIOH). Los productos del PCR fueron purificados utilizando Centri-Sepn Columns (Princeton Separation) y secuenciados utilizando Big Day Terminator (Applied Biosystems°, Foster City, CA) de acuerdo con las instrucciones del fabricante. Las muestras fueron analizadas con el software Secuencher 4.8 (Applied Biosystems°, Foster City, CA)
Para el virus de Influenza B se realizó un secuenciamiento parcial de la región de la hemaglutinina de 898 pares de bases (posiciones 61-959) posiciones (140-1097). La secuencia de nucleótidos fue analizada utilizando el programa Sequencer Genes Codes Corporation. Y luego fue ingresada en el GenBank para compararla con los subtipos circulantes de Influenza en el ámbito mundial. La elaboración de los árboles filogenéticos se realizaron con el Mega 4.0 utilizando el método de Neighbor-Joing.
Se analizaron los resultados consolidando por separado los casos de cada base militar (cuartel #1 y cuartel #2.)
Como parte del trabajo colaborativo con el Ministerio de Salud, un hisopo faríngeo se entregó al Laboratorio Referencial de Tumbes para su procesamiento por IFI.
RESULTADOS
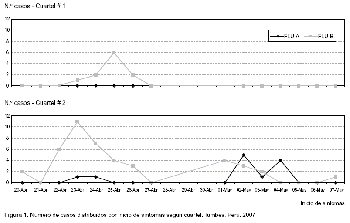
Un total de 350 casos de síndrome gripal fueron notificados de ambos cuarteles 200 del cuartel #1 y 150 en el cuartel #2. De ellos se obtuvieron 124 muestras de hisopado faríngeo, 20 del cuartel #1 y 104 en el cuartel #2. Las muestras se obtuvieron entre el 20 de abril hasta el 7 de mayo del 2007 (Figura 1).
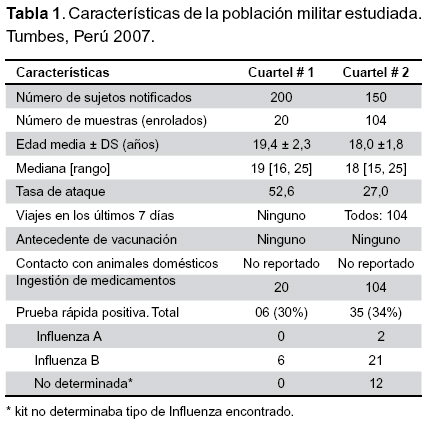
La edad promedio de los pacientes fue de 18,7 ± 1,8 años [15, 25] en el cuartel #1 y de 19,4 ± 2,3 [15, 25] en el cuartel # 2 (Tabla 1). Sólo una persona era del sexo femenino. El 16% de los casos se encontraba febril al momento de la visita y toma de muestra, pero todos tenían el antecedente febril dentro de los cinco días anteriores a la toma de muestra.
Un total de 41 (33%) muestras fueron positivas para Influenza por la prueba rápida, de ellos, 27 casos fueron del tipo B y 2 del tipo A. En otros 12 casos la prueba fue positiva para Influenza pero el kit usado no determinaba el tipo de virus Influenza encontrado (Tabla 1). Los resultados de la prueba rápida se compararon con los resultados obtenidos por aislamiento. Se obtuvo 41 muestras positivas por la prueba rápida, sin embargo se lograron aislar 66 cepas, siendo la sensibilidad de la prueba rápida de 62,1% (41/66) y la especificidad fue del 100% (58/58).
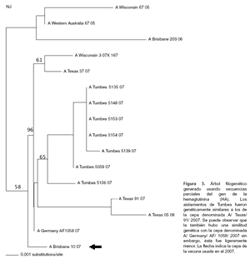
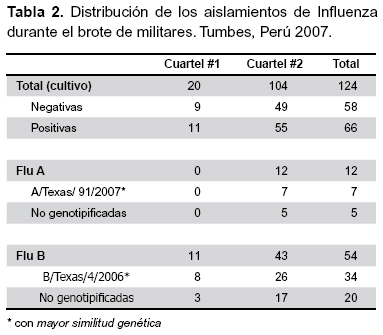
Por cultivo celular, 66 (53%) muestras fueron positivas. De ellas 54 fueron Influenza del tipo B y 12 del tipo A (Tabla 2). Se genotipificó al 63% de las muestras en las cuales se aisló Influenza de tipo B y el 58% de las muestras positivas a virus de Influenza A. La tipificación del tipo B mostró que las muestras de Tumbes fueron genéticamente similares con la cepa denominada B/Texas/4/2006 (n=34). Asimismo, hubo similitud genética con otros virus como B/Arizona/148/2005 y B/Johannesburg/ 501/ 2005; sin embargo, esta similitud fue menor a la cepa anteriomente mencionada. Los aislamientos del tipo A mostraron gran similitud genética con la cepa denominada A/ Texas/91/2007 (n=7). Las figuras 2 y 3 muestran los árboles filogenéticos de los virus de Influenza B y A, respectivamente, encontrados durante el brote.
El 100% de los casos del cuartel #2 había realizado viaje al cuartel #1 para actividades de entrenamiento. El tiempo promedio de duración de los síntomas fue de cinco a siete días. No se registraron complicaciones ni defunciones por Influenza. Los síntomas más frecuentes fueron fiebre o historia de fiebre 100%, cefalea 98%, malestar general 97%, tos 95%, dolor de garganta 95%, rinorrea 90%, mialgias 89%, faringe congestiva 85%. Otro signo menos frecuente encontrado fue la presencia de vómitos en tres casos (2%). (Tabla 2) Ninguno de los casos refirió haber tenido contacto o manipulado animales domésticos o silvestres
El tratamiento indicado para los casos fue paracetamol 500 mg VO 01 tableta qid, así como metamizol 1 g IM, más diclofenaco 75 mg IM si presentaban fiebre mayor o igual a 38,5 ºC. Ninguno de los militares recibió vacuna contra la Influenza ni como parte de las medidas de control, ni como profilaxis.
Se realizó aislamiento en habitaciones no individuales temporalmente acondicionadas para dicho fin sin un patrón específico para la distancia entre cama y cama. Al personal de salud y los pacientes se les alcanzó mascarillas de tipo quirúrgico.
DISCUSIÓN
La infección por el virus Influenza constituye un problema de salud pública importante que en el hombre se presenta como epidemias recurrentes y ocasionalmente como pandemias impredecibles (20).
En el departamento de Tumbes, las actividades de vigilancia centinela nos han permitido reconocer la continua circulación de virus Influenza durante todo el año con predominio de los meses de invierno, aunque como se sabe en climas tropicales como el nuestro puede darse luego de cambios bruscos de temperaturas (21). El presente brote tuvo como agente etiológico predominante al virus Influenza B, el cual está caracterizado por ser un virus causante de enfermedad menos grave que el A.
Desde el inicio de la vigilancia en los centros de salud de Pampagrande y Zarumilla, se han captado 948 hisopados faríngeos y 379 (40%) fueron positivas. El tipo de virus respiratorio predominantemente encontrado fue el de Influenza A en 174 casos (45,9%), seguido de Influenza B en 108 casos (28,5%) Otros virus respiratorios encontrados son adenovirus 8,8%, parainfluenza 1,2 ó 3 (5,6%), virus herpes simple (5,1%). En el área de Tumbes se ha aislado los serotipos de Influenza A A/H1N1, A/ H3N2. La distribución genotípica de virus Influenza B nos permitió reconocer a dos cepas circulantes prevalentes y que son parecidas genéticamente a la cepa circulante en HONG KONG y SHANGHAI.
La cepa que circuló en este brote, que también es muy parecida genéticamente a la de Texas, es de reciente reconocimiento en el departamento de Tumbes. Durante todo el brote el tipo de virus Influenza predominante fue el B, sin embargo, los casos de Influenza A fueron aumentando paulatinamente en el tiempo hasta casi igualarse al final del brote (Figura 1).
Los casos rápidamente se diseminaron de un cuartel a otro, ya que soldados de los distintos cuarteles de Tumbes tuvieron entrenamiento conjunto. Así mismo, el contacto que establecen durante el servicio de guardia de prevención facilitó la propagación entre soldados de distintos pabellones de un mismo cuartel.
Las personas afectadas presentaron un cuadro clínico moderado caracterizado por inicio brusco de fiebre, acompañada de síntomas generales a los cuales entre las 12 y 24 horas y que después se asociaron síntomas respiratorios, que restringieron parcialmente las actividades diarias pero que no presentaron complicaciones o defunciones. Esto probablemente por tratarse de una población joven entre 15 y 25 años de edad, aparentemente sanos, inmunocompetentes, sin enfermedades asociadas o que tenían inmunidad parcial a la cepa circulante. Un número mucho menor de pacientes (n=3) presentó vómitos en el curso de la enfermedad. La tasa de ataque fue de 52,6 y 27% para los cuarteles 1 y 2 respectivamente, valores que son comparables a estimaciones de otros autores en brotes de poblaciones que viven en contacto directo (22).
Las epidemias y brotes de Influenza están asociadas con un exceso de morbilidad y mortalidad, incluso en las epidemias de baja intensidad. Durante una epidemia moderada, alrededor del 10% de la población mundial contraería Influenza, ocasionado un fuerte impacto económico, debido a la pérdida de productividad laboral y a los costos derivados de la atención de pacientes (23).
Si bien existen cuatro fármacos aprobados para el tratamiento y prevención de Influenza A que son amantadina, rimantadina, oseltamivir y zanamivir; a los pacientes de este brote no se les aplicó tratamiento antiviral como está recomendado (24). El uso de profilaxis antiviral (amantadina o rimantadina), si se utiliza precozmente, reduce la morbilidad (TA) en 80%. Esta medida se ha utilizado con éxito para controlar brotes de gripe en residencias de la tercera edad, sin embargo estos fármacos, o bien no están disponibles o no se utilizan para el control de brotes en muchos países incluyendo el nuestro.
No fue posible, conforme está recomendado (25), aplicar la vacuna contra la Influenza debido a que no estuvo disponible antes ni durante el brote. No existe un programa de vacunación contra la Influenza para los militares.
Los casos fueron tratados con antinflamatorios, paracetamol, diclofenaco o metamizol por vía oral o intramuscular. La evolución fue favorable en 100% de los casos. Entendemos que esto es una lección aprendida para el personal que participó del tratamiento de este grupo de afectados.
La importancia del diagnóstico de Influenza está determinada por su alta capacidad de propagación y por la gravedad de sus posibles complicaciones. Las epidemias implican aumento significativo de morbilidad y mortalidad en la población general, con la consecuente demanda de atención ambulatoria y hospitalaria (24). El rápido diagnóstico e identificación de los casos permitió tomar medidas de control para disminuir el impacto del brote. El aislamiento temporal de algunos pacientes, el uso de mascarillas entre el personal de salud y pacientes así como la restricción de las salidas a la calle del personal militar disminuyó la diseminación a la población general. Es nuestra impresión que difícilmente este brote haya comenzado en la población militar. La circulación viral debe haber comenzado en la comunidad civil de donde ingresó a los cuarteles y se hizo evidente debido a que la tasa de ataque en estas poblaciones es mayor. Sería importante analizar los datos de cómo se comportaron las infecciones respiratorias agudas en los consultorios externos o en los servicios de emergencia de la ciudad de Tumbes, de tal manera que podríamos comprobar que hubo también un incremento.
Las regiones de hemaglutinina analizadas para Influenza A y B se encontraron pequeñas variantes en la secuencia de nucleótidos de las secuencias analizadas de Influenza A (H3N2), tres de siete cepas fueron exactamente iguales. En el caso de Influenza B el número de secuencias iguales correspondió a 21 cepas de 34. En ambos las cepas pertenecían a un solo cluster. Durante la presentación de estos datos estamos mostrando entonces la similititud genética de nuestros aislamientos con las cepas ya denominadas internacionalmente como B/Texas y A/Texas sin embargo con diferencias de algunos pares de bases la similitud también existe con otros como B/Arizona y A/Germany, sin embargo conforme mostramos en los árboles filogenéticos de las figuras 2 y 3 la similitud genética es mayor con los anteriormente mencionados. Para la comparación utilizamos las cepas vacunales del 2007.
Ya que el impacto de una pandemia de Influenza en nuestra región sería desastroso, recalcamos la importancia vital de mantener las actividades de vigilancia que el Perú ha desarrollado en su Plan de Respuesta basado en las recomendaciones internacionales de OMS, lo cual generará condiciones para mitigar las consecuencias de una eventual pandemia de Influenza. Sin embargo queda en la agenda de trabajo superar la limitada disponibilidad de recursos y lograr un mayor compromiso político y social frente a este problema. Por todo lo expuesto se hace urgente y necesario mantener e incrementar los esfuerzos hasta ahora realizados por el Ministerio de Salud en apoyo de la población para mantenernos alertas ante la posible emergencia de un brote de Influenza aviar.
AGRADECIMIENTOS
Disclaimer. Las opiniones y afirmaciones contenidas aquí son propias de los autores y no deben interpretarse como posición oficial o que reflejan la opinión del Departamento de la Marina o del servicio naval de los Estados Unidos. Financiamiento. Este trabajo ha sido financiado por la unidad de trabajo (Work Unit No. 847705 82000 25GB B0016). Comité de Ética. Este estudio (NMRCD.2002.0019) fue aprobado por el Comité de Ética del Centro se Investigación Médica Naval de acuerdo a todas las regulaciones federales que protegen a los sujetos humanos. Copyright. Algunos de los autores de este trabajo son personal que trabaja para el Ministerio de Defensa de los Estados Unidos y este trabajo ha sido preparado como parte de sus funciones oficiales.
REFERENCIAS BIBLIOGRÁFICAS
1. Cox NJ, Subbarao K. Influenza. Lancet. 1999; 354: 1277- 82.
2. Cross KJ, Burleigh LM, Steinhauer DA. Mechanisms of cell entry by Influenza virus. Expert Rev Mol Med. 2001; 3(21): 1-18.
3. Dietzman DE, Schaller JG, Ray CG, Reed ME. Acute myositis associated with Influenza B infection. Pediatrics. 1976; 57(2): 255-58.
4. Sperber S, Francis B. Toxic shock syndrome during an Influenza outbreak. JAMA. 1987; 257(8): 1086-87. 35-43. Saldarriaga T et al.
5. Schwarzmann SW, Adler JL, Sullivan RJ, Marine WM. Bacterial pneumonia during the Hong Kong Influenza epidemic of 1968-1969. Archives of Internal Medicine, 127. 1030-1041,1971
6. Mc Cullers JA. Insights into interaction between Influenza virus and pneumococcus. Clin Microbiol Rev. 2006; 19(3): 571-82.
7. Mc Cullers J.A, Bartmess KC. Role of neuraminidase in lethal synergism between Influenza virus and Streptococcus pneumoniae. J Infect Dis. 2003; 187(6): 1000-9.
8. Siegel JD, Rhinehart E, Jackson M, Chiarello L, Health Care Infection Control Practices Advisory Committee. 2007 Guideline for isolation precautions: Preventing transmission of infectious agents in healthcare settings. Am J Infect Control. 2007; 35(10 Suppl 2): S65-164.
9. Organización Mundial de la Salud. Respuesta a la amenaza de una pandemia de gripe aviar. Medidas estratégicas recomendadas. Ginebra: OMS; 2005. EHO/ CDS/CSR/GIP/2005.8
10. Perú, Ministerio de Salud. Plan nacional de preparación y respuesta frente a una potencial pandemia de Influenza. Lima: MINSA; 2006.
11. W orld Health Organization [página en Internet]. Influenza pandemic threat: current situacion. [Fecha de acceso: octubre 2007]. Disponible en: http://www.who.int/csr/disease/avian_Influenza/pandemic/en/index.html.
12. Dirección General de Epidemiología. Vigilancia epidemiológica en salud pública de la pandemia de Influenza aviar. Bol Epidemiol (Lima). 16(20): 288-89.
13. Senne DA. Avian Influenza in North and South America, 2002-2005. Avian Dis. 2007; 51(a Suppl): 167-73.
14. Suarez DL, Senne DA, Banks J, Brown IH, Essen SC, Lee CW, et al. Recombination resulting in virulence shift in avian Influenza outbreak, Chile. Emerg Infect Dis, 2004. 10(4): 693-99.
15. Mayca J, Torres de Yon Y, Capristano S, Farfán M. Brote de Influenza en Pucallpa, Ucayali, Perú. 2002. Rev Peru Med Exp Salud Publica. 2003; 20(4): 223-26.
16. Torres de Yon Y, Mayca J, Llanos-Zavalaga F, Velásquez J, Capristano S. Virus Influenza y el diagnóstico diferencial de sintomáticos febriles de la costa norte del Perú (mayo, 2001). Rev Peru Med Exp Salud Publica. 2004; 21(1): 12-17.
17. Ortiz EJ, Kochel TJ, Capuano AW, Setterquist SF, Gray GC. Avian Influenza and poultry workers, Peru 2006. Influenza Other Respir Viruses. 2007; 1(2): 65-69.
18. Perú, Ministerio de Salud. Vigilancia centinela de la Influenza y otros virus respiratorios. Lima: MINSA; 2005. Directiva 057 MINSA/OGE V.01
19. Daum L, Canas LC, Smith CB, Klimov A, Huff W, Barnes W, et al. Genetic and antigenic analysis of the first A/New Caledonia/20/99-like H1N1 Influenza isolates Reported in the Americas. Emerg Infect Dis. 2002; 8(4): 408-12.
20. Perú, Ministerio de Salud. Influenza aviar A H5N1 en humanos. Guía de práctica clínica. Lima: MINSA; 2007.
21. Viboud C, Alonso WJ, Simonsen L. Influenza in tropical regions. Plos Med. 2006; 3(4): e89.
22. Infuso A, Baron S, Fauveau H, Melon M, Fleury H, Desenclos JC. Value of Influenza vaccine during an outbreak of Influenza A in a nursing home, Pyrenees Atlantiques, France, November-December 1995. Euro Surveill. 1996; 1(5): 35-37.
23. Brammer L, Fukuda K, Klimov A, Cox N. Chapter 5: Influenza. In: Wharton M, Hughes H, Reilly M (eds), Manual for the surveillance of vaccine-preventable disease. 3rd ed. Atlanta: CDC; 2002.
24. Organización Mundial de la Salud. Plan mundial de la OMS de preparación para una pandemia de Influenza. Función y recomendaciones de la OMS para las medidas nacionales antes y durante las pandemias. Ginebra: OMS; 2005. WHO/CDS/CSR/GIP/2005.5
25. W orld Health Organization [página de interenet]. Recommendations for Influenza vaccines. [Fecha de acceso: septiembre 2007] Disponible en: www.who.int./csr/disease/Influenza/vaccinerecommendations.
Correspondencia:
Tatiana Saldarriaga / V. Alberto Laguna
Torres. Departamento de Virología, US Naval Medical Research Center Detachment. Lima, Perú.
Dirección: Av. Venezuela s/n Hospital Naval. Lima, Perú.
Teléfono: (511) 614-4166 anexo 166
Correos electrónicos: tasalva10@hotmail.com; alberto.laguna@med.navy.mil
Fecha de recepción: 31-10-07
Fecha de aceptación: 13-02-08