Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.27 n.1 Lima mar. 2010
ARTÍCULO ORIGINAL
Infecciones concurrentes por dos serotipos del virus dengue durante un brote en el noroeste de Perú, 2008
Concurrent infections by two dengue Virus serotypes during an outbreak in northwestern Peru, 2008
Enrique Mamani1,a,c, Dana Figueroa1,a, María Paquita García1,b, María del Carmen Garaycochea1,a, Edwar J. Pozo2,a
1 Laboratorio de Arbovirus, Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
2 Oficina de Epidemiología, Subregión de Salud Luciano Castilla. Sullana, Perú.
a Biólogo; b Tecnóloga Médico; c Magister en Microbiología.
RESUMEN
Objetivo. Describir la existencia de infecciones concurrentes por diferentes serotipos del virus dengue (DENV) en un brote ocurrido en el noroeste de Perú durante el 2008. Materiales y métodos. Se analizó 73 muestras séricas de pacientes con dengue en un brote en el noroeste de Perú entre mayo y junio de 2008. Para la serotipificación del DENV se utilizó técnicas de biología molecular; así, primero se realizó la extracción del ARN con el kit QIAamp viral RNA Mini, luego se realizó la transcripción inversa y amplificación de los fragmentos de ADNc viral mediante las técnicas de reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR multiplex) y de RT-Anidada PCR (RT-Nested PCR), y finalmente de realizó el secuenciamiento genético de los fragmentos de ADNc viral utilizando el kit Big Dye Terminator v.3,1. Resultados. Los 73 casos de dengue presentaron infecciones por diferentes serotipos: 34 (46,6%) por DENV-3, 29 (39,7%) por DENV-1, 4 (5,5%) por DENV-4 y 6 casos (8,2%) por DENV-1 y DENV-3. Las manifestaciones clínicas más frecuentes fueron fiebre y cefalea (100%), mialgia (94,5%), dolor ocular (83,6%), artralgia (78,1%), escalofríos (63,0%), nauseas/vómitos (38,4%), prueba de lazo positiva (30,1%) y erupción cutánea (20,5%). Los pacientes con infecciones concurrentes presentaron cuadros leves, excepto una paciente que presentó prueba de lazo positivo y sangrado genital. Conclusión. Es el primer reporte de pacientes peruanos con infecciones concurrentes por dos serotipos del DENV sin formas graves de la enfermedad.
Palabras clave: Dengue; Virus del dengue; Brote; Biología molecular; Perú.
ABSTRACT
Objetives. To establish the existence of concurrent infections by different dengue virus (DENV) serotypes in an outbreak in the Northwestern in Peru during 2008. Material and methods. 73 serum samples from patients with dengue were analyzed during an outbreak that occurred in Northwestern in Peru between May and June 2008. Molecular biology techniques were used to serotype the DENV, thus, firstly the viral RNA viral was extracted using Viral QIAamp RNA mini kit (Qiagen, Valencia, California, USA), then the viral cDNA fragments were reverse transcripted and amplified by means of the Reverse Transcriptase Polymerase Chain Reaction (RT-PCR) and the RT-Nested PCR region techniques and finally, genetic sequencing of the viral cDNA fragments were performed using the Big Dye Terminator v.3,1 kit. Results. The 73 dengue cases presented infections by different serotypes: 34 (46.6%) by DENV-3, 29 (39.7%) by DENV-1, 4 (5.5%) by DENV-4, and 6 (8.2%) concurrent infections by DENV-1 and DENV-3. The most frequent clinical manifestations observed among dengue patients were fever and headache (100%), myalgia (94.5%), ocular pain (83.6%), arthralgia (78.1%), shivers (63.0%), nausea/vomiting (38.4%), positive tourniquet test (30.1%), and rash (20.5%). All patients with concurrent infections presented light clinical course of dengue fever (DF) except one patient who had moderate hemorrhagic manifestations. Conclusion. This is the first Peruvian report of patients with concurrent infections of two DENV serotypes without severe clinical manifestations.
Key words: Dengue; Dengue virus; Outbreak; Molecular biology; Peru.
INTRODUCCIÓN
El dengue es un problema de salud pública producido por el virus dengue (DENV) que se presenta en áreas tropicales y subtropicales del mundo, predominando en áreas urbanas. Existen cuatro serotipos de virus dengue (DENV-1, 2, 3, y 4) los cuales están relacionados con la presencia de casos de dengue clásico (DC), casos de dengue hemorrágico (DH) y síndrome de choque por dengue (SCD). El DH es una complicación de la infección del dengue que fue reconocida en la década de 1950 y aún es considerada como principal causa de muerte infantil en varios países (1).
Las campañas realizadas en Centro y Sudamérica para erradicar el vector transmisor del dengue, Aedes aegypti, fueron exitosas por un tiempo, pero ahora son consideradas insuficientes. El reestablecimiento del vector en esta región a inicios de 1970 dio lugar al incremento del nivel de la actividad del virus y actualmente los cuatro serotipos del virus dengue están ampliamente distribuidos (2).
La infección concurrente de dengue está definida como la presencia de dos o más serotipos del DENV en un mismo individuo, el primer caso informado fue un paciente del cual se aislaron dos serotipos (DENV-1 y DENV-4) en Puerto Rico en 1982 (3). A partir de ese momento, los informes sobre las infecciones concurrentes se han incrementado; sin embargo, la asociación de infecciones concurrentes con las formas graves del dengue necesita ser más estudiado (4-9).
En Perú, el reingreso del dengue fue detectado en Iquitos en 1990 con el hallazgo del DENV-1, para el año 2000 los cuatro serotipos de DENV estaban circulando en el noroeste de Perú, así como en las regiones tropicales, se informó miles de casos de DC y algunos casos de DH/SCD, se aisló los virus de los cuatro serotipos y se determinó la circulación de dos genotipos del DENV-2 (10-16).
El brote descrito en este estudio se presentó en el distrito de Tambogrande, ubicado a 45 km al noroeste de la ciudad de Piura, a 04°5557 latitud sur y 80°2025 longitud oeste de Greenwich, a 68 msnm; su clima es cálido, muy seco, la temperatura anual media es 24 ºC y oscila entre 14 a 36 °C, con presencia del sol todo el año, la precipitación pluvial media y mediana es de 380 mm y 150 mm; tiene una población de 96 451 habitantes, 24,6% de las viviendas tiene agua potable y 51,4% servicio de desagüe, las principales actividades económicas son la agricultura, ganadería y comercio. En el año 2008 se notificó 604 casos, de ellos se confirmó a 102 casos. Se determinó los serotipos circulantes DENV-1, 3 y 4; el 90% (540/604) de los casos fueron informados en el área urbana y asentamientos humanos de la capital del distrito de Tambogrande. Se constató la presencia de Aedes aegypti intradomiciliario, los índices de infestación aédica en las viviendas variaron según los sectores entre 1,4 a 7,0 (17).
Este estudio es el primer informe de infecciones concurrentes por dos serotipos en el noroeste de Perú durante un brote de dengue donde se presentó la cocirculación de DENV-1, DENV-3 y DENV-4 durante el 2008.
MATERIALES Y MÉTODOS
PACIENTES Y MUESTRAS
Se realizó un estudio descriptivo transversal, se incluyó a todos los pacientes febriles que acudieron a los centros de salud y al hospital de la jurisdicción de Sullana a través del sistema de vigilancia epidemiológica del Ministerio de Salud, que presentaron sintomatología compatible con dengue según la definición de la OMS (18,19) durante el brote ocurrido entre mayo y junio de 2008 y a los que se tomó la muestra de sangre con menos de cinco días de iniciada la enfermedad (primer día de fiebre). Las muestras fueron centrifugadas a 2500 rpm, por 10 min, a 4 °C, el suero obtenido fue remitido en cadena de frío al Laboratorio de Arbovirus del Instituto Nacional de Salud para el diagnóstico virológico y molecular de dengue.
RT-PCR MÚLTIPLEX
El ARN viral de cada muestra fue extraído mediante el kit QIAamp viral RNA Mini de acuerdo con el protocolo del fabricante (Qiagen Inc, Valencia, California, EUA). Se realizó la RT-PCR múltiplex siguiendo el procedimiento previamente descrito por Harris et al. (20); este protocolo es una adaptación de la RT-PCR descrita por Lanciotti et al. (21).
Brevemente, se añadió 5 μL del ARN viral extraído a 20 mL de una mezcla RT-PCR constituida de cloruro de potasio (50 mM), Tris (10 mM, pH 8,5), gelatina 0,01%, deoxinucleótidos trifosfato (200 mM de cada uno), cloruro de magnesio (1,5 mM), cloruro de amonio tetrametilo (30 mM), betaina (0,5 M), ditriotreitol (15 mM), cebadores 5D1 y 3TS1 (1 mM cada uno), 3 TS2, TS3 y DENV-4 (0,5 mM de cada uno), transcriptasa inversa (MMLV 1U/ ml, Invitrogen, California, EUA), Taq DNA polimerasa (0,025 U/mL, Invitrogen, Brasil).
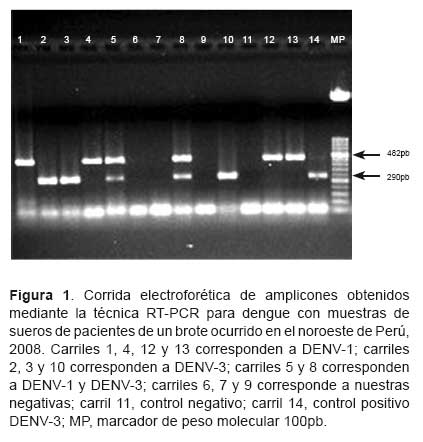
La transcripción inversa se realizó a 42 ºC por 60 minutos y la amplificación de los fragmentos de ADNc se hizo mediante 40 ciclos de 94 ºC por 30 s, 55 ºC por 1 min, y 72 ºC por 2 min, con una extensión final a 72 ºC por 5 min. La RT-PCR fue realizada en microtubos 0,2 mL en un termociclador MJ Research. La visualización de los amplicones se realizó mediante la electroforesis de 10 μL de cada amplicón en gel de agarosa al 1,5% en buffer TAE (Tris-Acetato 2M, EDTA 0,05 M pH 8,3). El tamaño esperado de los amplicones fueron: 482 pb para DENV-1, 119 bp para DENV-2, 290 bp para DENV-3, y 389 bp para DENV-4. El control positivo para la RT-PCR incluyó el extraído de ARN de DENV-3, el control negativo incluyó agua libre de masas.
RT-NESTED PCR
Se implementó la RT-Nested PCR para la región E/NS1 del genoma del virus dengue, específicamente desarrollado para la genotipificación del virus dengue a partir de muestras clínicas, siguiendo el procedimiento descrito por Domingo et al. (22). Así, se añadió 5 μL del ARN viral a 45 μL de la mezcla maestra del kit OneStep RT-PCR (Qiagen Inc, Valencia, California, EEUU). La mezcla maestra del kit contiene buffer, 400 mM de cada dNTP, 20 pmol de cebadores degenerados sentido y antisentido para DENV-1 (S1871DEN1, AS2622DEN1) y para DENV-3 (1871DEN3, AS2622DEN3) por separado.
Se realizó una transcripción inversa inicial a 41 °C por 45 min, seguido de una desnaturalización y activación de la Taq polimerasa (94 °C, 15 min) y, finalmente, 40 ciclos de desnaturalización (94 °C, 30 s), hibridización (55 °C, 1 min) y extensión (72 °C, 30 s). Se realizó una extensión final a 72 °C por 5 min. Seguidamente, se realizó una segunda amplificación (PCR anidada) fue realizada adicionando 1 μL del producto de amplificación inicial a una la mezcla de reacción que contenía buffer (60 mM Tris-HCl pH 8,5, 2 mM MgCl2, 15 mM (NH4)2SO4, 2,5 U de Taq DNA polimerasa (Invitrogen, Brasil) y 40 pmol de cada cebador sentido y antisentido para DENV-1 (S2176DEN1, AS2504DEN) y DENV-3 (S2176DEN3, AS2504DEN) por separado.
Se realizó una desnaturalización y activación de la Taq polimerasa (94 °C, 2 min) y 40 ciclos de desnaturalización (94 °C, 30 s), hibridización (57 °C, 4 min) y extensión (72 °C, 30 s). Finalmente, se realizó una extensión final a 72 °C por 5 min. El control positivo incluyó el extraído de ARN de DENV-3, el control negativo incluyó agua libre de masas.
SECUENCIAMIENTO DE LA REGIÓN E/NS1
Los amplicones de la región genómica E/NS1 fueron purificados utilizando el QIAquick PCR Purification Kit (Qiagen, Valencia, California, EEUU) para ser luego secuenciados usando el kit Big Dye Terminator v.3,1 (Applied Biosystems, Foster City, California, EEUU). Brevemente, para cada reacción de secuenciación se utilizó entre 50 a 100 ng de ADN con 3,2 pmol de cebador y la mezcla de la reacción que contenía los cuatro dideoxinucleótidos terminadores marcados. Los ciclos de secuenciación utilizados fueron los descritos en el protocolo del fabricante. El producto de reacción fue purificado en columna (NucleoSEQ, Macherey-Nagel GMBH & Co. KG) y el ADN fue secado en centrifuga al vacío por 20 min. El sedimento fue resuspendido en 10 μL de formamida, calentado a 95 °C por 2 min y mantenido en hielo hasta ser añadido al secuenciador Prism 310 (Applied Biosystems, Foster City, California, EEUU) utilizando un capilar de 47 cm por 50 μm con POP-6 (Applied Biosystems, Foster City, California, EEUU).
Los datos de las secuencias obtenidas fueron analizados inicialmente con el programa CHROMAS. Las secuencias sentido y antisentido de cada muestra fueron alineadas usando el software EDITSEQ (Dnastar Inc., Wisconsin, EEUU). La secuencias consensos fueron comparadas y alineadas con otras secuencias reportadas en el Genbank con el programa CLUSTAL X, versión 1,83 (23).
RESULTADOS
Se obtuvo 73 casos positivos para dengue mediante RT-PCR. El gel con los productos de la RT-PCR sometido a electroforesis presentó bandas individuales y dos bandas que correspondieron a los pacientes con infecciones concurrentes, 482pb para DENV-1 y 290pb para DENV-3 (Figura 1).
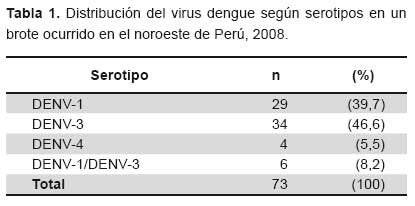
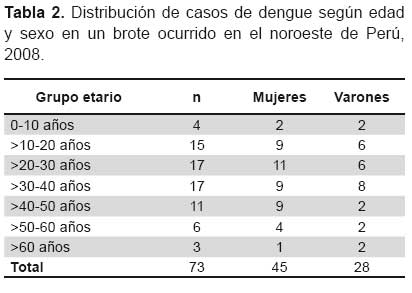
De los 73 casos positivos para dengue, 67 (91,8 %) casos correspondieron a infecciones por un solo serotipo (29 [39,7%] infecciones por DENV-1, 34 [46,6%] por DENV-3 y 4 [5,5%] por DENV-4) y seis casos a infecciones concurrentes por DENV-1 y DENV-3. En las infecciones por un solo serotipo, las ocasionadas por DENV-3 fueron las más prevalentes (46,6%), luego la ocasionadas por DENV-1 (39,7%) y por DENV-4 (5,5%). La prevalencia de infecciones concurrentes por DENV-1 y DENV-3 en el brote fue de 8,2 % (6/73) y no se observó por otras combinaciones (Tabla 1). Del total de casos, 62% fueron mujeres y 38% varones, siendo el grupo etario mas prevalente entre 20 a 40 años (Tabla 2).
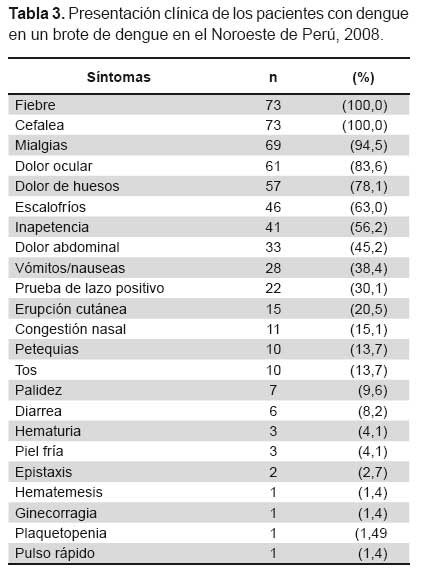
Los pacientes a los cuales se le detectó ARN viral del DENV mediante la técnica RT-PCR presentaron las siguientes manifestaciones clínicas: fiebre y cefalea 73 (100%), mialgias 69 (94,5%), dolor ocular 61 (83,6%), dolor articular 57(78,1%), escalofríos 46 (63%), inapetencia 41 (56,2%), vómitos/nauseas 28 (38,4%), prueba de lazo positiva 22 (30,1%), erupción cutánea 15 (20,5%), congestión nasal 11 (15,1%) y petequias 10 (13,7%). De los seis pacientes que tuvieron infecciones concurrentes, cinco cursaron con FD y un caso como dengue moderado con prueba de lazo positiva y sangrado genital (Tabla 3).
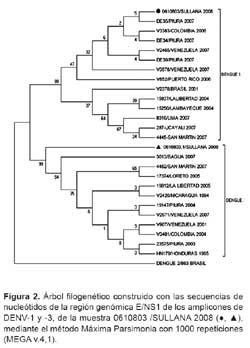
La caracterización genética de estos virus, tanto para DENV-1 y DENV-3 fue realizado amplificando un producto 328pb de la región E/NS1. Los genotipos encontrados correspondieron al genotipo América-África de DENV-1) y al genotipo India de DENV-3) (Figura 2).
DISCUSIÓN
Se tienen informes en varios países de la presencia de diferentes serotipos a través de los años e incluso se ha demostrado que en determinados brotes circulan dos o más serotipos (1,24), estos factores han dado lugar a la ocurrencia de una competencia en el proceso de infección viral y también a las infecciones concurrentes (3,8).
Desde el primer caso de infección concurrente en 1982 (3), se ha informado otros casos en diferentes países con todas las posibles dobles combinaciones de los cuatro serotipos del DEV: DENV-1/DENV-2, DEN-1/ DENV-3, DENV-1/DENV-4, DENV-2/DENV-3, DENV-2/ DENV-4, DENV-3/DENV-4 (4,8,9,25-27). Incluso, se ha llegado ha informar infecciones concurrentes por tres serotipos: DENV-1/DEN-2/DENV-3 y DENV-1/DENV-3/ DENV-4 (8,27). Algunas prevalencias de infecciones concurrentes se han informado en Taiwán (9,5%), México, Puerto Rico e Indonesia (5,5%), Indonesia (11%) e India (19%) (4,5,8,25,26), lugares endémicos donde periódicamente circulan dos o más serotipos del virus. Las infecciones concurrentes observadas muestran que este evento es fácilmente detectable por métodos de biología molecular respecto a la inmunofluorescencia (3,25-27).
En Perú, la circulación de cuatro serotipos en un mismo brote fue observada por primera vez en el noroeste de Perú, en Sullana, en el año 2001 (13) donde se informó los primeros casos de dengue hemorrágico con casos fatales; actualmente, esta misma región presenta casos durante todo el año por tres diferentes serotipos (DENV-1, DENV-3 y DENV-4). Los factores antes mencionados, como son la cocirculación de diferentes serotipos, presencia de casos de dengue durante todo el año, índices aédicos altos que van desde 1,4 a 7% (17), entre otros, probablemente condicionaron la presencia de las infecciones concurrentes por dos serotipos.
La infección concurrente descrita en este estudio está relacionada con los serotipos más predominantes informados en el brote descrito (DENV-1 y DENV-3), la mayoría de los casos correspondieron a pacientes residentes en área urbana (90%) y mujeres (61%). De los seis casos observados, una mujer presentó manifestaciones hemorrágicas moderadas, al respecto no se puede atribuir la gravedad de la enfermedad al evento de infección concurrente.
Los genotipos detectados en este brote para DENV-1 y DENV-3, genotipo América-África y genotipo India, respectivamente son los únicos informados en Perú para estos serotipos (13) y circularon en varias regiones del país como Amazonas, Huánuco, Lambayeque, San Martín, Ucayali y Loreto durante el 2008 (comunicación personal: Laboratorio de Metaxénicas Virales, Instituto Nacional de Salud, Lima, Perú).
Nuestros resultados corroboran que el Perú es una región hiperendémica para el dengue y es necesario fortalecer la vigilancia de los brotes de dengue y hacer un seguimiento de las características virales y su relación con la presentación clínica para establecer la significancia epidemiológica. Las futuras vigilancias de brotes en Perú deberían de ser conducidas con una nueva perspectiva para el entendimiento virológico y riesgo clínico epidemiológico de las infecciones concurrentes.
Fuente de Financiamiento
Centro Nacional de Salud Pública, Instituto Nacional de Salud, Perú.
Conflictos de Interés
Los autores declaran no tener conflictos de interés en la ejecución o publicación del presente estudio.
REFERENCIAS BIBLIOGRÁFICAS
1. Gubler DJ. Dengue y dengue hemorrhagic fever. Cin Microbiol Rev. 1998; 11(3): 480-96. [ Links ]
2. Monath TP. Dengue: the risk to developed and developing countries. Proc Natl Acad Sci USA. 1994; 91(7): 2395-400. [ Links ]
3. Gubler DJ, Kuno G, Sather GE, Waterman SH. A case of natural concurrent human infection with two dengue viruses. Am J Trop Med Hyg. 1985, 34(1): 170-3. [ Links ]
4. Laille M, Deubel V, Sinte-Marie FF. Demonstration of concurrent dengue 1 and dengue 3 infection in six patients by the polymerase chain reaction. J Med Virol. 1991, 34(1): 51-4. [ Links ]
5. Dos Santos CL, Bastos MA, Sallum MA, Rocco IM. Molecular characterization of dengue viruses type 1 and 2 isolated from a concurrent human infection. Rev Inst Med trop S Paulo. 2003; 45(1): 11-6. [ Links ]
6. Camacho DE, Alvarez M, Rodriguez-Henriquez F, Quintana M, Soler M, Chiarello A et al. Diagnóstico de laboratorio de infecciones por el virus dengue en el Estado Aragua; Venezuela: Octubre 1997 - Diciembre 1998. Invest Clin. 2003; 44(2): 91-103. [ Links ]
7. Gupta E, Dar L, Broor S. Concurrent infection by two dengue virus serotypes among dengue patients. Indian J Med Microbiol. 2008; 26(4): 402-3. [ Links ]
8. Loroño-Pino MA, Cropp CB, Farfán JA, Vorndam AV, Rodriguez- Angulo EM, Rosado-Paredes EP, et al. Common occurrence of concurrent infections by multiple dengue virus serotypes. Am J Trop Med Hyg. 1999; 61(5): 725-30. [ Links ]
9. Wang WK, Chao DY, Lin SR, King CC, Chang SC. Concurrent infections by two dengue virus serotypes among dengue patients in Taiwan. J Microbiol Immunol Infect. 2003; 36(2): 89-95. [ Links ]
10. Cabezas C. Dengue en el Perú: aportes para su diagnóstico y control. Rev Peru Med Exp Salud Publica. 2005; 22(3): 212-28. [ Links ]
11. Forshey BM, Morrison AC, Cruz C, Rocha C, Vilcarromero S, Guevara C, et al. Dengue virus serotype 4, northeastern Peru, 2008. Emerg Infect Dis. 2009; 15(11): 1815-18. [ Links ]
12. Chowell G, Torre CA, Munayco-Escate C, Suárez-Ognio L, López-Cruz RHyman JM, et al. Spatial and temporal dynamics of dengue fever in Peru: 1994-2006. Epidemiol Infect. 2008; 136(12): 1667-77. [ Links ]
13. Montoya Y, Holechek S, Caceres O, Palacios A, Burans J, Guevara C et al. Circulation of dengue viruses in northwestern Peru, 2000-2001. Dengue Bulletin. 2003; 27: 52-62. [ Links ]
14. Mostorino R, Rosas A, Gutiérrez V, Anaya E, Cobos M, García M. Manifestaciones clínicas y distribución geográfica de los serotipos del dengue en el Perú, año 2001. Rev Peru Med Exp Salud Publica. 2002; 19(4): 171-80. [ Links ]
15. Cobos M, Gutiérrez V, García M, Mamani E, Fernández R, Rimarachín R et al. Estudio serológico y virológico del brote de dengue en la provincia de Coronel Portillo Ucayali, Perú (2000-2001). Rev Peru Med Exp Salud Publica. 2004; 21(3): 139-45. [ Links ]
16. Mamani E, García M, Gutiérrez V, Cabezas C, Harris E. Tipificación molecular del virus dengue 3 durante el brote epidémico de dengue clásico en Lima, Perú, 2005. Rev Peru Med Exp Salud Publica. 2005; 22(3): 161-4. [ Links ]
17. Subdirección de Salud Luciano Coloma Castillo. Informe final de actividades de control del brote de dengue del distrito de Tambogrande. Sullana: Subdirección de Salud LCC; 2008. [ Links ]
18. World Health Organization. Guide for diagnosis, treatment and control of dengue hemorrhagic fever. 2nd ed. Geneva: WHO; 1997. [ Links ]
19. Juárez J, Soto P, Bernuy G, Alejo E, Valdivia M, Cosser J, et al. Evaluación de la definición de caso probable de dengue clásico durante el brote de dengue en Lima, 2005. Rev Peru Med Exp Salud Publica. 2005; 22(3): 205-11. [ Links ]
20. Harris E, Roberts TG, Smith L, Selle J, Kramer LD, Valle S et al. Typing of dengue viruses in clinical specimens and mosquitoes by single-tube multiplex reverse transcriptase PCR. J Clin Microbiol. 1998; 36(9): 2634-9. [ Links ]
21. Lanciotti R, Calisher CH, Gubler DJ, ChangGJ, Vorndam AV. Rapid detection and typing of dengue viruses from clinical samples by using reverse transcriptase-polymerase chain reaction. J Clin Microbiol. 1992; 30(3): 545-51. [ Links ]
22. Domingo C, Palacios G, Niedrig M, Cabrerizo M, Jabado O, Reyes N et al. A new tool for the diagnosis and molecular surveillance of dengue infections in clinical samples. Dengue Bull. 2004; 28: 87-95. [ Links ]
23. Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG. The Clustal_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 1997; 25(24): 4876-82. [ Links ]
24. Usme-Ciro JA, Mendez JA, Tenorio A, ReyGJ, Domingo C, Gallego-Gomez JC. Simultaneous circulation of genotypes I and III of dengue virus 3 in Colombia. Virol J. 2008; 5:101. [ Links ]
25. Araújo FM, Nogueira RM, de Araújo JM, Ramalho IL, Roriz ML, de Melo ME et al. Concurrent infection with dengue virus type-2 and DENV-3 in a patient from Ceará, Brazil. Mem Inst Oswaldo Cruz. 2006; 101(8): 925-8. [ Links ]
26. Bharaj P, Chahar HS, Pandey A, Diddi K, Dar L, Guleria R et al. Concurrent infections by all four dengue virus serotypes during an outbreak of dengue in 2006 in Delhi, India. Virol J. 2008; 5: 1. [ Links ]
27. Chinnawirotpisan P, Mammen MP Jr, Nisalak A, Thaisomboonsuk B, Narupiti S, Thirawuth V et al. Detection of concurrent infection with multiple dengue virus serotypes in Thai children by ELISA and nested RT-PCR assay. Arch Virol. 2008; 153(12): 2225-32. [ Links ]
Recibido: 25-01-10
Aprobado: 02-03-10
Correspondencia: Blgo. Enrique Mamani Zapana.
Dirección: Av. Defensores del Morro 2268 Chorrillos, Lima 9, Perú.
Teléfono: (511) 617-6200 anexo 1587; (511) 995-903-830.
Correo electrónico: emamani@ins.gob.pe