Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.27 no.4 Lima Oct./dec. 2010
ARTÍCULO ORIGINAL
Aplicación de las pruebas de PCR convencional simple y múltiple para la identificación de aislamientos de Leptospira spp. en Colombia
Application of conventional and multiplex PCR assays for identification of isolates of Leptospira spp. in Colombia
Natali Moreno 1,a, Piedad Agudelo-Flórez1,b
1 Grupo de Investigación en Medicina Tropical; Instituto Colombiano de Medicina Tropical, Universidad CES, Medellín, Colombia.
a Bacterióloga, M.Sc en Microbiología y Bioanálisis; b Bióloga, Ph.D en Ciencias Biomédicas.
RESUMEN
Debido a las dificultadas asociadas con la identificación serológica de aislamientos de Leptospira ssp, se genera gran interés en la pruebas moleculares por su poder discriminatorio, reproducibilidad y fácil interpretación. Objetivo. Aplicar y validar la prueba de PCR convencional, usando dos pares de iniciadores descritos previamente y dirigidos a los genes lipL32 (PCR simple) y secY/flaB (PCR múltiple), con el fin de evaluar su aplicación para identificar especies patógenas y saprófitas de Leptospira spp. Materiales y métodos. Para la estandarización de las pruebas de PCR se usó 22 cepas de referencia internacional y 12 aislamientos colombianos. Se determinó el nivel de detección de cada pareja de iniciadores, su especificidad frente a otros microorganismos causantes de enfermedades endémicas en Colombia y su capacidad de identificar especies dentro del grupo de Leptospira. Resultados. El límite de detección de la PCR simple lipL32 fue una dilución 1:10000 y para la PCR múltiple secY/flaB fue una dilución 1:100 para el gen secY y 1:1000 para flaB. La especificidad de todos los iniciadores fue de 100%. La PCR simple lipL32, mostró amplificado específico para 21/22 cepas de referencia mientras que la PCR multiple secY/flaB lo fue para 18/22 cepas. De los 12 aislamientos colombianos, siete fueron positivos por PCR lipL32 y seis lo fueron por PCR secY/flaB. Conclusiones. Los resultados más consistentes fueron obtenidos con la PCR simple lipL32 tanto en límite de detección, especificidad y utilidad para la identificación de Leptospira spp, por lo que esta prueba es aplicable a la identificación molecular de aislamientos patógenos de Leptospira spp de diversas fuentes.
Palabras clave: Leptospira; Reacción en cadena de la polimerasa; Técnicas de diagnóstico molecular; Colombia (fuente: Decs BIREME).
ABSTRACT
Serological identification of Leptospira ssp isolates is difficult to achieve. Thus, molecular testing may be of great interest thanks to its high discrimination power, reproducibility and easy interpretation. Objective. To implement and validate conventional and multiplex PCR methods (using primers directed against lipl32 and secY/flaB genes, respectively). To assess the capacity of PCR methods to identify pathogenic and saprophytic species of Leptospira ssp. Material and methods. 22 international reference strains and 12 colombian isolates were used. DNA was extracted with a commercial kit (Wizard). Specificity and sensitivity of both PCR methods were evaluated. Results. The maximum dilution of DNA samples allowing the detection of Leptospira ssp was determined to be 1:10000 for the PCR lipL32 and 1:100/1:1000 for the multiplex PCR secY/flaB. Both PCR didnt detect DNA from microorganisms unrelated to Leptospira ssp. The lipL32 PCR specifically amplified a 423bp fragment from all pathogenic Leptospira reference strains, while the secY/flaB PCR amplified both 285bp (secY) and 793bp (flaB) fragments from 18 reference strains. The lipL32 PCR detected 7/12 colombian isolates, while secY/flaB PCR detected both secY and flaB genes from 6/12 isolates. Conclusions. Best results were obtained with the lipL32 PCR, which displayed a better sensitivity and a better capacity to detect different strains than the multiplex PCR. The secY primers showed a poor specificity to pathogenic species and a poor sensitivity. Thus, lipL32 primers show high potential for molecular diagnosis of Leptospira spp in clinical and environmental samples.
Key words: Leptospira; Polymerase chain reaction; Molecular diagnostic techniques; Colombia (source: MeSH NLM).
INTRODUCCIÓN
La leptospirosis es una enfermedad infecciosa de distribución mundial que afecta al hombre y a diferentes especies de animales. Esta zoonosis es ocasionada por una espiroqueta del género Leptospira. La transmisión se da por contacto directo con orina de animales infectados con la bacteria o indirectamente con aguas y suelos contaminados con la orina de estos animales (1,2).
En la actualidad, la leptospirosis es reconocida como una enfermedad emergente que ha causado brotes epidémicos alrededor del mundo, como los registrados en Nicaragua, Brasil e India (1,2). En Colombia se ha presentado también de esta forma, como en el brote que se documentó en agosto de 1995 en Atlántico con 47 casos confirmados y 284 casos sospechosos, con una letalidad del 17%. El brote se asoció con las inundaciones que afectaron gran parte de la Costa Atlántica (3). Igualmente, se ha reportado en población general de diferentes regiones de Colombia con porcentajes de positividad del 12,5%, 18,5% y 23,3% (4-6).
Pero a pesar de haberse registrado para Colombia, es una enfermedad subestimada por las entidades de salud pública en general, debido, primero, a que las manifestaciones clínicas en la fase inicial son compartidas con otras enfermedades endémicas en Colombia, tales como influenza, dengue, malaria, salmonelosis, Brucelosis y Rickettsiosis. Esto hace que el diagnóstico basado solo en la clínica no sea suficiente.
La microaglutinación (MAT), que es la prueba de referencia de la Organización Mundial de la Salud, presenta una sensibilidad variable dependiente del número de serovares probados en el panel de evaluación serológica y de la inclusión o no de cepas locales; requiere además de sueros pareados para la detección de la seroconversión (que es el parámetro de confirmación de caso). Muestra reacciones cruzadas entre serogrupos principalmente en fases tempranas de la enfermedad, consume mucho reactivo debido a que es necesario mantener cultivos en repique constante, contando además, que al utilizar leptospiras vivas se presenta el riesgo ocupacional de infección para el personal de laboratorio. Igualmente, una de las dificultades de la prueba de MAT, es la determinación del punto de corte que permita la correcta interpretación de los resultados serológicos obtenidos por esta prueba. El punto de corte más utilizado es 1:100 para huéspedes susceptibles, pero no siempre resulta adecuado para poblaciones donde se presente alta o baja endemicidad de leptospirosis.
Teniendo en cuenta estas limitaciones, otras pruebas basadas en el diagnóstico molecular han sido desarrolladas en los últimos años para el diagnóstico de la leptospirosis. Como el género Leptospira consiste en un grupo diverso de espiroquetas patógenas y saprófitas, se ha usado la clasificación basada en estudios de hibridización del ADN que ha servido para clasificar las 17 especies conocidas (2,7,8). De las especies patógenas se reconocen alrededor de 200 serovares, esta diversidad ha sido atribuida a las diferencias en la estructura y composición de los lipopolisacáridos, especialmente en la variación de los carbohidratos, lo que refleja la diversidad antigénica entre especies de leptospiras patógenas (7,9,10).
Leptospira spp posee una estructura de doble membrana compuesta por la membrana citoplasmática, el periplasma y la membrana externa (9). En la membrana externa se ha identificado diferentes proteínas con diversos grados de expresión en la superficie como lo son Ompl1 (leptospiral outer membrane porin), LipL (leptospiral outer membrane lipoproteín) como LipL21, LipL36, LipL34, LipL41, LipL32 y proteinas Lig (leptospiral immunoglobulin-like proteins) entre otras (10,11). Además, existen diferentes proteínas que están ubicadas en el espacio periplásmico, como la proteína flagelar B (flaB) que se encuentra también relacionada con especies patógenas (12,13).
En los últimos años se ha evaluado la detección del genoma bacteriano por métodos moleculares basados en la reacción en cadena de la polimerasa (PCR) y se ha descritos diferentes iniciadores para su detección, usando una variedad de genes como blanco de amplificación, incluyendo flaB (12), lipL32 (14), OmpL1 (15), secY (16), Lig (17), gyrB (18), entre otros.
En general, las pruebas moleculares para Leptospira presentan alto poder discriminatorio, ofrecen resultados reproducibles, de fácil y exacta interpretación que permite disminuir los problemas asociados con la transferencia de tecnología y la reproducibilidad de pruebas entre laboratorios de zonas endémicas. Pese a las ventajas que representan el diagnóstico y la tipificación molecular para Leptospira, este último aspecto ha sido poco evaluado para Colombia.
El propósito de este estudio fue aplicar la prueba de PCR convencional, usando un par de iniciadores previamente descritos por Levett et al. (14) en una PCR convencional simple y dos pares de iniciadores descritos por Gravekamp et al. (19) y Kawabata et al. (12) en una PCR convencional múltiple. Todo esto con el propósito de evaluar su aplicación para identificar molecularmente especies saprófitas y patógenas de Leptospira spp de referencia y, posteriormente, su utilidad en la identificación de 12 aislamientos colombianos de diferentes fuentes: humanas, animales y ambientales.
MATERIALES Y MÉTODOS
Se realizó un estudio de estandarización y aplicación de pruebas basadas en la PCR, estandarizándolas con cepas de referencia y aplicándolas a la identificación de aislamientos colombianos de Leptospira spp.
CEPAS DE Leptospira spp
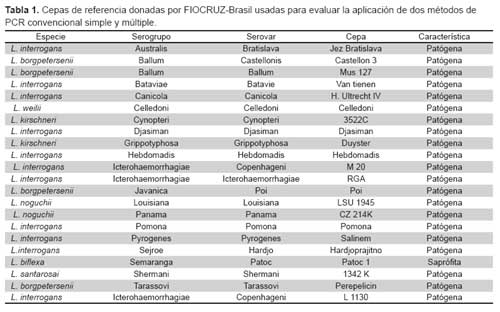
Se usó 22 cepas de referencia internacional, donadas por la Fundación Oswaldo Cruz (FIOCRUZ) de Brasil. Los datos de serogrupo, serovares y nombre de cada cepa utilizada se describen en la Tabla 1.
Las capas se incubaron a temperaturas entre 26 y 30 °C, hasta cuando alcanzaron crecimiento igual al estandar 0,5 de MacFarland en medio líquido EMJH (Elling-Hausen-McCullough-Johnson-Harris) (Becton- Dickinson, Sparks, MD. USA). Suplementado con 10% de medio de enriquecimiento (Becton-Dickinson-Biosciences).
AISLAMIENTOS COLOMBIANOS
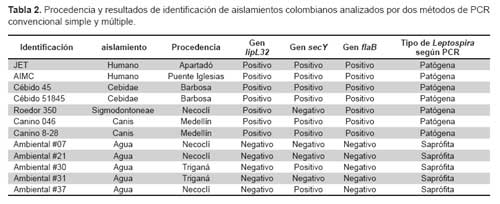
Para la aplicación de las pruebas de PCR con aislamientos colombianos de Leptospira spp se evaluó 12 aislamientos procedentes de dos pacientes con clínica compatible con leptospirosis, cinco de diferentes especies animales (dos monos Cebidae sp, un roedor y dos caninos) y cinco aislamientos de aguas ambientales. Todos estos aislamientos fueron mantenidos bajo las mismas condiciones que las cepas de referencia.
EXTRACCIÓN DE ADN
El ADN para las pruebas moleculares fue extraído con un kit comercial (Wizard Genomics Promega, Madison WI. USA), siguiendo el protocolo para bacterias Gram negativas recomendado por la casa comercial. Se utilizó como volumen inicial 1 mL de cultivo con crecimiento 0,5 MacFarland. Después de la extracción el ADN se rehidrató con buffer Tris-EDTA, y se almacenó a 4 °C hasta el momento de su uso.
REACCIÓN EN CADENA DE LA POLIMERASA SIMPLE
La reacción se realizó con los iniciadores descritos por Levett et al. (14), LipL32/270F (5´- CGCTGAAATGGGAGTTCGTATGATT-3´) y LipL32/692R (5´-CCAACAGATGCAACGAAAGATCCTTT-3´), dirigidos a una región ubicada entre las posiciones 270 y 692 del gen lipL32, que codifica para la lipoproteína de membrana LipL32. Esta secuencia está altamente conservada en las especies patógenas de Leptospira (Acceso GenBank L. interrogans serovar Lai str. 56601 AE010300.2 (20)).
El perfil térmico para esta PCR simple, fue estandarizado con una temperatura de desnaturalización de 95 °C durante cinco minutos, una fase de 35 ciclos a 94 °C durante un minuto, 55 °C durante un minuto y 72 °C durante dos minutos y, posteriormente, una fase de extensión final a una temperatura de 72 °C durante cinco minutos. En el proceso de PCR se utilizó los reactivos comerciales (Fermentas Taq DNA polymerase [recombinant], Foster City, CAL. USA) usando 35 μL como volumen final de la reacción, 3,5 μL de buffer de PCR (KCl) a una concentración 1X, 4,2 μL de MgCl2 a una concentración de 3,0 mM, 3,5 μL de cada uno de los iniciadores (forward - reverse) a una concentración 0,1 μM, 0,875 μL a una concentración 0,25 mM de dNTP y 0,28 μL de taq polimerasa con una concentración de 1U, y adicionando 5 μL de DNA molde.
REACCIÓN EN CADENA DE LA POLIMERASA MÚLTIPLE
A las muestras de ADN de todas las cepas se les realizó PCR múltiple con los iniciadores G1 (5`-CTGAATCGCTGTATAAAAGT- 3`) y G2 (5´-GGAAAACAAATGGTCGGAAG- 3`) descritos por Gravekamp et al. (19), dirigidos a una secuencia especifica del gen secY (Acceso GenBank L. interrogans serovar Lai str. 56601 AE010300.2 (20)) de especies patógenas de L. interrogans, L. borgpetersenii, L. weillii, L.noguchi, L. santarosai y L. meyerii, excepto L. kirschneri. Los iniciadores B64I/B64II no fueron utilizados como lo describe originalmente Gravekamp et al. (19), en este caso fueron reemplazados por los iniciadores L-flaBF1( 5´- TCTCACCGTTCTCTAAAGTTCAAC-3´) y L-flaBR1( 5´- CTGAATTCGGTTTCATATTTGCC-3´) que van dirigidos al gen que codifica para la proteína flagelar B (Acceso GenBank L. interrogans serovar Lai str. 56601 AE010300.2 (20)) presente en especies patógenas de Leptospira (12).
El perfil térmico se estandarizó con una temperatura de desnaturalización a 95 °C durante cinco minutos, una fase de 35 ciclos a 95 °C durante 30 segundos, 50 °C durante 30 segundos y 72 °C durante un minuto y, posteriormente, una fase de extensión final a una temperatura de 72 °C durante cinco minutos. En el proceso de PCR se utilizaron los reactivos comerciales (Fermentas Taq DNA polymerase [recombinant], Foster City, CAL. USA) usando 35 μL como volumen final de la reacción, 3,5μl de buffer PCR (KCl) a una concentración 1X, 3,5 μL de MgCl2 a una concentración de 2,5 mM, 3,5 μL de cada uno de los iniciadores (G1/G2, flaB-F1, flaB-R1) a una concentración 1,0 μM, 0,7 μL de dNTP a una concentración de 0,2 mM, y 0,2 μL de taq polimerasa con una concentración de 1U agregando 3 μL de DNA molde.
ELECTROFORESIS
Todos los productos de la PCR obtenidos se visualizaron utilizando la técnica de electroforesis en gel de agarosa, con una concentración de 1,5%, se usó buffer TBE (Tris- Base, EDTA, acido bórico) a una concentración de 1X, fue revelado con bromuro de etidio y se usó un GeneRuler 50bp DNA Ladder (Fermentas, Carlsbad CA. USA). Posteriormente se realizó la corrida electroforética a 80V durante una hora en cámara de electroforesis horizontal y, por último, se visualizó en un analizador de imagen Epichemi3.
LÍMITE DE DETECCIÓN Y ESPECIFICIDAD
Los límites de detección y especificidad de las pruebas de PCR fueron calculadas acorde al protocolo de validación de pruebas de PCR de la Organización Mundial de Sanidad Animal (16,21). Para el caso del límite de detección de ambos ensayos se utilizaron diluciones dobles de 1:10 a 1:1 000000, a partir de 120 ng de ADN extraído de la cepa de referencia L. interrogans Serovar Copenhageni Cepa M 20.
La especificidad de las pruebas fue establecida con las 22 cepas de referencia, comparando el resultado obtenido con cada prueba de PCR y los datos de la referencia de cada cepa suministrados por FIOCRUZ, Brasil. Igualmente fueron evaluadas las pruebas con ADN de especies diferentes a Leptospira spp. En este panel de evaluación de la especificidad se incluyó a Salmonella sp, Brucella sp, Plasmodium vivax, Toxoplasma gondii, Leishmania sp, Trypanosoma cruzi, Mycobacterium leprae y M. tuberculosis.
RESULTADOS
LÍMITE DE DETECCIÓN DE LA PCR
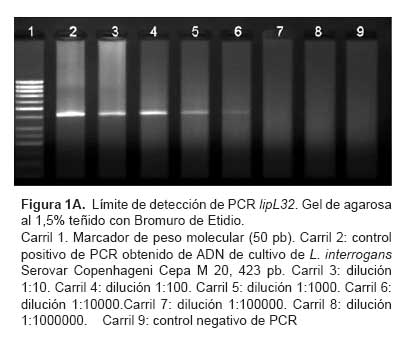
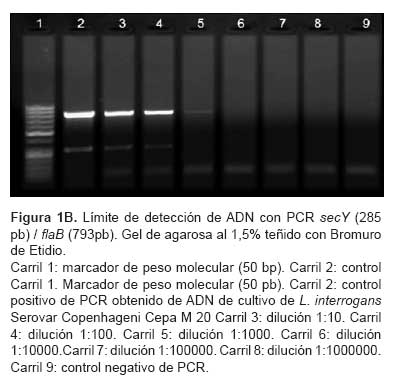
El límite de detección de la reacción de la PCR simple lipL32, fue verificada analizando diferentes concentraciones del ADN. Se obtuvo productos específicos de 423 pb de L. interrogans serovar Copenhageni cepa M 20 hasta la dilución 1:10 000 (Figura 1A). Para el caso del límite de detección de la reacción de la PCR múltiple secY/flaB se obtuvieron productos específicos de 285 pb y 793 pb de L. interrogans serovar Copenhageni cepa M 20, hasta la dilución 1:100 para secY y 1:1 000 para flaB (Figura 1B).
ESPECIFICIDAD DE LA PCR
Los ensayos de PCR simple y múltiple usando ADN de un panel de ocho agentes infecciosos propios de áreas endémicas colombianas fueron negativos, se mostró así la especificidad de estos iniciadores para Leptospira spp. Las figura 2A y figura 2B muestran esos resultados para tres de los agentes probados, Salmonella sp, Brucella sp, Plasmodium vivax.
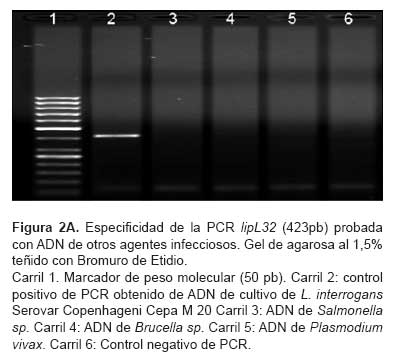
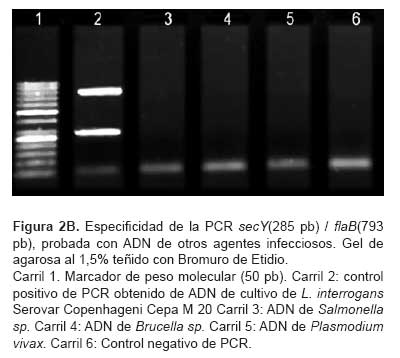
ANÁLISIS MOLECULAR DE LAS CEPAS DE REFERENCIA DE Leptospira spp
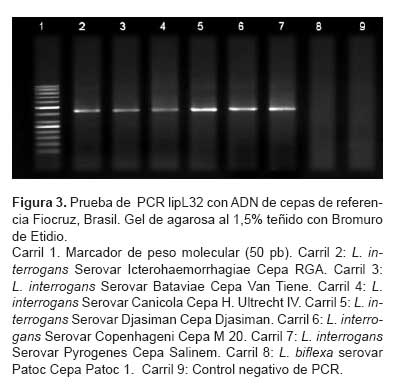
Los resultados de la prueba de PCR lipL32, mostraron amplificado específico en todas las 21 especies patógenas de referencia. En la que no se obtuvo amplificado fue en la especie saprofita L. biflexa serovar Patoc, resultado que estuvo de acuerdo con lo esperado, porque las especies saprofitas no tienen gen ortólogo de lipL32, lo que corrobora la especificidad de esta prueba de PCR para especies patógenas (Figura 3).
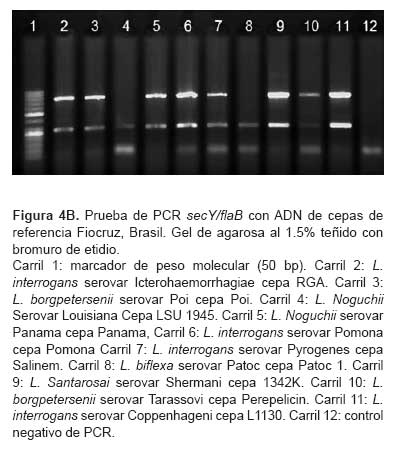
Para la PCR secY/flaB, se obtuvo amplificado específico de ambas bandas de 285 pb y de 793 pb en 18 de las 22 cepas de referencia, dos cepas amplificaron únicamente el gen secY (L. noguchii serovar Louisiana cepa LSU 1945 y L. biflexa serovar Patoc cepa Patoc 1) y dos de las 22 cepas amplificaron únicamente flaB, L. kirschneri serovar Cynopteri cepa 3522C y L. kirschneri serovar Grippotyphosa cepa Duyster (Figura 4A y figura 4B).

ANÁLISIS MOLECULAR DE LOS AISLAMIENTOS COLOMBIANOS
La PCR lipL32, fue positiva para los dos aislamientos procedentes de humanos y cinco aislamientos de animales; no se obtuvo amplificado para ninguno de los cinco aislamientos ambientales (Figura 5).
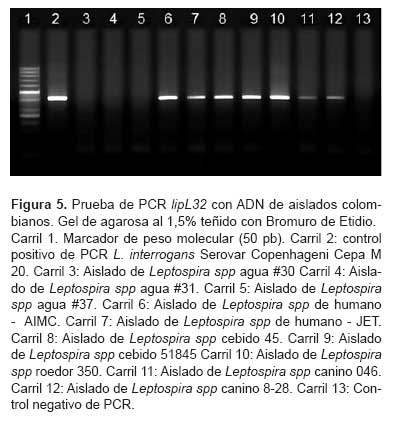
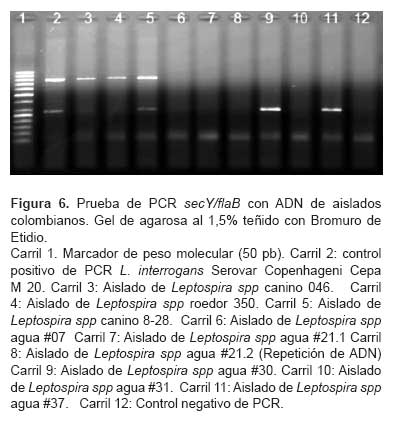
Para la PCR secY/flaB, se obtuvo amplificado en seis aislamientos de los 12 obtenidos, pertenecientes a los aislamientos de humanos, caninos y cébidos. Por su parte el aislamiento perteneciente al roedor 350 amplificó solo con flaB y no con secY. Dos aislamientos de aguas ambientales (#30 y # 37) amplificaron con secY y no con flaB (Figura 6). Los resultados obtenidos para cada aislamiento se describen en la Tabla 2.
DISCUSIÓN
Los resultados de este estudio muestran la utilidad de dos tipos de pruebas de PCR, la simple y la múltiple, para identificar especies de Leptospira de referencia y aislamientos colombianos a partir de iniciadores descritos en la literatura (12,14,19). El límite de detección de la PCR lipL32 (dilución 1:10 000) es similar a la que previamente se reportó en la publicación original usando PCR en tiempo real, corroborando que la sensibilidad de la prueba es reproducible en condiciones de PCR convencional (14). Por su parte, la PCR secY/flaB, difiere en el límite de detección para cada uno de los iniciadores, debido a que para el gen secY el límite de detección fue diez veces menor que el obtenido para el gen flaB. Por lo tanto, en términos de límite de detección, la PCR lipL32, es más sensible que la PCR secY/flaB, lo que además la posiciona como de elección para estudios posteriores que busquen validar su uso potencial para el diagnóstico de leptospirosis en muestras clínicas de pacientes que se encuentren en fases tempranas de la infección o incluso que hubieran iniciado la terapia antibiótica (14).
La especificidad de las dos pruebas de PCR se encuentra en concordancia con otros estudios realizados en otras regiones geográficas usando estos iniciadores (22,23). Frente a la especificidad con cepas de referencia por la prueba de PCR lipL32, se encontró una concordancia total frente a los registros de cada cepa, suministrados por Fiocruz , Brasil. Lo que confirma que es un factor de virulencia putativo altamente conservado en especies patógenas y no tiene genes ortólogos en especies saprofitas que codifiquen para esta lipoproteína (11).
Por su parte la PCR secY/flaB, muestra resultados discordantes con las cepas L. noguchii Serovar Louisiana Cepa LSU1945 y L. biflexa Serovar Patoc Cepa Patoc I. Esto puede sugerir que la región del gen secY no es especifica de especies patógenas, ya que se obtuvo amplificado con L. biflexa. Esto se debe a que este locus es conservado en el género Leptospira, ya que análisis de PCR dirigidos al secY tienen la capacidad de generar resultados variados en ensayos con especies saprofitas e intermedias (24,25).
Mediante análisis de BLAST, se demuestra que estos iniciadores tienen complementariedad tanto con especies patógenas y saprofitas como L. biflexa serovar Patoc 1 (Acceso GenBank CP000786.1). Las dos únicas cepas que fueron amplificadas solo con flaB, pertenecen a la especie de L. kirschneri, que no es amplificada con iniciadores G1/G2. Por esta razón en la publicación original de Gravekamp (19), se recomendó el uso de otros iniciadores (B64I/B64II) que también están asociados con el gen flaB que se encuentra en la especie L. kirschneri (19,26). Esto reafirma que el análisis molecular llevado a cabo por esta PCR múltiple sea inconcluso, al dejar especies patógenas de Leptospira sin identificar y sea necesaria la utilización de otro par de iniciadores.
La PCR lipL32, probada para identificar los aislamientos colombianos, ha demostrado especificidad para la amplificación de muestras de animales y humanos procedentes de Colombia (27,28), corroborando lo ya obtenido con cepas de referencia. La presencia de este marcador en cultivos de hospederos susceptibles, verifica en forma objetiva la virulencia de los aislamientos colombianos. Si bien en este trabajo no se llevaron a cabo estudios de virulencia in vivo de estos aislamientos, este hallazgo cobra importancia, debido a que hasta donde llega nuestro conocimiento, es la primera oportunidad en que se demuestra la presencia, por métodos moleculares, del marcador de virulencia, lipL32, en aislamientos de origen colombiano. Aunque es muy poco conocido el papel de esta proteína de membrana, en la patogénesis de leptospirosis, se sabe por estudios experimentales que niveles significativos de expresión de LipL32 están presentes durante la infección aguda letal en infecciones in vitro de cultivos de células de cobayo (29). Es recomendable por lo tanto, continuar explorando marcadores de virulencia para los aislamientos de Leptospira spp. Con el fin de conocer aspectos de la patogénesis de esta enfermedad en huéspedes adaptados o en reservorios, que ayuden a caracterizar la presentación clínica de esta infección en Colombia.
Por su parte, los resultados de los aislamientos ambientales probados por la PCR lipL32, sugieren que muy posiblemente sí pertenecen a especies saprofitas que, como ya se dijo, no codifican para esta lipoproteína de membrana externa (9-11). Por ser la leptospirosis una enfermedad de transmisión ambiental y que se ha asociado a brotes posteriores a inundaciones, como el caso del presentado en la costa atlántica colombiana (3) y conociendo que existen especies de Leptospira de carácter intermedio en donde su capacidad patogénica no es clara, es recomendable continuar explorando otros marcadores genéticos de especies saprofitas e intermedias, adicionales a los usados como blancos de amplificación en las pruebas descritas en el presente artículo, como es el caso del gen 16S que permitan identificar la alta variedad de especies de Leptospira que pueden encontrarse en muestras de origen ambiental (30). Debido a que es ampliamente conocido que especies patógenas de Leptospira son capaces de sobrevivir en ambientes acuáticos por períodos variables de tiempo y ser por lo tanto, un foco potencial de infección para humanos y animales (31,32).
Con respecto a los resultados de los aislamientos colombianos procesados por PCR secY/flaB, se demostró igualmente discordancia con respecto a los resultados esperados. Por un lado el gen secY fue reconocido en dos muestras de aguas, que habían sido negativas por la PCR lipL32, demostrando la inespecificidad de los iniciadores G1/G2, al ser positiva en posibles especies saprofitas y en ser negativa en el caso de la cepa procedente de roedor 350. Que había sido claramente identificada como patógena tanto por los iniciadores lipL32 como flaB. La ambigüedad de los iniciadores G1/G2 ya había sido reportada por otros investigadores, los que también reportan discordancia en la identificación de especies saprofitas de patógenas (24,25). Esto hace poco recomendable el uso de estos iniciadores cuando se requiere diferenciar entre especies patógenas de saprofitas, especialmente, como se describió anteriormente, en muestras ambientales.
En conclusión, este estudio presenta la evaluación de pruebas moleculares para la identificación de aislamientos patógenos de Leptospira provenientes de diferentes fuentes. Según los criterios de límite de detección y especificidad evaluados en este estudio, la prueba de PCR lipL32, es un método que permite caracterizar un área geográfica permitiendo identificar posibles factores eco-epidemiológicos de riesgo. La prueba tiene potencial para ser validada en trabajos futuros para el diagnóstico molecular de la leptospirosis en diferentes muestras clínicas o ambientales.
AGRADECIMIENTOS
Al doctor Albert Ko de Fiocruz, Brasil, por la donación de las cepas de referencia. Al médico veterinario Andrés Felipe Londoño por su colaboración en la obtención de las muestras ambientales.
Financiamiento
Este estudio recibió financiación del Departamento Administrativo de Ciencia, Tecnología e innovación COLCIENCIAS, Colombia (Cod 325645221265 - 352-2008) y el Instituto Colombiano de Medicina Tropical-Universidad CES.
Conflictos de Interés
Los autores declaramos no tener conflictos de interés en la publicación de este artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. McBride A, Athanazio D, Reis M, Ko A. Leptospirosis. Curr Opin Infect Dis. 2005;18(5):376-86. [ Links ]
2. Bharti A, Nally J, Ricaldi J, Matthias M, Diaz M, Lovett M, et al. Leptospirosis: a zoonotic disease of global importance. Lancet Infect Dis. 2003;3(12):757-71. [ Links ]
3. Epstein P, Calix Pena O, Blanco Racedo J. Climate and disease in Colombia. Lancet. 1995;346(8985):1243-44. [ Links ]
4. Agudelo-Flórez P, Restrepo-Jaramillo B, Arboleda-Naranjo M. Leptospirosis in Uraba, Antioquia, Colombia: a seroepidemiological and risk factor survey in the urban population. Cad Saude Publica. 2007;23(9):2094-102. [ Links ]
5. Sebek Z, Sixl W, Valova M, Marth E, Köck M, Reinthaler F. Serological investigations for leptospirosis in humans in Columbia. Geogr Med Suppl. 1989;3:51-60. [ Links ]
6. Ferro B, Rodríguez A, Pérez M, Travi B. Seroprevalence of Leptospira infection in habitants of peripheral neighborhoods in Cali, Colombia. Biomedica. 2006;26(2):250-57. [ Links ]
7. Haake D, Suchard M, Kelley M, Dundoo M, Alt D, Zuerner R. Molecular evolution and mosaicism of leptospiral outer membrane proteins involves horizontal DNA transfer. J Bacteriol. 2004;186(9):2818-28. [ Links ]
8. de la Peña-Moctezuma A, Bulach D, Adler B. Genetic differences among the LPS biosynthetic loci of serovars of Leptospira interrogans and Leptospira borgpetersenii. FEMS Immunol Med Microbiol. 2001;31(1):73-81. [ Links ]
9. Xue F, Yan J, Picardeau M. Evolution and pathogenesis of Leptospira spp.: lessons learned from the genomes. Microbes Infect. 2009;11(3):328-33. [ Links ]
10. Koizumi N, Watanabe H. Leptospirosis vaccines: past, present, and future. J Postgrad Med.51(3):210-4. [ Links ]
11. Ko A, Goarant C, Picardeau M. Leptospira: the dawn of the molecular genetics era for an emerging zoonotic pathogen. Nat Rev Microbiol. 2009;7(10):736-47. [ Links ]
12. Kawabata H, Dancel L, Villanueva S, Yanagihara Y, Koizumi N, Watanabe H. flaB-polymerase chain reaction (flaB-PCR) and its restriction fragment length polymorphism (RFLP) analysis are an efficient tool for detection and identification of Leptospira spp. Microbiol Immunol. 2001;45(6):491-96. [ Links ]
13. Lin M, Surujballi O, Nielsen K, Nadin-Davis S, Randall G. Identification of a 35-kilodalton serovar-cross-reactive flagellar protein, FlaB, from Leptospira interrogans by N-terminal sequencing, gene cloning, and sequence analysis. Infect Immun. 1997;65(10):4355-9. [ Links ]
14. Levett P, Morey R, Galloway R, Turner D, Steigerwalt A, Mayer L. Detection of pathogenic leptospires by real-time quantitative PCR. J Med Microbiol. 2005;54(Pt 1):45-9. [ Links ]
15. Dong H, Hu Y, Xue F, Sun D, Ojcius D, Mao Y, et al. Characterization of the ompL1 gene of pathogenic Leptospira species in China and cross-immunogenicity of the OmpL1 protein. BMC Microbiol. 2008;8:223. [ Links ]
16. Ahmed A, Engelberts M, Boer K, Ahmed N, Hartskeerl R. Development and validation of a real-time PCR for detection of pathogenic leptospira species in clinical materials. PLoS One. 2009;4(9):e7093. [ Links ]
17. Cerqueira G, McBride A, Picardeau M, Ribeiro S, Moreira A, Morel V, et al. Distribution of the leptospiral immunoglobulin-like (lig) genes in pathogenic Leptospira species and application of ligB to typing leptospiral isolates. J Med Microbiol. 2009;58(Pt 9):1173-81. [ Links ]
18. Slack A, Symonds M, Dohnt M, Smythe L. Identification of pathogenic Leptospira species by conventional or real-time PCR and sequencing of the DNA gyrase subunit B encoding gene. BMC Microbiol. 2006;6:95. [ Links ]
19. Gravekamp C, Van de Kemp H, Franzen M, Carrington D, Schoone G, Van Eys G, et al. Detection of seven species of pathogenic leptospires by PCR using two sets of primers. J Gen Microbiol. 1993;139(8):1691-700. [ Links ]
20. Ren S, Fu G, Jiang X, Zeng R, Miao Y, Xu H, et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 2003;422(6934):888-93. [ Links ]
21. World Organization for Animal Health. Principles of validation of diagnostic assays for infectious diseases. Chapter 1.1.4. In: Manual of diagnostic tests and vaccines for terrestrial animals. Geneva: OIE; 2009. [ Links ]
22. Branger C, Blanchard B, Fillonneau C, Suard I, Aviat F, Chevallier B, et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiol Lett. 2005;243(2):437-45. [ Links ]
23. Wangroongsarb P, Saengsongkong W, Petkanjanapong W, Mimgratok M, Panjai D, Wootta W, et al. An application of duplex PCR for detection of Leptospira spp. and Orientia tsutsugamushi from wild rodents. Jpn J Infect Dis. 2008;61(5):407-9. [ Links ]
24. Victoria B, Ahmed A, Zuerner R, Ahmed N, Bulach D, Quinteiro J, et al. Conservation of the S10-spc-alpha locus within otherwise highly plastic genomes provides phylogenetic insight into the genus Leptospira. PLoS One. 2008;3(7):e2752. [ Links ]
25. Zuerner R, Hartskeerl R, van de Kemp H, Bal A. Characterization of the Leptospira interrogans S10-spc-alpha operon. FEMS Microbiol Lett. 2000;182(2):303-8. [ Links ]
26. Brown P, Gravekamp C, Carrington D, van de Kemp H, Hartskeerl R, Edwards C, et al. Evaluation of the polymerase chain reaction for early diagnosis of leptospirosis. J Med Microbiol. 1995;43(2):110-14. [ Links ]
27. Agudelo-Flórez P, Londoño A, Quiroz V, Angel J, Moreno N, Loaiza E, et al. Prevalence of Leptospira spp. in urban rodents from a groceries trade center of Medellin, Colombia. Am J Trop Med Hyg. 2009;81(5):906-10. [ Links ]
28. Szonyi B, Agudelo-Flórez P, Ramírez M, Moreno N, Ko A. An outbreak of severe leptospirosis in capuchin (Cebus) monkeys. Vet J. 2010 May 27 [Epub ahead of print]. [ Links ]
29. Nally J, Whitelegge J, Bassilian S, Blanco D, Lovett M. Characterization of the outer membrane proteome of Leptospira interrogans expressed during acute lethal infection. Infect Immun. 2007;75(2):766-73. [ Links ]
30. Vital-Brazil J, Balassiano I, Oliveira F, Costa A, Hillen L, Pereira M. Multiplex PCR-based detection of Leptospira in environmental water samples obtained from a slum settlement. Mem Inst Oswaldo Cruz. 2010;105(3):353-55. [ Links ]
31. Romero E, Billerbeck A, Lando V, Camargo E, Souza C, Yasuda P. Detection of Leptospira DNA in patients with aseptic meningitis by PCR. J Clin Microbiol. 1998;36(5):1453-55. [ Links ]
32. Merien F, Baranton G, Perolat P. Comparison of polymerase chain reaction with microagglutination test and culture for diagnosis of leptospirosis. J Infect Dis. 1995;172(1):281-85. [ Links ]
Correspondencia: Piedad Agudelo Flórez.
Dirección: Cra 43A N.º 52 Sur-99, Sabaneta-Colombia.
Teléfono 574-3053500 extensión 2314. Fax 574-3014258
Email pagudelo@ces.edu.co
Recibido: 14-07-10
Aprobado: 27-10-10