Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.28 n.1 Lima mar. 2011
ORIGINAL BREVE
Circulación de un linaje diferente del virus dengue 2 genotipo América / Asia en la región amazónica de Perú, 2010
Circulation of a different lineage of dengue virus serotype 2 American / Asian genotype in the Peruvian amazon, 2010
Enrique Mamani1,a, Carlos Álvarez 2,b, María García M.1, c, Dana Figueroa1,a, Milady Gatti2,a, Heinner Guio3,b, Susy Merino1,c, Pedro Valencia4,b, Carlos Calampa2,b , Leticia Franco5,d, César Cabezas6,b
1 Laboratorio de Arbovirus – Instituto Nacional de Salud, Perú.
2 Dirección Regional de Salud Loreto.
3 Laboratorio de Biología Molecular y Biotecnología – Instituto Nacional de Salud.
4 Centro Nacional de Salud Pública - Instituto Nacional de Salud, Perú.
5 Laboratorio de Arbovirus y virus importados. Centro Nacional de Microbiología. Instituto de Salud Carlos III – Madrid-España.
6 Instituto Nacional de Salud, Perú.
a Biólogo; b Médico; c Tecnólogo Médico; d Bioquímica.
RESUMEN
El objetivo del estudio fue determinar el genotipo del virus dengue tipo 2 (DENV-2) que circuló en la región Amazónica de Perú entre noviembre de 2010 y enero de 2011. Se analizaron ocho muestras de pacientes captados durante la vigilancia para dengue en las ciudades de Iquitos, Yurimaguas, Trujillo, Tarapoto y Lima entre noviembre de 2010 y enero de 2011 que fueron remitidas al Instituto Nacional de Salud. Se realizó el aislamiento viral en la línea C6/36 HT y la extracción del ARN viral. Se aplicaron técnicas de biología molecular para establecer el serotipo (RT – PCR múltiple) y genotipo (RT-Nested PCR de la región E/NS1) seguidas de secuenciación y análisis filogenético. El análisis filogenético reveló la introducción de un linaje diferente que ingresó a Perú a finales del 2010. Estos aislamientos encontrados en Iquitos y otras ciudades de Perú están muy relacionados con aislamientos de DENV-2 que circularon en Brasil durante el 2007 y 2008 asociados con casos de dengue grave y muertes. En conclusión se detectó la introducción de un linaje diferente del DENV-2 genotipo América/Asia en Perú que podría estar asociado con la presencia de casos más graves de dengue.
Palabras clave: Dengue, Genotipo, Perú (fuente: DeCS BIREME).
ABSTRACT
Our objective was to determine the genotype of the dengue virus type 2 (DENV-2) that circulated in the Amazon region of Peru between November 2010 and January 2011. We analyzed eight samples collected during dengue surveillance activities in the cities of Iquitos, Yurimaguas, Trujillo, Tarapoto and Lima between November 2010 and January 2011 that were sent to Insitituto Nacional de Salud. The viruses were isolated in C6/36 HT cell line. Viral RNA was extracted and the serotype (RT - PCR multiplex) and genotype (RT-Nested PCR of the region E/NS1) were determined. Finally, the E/ NS1 amplicons were sequenced and analyzed by phylogeny. The phylogenetic analysis revealed the introduction of a different lineage which entered in Peru by the end of 2010. These isolates found in Iquitos and other cities in Peru are closely related to DENV-2 isolates that circulated in Brazil during 2007 and 2008, associated with severe dengue cases and deaths. In conclusion, we detected the introduction of a different lineage of DENV-2 America / Asia genotype in Peru that could be associated with the presence of more severe cases.
Keywords: Dengue Virus Type 2, Genotype, Peru (source: MeSH NLM).
INTRODUCCIÓN
El dengue sigue siendo un problema para la salud pública en las Américas. Está causado por el virus dengue (DENV), perteneciente a la familia flaviviridae, género flavivirus. La distribución de la enfermedad es predominante en las áreas urbanas de zonas tropicales y subtropicales en todo el mundo. Existen cuatro serotipos de virus dengue (DENV-1, 2, 3, y 4) manteniéndose en la naturaleza tanto en ciclos endémicos, a través del mosquito Ae. aegypti principalmente, y en ciclos selváticos enzoóticos entre primates no humanos y varias especies de mosquitos Aedes. La infección con cualquiera de los cuatro serotipos del DENV produce una sintomatología clínica variada que va desde una infección inaparente, pasando por el dengue clásico (DC) hasta formas más graves como el dengue hemorrágico (DH) y el síndrome de shock por dengue (SCD), este último puede producir la muerte del paciente (1). Actualmente, con la aparición de manifestaciones atípicas de la enfermedad incluyendo presentaciones neurológicas, gastrointestinales o cardiacas sin signos hemorrágicos, la Organización Mundial de la Salud OMS junto con el TDR (Programa Especial de Entrenamiento en Enfermedades Tropicales), ha actualizado la clasificación clínica del dengue según la presencia o ausencia de signos de alarma y formas graves. Esta última categoría, además de los casos hemorrágicos incluye aquellas manifestaciones no usuales (2). Las formas graves de dengue (dengue hemorrágico y síndrome de shock) reconocidas desde la década de 1950 siguen siendo consideradas como principal causa de muerte infantil en varios países (1,2).
En Perú, la reemergencia del dengue fue detectada en la ciudad de Iquitos en 1990 con el hallazgo del DENV-1. Para el año 2000, los cuatro serotipos de DENV estaban circulando en el noroeste de Perú, así como en otras regiones tropicales, se informaron miles de casos de DC y casos de DH/SCD, se aislaron los cuatro serotipos del virus dengue y se determinó la circulación de dos genotipos del DENV-2, el americano nativo y el genotipo América - Asia (3-10).
El DENV-2 genotipo América ingresó a Iquitos en el año 1995, causando un brote sin presencia de casos graves, pese a que cinco años antes se tuvo un brote con muchos casos asociados el DENV-1 (11,12). El ingreso del DENV-3 fue en el año 2001 (13), el DENV-2 genotipo asiático ingresó en el 2002 y el DENV-4 en el 2008 (5).
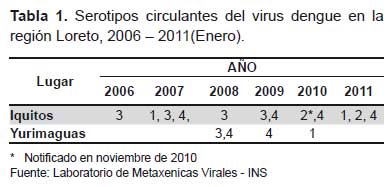
En lo que va del año 2011 se tiene el reporte de la circulación de los serotipos DENV-1, DENV-2 y DENV-4 (Tabla 1).

Durante el 2010, la Oficina de Epidemiología de la región Loreto, notificó 2135 casos de dengue sin señales de alarma; 799 (37,42%) confirmados y 1336 (62,58%) permanecen como probables. En el canal endémico se observó (14) incremento de casos a partir de la semana epidemiológica (SE) 42, llegando a notificarse 138 casos en la SE 51, considerado valor máximo hasta la SE 2 del 2011, donde se ubica en la zona de epidemia. Durante este periodo se notificaron 59 casos de dengue con señales de alarma (dos fueron confirmados) y siete casos de dengue grave (tres confirmados). Dadas las características del brote descrito, consideramos importante la tipificación del virus circulante siendo por tanto el objetivo del presente estudio determinar el genotipo del virus dengue tipo 2 (DENV-2) que circuló en la región Amazónica de Perú entre noviembre de 2010 y enero de 2011.
EL ESTUDIO
MUESTRAS
Ocho sueros de pacientes febriles con inicio de síntomas menor a cinco días, que fueron atendidos en los establecimientos del Ministerio de Salud durante la vigilancia de dengue que se realiza a nivel nacional con la participación del Área de Epidemiología y los Laboratorios de Salud Pública de cada región. Las ocho muestras en estudio fueron obtenidas, además de Iquitos (3), en las ciudades de Yurimaguas (1) y Tarapoto (1), ubicadas en la Amazonía peruana; las restantes fueron obtenidas en las ciudades costeras de Trujillo (1) y Lima (2). Los pacientes procedentes de las últimas cuatro ciudades indicaron que habían viajado a la ciudad de Iquitos antes de presentar los signos y síntomas de la enfermedad. Las muestras fueron enviadas en cadena de frío y procesadas en el Laboratorio de Arbovirus y Biología Molecular del INS.
AISLAMIENTO VIRAL
Se aisló DENV-2 en línea celular de Aedes albopictus C6/36 HT.
EXTRACCIÓN DE ÁCIDOS NUCLEICOS
La extracción del RNA viral fue realizada siguiendo el protocolo del QIAamp Viral RNA Mini Kit (Qiagen, Alemania)
RT-PCR MÚLTIPLEX
Se realizó RT-PCR múltiplex (9). Cinco μL del ARN viral extraído fue adicionado en 20 μL de una mezcla RTPCR constituido de cloruro de potasio 50mM, Tris 10mM (pH 8,5), gelatina 0,01%, 200 mM de cada deoxinucleótidos trifosfato, cloruro de magnesio 1,5 mM, tetrametil amonio clroruro 30mM, betaina 0,5 M, ditriotreitol l5 mM, primers 5D1 y 3TS1 1mM cada uno, primers 3 TS2, TS3 y DENV-4 0,5 mM cada uno, transcriptasa reversa 1U/ mL MMLV (Invitrogen), Taq DNA polimerasa 0,025 U/mL (Invitrogen). La transcripción reversa se realizó a 42 °C por 60 minutos, seguido por 40 ciclos de amplificación a 94 °C por 30 s, 55 °C por 1 min, y 72 °C por 2 min, una extensión final a 72 °C por 5 min. La RT-PCR fue realizada en tubos 0,2 mL en un termociclador JM Research. 10μL de cada producto amplificado fue analizado en electroforesis en gel de agarosa al 1.5% en buffer TAE (tris-acetato 2M, EDTA 0,05 M pH 8,3). El tamaño de los productos esperados fueron: 482 pb para DENV-1, 119 bp para DENV-2, 290 bp para DENV-3, y 389 bp para DENV-4. EL control positivo para la RT-PCR incluyó el extraído de ARN de las cepas referenciales de los cuatro serotipos.
RT-NESTED PCR DE LA REGIÓN E/NS1
Se realizó la RT-Nested PCR en la región E/NS1 del genoma del virus dengue aplicado para la genotipificación del virus a partir de muestras clínicas. Cinco μL del ARN viral se adicionaron a 45 μL del kit (OneStep RT-PCR Qiagen).La mezcla maestra del kit contiene buffer, 400 mM de cada dNTP, 20 pmol de cada primer degenerado sentido y antisentido para DENV-2, (S1871DEN2, AS2622DEN2) y enzimas. Se realizó una transcripción reversa inicial a 41 °C por 45 min seguido de una desnaturalización y activación de la Taq polimerasa (94 °C, 15 min) y 40 ciclos de desnaturalización (94 °C, 30 s), hibridación (55 °C, 1 min), y extensión (72 °C, 30 s), se realizó una extensión final a 72 °C por 5 min. Una segunda amplificación (Nested PCR) fue realizada adicionando 1 μL del producto de amplificación inicial. La mezcla de reacción contiene buffer (60 mM Tris- HCl pH 8.5, 2 mM) MgCl2, 15 mM (NH4)2SO4; 2,5 U de Taq DNA polimerasa (Invitrogen) y 40 pmol de cada primer sense y antisense para DENV-2 (S2176DEN2, AS2504DEN). Se realizó una desnaturalización activación de la Taq polimerasa (94 °C, 2 min) y 40 ciclos de desnaturalización (94 °C, 30 s), hibridación (57 °C, 4 min), y extensión (72 °C, 30 s). Se realizó una extensión final a 72 °C por 5 min.
SECUENCIACIÓN DE LA REGIÓN E/NS1
Los productos amplificados fueron purificados (QIAquick PCR Purification Kit, Qiagen.) y secuenciados con el método Big dye terminator (kit Big Dye Terminator v. 3.1, Applied Biosystems). Para cada reacción de secuenciación se utilizó entre 50 a 100 ng de ADN con 3°2 pmol de primer y la mezcla de la reacción conteniendo cuatro dideoxinucleótidos terminadores marcados. Los ciclos de amplificación utilizados fueron los descritos en el protocolo del fabricante. El producto de reacción fue purificado en columna (Macherey-Nagel) y el ADN fue secado en centrifuga al vacío por 20 min. El pellet fue resuspendido en 10 μL de formamida, calentado a 95 °C por 2 min y mantenido en hielo hasta el cargado en el secuenciador Prism 310 (Applied Biosystems) utilizando un capilar de 47 cm por 50 μm con POP-6 (Applied Biosystems).
Las secuencias sentido y antisentido de cada muestra fueron editadas usando el software SeqMan Pro (Dnastar Inc., Wisconsin, E.U.A.). La secuencia consenso de 306 bp fue alineada y comparada con otras reportadas en el Genbank que incluye los genotipos America/Asia , Asia y América, respectivamente utilizando el programa CLUSTAL X, versión 1,83 (15). El análisis filogenético se realizó con el programa MEGA v. 4.0 (16), mediante el método Neighbor Joining, modelo Tamura-Nei con 1000 repeticiones.
HALLAZGOS
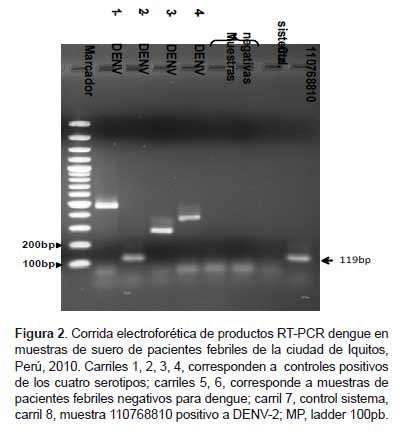
Las muestras analizadas mediante RT-PCR multiplex y electroforesis en gel de agarosa presentaron un producto de amplificación de 119 pb que corresponde al DENV-2 (Figura 2).
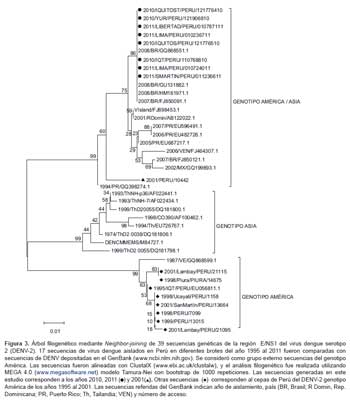
Posteriormente, las secuencias de DENV-2 resultantes de la amplificación de la región E/NS1 (306 pb) de las ocho muestras analizadas fueron comparadas mediante análisis filogenético, junto con otras secuencias obtenidas de GenBank (http://www.ncbi.nlm.nih.gov/genbank/GenbankSearch.html), agrupándose dentro del genotipo America/Asia. Dentro de este genotipo, las ocho muestras analizadas en este estudio forman un grupo definido con las secuencias reportadas de Brasil del 2007 (FJ850091.1) y 2008 (HM181971.1, GQ868551.1, GU131882.1) (21), con un valor de bootstrap mayor a 80%. Mientras que las secuencias reportadas de otros países como República Dominicana (AB122022.1), México (GQ199893.1), Venezuela (J464307.1) y Puerto Rico (EU687217.1, EU482726.1, EU596491.1) formaron un clado distinto. Interesantemente la secuencia de DENV-2 de la muestra 10442 de Perú detectada en el año 2001 se agrupó en una rama diferente a las muestras en estudio.
Dentro del análisis filogenético también se consideró ocho secuencias de Perú que corresponden a cepas DENV-2 de diferentes áreas geográficas: Iquitos (1995), costa norte (Piura, 1998; Lambayeque, 2001), Ucayali (1998), San Martín (2001), además de una secuencia de Venezuela (GQ868599.1) del año 1987. Todas las secuencias mencionadas se agruparon en una rama diferente y característica del Genotipo América (Figura 3).
DISCUSIÓN
El DENV-1 se detectó en Iquitos el año 1990 observándose los primeros casos de dengue (3). En 1995 se reportó en esta ciudad la circulación del DENV-2, muchos de los pacientes cursaron una infección secundaria pero no se presentaron casos de dengue hemorrágico o grave (11). DENV-3 y DENV-4 ingresaron a Iquitos el 2001 y 2004, respectivamente(13).
El DENV-2 es el único serotipo que presentó dos genotipos circulantes en Perú, el genotipo América que ingreso en 1995 en Iquitos y el genotipo América/Asia que ingresó el 2000-2001 en la costa norte del país y luego se dispersó por otras áreas geográficas incluido Iquitos donde circuló el año 2002 (17).
El ingreso del genotipo América/Asia en el 2000-2001 en la costa norte de Perú estuvo asociado con un brote donde se reportó los primeros casos de dengue hemorrágico con casos fatales. Este brote, además, se caracterizó por la circulación de los cuatro serotipos; el DENV-2 presentó los dos genotipos mencionados (5). Los factores como cocirculación de diferentes serotipos, presencia de casos de dengue durante todo el año, índices aédicos altos, entre otros, condicionaron la presencia de los casos graves y la mayor cantidad de diferencias del DENV-2 respecto a los otros serotipos y la mayor proporción de pacientes con prueba de lazo positivo (manifestación hemorrágica más frecuentemente encontrada en la mayoría de epidemias) por lo que se señalaría a este serotipo como el responsable de mayor sintomatología y gravedad en el Perú, independientemente de la condición de infección primaria y secundaria (8).
En el presente trabajo se pudo determinar que un linaje diferente de DENV-2 está circulando en la ciudad de Iquitos y que esta cepa viral está siendo importada desde Iquitos hacia al menos otras cuatro ciudades de Perú (Yurimaguas, Tarapoto, Trujillo y Lima).
La reintroducción de este linaje diferente del DENV-2 genotipo América/Asia en la ciudad de Iquitos a finales del 2010 estaría asociado con un brote de dengue con casos graves y muertes, y su secuencia presenta alta homología con la secuencia de aislados de Brasil (18) del 2007 y 2008 asociada con casos graves y muertes, formando un linaje muy diferente respecto a los virus circulantes reportados en otros países de centro y Sudamérica. Por otro lado, queda claramente definido que el DENV-2 genotipo América/Asia que circuló en el año 2001 en Perú es genéticamente distinto al del 2010 y no está asociado con casos graves (7). Sin embargo, es necesario indicar que varios factores cumplen un papel importante en el desarrollo de la patogenia de del dengue, como infección secundaria, edad, inóculo viral, entre otros.
Por la aparente importación de casos de DENV-2 pertenecientes a este linaje, es necesario fortalecer la vigilancia del síndrome febril en esta y otras poblaciones de la Amazonía peruana, así como también en brotes de dengue en otras áreas del país. También se debería hacer un seguimiento de la situación acontecida en países fronterizos para conocer la presencia de este linaje y su relación con la presentación clínica. Las futuras vigilancias de brotes en Perú deberían de ser conducidas con una nueva perspectiva para el entendimiento virológico y riesgo clínico epidemiológico de las infecciones por virus asociados con gravedad de la enfermedad.
AGRADECIMIENTOS
Antonio Tenorio del ISCIII – España por su colaboración con los cebadores utilizados para la secuenciación de la región E/NS1, Rosa Palacios y Miguel Farfán por realizar el aislamiento del virus dengue en el Laboratorio de Metaxénicas virales del INS, Carlos Padilla por realizar la secuenciación en el Laboratorio de Biología Molecular y Biotecnología del INS.
Fuentes de Financiamiento
Instituto Nacional de Salud.
Conflictos de Interés
Los autores declaran no tener conflictos de interés en la publicación del presente estudio.
REFERENCIAS BIBLIOGRÁFICAS
1. Gubler DJ. Dengue and dengue hemorrhagic fever. Cin Microbiol Rev. 1998; 11(3):480-96. [ Links ]
2. World Health Organization. Dengue Guidelines for Diagnosis, Treatment and Control. Geneva: WHO; 2009 Disponible en: http://whqlibdoc.who.int/publications/2009/9789241547871_eng.pdf [ Links ]
3. Giraldo A. Primer brote de Dengue documentado en la región Amazónica del Perú. Bol Of Sanit Panam. 1993; 114(6):513-19. [ Links ]
4. Cabezas C. Dengue en el Perú: aportes para su diagnóstico y control. Rev Peru Med Exp Salud Publica. 2005;22(3): 212-28. [ Links ]
5. Forshey BM, Morrison AC, Cruz C, Rocha C, Vilcarromero S, Guevara C, et al. Dengue virus serotype 4, northeastern Peru, 2008. Emerg Infect Dis. 2009;15(11):1815-8. [ Links ]
6. Chowell G, Torre CA, Munayco-Escate C, Suárez-Ognio L, López-Cruz RHyman JM, et al. Spatial and temporal dynamics of dengue fever in Peru: 1994-2006. Epidemiol Infect. 2008;136(12):1667-77. [ Links ]
7. Mostorino R, Rosas A, Gutiérrez V, Anaya E, Cobos M, García M. Manifestaciones clínicas y distribución geográfica de los serotipos del dengue en el Perú, año 2001. Rev Peru Med Exp Salud Publica. 2002;19(4):171-80. [ Links ]
8. Cobos M, Gutiérrez V, García M, Mamani E, Fernández R, Rimarachín R et al. Estudio serológico y virológico del brote de dengue en la provincia de Coronel Portillo Ucayali, Perú (2000-2001). Rev Peru Med Exp Salud Publica. 2004; 21(3):139-45. [ Links ]
9. Mamani E, García M, Gutiérrez V, Cabezas C, Harris E. Tipificación molecular del virus dengue 3 durante el brote epidémico de dengue clásico en Lima, Perú, 2005. Rev Peru Med Exp Salud Publica. 2005;22(3):161-4. [ Links ]
10. Mamani E, Figueroa D, Garcia M. Garaycochea M, Pozo E. Infecciones concurrentes por dos serotipos del virus dengue durante un brote en el noroeste de Perú, 2008. Rev Peru Med Exp Salud Publica. 2010; 27(1):16-21. [ Links ]
11. Watts DM, Porter KR, Putvatana P, Vasquez B, Calampa C, Hayes CG, Halstead SB. Failure of secondary infection with American genotype dengue 2 to cause dengue haemorrhagic fever. Lancet. 1999;354(9188):1431-4. [ Links ]
12. Hayes CG, Phillips IA, Callahan JD, Griebenow WF, Hyams KC, et al. The epidemiology of dengue virus infection among urban, jungle, and rural populations in the Amazon region of Peru. Am J Trop Med Hyg. 1996;55:459–463. [ Links ]
13. Kochel T, Aguilar P, Felices V, Comach G, Cruz C, et al. Molecular epidemiology of dengue virus type 3 in Northern South America: 2000–2005. Infect Genet Evol. 2008;8:682–688. [ Links ]
14. Ministerio de Salud del Perú, DIRESA Loreto, Oficina de Epidemiología. Informe técnico: situación del brote de dengue en Iquitos, al 11 de enero del 2011. Lima: MINSA; 2011. [ Links ]
15. Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG. The Clustal_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 1997;25(24):4876-82. [ Links ]
16. Tamura K, Dudley J, Nei M, Kumar S MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol. 2007; 24(8):1596-9. [ Links ]
17. Morrison A, Minnick S, Rocha C, Forshey B, Stoddard S, Getis A, Focks D, et al. Epidemiology of dengue virus in Iquitos, Peru 1999 to 2005: interepidemic and epidemic patterns of transmission. PLoS Negl Trop Dis. 2010 May; 4(5):e670. [ Links ]
18. Michelli Faria Oliveira MF, Galvão Araújo JM, Ferreira OC Jr., Ferreira DF, Lima DB, Santos FB. Two Lineages of Dengue Virus Type 2, Brazil. Emerg Infect Dis. 2010; 16(3):576-578. [ Links ]
Correspondencia: Blgo. Enrique Mamani Zapana.
Dirección: Av. Defensores del Morro 2268 Chorrillos, Lima 9, Perú.
Teléfono: (511) 617-6200; Anexo 1545; (511) 995-903830.
Correo electrónico: emamani@ins.gob.pe; e_mamani@hotmail.com
Recibido: 17-01-11
Aprobado: 09-03-11