Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.30 no.2 Lima Apr. 2013
Original Breve
Variabilidad genética del Aedes aegypti determinada mediante el análisis del gen mitocondrial Nd4 en once áreas endémicas para dengue en el Perú
Genetic variability of Aedes Aegypti determined by mitochondrial gene ND4 analysis in eleven endemic areas for dengue in Peru
Pamela Yáñez1,a, Enrique Mamani2,a, Jorge Valle3,a, María Paquita García2,b, Walter León3,a, Pablo Villaseca3,a, Dina Torres2,a, César Cabezas4,c
1 Universidad Nacional de San Antonio Abad del Cusco. Cusco, Perú.
2 Laboratorio de Metaxénicas Virales, Instituto Nacional de Salud. Lima, Perú.
3 Laboratorio de Entomología, Instituto Nacional de Salud. Lima, Perú.
4 Instituto Nacional de salud. Lima. Perú
a Biólogo, b tecnólogo médico, c médico cirujano.
RESUMEN
Con el objetivo de establecer la variabilidad genética de Aedes aegypti determinada por el análisis del gen mitocondrial ND4, se analizaron 51 especímenes de Ae. aegypti en once regiones endémicas para dengue en el Perú. La variabilidad genética se determinó mediante la amplificación y secuenciación de un fragmento de 336 pares de bases del gen mitocondrial ND4. El análisis de filogenia intraespecífica se realizó con el programa Network Ver. 4.6.10; y el análisis filogenético, con el método de distancia Neighbor Joining. Se identificó la presencia de cinco haplotipos de Ae. aegypti agrupados en dos linajes: el primero agrupa a los haplotipos 1, 3 y 5 y el segundo agrupa los haplotipos 2 y 4, se muestra además la distribución geográfica de cada uno de los haplotipos encontrados. Se concluye que esta variabilidad se debe tanto a la migración activa de este vector como a la migración pasiva mediada por la actividad humana.
Palabras clave: Aedes; Haplotipos; Linaje; ADN Mitocondrial; Perú (fuente: DeCS BIREME).
ABSTRACT
In order to establish the genetic variability of Aedes aegypti determined by the analysis of the MT-ND4 gene, in eleven endemic regions for dengue in Peru, 51 samples of Ae. Aegypti were tested. The genetic variability was determined through the amplification and sequencing of a fragment of 336 base-pairs of MT ND4, the analysis of intra-specific phylogeny was conducted with the Network Ver. 4.6.10 program; and the phylogenetic analysis, with the Neighbor Joining distance method. The presence of five haplotypes of Ae. Aegypti grouped in two lineages was identified: the first one includes haplotypes 1, 3 and 5, and the second one comprises haplotypes 2 and 4. The geographic distribution of each of the haplotypes found is also shown. It is concluded that this variability is caused by the active migration of this vector and the human activity-mediated passive migration.
Key words: Aedes; Haplotypes; Pedigree; DNA, Mitochondrial; Peru (source: MeSH NLM).
INTRODUCCIÓN
El Aedes aegypti es el vector de los cuatro serotipos del virus de dengue (DENV), se encuentra en casi 100 países tropicales y se calcula que 2,5 billones de personas habitan en áreas donde existe el riesgo de transmisión de la epidemia (1). Es considerado como uno de los vectores más eficientes en la transmisión del dengue debido a que es altamente antropofílico (2).
En el Perú, el Aedes aegypti fue detectado por primera vez en el año 1852, habría ingresado por la frontera norte desde la región de Guayaquil (Ecuador), extendiéndose progresivamente a lo largo de la costa del Perú (3). En 1938, los resultados de una encuesta nacional mostraron que 11 de los 24 departamentos del Perú estaban infestados con Aedes aegypti (4), anunciándose su erradicación para 1958, como resultado de la intensa campaña de control ejecutada por el Ministerio de Salud del Perú, con el apoyo de la Organización Panamericana de la Salud (5). Sin embargo, una reinfestación de Aedes aegypti fue detectada en Loreto en 1984, a partir de la cual se expandió a varios departamentos del Perú. En el 2001 se informó de su presencia en la ciudad de Lima (6) y esta se asoció, en 2005, a una epidemia de dengue en el distrito de Comas (7). En el 2012, la Dirección General de Epidemiologia del Perú informó que el Aedes aegypti estaba presente en 17 departamentos del Perú y que el DENV circulaba en 15 de ellos (8).
La amplia distribución de este vector en el territorio peruano, sumado a las graves repercusiones que traen las epidemias de dengue, hacen necesario el desarrollo y la elaboración de estrategias de control. Estudios previos señalan que el conocimiento de la variabilidad genética de este vector puede contribuir en el diseño de estrategias de control vectorial (9). Una de alternativas viables para el estudio de la variabilidad genética del Aedes aegypti se realiza a través del ADN mitocondrial. El cual posee numerosas ventajas para el estudio de las relaciones evolutivas, ya que su longitud es mucho menor que el ADN hallado en los cromosomas del núcleo, es abundante, tiene una mayor tasa de evolución, posee una gran variación intraespecífica y es de herencia uniparenteral (10). Entre los genes mitocondriales, el gen de la nicotinamida adenín dinucleótido subunidad 4 (ND4) ha demostrado ser un excelente marcador para el análisis de la estructura genética de la población y eventos de colonización de Aedes aegypti (11). El estudio de las variaciones del gen ND4 permite determinar los haplotipos de Aedes aegypti existentes en diferentes áreas geográficas, con lo cual se podría además tomar medidas específicas. El objetivo del presente estudio fue establecer la variabilidad genética de Aedes aegypti mediante el gen mitocondrial ND4 en algunas áreas endémicas para dengue de Perú.
EL ESTUDIO
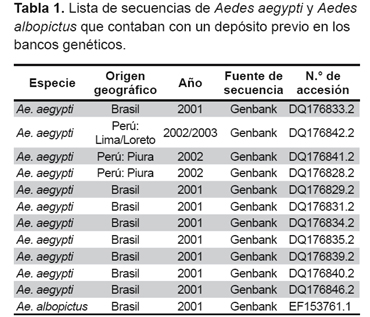
Se realizó un estudio transversal, para el cual se incluyeron 49 especímenes de Aedes aegypti (larvas o adultos) seleccionados de manera aleatoria entre el 2006 y el 2013 colectados de once regiones del Perú endémicas para dengue, y que cuentan además con un sistema de vigilancia entomológica: Amazonas (Bagua); Cusco (Quillabamba y Camanti); Loreto (Iquitos); Cajamarca (Jaén); Lima (Comas y Jicamarca); Madre de Dios (Puerto Maldonado); Piura (Sullana y Castilla); Ucayali (Pucallpa); San Martin (Tarapoto); La Libertad (Trujillo), y Tumbes (La Cruz y Villa Primavera), donde previamente se reportó tanto la presencia de Aedes aegypti así como casos de dengue. Los especímenes de Aedes aegypti fueron capturados en el área urbana, intradomiciliaria y conservados en alcohol de 70° para ser transportados al Instituto Nacional de Salud (Anexo 1). Además de estos especímenes se incluyeron en el análisis doce secuencias previamente reportadas en el GenBank, las cuales fueron utilizadas como patrones de comparación con las secuencias halladas en el presente estudio (9). (Tabla 1).
AISLAMIENTO DEL ADN TOTAL DE Aedes aegypti
El aislamiento de ADN se realizó empleando membranas de sílice (DNeasy Blood & Tissue Kit®, Qiagen, Hilden, Germany), protocolo diseñado para la purificación del ADN total de insectos de más de 50 mg de peso (12).
AMPLIFICACIÓN DEL FRAGMENTO DEL GEN MITOCONDRIAL ND4 DE Aedes aegypti
Un fragmento de 377 pares de bases del gen mitocondrial ND4 de Ae. aegypti fue amplificado mediante PCR, el volumen de la reacción fue de 25 µL, con una concentración final de Buffer PCR 1x (Invitrogen), MgCl2 3 mM (Invitrogen), mix de dNTPs 400 µM (Applied Biosystems), 0,4 µM de cada primer reportado previamente (9), 1 U/ µL de Taq Polimerasa (Invitrogen) y 2 µL de la muestra de ADN.
La PCR fue realizada con un ciclo inicial a 94 °C por 2 min; 40 ciclos a 94 °C por 1 min; 62 °C por 30 s y 72 °C por 1 min; la extensión final fue de 72 °C por 7 min; 10 µL del producto de amplificación fue sometido a electroforesis horizontal en gel de agarosa al 1,5% con 2 µL del buffer loading 6x en buffer TAE 1x a 110v por 60 min. El gel fue teñido con 0,5 µg/mL de bromuro de etidio por 10 min y visualizado en un transiluminador de luz UV. Finalmente, se documentaron las imágenes con fotodocumentador de geles BioRad modelo ChemiDoc XRS+.
SECUENCIACIÓN DEL FRAGMENTO DEL GEN MITOCONDRIAL ND4 DE Aedes aegypti
Los productos de PCR fueron purificados mediante la utilización del QIAquick PCR Purification Kit® (Qiagen, Hilden, Germany) (13). La secuenciación de los fragmentos amplificados se llevó a cabo utilizando el kit BigDye® Terminator v3.1 Cycle Sequencing Kit. El volumen final de la reacción fue de 15 µL conteniendo 4 µL de RR 100 BigDye; 2 µL de buffer 5X BygDye; 5 µL agua grado molecular; 2 µL del primer 2 mM y 2 µL de producto de PCR purificado. El termociclador se programó para un paso de desnaturalización inicial de 94 °C por 3 min; seguido por 25 ciclos de 96 °C por 10 s; 50 °C por 5 s y 60 °C por 4 min. Los productos de secuenciación fueron purificados según especificaciones técnicas del Kit Centri Sep (Princeton Separations) (14), utilizándose luego el secuenciador 3500xL (Applied Biosystems). Las secuencias consenso se obtuvieron a través del análisis de las secuencias sentido y antisentido de cada fragmento de PCR obteniéndose secuencias de 336 pb, mediante el uso del programa BioEdit ver. 7.0.9.0 y el software SeqMan II (Dnastar Inc).
ANÁLISIS DE SECUENCIAS
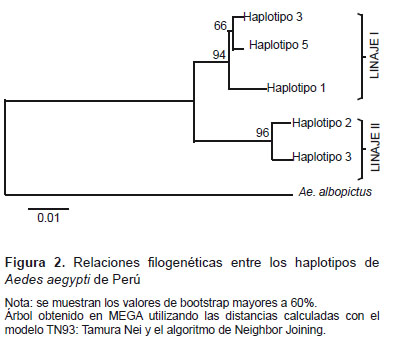
Las secuencias fueron alineadas con el programa CLUSTAL X ver. 2.1, junto con otras secuencias previamente reportadas en el GenBank que corresponde a Brasil y Perú, además de una secuencia de Aedes albopictus, obtenida por Costa, et al., 2006 (15) que se utilizó como grupo externo . Para hallar los valores de diversidad genética (H) y nucleotídica (Pi) se utilizó el programa DNAsp ver. 5.10.01. El análisis filogenético de los haplotipos encontrados se realizó mediante la utilización del programa MEGA ver. 5, con el algoritmo de Neighbor Joining (NJ), se hizo mediante la construcción de un árbol con un bootstrap de 1000 autoréplicas, utilizando como grupo externo a Aedes albopictus (16). El análisis de filogenia intraespecífica utilizando redes de haplotipos fue realizado con el programa Network ver. 4.6.1.0 con el algoritmo Median joining.
HALLAZGOS
Se obtuvieron 49 secuencias del gen ND4, cada una de 336 pb, con 321 sitios conservados, 15 posiciones variables, todas de carácter informativo y mutaciones sinónimas. Al analizar las secuencias encontradas para Perú se encontraron cinco haplotipos, en la Figura 1 se muestra la distribución de cada uno de ellos en relación a los puntos en los que fueron colectados.
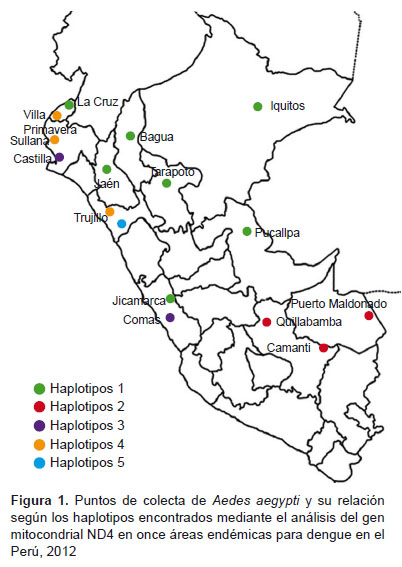
El análisis filogenético de los haplotipos determinó que ellos se encuentran agrupados en dos linajes. El primer linaje incluyó a los haplotipos 1, 3 y 5, el haplotipo 1 estuvo separado por tres mutaciones del haplotipo 3 y el haplotipo 5 estuvo separado del haplotipo 3 por una mutación. El segundo linaje, separado del primero por nueve mutaciones, incluyó los haplotipos 2 y 4; el haplotipo 2 estuvo separado del haplotipo 4 por tres mutaciones (Figura 2, Anexo 2).
DISCUSIÓN
El hallazgo de cinco haplotipos agrupados en dos linajes y distribuidos en once regiones del Perú descrito en el presente estudio, coincide con estudios previos que informan de la presencia de tres haplotipos, dos de ellos en la región Piura, y uno presente en Lima y Loreto (Iquitos) (17). De igual manera, otro estudio en el 2005 señaló que la variabilidad genética de Aedes aegypti en América era 0,82; y el valor de diversidad nucleotídica, 0,02 (9). Por su parte, la presencia en Perú de dos linajes de Aedes aegypti, concuerdan con estudios previos que dividen a la población de Aedes aegypti en dos grandes grupos, la variante costera y la variante sierra-selva (18, 19). La existencia de estos dos linajes estaría explicada por los resultados de otra investigación que señaló la introducción de, al menos, dos linajes genéticos de Aedes aegypti al Perú (17); a ello se suma la investigación de Bracco, et al. quienes señalan la presencia de solo dos linajes genéticos de Aedes aegypti en todo el continente americano (9). La diferencia encontrada entra ambos linajes en nueve mutaciones, es similar a un estudio que indicó la existencia de tres haplotipos, el primero de ellos presente en Lima e Iquitos, que distaba de un haplotipo presente en Piura por cuatro mutaciones, y de un tercer haplotipo (también en Piura) en diez mutaciones (17).
La variabilidad genética de Aedes aegypti en las regiones geográficas de Perú se debería a la migración activa que realiza la hembra del Aedes aegypti en busca de donde poner los huevos (2); sin embargo, este tipo de migración presenta restricciones geográficas y climáticas. Entonces, la variabilidad genética de Aedes aegypti también estaría explicada por una migración pasiva o forzada mediada por la actividad del ser humano, expresada en su actividades comerciales y en los sistemas de trasporte. Este tipo de migración, adicionalmente, explicaría la existencia de un mismo haplotipo en regiones geográficas marcadamente distantes, como los casos de Lima e Iquitos. Lugares en los cuales estudios previos también han señalado que comparten un mismo haplotipo (17).
Estudios previos han señalado la existencia en Perú de dos variantes de Aedes aegypti, la variante costera y la variante sierra-selva, separadas por la cordillera de los Andes, afirmando que esta serviría como una barrera natural que evitaría la dispersión de este vector. Nuestros resultados muestran que esta barrera natural ha sido vulnerada, pues se ha encontrado un mismo haplotipo a ambos lados de la cordillera. Este hecho, como se explicó líneas arriba, se debería a la migración pasiva del vector mediada por la actividad humana. En consecuencia, no podemos usar, para agrupar los haplotipos encontrados, las denominaciones de variantes costera y de sierra-selva (17, 19, 20).
Si bien el gen mitocondrial es utilizado para el análisis intraespecífico de Aedes aegypti, recientemente se ha reportado en esta especie la presencia de pseudocopias nucleares (NUMT) que pueden alterar la divergencia inferida si son indistintamente analizados como copias mitocondriales (20). En tal sentido, será de gran utilidad utilizar en futuros estudios, marcadores nucleares, además del marcador mitocondrial para discriminar la posibilidad de NUMT
La principal limitación de nuestro estudio radicó en que el tiempo empleado para la colecta de especímenes fue demasiado amplio (2006-2012), con lo cual existe la posibilidad que la variabilidad en las zonas geográficas haya sufrido modificaciones. A pesar de ello, consideramos que nuestro estudio constituye un aporte en el desarrollo de líneas de investigación destinadas a evaluar la interacción o asociación del Aedes aegypti con los serotipos de dengue circulantes, con la resistencia de este vector a insecticidas, y con sus niveles de dispersión. Para así contribuir con el desarrollo de medidas de intervención para la prevención y control del dengue.
En conclusión, se han descrito cinco haplotipos de Aedes aegypti, agrupados en dos linajes, en once zonas endémicas de dengue en el Perú. Esta variabilidad se explicaría tanto por la migración activa del vector como por su migración pasiva, mediada por la actividad humana. De igual manera, debido a la migración pasiva del Aedes aegypti se debe reconsiderar la clasificación tradicional de variantes costera y sierra-selva, ya que se ha encontrado un mismo haplotipo en ambas regiones.
Agradecimientos: al Laboratorio de Entomología del Instituto Nacional de Salud por el apoyo en la identificación morfológica de los especímenes analizados, así como proveernos de especímenes de Madre de Dios y Lima. A los señores: Enrique Purisaca (Amazonas); Dina Torres y Luis Ayma (Cusco); Milady Gatti (Loreto); Lucinda Troyes (Cajamarca); Edwin Tineo (Madre de Dios); Miguel Castro (Piura); Sadith Arévalo (Ucayali); Etty Lopez (San Martin); Bertha Moreno (La Libertad) y Micsuco Astudillo (Tumbes) por el apoyo en el envío de los especímenes analizados en este estudio.
Contribuciones de autoría: PY diseño el estudio; PY y WC recolectaron los especímenes; PY, EM, JV y DT analizaron e interpretaron los datos; PY, EM, DT y CC redactaron; PY, EM y MPG intervinieron para la aprobación de la versión final; JV y PV aportaron con material de estudio; EM y MP obtuvieron el financiamiento; todos contribuyeron para la revisión critica
Fuentes de financiamiento: Instituto Nacional de Salud. Lima, Perú.
Conflictos de interés: los autores declaran no tener conflictos de interés en la publicación del presente estudio.
Referencias Bibliográficas
1. World Health Organization. Dengue. Guías para el diagnóstico, tratamiento, prevención y control . La Paz; OPS/OMS; 2009. [ Links ]
2. Nelson MJ. Aedes aegypti: Biología y ecología. Washington, D.C.: OPS; 1986. [ Links ]
3. Griffitts DHT. Fiebre amarilla: importancia de algunas fases relativamente nuevas en relación con su profilaxia en los focos antiguos y nuevos . Bol Of Sanit Panam. 1934;13(6):511-8. [ Links ]
4. Oficina Sanitaria Panamericana. Informe mensual sobre la campaña de erradicación del Aedes aegypti en los países americanos. Bol Of Sanit Panam. 1949;28:62-6. [ Links ]
5. Severo OP. Evolución de la campaña anti-Aegyti en los últimos diez años. Bol Of Sanit Panam. 1958;45:375-86. [ Links ]
6. Sevilla Andrade C, Cáceres AG, Vaquerizo A, Ibañez-Bernal S, Sulca Cachay L. Reappearance of Aedes aepypti (Díptera: Culicidae) in Lima, Peru . Mem Inst Oswaldo Cruz. 2001;96(5):657-8. [ Links ]
7. Cabezas C, Solari L, Solano E, Suárez V, León-Cueto W, Cobos M, et al. Emergencia de Dengue en Lima durante una campaña de prevención . Bol Inst Nac Salud. 2005;11(5-6):132. [ Links ]
8. Arrasco J. Situación del dengue en el Perú [editorial]. Bol Epidemiol (Lima). 2012;21(3):35-6. [ Links ]
9. Bracco JE, Capurro ML, Lourenço-De-Oliveira R, Mureb MA. Genetic variability of Aedes aegypti in the Americas using a mitochondrial gene: evidence of multiple introductions . Mem Inst Oswaldo Cruz. 2007;102(5):573-80. [ Links ]
10. Lima Júnior RS, Scarpassa VM. Evidence of two lineages of the dengue vector Aedes aegypti in the Brazilian Amazon, based on mitochondrial ADN ND4 gene sequences . Genet Mol Biol. 2009;32(2):414-22. [ Links ]
11. Urdaneta-Marquez L, Bosio C, Herrera F, Rubio-Palis Y, Salasek M, Black WC. Genetic Relationships among Aedes aegypti collections in venezuela as determined by mitochondrial ADN variation and nuclear single nucleotide polymorphisms . Am J Trop Med Hyg. 2008;78(3):479-91. [ Links ]
12. QIAGEN®. QIAGEN Supplementary Protocol: Purification of total DNA from insects using the DNeasy® Blood & Tissue Kit [Internet]. Germantown, MD: QIAGEN®; 2006 [citado el 28 de agosto del 2012]. Disponible en: http://www.qiagen.com/resources/Download.aspx?id=%7BCABD47A4-CB5A-4327-B10D-D90B8542421E%7D&lang=en&ver=1 [ Links ]
13. QIAGEN®. QIAquick® PCR Purification Kit [Internet]. Germantown, MD: QIAGEN®; 2010 [citado el 29 de agosto del 2012]. Disponible en: http://www.qiagen.com/resources/Download.aspx?id=%7B390A728A-E6FC-43F7-BF59-B12091CC4380%7D&lang=en&ver=1 [ Links ]
14. Princeton Separations. Centri-sep columns. For Research use only [Internet].. Adelphia, NJ: Princeton Separations, Inc.; 2011 [citado el 2 de septiembre del 2012]. Disponible en: http://www.prinsep.com/sites/prinsep.rpdesign.com/files/Centri-Sep%20Procedure_0.pdf [ Links ]
15. NCBI [base de datos en Internet]. Bethesda: National Center for Biotechnology [citado el 1 de septiembre del 2012]. Costa MCV, Paduan KS, Ribolla PEM. Temporal analysis of mitochondrial gene (NDH4) in Aedes aegypti populations from endemic and non-endemic areas in Brazil. Acceso: EF153761.1 [aprox. 2 pantallas]. Disponible en: http://www.ncbi.nlm.nih.gov/nucleotide/119656928?report=genbank&log$=nucltop&blast_rank=1&RID=48KUDZE301N [ Links ]
16. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods . Mol Biol Evol. 2011;28(10): 2731-9. [ Links ]
17. Costa-da-Silva, AL; Capurro ML, Bracco JE. Genetic lineages in the yellow fever mosquito Aedes (Stegomyia) aegypti (Diptera: Culicidae) from Peru . Mem Inst Oswaldo Cruz. 2005;100(6):539-44. [ Links ]
18. Cáceres OA, León W. Variantes Genéticas de Aedes aegypti y su asociación con el serotipo del virus dengue en una área endémica del Perú [Internet]. Lima: Instituto Nacional de Salud; 2007 [citado el 06 de agosto del 2012]. Disponible en: http://www.bvs.ins.gob.pe/insprint/cindoc/informes_tecnicos/96.pdf [ Links ]
19. Leiva N, Cáceres O. Variabilidad genética de Aedes aepypti en algunas áreas del Perú usando Single Stranded Conformational Polymorphism (SSCP) . Rev Peru Med Exp Salud Publica. 2004;21(3):157-66. [ Links ]
20. Hlaing T, Tun-Lin W, Somboon P, Socheat D, Setha T, Min S, et al. Mitochondrial pseudogenes in the nuclear genome of Aedes aegypti mosquitoes: implications for past and future population genetic studies . BMC Genet. 2009;10:11. [ Links ]
Correspondencia: Enrique Mamani Zapana
Dirección: Av. Defensores del Morro 2268, Chorrillos. Lima, Perú.
Teléfono: (511) 995 903 830
Correo electrónico: emamani@ins.gob.pe
Recibido: 19-12-12
Aprobado: 17-04-13













