Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.31 no.2 Lima abr. 2014
Artículo Original
Comparación de dos protocolos de extracción de ADN de Trypanosoma cruzi cultivados en medio axénico
Comparing two protocols of DNA extraction of Trypanosoma cruzi cultured in axenic medium
Mariela López1,2,a, María G. Rivera1,a, Mercedes Viettri1,a, María Lares1,b, Antonio Morocoima 3,c, Leidi Herrera4,d, Elizabeth Ferrer1,5,e
1 Instituto de Investigaciones Biomédicas “Dr. Francisco J. Triana Alonso”, Facultad de Ciencias de la Salud, Universidad de Carabobo. Maracay, Venezuela.
2 Escuela de Bioanálisis, Facultad de Ciencias de la Salud, Universidad de Carabobo. Maracay, Venezuela.
3 Centro de Medicina Tropical de Oriente, Facultad de Medicina, Universidad de Oriente. Anzoátegui, Venezuela.
4 Instituto de Zoología y Ecología Tropical, Facultad de Ciencias, Universidad Central de Venezuela. Caracas, Venezuela.
5 Departamento de Parasitología, Facultad de Ciencias de la Salud, Universidad de Carabobo. Maracay, Venezuela.
a Licenciada en Bioanálisis; b técnico superior universitario en Química; c médico cirujano, magíster en Parasitología; d biólogo, doctora en Ciencias; e licenciada en bioanálisis, doctora en Biología Molecular.
RESUMEN
Objetivos. Comparar dos protocolos de extracción de ADN de Trypanosoma cruzi para su uso en la amplificación de ADN de minicírculos de kinetoplasto (ADNk) mediante la técnica de Reacción en Cadena de Polimerasa (PCR). Materiales y métodos. Se cultivaron epimastigotas de T. cruzi en condiciones exénicas obteniéndose masas entre 1,5 hasta 100 x 106 parásitos. A partir de estas se procedió a la extracción de ADN mediante dos protocolos: extracción con solventes orgánicos (fenol/cloroformo), y empleo de resina (Chelex®100), a partir de los diferentes sedimentos parasitarios. La concentración y pureza del ADN se determinó por espectrofotometría y la integridad se evaluó mediante electroforesis en geles de agarosa. Se realizó el análisis de varianza y comparaciones de medias mediante la prueba de Tukey, utilizando el software Statistix 8.0. Resultados. Se realizaron diez extracciones de ADN de cada una de las diferentes cantidades de parásitos sedimentados. En la extracción de ADN con la resina Chelex®100 se obtuvo mayor rendimiento, pero menor pureza e integridad respecto a la extracción con solventes orgánicos. Sin embargo, permitió la amplificación del producto de 330 pb de ADNk de T. cruzi. Conclusiones. Aun cuando la técnica de Chelex®100 proporcionó menor pureza e integridad del ADN, permitió la amplificación con éxito de ADNk por PCR, evitando el uso de técnicas laboriosas y solventes orgánicos tóxicos.
Palabras clave: Trypanosoma cruzi; Enfermedad de Chagas; ADN; Diagnóstico; Reacción en cadena de la polimerasa (fuente: DeCS BIREME).
ABSTRACT
Objectives . To compare two extraction protocols of Trypanosoma cruzi DNA for use in DNA amplification of kinetoplast minicircles (kDNA) through the technique of Polymerase Chain Reaction (PCR). Materials and methods. Epimastigotes of T. cruzi were cultured in axenic conditions and masses from 1.5 to 100 x 106 parasites were obtained. DNA extraction was performed using two protocols: extraction with organic solvents (phenol/chloroform), and with resin (Chelex®100), from different parasitic sediments. Concentration and purity of DNA was determined by spectrophotometry, and integrity was assessed by agarose gel electrophoresis. Analysis of variance and comparisons of means were performed through Tukey’s test, using the Statistix 8.0 software. Results. Ten DNA extractions were done of each one of the different amounts of parasitic sediments. In the DNA extraction with Chelex®100 resin, a higher performance was obtained but a lower purity and integrity compared to the extraction with organic solvents. However, it allowed a product amplification of 330 bp of T. cruzi kDNA. Conclusions. Although the technique of Chelex®100 provided less purity and integrity of DNA, it allowed a successful amplification of kDNA by PCR, avoiding the use of laborious techniques and toxic organic solvents.
Key words: Trypanosoma cruzi; Chagas disease; DNA; Diagnosis; Polymerase chain reaction (source: MeSH NLM).
INTRODUCCIÓN
La técnica de reacción en cadena de la polimerasa (PCR, por sus siglas en inglés, Polymerase Chain Reaction), ha permitido el desarrollo de novedosos y oportunos protocolos diagnósticos, que han sido estandarizados y validados, pues presentan ventajas en cuanto a sensibilidad, especificidad, rapidez y versatilidad (1-5).
En la enfermedad de Chagas o tripanosomiasis americana, la cual es producida por el protozoario T. cruzi, las técnicas diagnósticas por emplear varían en función de la fase en que se encuentre la enfermedad. El diagnóstico en la fase aguda se realiza principalmente por métodos parasitológicos con baja sensibilidad, mientras que en la fase crónica se emplean generalmente métodos inmunológicos que pueden tener baja especificidad (6). La PCR es hoy en día una de las técnicas más utilizadas para la detección de ADN de T. cruzi, siendo útil en el diagnóstico de las fases aguda o crónica temprana ya que detecta cantidades muy pequeñas del ADN del parásito (7-9).
La técnica de PCR tiene gran versatilidad en cuanto a las muestras por emplear, pues se puede analizar todo tipo de fluidos o tejidos biológicos, principalmente sangre, suero (tanto muestras líquidas como secas en papel de filtro) (8-11), biopsias (tejidos frescos, congelados o incluidos en parafina) (12-13), heces de triatominos (muestras líquidas o secas en papel de filtro) (14). Además, se puede aplicar en muestras de organismos muertos, momificados o restos fósiles (15).
El éxito de las técnicas de amplificación del ADN depende de su adecuada extracción, afectando su sensibilidad y reproducibilidad. En este aspecto, son relevantes los reactivos utilizados y los métodos de extracción de ADN (16). El aislamiento de ADN intacto, con un menor número de contaminantes y con buen rendimiento es esencial para la obtención de un resultado adecuado, y es muy importante cuando se usa para diagnóstico en pacientes. Además, cuando se trabaja con elevado número de muestras, es necesario garantizar un protocolo que brinde sencillez, facilidad, rapidez, eficiencia y eficacia en su operatividad (17).
Por estos motivos, el objetivo de este trabajo fue comparar dos protocolos de extracción de ADN, tanto extracción con solventes orgánicos (fenol/cloroformo), como con el uso de resinas, a partir de T. cruzi en cultivo, a fin de establecer cuál de las dos proporciona mejor rendimiento, pureza e integridad del ADN y que permita una mejor amplificación de secuencias de minicírculos de kinetoplasto.
MATERIALES Y MÉTODOS
CULTIVO DE PARÁSITOS
Se cultivaron epimastigotas de T. cruzi, clon Dm28c (18) en medio LIT (Liver-Infusion-Tryptose) suplementado con suero fetal bovino (SFB) al 10% a 28 ºC. Los parásitos fueron sedimentados a 14 000 rpm (microcentrífuga Spectrafuge 16M Labnet) durante 5 min, a temperatura ambiente. Se lavaron con tampón fosfato salino (PBS) y se obtuvieron masas de 1,5, 3, 6, 10, 20, 50 y 100 x 106 epimastigotas, los cuales fueron almacenados a -80 °C hasta su uso.
MÉTODOS DE EXTRACCIÓN DE ADN
Se realizó la extracción de ADN a partir de masas con diferentes cantidades de parásitos (1,5, 3, 6, 10, 20, 50 y 100 x 106 epimastigotas) mediante la técnica de extracción de ADN con solventes orgánicos, fenol/cloroformo, según Sambrook y Russell, 2001 (19) y la técnica de extracción de ADN con resina, Chelex®100, según instrucciones del fabricante.
TÉCNICA DE EXTRACCIÓN DE ADN CON SOLVENTES ORGÁNICOS (FENOL/CLOROFORMO)
Las diferentes masas de parásitos recolectadas a partir de los cultivos de T. cruzi fueron lisados en 500 µL de tampón de lisis (NaCl 0,02 M, EDTA 0,5 M pH 8, Tris-HCL 1 M, pH 7,4) y agitación en vortex. A esta suspensión se le añadió 25 µL de dodecil sulfato de sodio (SDS) (10%) y 10 µL de ARNasa (Promega, Madison, EE. UU) (10 mg/mL) y se incubó a 37 °C durante 30 min. Luego, se agregó 5 µL de proteinasa K (Promega, Madison, EE. UU.) (20 mg/mL) y se incubó durante 3 h a 37 °C. Posteriormente, se añadió igual volumen de fenol/cloroformo (1:1), se centrifugó a 14 000 rpm durante 10 min a temperatura ambiente. La fase acuosa obtenida se transfirió a un nuevo tubo, en el cual se precipitó el ADN añadiendo 2,5 volúmenes de etanol a -20 ºC, y 0,1 volumen de acetato de sodio 3 M, mezclando suavemente para luego dejar la muestra toda la noche a -20 °C. Posteriormente, se centrifugó a 14 000 rpm durante 30 min a temperatura ambiente, y luego se descartó el sobrenadante; el precipitado fue lavado añadiendo 1 mL de etanol al 70% frío, sedimentado y se descartó el etanol. Se dejó secar el sedimento que contiene el ADN y se resuspendió en 50 µL de agua destilada. Las muestras se mantuvieron a -20 º C hasta su uso.
TÉCNICA DE EXTRACCIÓN DE ADN CON RESINA, CHELEX ® 100 (BIORAD)
Las diferentes masas de parásitos fueron resuspendidas en 1 mL de agua destilada e incubados 30 min a temperatura ambiente. Posteriormente, se centrifugó durante 3 min a 14 000 rpm, y se retiró el sobrenadante, dejándose un volumen de 50 μL, a los cuales se les agregó 200 mL de la resina Chelex®100 al 5%, y se incubó durante 30 min a 56 ºC. La muestra se agitó por 10 s, y se hirvió durante 10 min para luego mezclarse durante 10 s nuevamente. La resina y las proteínas desnaturalizadas fueron separadas mediante centrifugación por 3 min a 14 000 rpm, y se recolectó el sobrenadante, el cual se mantuvo a -20 °C hasta su uso.
DETERMINACIÓN DE LA CONCENTRACIÓN Y PUREZA DEL ADN
Se siguieron los métodos descritos por Sambrook y Russell (19). La concentración de ADN fue estimada en un espectrofotómetro UV/Visible Gene Quantro, se midió la absorbancia a 260 nm (A260) y 280 nm (A280) utilizando la fórmula siguiente: [ADN]= A260 nm x D x 50 μg/mL D= factor de dilución. El grado de pureza se calculó dividiendo la absorbancia a 260 nm entre la absorbancia a 280 nm.
TÉCNICA DE PCR PARA LA AMPLIFICACIÓN DE ADN DE MINICÍRCULO DE KINETOPLASTO DE T. cruzi
Se utilizó el protocolo de Wincker et al. (20), empleando los cebadores para la secuencia de minicírculos del kinetplasto 121 5`-AAA TAA TGT ACG GGT GAG ATG CAT GA-3`, y 122 5`-GGG TTC GAT TGG GGT TGG TGT-3`. Dicho protocolo fue modificado para adaptarlo a las condiciones del laboratorio (21). Las condiciones de reacción fueron: desoxinucleótidos tri-fosfato (dNTP), 200 μM, cebadores 0,5 μM, cloruro de magnesio 2 μM y ADN Polimerasa (GoTaq Promega) 1U. Como ADN molde se utilizó 10 picogramos de cada extracción. En los controles negativos se utilizó la mezcla de reacción añadiendo agua destilada. La PCR se llevó a cabo mediante el siguiente programa: Desnaturalización inicial a 94 ºC durante 10 min, seguido de 30 ciclos de desnaturalización a 94 ºC, por 30 s, hibridación a 63 ºC por 30 s y extensión a 72 ºC por 30 s, terminando con una extensión final a 72 ºC, durante 10 min.
ELECTROFORESIS DE ADN EN GELES DE AGAROSA
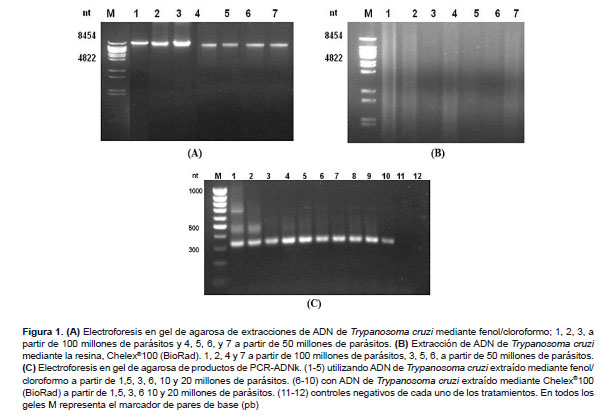
La calidad e integridad del ADN extraído, se analizó por electroforesis en gel de agarosa (Prona), al 1 %, utilizando el tampón TAE (tampón tris-ácido acético 40 mM, EDTA 0,5 M pH 8,0). En los geles se cargó 2 µL de cada extracción por fenol/cloroformo y 8 µL por Chelex®100 y, en el caso de los geles de las PCR se cargó los 25 µL de los productos. La separación del ADN se realizó en una cámara de electroforesis (Minicell® EC 370M), empleando un voltaje de 60-100 V. Los geles fueron teñidos con bromuro de etidio (0,5 μg/mL). Las bandas de ADN se visualizaron mediante el sistema Gel Doc 1000 (BioRad). El tamaño de las bandas de ADN se estimó comparándolas con el marcador de tamaño molecular λ-BstEII (New England Biolabs) y 100 pb (Promega) para los productos de PCR.
ANÁLISIS ESTADÍSTICO
Se comparó el rendimiento y la pureza del ADN mediante un arreglo factorial de tratamientos 2 x 7 con desigual número de repeticiones; donde el primer factor fueron los métodos de extracción a evaluar y el segundo factor correspondió a la cantidad de parásitos desde 1,5 hasta 100 x 106 parásitos. A los resultados se les aplicó el análisis de varianza y se realizaron las comparaciones de medias correspondientes mediante la prueba de Tukey, utilizando para ello el software Statistix 8.0 para Windows.
RESULTADOS
Se realizaron diez extracciones de ADN de cada una de las diferentes cantidades de parásitos sedimentados (1,5, 3, 6, 10, 20, 50 y 106 millones de parásitos) para un total de setenta extracciones para cada una de las técnicas empleadas. Los valores de rendimiento y pureza se promediaron y se muestran en la Tabla 1.
Con respecto al rendimiento, el protocolo con la resina Chelex®100 permitió extraer mayor cantidad de ADN que el protocolo con solventes orgánicos, mostrando una diferencia significativa (p<0,05) en todos los casos, además que su rendimiento aumenta proporcionalmente a medida que aumenta la cantidad de parásitos utilizados en la extracción (Tabla 1).
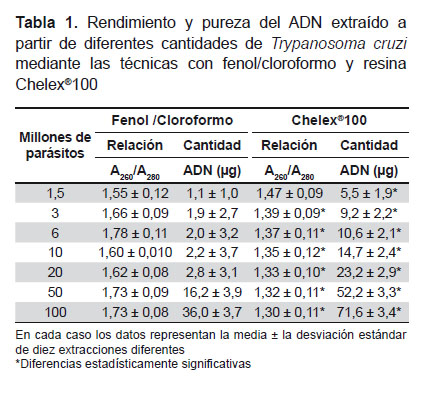
La determinación del índice de pureza del ADN extraído por los protocolos descritos, fue evaluada a partir de los promedios de las relaciones A260 nm/A280 nm. Los resultados muestran que para el protocolo con fenol/cloroformo las relaciones de absorbancia estuvieron, en varias de las cantidades ensayadas dentro de los rangos adecuados (1,7-1,9), a diferencia del protocolo con Chelex®100 que todos los promedios de las relaciones obtenidas estaban por debajo de los valores esperados para obtener un ADN puro (Tabla 1). Resaltando que a medida que se realizaban las extracciones con una mayor cantidad de parásitos, la pureza tendía a disminuir, evidenciándose una diferencia significativa (p<0,05) entre el ADN extraído en ambos protocolos en casi todos los casos (Tabla 1).
En cuanto a integridad, el protocolo fenol/cloroformo, permitió obtener un ADN con mayor integridad, notándose una banda única de gran tamaño a diferencia del protocolo Chelex®100, donde se observó en el gel de agarosa un extendido a lo largo de todo el carril, evidenciando degradación (Figura 1A y 1B).
Se empleó la técnica de PCR-ADNk previamente estandarizada y validada (21,22) con las muestras de ADN extraídas con ambas técnicas descritas. Como puede observarse, en la Figura 1C en todos los casos se obtuvo la amplificación del fragmento esperado de 330 pb, con pocas diferencias entre las distintas muestras. Es importante resaltar que pueden observarse otras bandas de otros tamaños (Figura 1C) ya que cada minicírculo contiene cuatro repeticiones continuas de la secuencia diana, por lo que podrían observarse bandas con el doble o triple del tamaño (660 o 990 pb) o, incluso bandas de tamaños intermedios entre estas, siendo considerado como diagnóstico la aparición de la banda de 330 pb.
DISCUSIÓN
La elección de un buen protocolo para la extracción de ADN es un punto crítico para la adecuada amplificación en la técnica de PCR. La mayoría de las técnicas están basadas en extracción con solventes orgánicos, que presentan inconvenientes en cuanto a tiempo, laboriosidad, costos y toxicidad. Los ensayos de extracción deben ser sencillos, rápidos, reproducibles, económicos, seguros y que permitan el manejo de gran cantidad de muestras (17, 21,22).
Por tal motivo, se estandarizaron y evaluaron dos protocolos de extracción a partir de parásitos de cultivo, uno basado en el clásico procedimiento de extracción con solventes orgánicos utilizando fenol/cloroformo y otro, utilizando una resina quelante de iones metálicos polivantes, Chelex® 100, con el propósito de comparar un protocolo clásico muy elaborado, con un protocolo sencillo, rápido y económico.
En la extracción de ADN con la resina Chelex®100 (BioRad) se obtuvo mayor rendimiento que en la extracción con solventes orgánicos, permitiendo extraer tres o hasta cuatro veces más ADN, dependiendo de la cantidad de parásitos utilizados. No excluyendo la posibilidad que estos valores estén sobrestimados por contaminación con ARN que pudiese estar en la muestra.
El bajo rendimiento mostrado por el protocolo clásico con solventes orgánicos podría atribuirse a la posible oxidación del fenol utilizado (aun siendo nuevo y de buena calidad). Además, el protocolo fenol/cloroformo incluye gran cantidad de pasos que lo hacen largo y laborioso, por lo que puede ocurrir pérdida del ADN en los diferentes pasos de la técnica, lo que hace la técnica sea poco reproducible, además que se utilizan solventes contaminantes y tóxicos (22-24).
Por otro lado, el alto rendimiento de la técnica con la resina Chelex®100, con respecto a la técnica de fenol/cloroformo, fue más notorio en cantidades pequeñas de parásitos, ya que empleando 1,5; 3; 6; 10 y 20 millones de parásitos, se obtuvo por Chelex®100 aproximadamente entre 5 y 10 veces a lo obtenido por fenol/cloroformo, mientras que al emplear cantidades mayores de parásitos (50-100 millones), el rendimiento por Chelex ®100 fue aproximadamente entre 2 y 3 veces el obtenido con la técnica de fenol/cloroformo. Este aspecto es de suma importancia ya que en las muestras de pacientes, sobre todo en la fase crónica de la enfermedad de Chagas, la cantidad de ADN del parásito en la muestra de sangre es muy escasa.
Cuando se evaluó la pureza del ADN extraído, se logró obtener mejores resultados con el protocolo fenol/cloroformo, que con el protocolo Chelex ®100, y esto puede ser debido a que el protocolo clásico se empleó tratamiento ARNasas y proteinasa K que ayudan a obtener muestras de ADN más puras (libres de proteínas y ARN), pero dichos tratamientos producen un aumento significativo s en el tiempo y costo del protocolo. Además, estos procesos tan largos y laboriosos son difíciles de realizar a gran escala en la práctica clínica, para el manejo de gran cantidad de muestras (22-24).
En el caso de la integridad del ADN, se observó como resultado de la extracción con solventes orgánicos una banda única de ADN, mientras que el ADN obtenido con el protocolo Chelex®100 (BioRad), se podría observar signos de degradación, ya que se observa un “extendido” o “barrido”.
Si bien es cierto que, con la técnica con Chelex®100 (BioRad) se obtuvo, una menor pureza e integridad, sin embargo, fue eficiente para lograr obtener un ADN que permitió producir amplificación de producto de 330 pb de la secuencia del ADN de minicírculos de kinetoplasto de T. cruzi utilizada como técnica molecular para el diagnóstico (20,25,26). Es importante mencionar que a pesar de que el ADN haya quedado aparentemente fragmentado, al ser la diana a amplificar un fragmento de tamaño pequeño y altamente repetitivo, se aumenta la probabilidad de que se produzca la amplificación por PCR.
Algunos estudios especifican que es posible amplificar fragmentos de hasta 650 pb en muestras tratadas solo con la resina Chelex®100, y fragmentos de hasta 1000 pb en las que se ha utilizado la resina y digestión con proteinasa K, proporcionando de esta manera una adecuada cantidad y calidad de ADN para amplificar (24). Además, otros estudios indican algunos límites parecidos, considerando el tamaño de los productos que pueden ser amplificados, en virtud que a medida que aumenta el tamaño de la diana a amplificar, debe mejorarse tanto la integridad como la pureza del ADN obtenido para así garantizar que exista una adecuada y óptima amplificación (27).
La resina Chelex®100 también ha sido utilizada para extracción de ADN de otros parásitos, tales como; Acantha moeba sp (28) y Trypanosoma evansi (29) y su empleo posterior en PCR ha evidenciado buena amplificación. Por otro lado, en un estudio para el diagnóstico de tripanosomiasis africana, la PCR mejoró su sensibilidad cuando se hizo extracción de ADN con la resina Chelex®100, en comparación al uso de la muestra de sangre directamente (30).
En conclusión, se pudo evidenciar que la técnica de extracción de ADN del parásito T. cruzi, mediante el uso de la resina Chelex® 100 (BioRad), es una técnica que mostró ventajas en cuanto a sencillez, rapidez, reproducibilidad y seguridad para el operador, ya que no requiere la exposición a solventes orgánicos peligrosos al personal que realiza estos procedimientos. Además, dicha técnica permite el mejor manejo de un gran número de muestras y es más económica, por lo que podría sugerirse su uso como técnica de extracción de rutina en laboratorios que estén orientados al diagnóstico de la enfermedad de Chagas.
Fuentes de financiamiento: ayuda menor CDCH-UC 0440-10, Universidad de Carabobo y Proyecto N.º 2008000911-6. Misión Ciencia, Ministerio de Ciencia y Tecnología FONACIT.
Conflictos de interés: los autores declaran no tener conflictos de interés
Referencias Bibliográficas
1. Barken KB, Haagensen JA, Tolker-Nielsen T. Advances in nucleic acid base diagnostics of bacterial infections . Clin Chim Acta. 2007 Sep;384(1-2):1-11. [ Links ]
2. Hernández-Hernández FC, Rodríguez MH. Avances biotecnológicos en el diagnóstico de enfermedades infecciosas . Salud Pública Méx. 2009;51 Supl 3:424-38. [ Links ]
3. Tang YW, Procop GW, Persing DH. Molecular diagnosis of infectious diseases . Clin Chem. 1997 Nov;43(11):2021-38. [ Links ]
4. Millar BC, Xu J, Moore JE. Molecular diagnostics of medically important bacterial infections . Curr Issues Mol Biol. 2007 Jan;9(1):21-39. [ Links ]
5. Ieven M. Currently used nucleic acid amplification tests for the detection of viruses and atypicals in acute respiratory infections . J Clin Virol. 2007 Dec;40(4):259-76. [ Links ]
6. Atias A. Enfermedad de Chagas. En: Parasitología Médica. 2da edición. Santiago: Publicaciones técnicas Mediterráneo Ltda; 1998. p. 251-64. [ Links ]
7. Andersson, J. Molecular diagnosis of experimental Chagas diseases . Trends Parasitol. 2004 Feb;20(2):52-3. [ Links ]
8. Avila HA, Pereira JB, Thiemann O, De Paiva E, DeGrave W, Morel CM, et al. Detection of Trypanosoma cruzi in blood specimens of chronic chagasic patients by polimerase chain reaction amplification of kinetoplast minicircle DNA: comparison with serology and xenodiagnosis . J Clin Microbiol. 1993 Sep;31(9):2421-6. [ Links ]
9. Britto C, Cardoso MA, Vanni CM , Hasslocher-Moreno A , Xavier SS , et al. Polymerase chain reaction detection of Trypanosoma cruzi in human blood samples as a tool for diagnosis and treatment evaluation . Parasitology. 1995 Apr;110 (Pte 3):241-7. [ Links ]
10. Russomando G, Figueredo A, Almiron M, Sakamoto M, Morita K. Polymerase chain reaction-based detection of Trypanosoma cruzi DNA in serum . J Clin Microbiol. 1992 Nov;30(11):2864-8. [ Links ]
11. Kirchhoff LV, Votava JR, Ochs DE, Moser DR. Comparison of PCR and microscopic methods for detecting Trypanosoma cruzi. J Clin Microbiol. 1996 May;34(5):1171-5. [ Links ]
12. Cao W, Hashibe M, Rao JY, Morgenstern H, Zhang ZF. Comparison of methods for DNA extraction from paraffin-embedded tissues and buccal cells . Cancer Detect Prev. 2003;27(5):397-404. [ Links ]
13. Chan PK, Chan DP, To KF, Yu MY, Cheung JL, Cheng AF. Evaluation of extraction methods from paraffin wax embedded tissues for PCR amplification of Human and viral DNA . J Clin Pathol. 2001 May;54(5):401-3. [ Links ]
14. Moser DR, Kirchhoff LV, Donelson JE. Detection of Trypanosoma cruzi by DNA amplification using the polymerase chain reaction . J Clin Microbiol. 1989 Jul;27(7):1477-82. [ Links ]
15. Guhl F, Jaramillo C, Carranza JC, Vallejo GA. Molecular characterization and diagnosis of Trypanosoma cruzi and Trypanosoma rangeli . Arch Med Res. 2002 Jul-Aug;33(4):362-70. [ Links ]
16. De Armas R, Rodríguez MM, Bisset JA, Fraga J. Modificación de un método de extracción de ADN genómico de Aedes aegypti (Diptera: Culicidae) . Rev Colomb Entomol. 2005 Jul;31:203-6. [ Links ]
17. De Armas Y, Capo V, López L, Mederos L, Díaz R. Comparison of three methods for DNA extraction from paraffin-embedded tissues . Biotecnol Apl. 2011 Ene-Mar;28(1):40-3. [ Links ]
18. Contreras VT, Araujo-Jorge TC, Bonaldo MC, Thomaz N, Barbosa HS, Meirelles Mde N, et al. Biological aspects of the Dm 28c clone of Trypanosoma cruzi after metacyclogenesis in chemically defined media . Mem Inst Oswaldo Cruz. 1988 Jan-Mar;83(1):123-33. [ Links ]
19. Sambrook J, y Russell D. Molecular Cloning. A Laboratory Manual . 3ra Ed. New York:Cold Spring Harbor Laboratory Press; 2001. [ Links ]
20. Wincker P, Britto C, Pereira JB, Cardoso MA, Oelemann W, Morel CM. Use of a simplified polymerase chain reaction procedure to detect Trypanosoma cruzi in blood samples from chronic chagasic patients in a rural endemic area . Am J Trop Med Hyg. 1994 Dec;51(6):771-7. [ Links ]
21. Ferrer E, Da Conceicão F, Campioli P, Rivera MG, López M, Lares M, et al. Estandarización de técnicas moleculares para la identificación y caracterización de Trypanosoma cruzi . En: Memorias del VI Congreso de Investigación “La Investigación en el Siglo XXI: Oportunidades y Retos”. Carabobo: Consejo de Desarrollo Científico y Humanístico; 2008. p. 363-7. [ Links ]
22. Ferrer E, Da Conceicão F, Campioli P, Lares M, López M, Rivera MG, et al. Validación de protocolos de PCR para el diagnóstico molecular de la Enfermedad de Chagas. Salus online [Internet]. 2009 [citado el 20 de enero del 2014];12(S1):163-74. Disponible en: http://salus-online.fcs.uc.edu.ve/pcr_diag_mol_chagas.pdf
23. Lopera-Barrero N, Povh JA, Ribeiro RP, Gomes PC, Jacometo CB, Da Silva T. Comparación de dos protocolos de extracción de ADN con muestras de aletas y larvas de peces extracción modificada en cloruro de sodio . Cien Inv Agr. 2008;35(1):77-86. [ Links ]
24. García González LA , Rodrigo Tapia JP , Sánchez Lazo P , Ramos S , Suárez Nieto C . Extracción de ADN con resina Chelex en el análisis de la amplificación oncogénica en carcinomas de cabeza y cuello . Acta Otorrinolaringol Esp. 2004 Mar;55(3):139-44. [ Links ]
25. Telleria J, Lafay B, Virreira M, Barnabé C, Tibayrenc M, Svoboda M. Trypanosoma cruzi : sequence analysis of the variable region of kinetoplast minicircles . Exp Parasitol. 2006 Dec;114(4):279- 88. [ Links ]
26. Mejía AM, Triana O. Análisis por LSSP-PCR de la variabilidad genética de Trypanosoma cruzi en sangre y órganos de ratones . Biomédica. 2005;25(1):76-86. [ Links ]
27. Sepp R, Szabó I, Uda H, Sakamoto H. Rapid Technique for DNA extraction from routinely processed archival tissue for use in PCR . J Clin Pathol. 1994 Apr;47(4):318-23. [ Links ]
28. Iovieno A, Miller D, Lonnen J, Kilvington S, Alfonso EC. Extraction of Acanthamoeba DNA by use of Chelex resin . J Clin Microbiol. 2011 Jan;49(1):476-7. doi: 10.1128/JCM.01795-10. [ Links ]
29. Pruvot M , Kamyingkird K , Desquesnes M , Sarataphan N , Jittapalapong S . The effect of the DNA preparation method on the sensitivity of PCR for the detection of Trypanosoma evansi in rodents and implications for epidemiological surveillance efforts . Vet Parasitol. 2013 Jan 31;191(3-4):203-8. doi: 10.1016/j.vetpar.2012.09.010. [ Links ]
30. Ahmed HA, MacLeod ET, Hide G, Welburn SC, Picozzi K. The best practice for preparation of samples from FTA®cards for diagnosis of blood borne infections using African trypanosomes as a model system . Parasit Vectors. 2011 May 7;4:68. doi: 10.1186/1756-3305-4-68. [ Links ]
Correspondencia: Elizabeth Ferrer
Dirección: Instituto de Investigaciones Biomédi cas “Dr. Francisco J. Triana Alonso” (BIOMED), calle Cecilio Acosta, Urb. La Rinconada, Las De licias, Maracay, estado Aragua, Venezuela.
Teléfono: +58-243-2425822
Correo electrónico: elizabeth.ferrer@gmail.com
Recibido: 14-04-14
Aprobado: 28-05-14