Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.32 no.4 Lima Oct. 2015
Original Breve
Variantes alélicas de CYP2D6: *4, *6 y *10 en una muestra de residentes del estado Aragua, Venezuela
Allelic variants of the CYP2D6: *4, *6 and *10 in a sample of resident from the Aragua state, Venezuela
Carlos Flores-Angulo1,a, Cecilia Villegas1,b, Yuselin Mora1,b; José Antonio Martínez1,c, Teresa Oropeza1,d, Nancy Moreno1,e
1 Instituto de Investigaciones Biomédicas Dr. Francisco J. Triana Alonso (BIOMED). Facultad de Ciencias de la Salud Universidad de Carabobo Sede Aragua. Maracay, estado Aragua, Venezuela
a Médico cirujano; b licenciada en Bioanálisis; c magíster scientiarum; d especialista en Salud Pública; e doctora en Biología Molecular
RESUMEN
El objetivo del estudio fue determinar la frecuencia de las variantes del gen CYP2D6: *4, *6 y *10 y predecir el fenotipo metabolizador en una muestra de 145 individuos no consanguíneos, aparentemente sanos, residentes del estado Aragua, Venezuela. Los genotipos fueron determinados mediante ensayos de reacción en cadena de la polimerasa seguidos de digestión con endonucleasas de restricción. La predicción del fenotipo metabolizador se realizó con base al sistema Activity score. Las frecuencias de CYP2D6 *4, *6 y *10 fueron de 14,5%, 0,3% y 1%, respectivamente; un porcentaje significativo de individuos fueron categorizados como metabolizador rápido heterocigoto/metabolizador intermedio (23,5%) y metabolizador lento (4,1%). Esta información tiene impacto clínico potencial, porque CYP2D6 interviene en el metabolismo de fármacos de prescripción frecuente como: carvedilol, captopril, cloroquina, codeína, fluoxetina, fluvastatina, haloperidol, idarrubicina, indinavir, imatinib, loperamida, nifedipina, ondansetrón y tamoxifeno.
Palabras clave: CYP2D6; Genotipo; Fenotipo; Farmacogenética, Venezuela (fuente: DECS/Bireme)
ABSTRACT
The aim of this study was to determine the CYP2D6: * 4, * 6 and * 10 gene variants frequency and to predict the metabolizer phenotype in a sample of 145 unrelated apparently healthy individuals residing in the state of Aragua, Venezuela. Genotypes were determined by Polymerase chain reaction assays followed by restriction endonucleases digestion. The metabolizer phenotype prediction was made based on the activity score system. The frequencies of CYP2D6 * 4, * 6 and * 10 allelic variants were 14.5%, 0.3% and 1%. A significant percentage of individuals were categorized as heterozygote-extensive/intermediate (23.5%) and poor metabolizers (4.1%), this information has potential clinical impact, because the CYP2D6 protein is involved in the metabolism of drugs frequently prescribed as: carvedilol, captopril, chloroquine, codeine, fluoxetine, fluvastatin, haloperidol, idarubicin, indinavir, imatinib, loperamide, nifedipine, ondansetron and tamoxifen.
Key words: CYP2D6; Genotype; Phenotype; Pharmacogenetics, Venezuela (source: MeSH/NLM)
INTRODUCCIÓN
La terapia farmacológica es uno de los pilares más importantes del tratamiento en la medicina moderna; sin embargo, la respuesta terapéutica evidenciada no siempre es homogénea, existiendo un amplio espectro de respuestas. Esta variabilidad interindividual se debe a factores diversos, incluyendo a los polimorfismos en genes que codifican enzimas metabolizadoras de fármacos, como la superfamilia del citocromo P450 (CYP) (1). Dentro de este grupo se encuentra la proteína CYP, familia 2, subfamilia D e isoforma 6 (CYP2D6), la cual es expresada principalmente en el hígado y es responsable del metabolismo del 25% de los fármacos (1).
En el gen CYP2D6 se han descrito más de 100 alelos, los cuales son clasificados en cuatro categorías de acuerdo al impacto sobre la actividad enzimática: los que generan una proteína sin función, con actividad disminuida, normal o incrementada (http://www.cypalleles.ki.se). Las variantes CYP2D6*4 y *6 generan una proteína truncada sin actividad y CYP2D6*10 una enzima con actividad disminuida (2). La identificación de los genotipos de CYP2D6 permite predecir la capacidad de hidroxilación y, por ende, el fenotipo metabolizador (3-5).
Debido a la existencia de diversos tipos de metabolizadores, la Food and Drug Administration (FDA) aprobó describir en el prospecto de 137 fármacos que individuos con determinadas características fenotípicas, bien sea como consecuencia de la presencia de variantes genéticas o por interacción medicamentosa, tienen el riesgo de experimentar efectos adversos y responder o no a un tratamiento específico (http://www.fda.gov).
Diversos estudios poblacionales han demostrado que la frecuencia de los alelos del gen CYP2D6 varía considerablemente de una población a otra (6); en Venezuela se han reportado datos sobre CYP2D6 en muestras poblacionales de la región centro-occidental y de varias poblaciones amerindias nativas (7). En virtud que el grado de mestizaje varía de acuerdo con las diferentes zonas geográficas venezolanas, es necesario generar conocimiento sobre la frecuencia de variantes de dicho gen en otras regiones del país, para incrementar la información básica de importancia en el diseño de estudios de fármacogenética. En este trabajo se determinaron las frecuencias de variantes de CYP2D6: *4, *6 y *10 con el fin de predecir el fenotipo metabolizador, en una muestra de individuos residentes del estado Aragua, Venezuela.
EL ESTUDIO
La muestra estuvo constituida por 145 individuos sin relación de parentesco, residentes en diversas localidades del estado Aragua, compuesta por 72,4% (n=105) de mujeres y 27,6% (n=40) de hombres, todos mayores de edad, con una media de 32,5 ± 10,6 años y aparentemente sanos. El estado Aragua está ubicado en la región central del país (9º20 a 10º29N y 66º40 a 67º45O). Todos los participantes dieron el consentimiento informado, el cual fue aprobado por el Comité de Bioética del BIOMED. Se tomó una muestra de 1 mL de sangre periférica a partir de la cual se aisló el ADN genómico mediante una modificación del método de precipitación salina (8).
En función de los polimorfismos de un solo nucleótido (SNP) y de acuerdo con lo descrito en la CYP Allele Nomenclature Database (http://www.cypalleles.ki.se), se indagó la presencia de los alelos CYP2D6*4 (1846G>A y 100C>T), CYP2D6*4M (1846G>A), CYP2D6*6 (1707delT) y CYP2D6*10 (100C>T). Estas variantes fueron seleccionadas en virtud de haber sido investigadas en muestras de población de la región centro-occidental y de grupos amerindios venezolanos (7).
Las secuencias que contienen los SNP de interés fueron amplificadas mediante ensayos reacción en cadena de la polimerasa empleando los oligonucleótidos cebadores siguientes: a) Polimorfismo 1846G>A (rs3892097): 5´- GCT TCG CCA ACC ACT CCG - 3´ (directo) y 5´- AAA TCC TGC TCT TCC GAG GC - 3´(reverso) (9); b) Polimorfismo 1707delT (rs5030655): 5´- CCT GGG CAA GAA GTC GCT GGA CCA G - 3´ (directo) y 5´- GAG ACT CCT CGG TCT CTC G - 3´(reverso) (10); c) polimorfismo 100C>T (rs1065852): 5´- AAC GCT GGG CTG CAC GGT AC - 3´ (directo) y 5´- TGA TGG TCC ATG TCG GTG AGC A - 3´ (reverso) (EGT, San Diego, CA, USA) estos dos últimos fueron diseñados empleando el programa Primer3 Versión 0.4.0 (Cambridge, MA, USA).
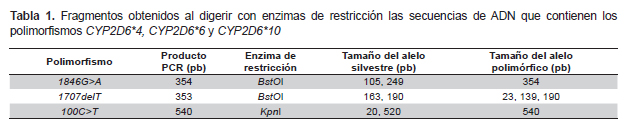
Los SNP fueron determinados empleando la técnica del polimorfismo de la longitud de los fragmentos de restricción (RFLP) (Tabla 1) y las condiciones de incubación para la digestión fueron las recomendadas por el fabricante de las enzimas de restricción (Promega®, Madison, WI, USA). Los genotipos para cada polimorfismo fueron determinados en función del perfil de los fragmentos de restricción, evidenciados mediante electroforesis en gel de poliacrilamida. Los sujetos con ausencia de los SNP investigados fueron catalogados como CYP2D6*1.
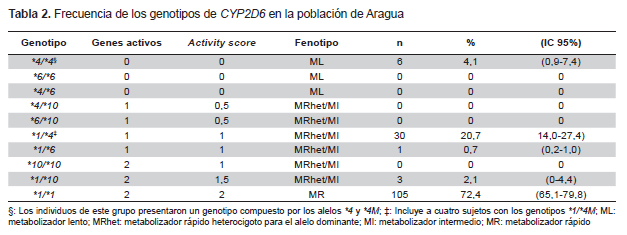
El fenotipo metabolizador fue inferido de acuerdo con los genotipos empleando el modelo de Activity Score (AS) (3,4). Se asignó el valor de 0 a las variantes CYP2D6*4 y *6, un valor de 0,5 a CYP2D6*10 y de 1 a CYP2D6*1 (3,4). Los individuos con un AS de 0, quienes presentaron genotipos compuestos por dos alelos inactivos (*4 y *6) fueron catalogados como metabolizadores lentos (ML), aquellos con un AS de 0,5 a 1 o con los genotipos *4/*10, *6/*10 y*10/*10 fueron considerados como metabolizadores intermedios (MI), sujetos con un AS de 1 a 1,5 heterocigotos para el alelo dominante (*1): *1/*4, *1/*6 y *1/*10, fueron denominados metabolizadores rápidos heterocigotos para el alelo dominante (MRHet) y los individuos con un AS de 2 u homocigotos para el alelo *1 fueron considerados como metabolizadores rápidos (MR) (5). Los fenotipos MI y MRHet fueron incluidos en un mismo grupo en función de lo propuesto por Saladores et al. (5).
La frecuencia alélica encontrada fue comparada con las de otras poblaciones mediante la prueba de chi cuadrado con la corrección de Yate, se consideró significativo un valor de p<0,05. Se calculó el intervalo de confianza con una seguridad del 95% (IC 95%) con el programa SPSS Versión 19 (IBM®, Armonk, NY, USA). Para cada polimorfismo se investigó el equilibrio de Hardy-Weinberg mediante el programa POPGENE Versión 1.31 (Edmonton, Canadá).
HALLAZGOS
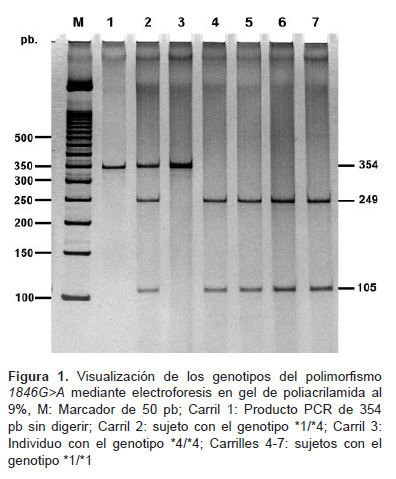
De los tres polimorfismos analizados en este trabajo, solo en el caso de 1846G>A se observaron los tres genotipos esperados (Figura 1). El SNP 1846G>A en estado homocigoto y heterocigoto se encontró con una frecuencia de 4,1% (IC 95%: 0,9-7,4) y 20,7% (IC 95%: 14,0-27,4), respectivamente. Los polimorfismos 1707delT y 100C>T fueron encontrados solo en forma heterocigota con una frecuencia de 0,7% (IC 95%: 0-2,1) y 24,1% (IC 95%: 17,-31,2) para cada uno. Los SNP estudiados se encontraron en equilibrio de HardyWeinberg (p>0,05).
En la Tabla 2 se muestra la frecuencia de los genotipos de las variantes de CYP2D6 y la predicción del fenotipo metabolizador. Los genotipos identificados como *1/*1 corresponden a la forma silvestre para cada uno de los polimorfismos analizados en este trabajo, sin embargo, en este grupo de individuos también están incluidos metabolizadores lentos, intermedios y ultrarrápidos, los cuales se evidenciarán cuando se analicen otros polimorfismos de CYP2D6.
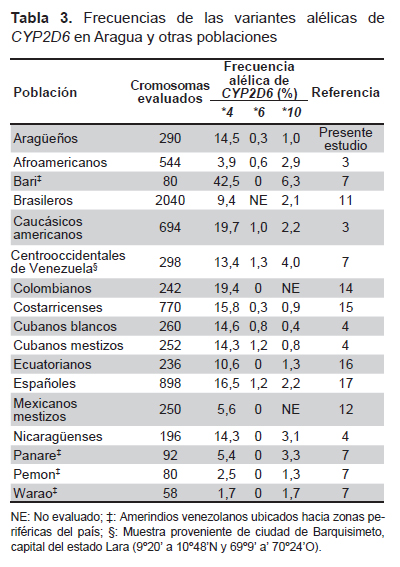
En la Tabla 3 se muestra la comparación de la frecuencia de las variantes alélicas CYP2D6*4, *6 y *10 obtenidas en este estudio, con las reportadas en poblaciones venezolanas y de otros países. Del total de cromosomas analizados (n=290) se determinó que el alelo más frecuente fue el CYP2D6*1 (84,2%; IC 95%: 79,9-88,4). La frecuencia del alelo *4 fue de 14,5% (IC 95%: 10,4-18,6), encontrándose dentro de este grupo al subtipo *4M con una frecuencia de 3,5% (IC 95%: 1,3- 5,6). Los alelos *6 y *10 presentaron una distribución de frecuencia de 0,3% (IC 95%: 0-1,0) y 1% (IC 95%: 0-2,2), respectivamente.
DISCUSIÓN
La distribución de los alelos de CYP2D6 muestra una gran variabilidad poblacional, en esta investigación se pone de manifiesto que la muestra en estudio exhibe diferencias significativas (p<0,05) en relación a las frecuencias del alelo *4 con muestras de población de brasileros (9,4%) (11), mexicanos mestizos (5,6%) (12) y amerindios venezolanos: panare (5,4%) (7), pemon (2,5%) (7), warao (1,7%) (7) y barí (42,5%) (7) (Tabla 3). La diferencia en la distribución de los alelos de CYP2D6 puede ser explicada por las particularidades evolutivas de las poblaciones, lo cual se pone de manifiesto en la actualidad por la variabilidad en el metabolismo y efecto biológico de múltiples xenobióticos (13).
Por otra parte, la frecuencia del alelo *4 encontrada en la muestra de población de Aragua (14,5%) es estadísticamente similar (p>0,05) a lo reportado en individuos de la región centro-occidental de Venezuela (13,4%) (7) y en poblaciones de caucásicos americanos (19,7%) (3), colombianos (19,4%) (14), costarricenses (15,8%) (15), cubanos blancos (14,6%) (4), cubanos mestizos (14,3%) (4), ecuatorianos (10,6%) (16), españoles (16,5%) (17), y nicaragüenses (14,3%) (4) (Tabla 3).
En cuanto al alelo *6, no se evidenciaron diferencias significativas (p>0,05) al compararlo con otras poblaciones (3,4,7,15,17) (Tabla 3); de igual forma ocurre con CYP2D6*10 cuya frecuencia (1%) es similar (p>0,05) a lo descrito previamente en caucásicos americanos, españoles y en poblaciones latinoamericanas (3,4,11,15-17) (Tabla 3): no obstante, sí existen diferencias significativas (p<0,05) con dos muestras de poblaciones venezolanas: la de centrooccidentales (4%) y amerindios bari (6,3%) (7). Las diferencias estadísticas observadas al comparar la frecuencia de población aragüeña con la reportada en poblaciones de amerindios venezolanos (7) son concordantes con datos históricos, los cuales revelan que durante los primeros 150 años de la conquista española la población de amerindios fue reducida en un 95%, por lo tanto, las características actuales de la población mestiza venezolana evidencia un menor aporte genético de estos grupos en relación al aporte español (18), está reportado que en la región central donde se encuentra ubicado el estado Aragua, el aporte español es predominante (0,604 ± 18) sobre el amerindio (0,235 ± 5) y el africano (0,161 ± 22) (19). Este hecho aunado a los movimientos migratorios desde Europa a América (18) ha generado que la población venezolana, tenga una frecuencia considerable del alelo CYP2D6*4, característico de la población caucásica, y similar, estadísticamente, a poblaciones de caucásicos americanos (p=0,063) y españoles (p=0,475) (Tabla 3).
Con relación a la frecuencia del fenotipo ML, se encontró que tiene un valor de 4,1%, siendo el doble que la reportada en muestras de población de venezolanos mestizos de la ciudad de Barquisimeto (7) y también es mayor a frecuencias observadas en varias poblaciones mestizas latinoamericanas (11,12,15,16), en contraste con el grupo de amerindios barí, donde se reportó 25% (7). Esta información tiene una aplicación potencial en la práctica médica, ya que es probable que el grupo de ML presente efectos adversos o baja respuesta a determinados medicamentos de prescripción frecuente y que son metabolizados por la enzima CYP2D6, tales como: amiodarona, amitriptilina, bisoprolol, carvedilol, captopril, cloroquina, codeína, domperidona, fluoxetina, fluvastatina, haloperidol, indinavir, imatinib, loperamida, nifedipina, ondansetrón, tamoxifeno, venlafaxina; para varios de estos fármacos existen guías de dosificación fundamentadas en las características genéticas de los pacientes, información que está disponible en la base de datos PharmGKB®
En este orden de ideas, la guía de dosificación para el fármaco metoprolol, de prescripción frecuente en arritmias cardiacas e hipertensión arterial sistémica, recomienda reducir en 75% la dosis estándar en los sujetos con características genotípicas de ML (20); porque pudieran estar sometidos al riesgo de desarrollar efectos adversos característicos de intoxicación como: visión borrosa, dolor torácico, mareos, síncopes, debilidad, oliguria, disnea. Este ejemplo resalta la importancia de continuar con las investigaciones sobre el gen CYP2D6, donde incluya un número mayor de individuos, tanto de la región central como de otras regiones del país y se analicen otros alelos que han sido reportados en Venezuela y en poblaciones latinoamericanas.
En Venezuela existen pocos estudios sobre CYP2D6 y otros genes involucrados en la variabilidad de respuesta a fármacos; por lo tanto, aún no es posible integrar conocimientos de farmacogenética a la práctica clínica. Los resultados de esta investigación son importantes para el diseño de estudios clínicos en virtud que el conocimiento de la frecuencia alélica es un requisito para un adecuado cálculo del tamaño muestral en estudios de casos y controles, de cohorte y de supervivencia y así generar conocimientos que contribuyan a lograr el ajuste de la dosis de un fármaco determinado, de acuerdo con las características de cada individuo, lo cual pudiera reducir la incidencia de efectos adversos y aumentar el efecto terapéutico deseado, en pacientes con enfermedades propias de la población venezolana.
Agradecimientos: a los voluntarios que participaron en este estudio. Gracias a los doctores Flor Herrera y Heriberto Correia del BIOMED-UC, por permitirnos realizar algunos experimentos en sus laboratorios y cedernos algunos reactivos necesarios para la investigación.
Contribuciones de autoría: NM y CFA participaron en la concepción y diseño del artículo, CFA, CV, YM y TO participaron en la recolección y obtención de resultados. NM, CFA, CV y JM participaron en el análisis e interpretación de datos. CFA participo en la redacción del artículo. NM y JM participaron en la revisión crítica del artículo. NM, CFA, CV, YM, JM y TO participaron en la aprobación de la versión final. CFA, CV y TO participaron en el aporte de pacientes o material de estudio. NM participo en la obtención de financiamiento.
Fuentes de financiamiento: se recibió financiamiento del Fondo Nacional de Ciencia, Tecnología e Innovación (FONACIT) a través de los proyectos 2012001248 y 2012002328. Parte de los equipos fueron financiados por el proyecto Misión Ciencia 2008000911-1
Conflictos de interés: los autores declaramos que la investigación fue conducida en ausencia de cualquier relación comercial y de financiamiento que pueda originar un posible conflicto de interés.
Referencias Bibliográficas
1. Samer CF, Lorenzini KI, Rollason V, Daali Y, Desmeules JA. Applications of CYP450 testing in the clinical setting. Mol Diagn Ther. 2013 Jun;17(3):165- 84. doi: 10.1007/s40291-013-0028-5. [ Links ]
2. Zhou SF, Sneed KB. Drug response heterogeneity and the genetic variability of cytochrome P450- Metabolizing enzymes. En: Vizirianakis I, ed. Handbook of Personalized Medicine: Advances in Nanotechnology, Drug Delivery, and Therapy. Florida: CRC Press; 2013. p. 375-606. [ Links ]
3. Gaedigk A, Simon SD, Pearce RE, Bradford LD, Kennedy MJ, Leeder JS. The CYP2D6 activity score: translating genotype information into a qualitative measure of phenotype. Clin Pharmacol Ther. 2008 Feb;83(2):234-42. [ Links ]
4. Llerena A, Dorado P, Ramírez R, González I, Alvarez M, PeñasLledó EM, et al. CYP2D6 genotype and debrisoquine hydroxylation phenotype in Cubans and Nicaraguans. Pharmacogenomics J. 2012 Apr;12(2):176-83. doi: 10.1038/ tpj.2010.85. [ Links ]
5. Saladores P, Mürdter T, Eccles D, Chowbay B, Zgheib NK, Winter S, et al. Tamoxifen metabolism predicts drug concentrations and outcome in premenopausal patients with early breast cancer. Pharmacogenomics J. 2015 Feb;15(1):84-94. doi: 10.1038/ tpj.2014.34. [ Links ]
6. Teh LK, Bertilsson L. Pharmacogenomics of CYP2D6: molecular genetics, interethnic differences and clinical importance. Drug Metab Pharmacokinet. 2012;27(1):55-67. [ Links ]
7. Griman P, Moran Y, Valero G, Loreto M, Borjas L, Chiurillo MA. CYP2D6 gene variants in urban/admixed and Amerindian populations of Venezuela: pharmacogenetics and anthropological implications. Ann Hum Biol. 2012 Mar;39(2):137-42. doi: 10.3109/03014460.2012.656703.
8. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extraction DNA from human nucleated cells. Nucleic Acids Res. 1988 Feb;16(3):1215. [ Links ]
9. Hanioka N, Kimura S, Meyer UA, Gonzalez FJ. The human CYP2D locus associated with a common genetic defect in drug oxidation: a G1934--- -A base change in intron 3 of a mutant CYP2D6 allele results in an aberrant 3 splice recognition site. Am J Hum Genet. 1990 Dec;47(6):994-1001. [ Links ]
10. Sachse C, Brockmöller J, Bauer S, Roots I. Cytochrome P450 2D6 variants in a Caucasian population: allele frequencies and phenotypic consequences. Am J Hum Genet. 1997 Feb;60(2):284-95. [ Links ]
11. Friedrich DC, Genro JP, Sortica VA, Suarez-Kurtz G, de Moraes ME, Pena SD, et al. Distribution of CYP2D6 alleles and phenotypes in the Brazilian population. PLoS One. 2014 Oct 20;9(10):e110691. doi: 10.1371/ journal.pone.0110691. [ Links ]
12. Salazar-Flores J, Torres-Reyes LA, Martínez-Cortés G, Rubi-Castellanos R, Sosa-Macías M, Muñoz-Valle JF, et al. Distribution of CYP2D6 and CYP2C19 polymorphisms associated with poor metabolizer phenotype in five Amerindian groups and western Mestizos from Mexico. Genet Test Mol Biomarkers. 2012 Sep;16(9):1098-104. doi: 10.1089/gtmb.2012.0055. [ Links ]
13. Ingelman-Sundberg M. Genetic polymorphisms of cytochrome P450 2D6 (CYP2D6): clinical consequences, evolutionary aspects and functional diversity. Pharmacogenomics J. 2005;5(1):6-13. [ Links ]
14. Isaza CA, Henao J, López AM, Cacabelos R. Isolation, sequence and genotyping of the drug metabolizer CYP2D6 gene in the Colombian population. Methods Find Exp Clin Pharmacol. 2000 Nov;22(9):695-705. [ Links ]
15. Céspedes-Garro C, JiménezArce G, Naranjo ME, Barrantes R, Llerena A, CEIBA. FP Consortium of the Ibero-American Network of Pharmacogenetics & Pharmacogenomics RIBEF. Ethnic background and CYP2D6 genetic polymorphisms in Costa Ricans. Rev Biol Trop. 2014;62(4):1659-71. [ Links ]
16. Dorado P, Heras N, Machín E, Hernández F, Teran E, Llerena A. CYP2D6 genotype and dextromethorphan hydroxylation phenotype in an Ecuadorian population. Eur J Clin Pharmacol. 2012 May;68(5):637-44. doi: 10.1007/ s00228-011-1147-8. [ Links ]
17. Almoguera B, Riveiro-Alvarez R, Gomez-Dominguez B, LopezRodriguez R, Dorado P, VaqueroLorenzo C, et al. Evaluating a newly developed pharmacogenetic array: screening in a Spanish population. Pharmacogenomics. 2010 Nov;11(11):1619-25. doi: 10.2217/ pgs.10.131. [ Links ]
18. Castro de Guerra D, Suárez M. Sobre el proceso de mestizaje en Venezuela. Interciencia. 2010;35(9):654-8. [ Links ]
19. Rodríguez-Larralde Á, Castro de Guerra D, González-Coira M, Morales J. Frecuencia génica y porcentaje de mezcla en diferentes áreas geográficas de Venezuela, de acuerdo a los grupos RH y ABO. Interciencia. 2001;26(1):8-12. [ Links ]
20. Whirl-Carrillo M, McDonagh EM, Hebert JM, Gong L, Sangkuhl K, Thorn CF, et al. Pharmacogenomics knowledge for personalized medicine. Clin Pharmacol Ther. 2012 Oct;92(4):414-7. doi: 10.1038/ clpt.2012.96. [ Links ]
Correspondencia: Nancy Moreno.
Dirección: BIOMED Final de la calle Cecilio Acosta Las Delicias - Maracay Estado Aragua Venezuela
Teléfono: 58-243- 2420577 y 58-243-2425822.
Correo electrónico: nanmorja@hotmail.com
Recibido: 02-06-15
Aprobado: 07-10-15