Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.32 no.4 Lima oct. 2015
Simposio
Rol de la farmacogenómica en el régimen de tratamiento de tuberculosis
The role of pharmacogenomics in the tuberculosis treatment regime
Heinner Guio1,a, Kelly S. Levano1,b, Cesar Sánchez1,c, David Tarazona1,d
1 Instituto Nacional de Salud. Lima, Perú
a Médico PhD en Ciencias Médicas; b bióloga PhD en Bioquímica; c médico; d genetista-biotecnólogo, máster en Biología Molecular
RESUMEN
La tuberculosis es un problema de salud Pública a nivel mundial con un tercio de la población infectada por el bacilo Mycobacterium tuberculosis. El tratamiento de primera línea incluye a las drogas isoniazida (INH) y rifampicina (RIF) metabolizadas en el hígado. La metabolización de drogas está directamente relacionada con la variación genética de NAT2 y CYP2E1 (asociados a metabolismo de INH) y AADAC (asociados a metabolismo de RIF), y los efectos pueden producir que un individuo sea metabolizador rápido, intermedio o lento. Los polimorfismos en genes de personas con tratamiento estándar de tuberculosis pueden ocasionar efectos en el metabolismo de drogas con consecuencias de hepatoxicidad e, incluso, posible drogorresistencia. Algunos países han empezado ensayos clínicos enfocados en la personalización del tratamiento a tuberculosis para reducir las consecuencias en pacientes en tratamiento. En países como el Perú, donde se registran altos índices de tuberculosis y, por consiguiente, más población en tratamiento, la farmacogenómica de individuos se convierte en una herramienta crucial para un óptimo tratamiento. La presente revisión destaca la importancia de tener estudios en farmacogenómica e identificar los polimorfismos asociados al metabolismo de las drogas antituberculosas en nuestra población peruana.
Palabras clave: Farmacogenética; Mycobacterium tuberculosis; Variación genética (fuente: DECS/Bireme)
ABSTRACT
Tuberculosis is a health problem worldwide with one-third of the population infected with the Mycobacterium tuberculosis bacilli. The first-line of treatment for tuberculosis includes the drugs Isoniazid (INH) and Rifampicin (RIF) metabolized in the liver. Drug metabolism is directly related to the genetic variation of NAT2 and CYP2E1 (associated with INH metabolism) and AADAC (associated with RIF metabolism), and the effects produced in an individual may be a fast, intermediate or slow metobolizer. Polymorphisms in genes of people in standard tuberculosis treatment can cause effects on drug metabolism with consequences of hepatotoxicity and even drug resistance. Countries have began clinical trials focusedon personalization of tuberculosis treatment to reduce the consequences for patients in treatment. In countries like Peru, where high rates of tuberculosis are recorded and therefore more people in treatment, the pharmacogenomic of individuals becomes a crucial tool for an optimum tuberculosis treatment. This review highlights the importance of having pharmacogenomic studies and having the identification of polymorphisms associated to the metabolism of the anti-tuberculosis drugs in our Peruvian population.
Key words: Pharmacogenetics; Mycobacterium tuberculosis; Genetic variation (source: MeSH/NLM)
LA GENÓMICA
La genómica, definido por Guttmacher y Collins como el estudio de genes individuales en relación a sus efectos en la fisiología y la interacción con otros genes del genoma, está tomando cada vez más importancia en los estudios biomédicos (1). De acuerdo con el articulo “A vision for the future of genomics research” (una visión del futuro de la investigación genómica) escrito por Collins et al., la genómica inició con el descubrimiento de las leyes de la herencia de Gregor Mendel en 1865 (2, 3) y junto con el reconocimiento de ADN como el material hereditario en 1944 por Oswald Avery, Collin MacLeod y Maclyn McCarty (4), el establecimiento de la estructura de doble hélice del ADN en 1953 por Rosalind Franklin, James Watson y Francis Crick (5), el desarrollo de la tecnología de ADN recombinante en 1972 por Stanley Cohen y Herbert Boyer (6), la invención del método de secuenciamiento en 1977 por Frederick Sanger y Allan Maxam (6, 7), el desarrollo de la técnica de la reacción en cadena de la polimerasa (PCR) en 1985 (8), y el desarrollo de la primera generación de un mapa genético humano en 1987 (9) en conjunto, llevaron al desarrollo del Proyecto del Genoma Humano en 1990 y culminaron con el secuenciamiento total del genoma humano en el año 2003. La genómica está llevando la investigación biomédica a una nueva gran escala (Big data) permitiendo la aparición de nuevas perspectivas y aplicaciones de este conocimiento profundo de la interacción de genes y medioambiente.
LA FARMACOGENÓMICA
Estamos viviendo en la era de las “omicas”, con nuevas áreas de investigación, incluyendo, la genómica humana, metagenómica, nutrígenómica, transcriptomica, proteomica y farmacogenómica. Esta última es una de las más prometedoras para la llamada “medicina personalizada”. La genómica humana ha generado mucha información, en especial entre las interacciones de genoma-enfermedad y genoma-droga, la cual ha transformado la farmacogenética a la farmacogenómica (10,11). La farmacogenética se enfoca en estudiar el efecto de polimorfismos de un gen en el metabolismo y la acción de medicamentos en cada individuo para poder así optimizar el tratamiento, aumentando la eficacia y reduciendo los efectos adversos. La farmacogenómica, nos traslada al siguiente paso, encontrar nuevas y potenciales dianas, evaluando la respuesta no solo de un gen sino de múltiples genes, y así poder proponer nuevas estrategias para evaluar y desarrollar medicamentos (12).
TUBERCULOSIS, FARMACOGENÓMICA Y DROGORRESISTENCIA
Se conoce que la farmacogenómica desempeña un papel importante en el estudio del metabolismo de las drogas antituberculosas y así en las aplicaciones clínicas en términos de la eficacia al tratamiento (13), y es aquí donde nos centraremos en el presente simposio.
Como se conoce, la tuberculosis (TB) es un problema de salud pública a nivel mundial. De acuerdo con la Organización Mundial de la Salud (OMS), en el 2013 se reportaron nueve millones de casos de tuberculosis, y 1,5 millones de muertes. Más del 95% de muertes por TB ocurren en los países de bajo y mediados ingresos como el Perú, el cual tuvo una incidencia de TB estimada a 124 000 en el 2013. Casi al 100% de los casos de TB sensible pueden ser curados si la terapia es administrada adecuadamente y a tiempo. Adicionalmente a este problema, el incremento de casos de drogorresistencia se está haciendo cada vez más evidente, en el 2013, la OMS reporto 480 000 casos de TB multidrogorresistente (TB-MDR) a nivel mundial, y en el Perú 3,9% de casos de TB fueron TB-MDR.
La drogorresistencia en TB puede ocurrir debido a la administración inadecuada de los medicamentos. La dosis o concentración adecuada de un medicamento depende de su tasa de absorción, distribución, biotransformación y excreción dentro del organismo. La farmacocinética se enfoca en esta fase estudiando el curso y distribución del medicamento/droga y sus metabolitos. La farmacodinámica se encarga de estudiar los mecanismos de acción, y los efectos bioquímicos y fisiológicos de la droga (14). La farmacocinética y la farmacodinámica son afectadas por la variabilidad genética que existe entre distintas poblaciones e individuos. La presencia de polimorfismos en genes que codifican las enzimas que procesan los medicamentos puede afectar su actividad metabólica y, por consiguiente, la concentración del medicamento en sangre, llevando a un tratamiento fallido. La dinámica de concepción en el tratamiento médico está cambiando desde la medicina curativa (diagnóstico y tratamiento), la medicina preventiva (intervención para evitar o retrasar la aparición de la enfermedad) a la medicina predictiva (identificación del perfil genético para un tratamiento personalizado). Por consiguiente, vemos la necesidad de incluir un análisis personalizado de los polimorfismos en los genes asociados al metabolismo de los medicamentos antes de la prescripción y dosificación de un tratamiento indicado.
A la fecha, todavía están pendientes los estudios del perfil completo de polimorfismos presentes en los genes que codifican las enzimas que procesan los medicamentos de tratamiento de primera línea para TB en la población peruana. Las drogas de primera línea para tratar TB son isoniazida (INH) y rifampicina (RIF) y la resistencia a ellas no solamente sería debido a una falta de adherencia al tratamiento, sino también a un factor genético (15). Estudios han reportado que deleciones y mutaciones en el gen katG, el cual codifica una catalasa-peroxidasa que convierte la INH a su forma activa, fueron asociados con resistencia a INH (15,16). La resistencia a RIF es conferida por mutaciones puntuales, deleciones de nucleótidos o inserciones en una región del gen rpoB, la cual codifica la β-subunidad de la ARN polimerasa (15,17). La sensibilidad y resistencia a drogas esta relacionado al genoma del patógeno; sin embargo, como ya se mencionó, polimorfismos en los genes del huéspedes involucrados en el procesamiento y disponibilidad de las drogas antituberculosas pueden llevar a la adquisición de resistencia, así como en los genes que codifican las enzimas que procesan INH, N-acetyltransferasa 2 (NAT2) y el citocromo P4502E1 (CYP2E1) en humanos (18-21).
Estas dos enzimas se encuentran en el hígado y trabajan en conjunto para metabolizar INH a acetilisoniazida, seguido por hidrólisis a acetilhidrazina y oxidación a metabolitos tóxicos (Figura 1). Aunque se conoce menos sobre las enzimas que metabolizan a RIF, estudios previos han reportado que polimorfismos en la enzima arilacetamida desacetilasa (AADAC) (22), el cual desacetiliza a RIF, ocasionando cambios en su actividad farmacológica (Figura 1). Polimorfismos en estos genes pueden causar un incremento en la actividad metabólica llevando a una disminución en la concentración mínima inhibitoria para eliminar el Mycobaterium tuberculosis, la cual podría resultar eventualmente en drogorresistencia. Por el contrario, los polimorfismos también pueden causar una disminución en la activad metabólica llevando a un incremento en la concentración sérica de las drogas, resultando en hepatotoxicidad inducida por drogas antituberculosas.
NAT2
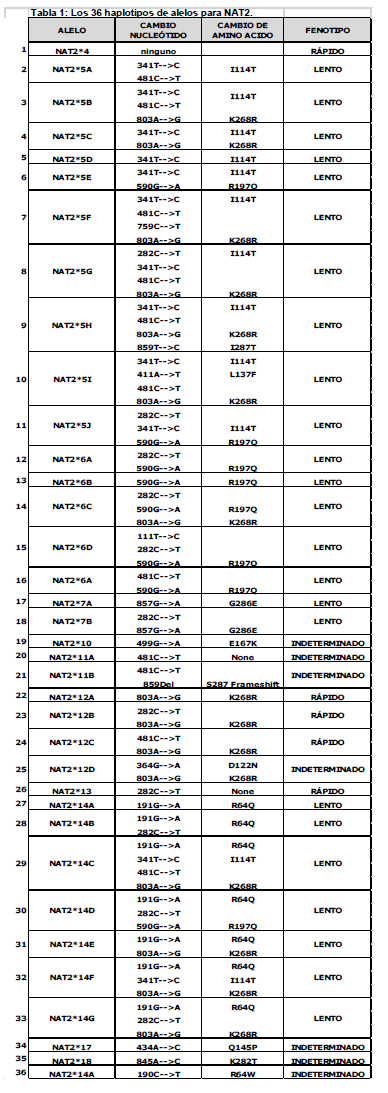
La enzima NAT2 ha sido extensamente estudiada. Es una enzima detoxificante expresada principalmente en el hígado. Acetiliza varias drogas actualmente en el mercado, incluyendo, INH, sulfadmidina, hidralazina, dapsona, procaina amida, sulfapiridina y nitrazepam (23). El gen NAT2, el cual contiene un solo exon de 870 pares de bases, codifica 290 aminoácidos, y se conocen 16 polimorfismos de nucleótido único (SNP) y una deleción de un par de bases, los cuales han sido asociados con una alteración en la actividad metabólica (24-26). Existen 36 haplotipos de alelos conocidos, y cada haplotipo individual tiene un fenotipo predictivo de lento, intermedio o rápido (Tabla 1). El fenotipo de un individuo (metabolizador lento, intermedio o rápido) está basado en la identificación de los SNP y los aplotipos encontrados en NAT2. Durante la renotificación se identificará dos haplotipos. La identificación de dos haplotipos rápidos nos lleva a fenotipificar como un metabolizador rápido, mientras que genotipificar dos haplotipos lentos fenotipifíca un metabolizador lento. En el caso de encontrarse un haplotipo rápido y el otro lento el fenotipo resultante será catalogado como intermedio (27). Kuznetsov et al. han desarrollado un servidor web de acceso libre para la determinación rápida de los fenotipos de NAT2 en un tamizaje genético (http:// nat2pred.tit.albany.edu) (27).
Los estudios de diversidad genética en las poblaciones son importantes porque identifica los polimorfismos propios para cada población. La base de la farmacogenómica es que no se puede extrapolar el genotipo obtenido en otras poblaciones para la elección y dosificación de un determinado fármaco. Basado en estos conceptos, el Instituto Nacional de Salud del Perú en colaboración con la Estrategia Sanitaria Nacional de Prevención y Control de la Tuberculosis (ESNPCT) está identificando los polimorfismos en los genes que codifican a las enzimas (NAT2, AADAC y CYP2E1) responsables de metabolizar INH y RIF en la población peruana con diagnóstico de tuberculosis. Fuselli et al. en el 2007, analizaron los polimorfismos en NAT2 en 456 participantes de 12 poblaciones nativo americanas (Chipewyan y Cree de Canadá; Pima y Maya de México; Piapoco y Curripaco de Colombia; Cayapa de Ecuador; San Martín y Tayacaja de Perú; Karitiana, y Surui, Xavante de Brasil.
Los polimorfismos en NAT2 (26). En los resultados no se observó ningún polimorfismo único o propio para las poblaciones americanas, y el promedio de frecuencias de metabolizadotes rápidos, intermedios y lentos fue de 18, 56, 25% respectivamente, en todas las poblaciones nativas. Este primer estudio de polimorfismos en varias poblaciones basado en 66 muestras de Perú, no provee suficiente evidencia para optimizar la concentración de la droga y disminuir los efectos secundarios de la isoniazida en la población peruana con diagnóstico de tuberculosis. El estudio “Frecuencia de los genotipos NAT2, CYP2E1 y AADAC en pacientes peruanos con diagnóstico de tuberculosis pulmonar” que se está planteando desde el Instituto Nacional de Salud analizará 500 pacientes con diagnóstico de TB en Lima, la cual concentra el 70% de pacientes del Perú.
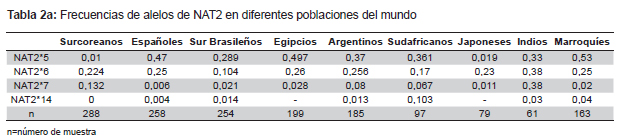
Otros países ya han reportado los genotipos de NAT2 en sus poblaciones, entre ellos están, China (28), México (28-30), Morocco (31), India (32) y Brasil (33), encontrado diferencias en sus genotipos comparados con los de otras poblaciones. La Tabla 2a muestra un metaanálisis de las frecuencias de los alelos de NAT2 en nueve poblaciones (31).
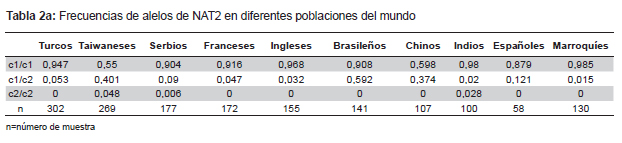
CYP2E1 El gen CYP2E1 es miembro de la familia de enzimas del citocromo P450, su función es importante en el metabolismo y bioactivación de compuestos de bajo peso molecular (etanol y acetona), procarcinógenos (benceno, N-nitrosodimetilamina y estireno), y drogas (INH, acetaminofeno, clorzoxazona y anestésicos) (34). Se ha reportado que el gen CYP2E1 tiene varios polimorfismos, pero solo uno (rs2031920 (-1053 C>T)) ha sido extensamente evaluado en asociación con las drogas antituberculosas, este polimorfismo puede afectar la actividad de la enzima (31,35). El genotipo (C/C) ha sido asociado a la reducción de actividad metabólica (metabolizador lento) de CYP2E1 y así causando un alto riesgo de hepatotoxicidad. La Tabla 2b muestra un metaanálisis de las frecuencias de los genotipos reportados para CYP2E1, en el cual se demuestra la variación genética entre diferentes poblaciones (31).
AADAC
Actualmente no existe mucha información de frecuencias de genotipos en la enzima AADAC. Se ha reportado que es una enzima relacionada al metabolismo de RIF, miembro de la superfamilia de serina esterasa, la cual se expresa en el hígado y en el tracto gastrointestinal (36). Uno de los polimorfismos que ha sido reportado por afectar la actividad de esta enzima es AADAC*3: 14008T>C. Esta enzima está siendo considerada y analizada en un estudio actual del Instituto Nacional de Salud en pacientes con tuberculosis, trabajo en el que reportarán los genotipos presentes en nuestra población.
TERAPIA BASADA EN LA FARMACOGENÓMICA
Como hemos venido mencionando, la importancia de determinar la variabilidad genética de la población peruana con el fin de rediseñar el régimen de tratamiento antituberculoso y administrar la dosis adecuada. En Japón ya se han empezado ensayos clínicos para evaluar el cambio de régimen de tratamiento basado en los polimorfismos del gen NAT2 (37), Azuma et al., se basaron en estudios previos de farmacocinética para determinar la dosis clínica apropiada para cada genotipo. De acuerdo con estos estudios, individuos con fenotipo lento deben recibir mitad de la dosis estándar, mientras que individuos con fenotipo rápidos deben recibir 1,5 veces la dosis estándar (38). De acuerdo con la Norma Técnica de salud para la atención integral de las personas afectadas por tuberculosis/Ministerio de Salud del 2013 y a nivel mundial, la dosis actual de los medicamentos antituberculosis de primera línea, isoniazida (INH) y rifampicina (RIF) para pacientes con diagnóstico de TB mayores de 15 años son: para la fase inicial, 5 mg/kg (máximo 300 mg) de INH y 10 mg/kg (máximo 600 mg) de RIF de forma diaria, y para segunda fase 10 mg/kg (máximo 900 mg) de INH y 10 mg/kg (máximo 600mg) de RIF tres veces por semana. Debemos de considerar la reevaluación de estas dosis basándonos en los genotipos de nuestra población y/o de cada individuo. Los resultados del ensayo clínico de Azuma et al. mostraron que hubo una incidencia baja de lesión hepática del 15% para los metabolizadores rápidos que recibieron la dosis modificada basada en el genotipo de NAT2 comparada con una incidencia de 38% de los que recibieron la dosis estándar. Asimismo, un 78% de los metabolizadores lentos que recibieron la dosis estándar tuvieron una lesión hepática relacionada a INH, mientras que ninguno de los metabolizadores lentos que recibieron la dosis modificada basado en el genotipo de NAT2 tuvieron problemas hepáticos. El ensayo clínico demuestra el beneficio en la reducción de los efectos adversos de pacientes en tratamiento estándar y pacientes en tratamiento modificado basado en los genotipos de metabolización de drogas INH.
En países con población homogénea y heterogénea, se observa individuos con genotipos distintos en la metabolización de drogas. ¿Cual será la distribución en nuestra población? En nuestro estudio “Frecuencia de los genotipos NAT2, CYP2E1 y AADAC en pacientes peruanos con diagnóstico de tuberculosis pulmonar”, ampliaremos nuestro conocimiento de la distribución alélica para los genes involucrados en la metabolización de drogas INH y RIF en nuestra población.
COMENTARIOS FINALES
Las concentraciones en sangre de las drogas antituberculosas varían significativamente entre poblaciones y/o individuos. La estrategia de “una sola dosis para todos” en el tratamiento contra TB debería reevaluarse. Como hemos venido enfatizando en este simposio, si el tratamiento es inadecuado puede fracasar, provocar hepatotoxicidad y drogorresistencia. Los polimorfismos en las enzimas responsables del metabolismo de INH y RIF son uno de los factores más comunes que causa variabilidad interindividual. En un futuro cercano, la determinación del genotipo metabolizador debe de incluirse en el protocolo de evaluación para así dar un tratamiento personalizado adecuado que incremente la eficacia y disminuya los efectos secundarios (Figura 2). El análisis de los genotipos de NAT2, CYP2E1 y AADAC en personas con diagnóstico de TB en el Perú será de gran importancia para determinar los polimorfismos presentes en nuestra población y así determinar nuestro fenotipo metabolizador.
Contribuciones de autoría: HG y KL han participado en la concepción del artículo. HG, KL, CS y DT participado en la recolección de datos, análisis e interpretación del artículo, redacción del artículo, revisión crítica del artículo.
Fuentes de financiamiento: Instituto Nacional de Salud, CNSP.
Conflictos de interés: ninguna.
Referencias Bibliográficas
1. Guttmacher AE, Collins FS. Genomic medicine--a primer. N Engl J Med. 2002 Nov 7;347(19):1512-20. [ Links ]
2. Mendel G. Versuche über PflanzenHybriden. Verhandlungen des naturforschenden Vereines, Abhandlungen, Brünn. 1866;4:3-47. [ Links ]
3. Collins FS, Green ED, Guttmacher AE, Guyer MS; US National Human Genome Research Institute. A vision for the future of genomics research. Nature. 2003 Apr 24;422(6934):835-47. [ Links ]
4. Avery OT, Macleod CM, McCarty M. Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types : Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III. J Exp Med. 1944 Feb 1;79(2):137-58. [ Links ]
5. Watson JD, Crick FH. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid. Nature. 1953;171(4356):737-8. [ Links ]
6. Cohen SN, Chang AC, Boyer HW, Helling RB. Construction of biologically functional bacterial plasmids in vitro. Proc Natl Acad Sci U S A. 1973 Nov;70(11):3240-4. [ Links ]
7. Sanger F, Coulson AR. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 1975 May;94(3):441-8. [ Links ]
8. Smith LM, Sanders JZ, Kaiser RJ, Hughes P, Dodd C, Connell CR, et al. Fluorescence detection in automated DNA sequence analysis. Nature. 1986 Jun 12-18;321(6071):674-9. [ Links ]
9. Committee on Mapping and Sequencing the Human Genome, Commission on Life Sciences. Mapping and Sequencing the Human Genome. Washington D.C: National Academies Press; 1988. [ Links ]
10. Ginsburg GS, Willard HF. Genomic and personalized medicine: foundations and applications. Transl Res. 2009 Dec;154(6):277-87. doi: 10.1016/j.trsl.2009.09.005. [ Links ]
11. Ma Q, Lu AY. Pharmacogenetics, pharmacogenomics, and individualized medicine. Pharmacol Rev. 2011 Jun;63(2):437-59. doi: 10.1124/pr.110.003533. [ Links ]
12. Soto Alvarez J. [Economic evaluation in the pharmacogenetics and pharmacogenomics era: a light in the darkness?]. Med Clin (Barc). 2006 Nov 4;127(17):657-9. [Artículo en español] [ Links ]
13. Ramachandran G, Swaminathan S. Role of pharmacogenomics in the treatment of tuberculosis: a review. Pharmgenomics Pers Med. 2012;5:89- 98. [ Links ]
14. Teixeira R, Quinhones M, Noel P, Rezende A. Tuberculosis Pharmacogenetics: State of The Art. Croacia: InTech; 2013. [ Links ]
15. Nachega JB, Chaisson RE. Tuberculosis drug resistance: a global threat. Clin Infect Dis. 2003 Jan 15;36(Suppl 1):S24-30. [ Links ]
16. Zhang Y, Heym B, Allen B, Young D, Cole S. The catalase-peroxidase gene and isoniazid resistance of Mycobacterium tuberculosis. Nature. 1992 Aug 13;358(6387):591-3. [ Links ]
17. Telenti A, Imboden P, Marchesi F, Lowrie D, Cole S, Colston MJ, et al. Detection of rifampicin-resistance mutations in Mycobacterium tuberculosis. Lancet. 1993 Mar 13;341(8846):647-50.
18. Lauterburg BH, Smith CV, Todd EL, Mitchell JR. Pharmacokinetics of the toxic hydrazino metabolites formed from isoniazid in humans. J Pharmacol Exp Ther. 1985 Dec;235(3):566-70. [ Links ]
19. Calver AD, Falmer AA, Murray M, Strauss OJ, Streicher EM, Hanekom M, et al. Emergence of increased resistance and extensively drugresistant tuberculosis despite treatment adherence, South Africa. Emerg Infect Dis. 2010 Feb;16(2):264-71. doi: 10.3201/eid1602.090968. [ Links ]
20. Srivastava S, Pasipanodya JG, Meek C, Leff R, Gumbo T. Multidrugresistant tuberculosis not due to noncompliance but to between-patient pharmacokinetic variability. J Infect Dis. 2011 Dec 15;204(12):1951-9. doi: 10.1093/infdis/jir658. [ Links ]
21. Pasipanodya JG, Srivastava S, Gumbo T. Meta-analysis of clinical studies supports the pharmacokinetic variability hypothesis for acquired drug resistance and failure of antituberculosis therapy. Clin Infect Dis. 2012 Jul;55(2):169-77. doi: 10.1093/cid/cis353. [ Links ]
22. Nakajima A, Fukami T, Kobayashi Y, Watanabe A, Nakajima M, Yokoi T. Human arylacetamide deacetylase is responsible for deacetylation of rifamycins: rifampicin, rifabutin, and rifapentine. Biochem Pharmacol. 2011 Dec 1;82(11):1747-56. doi: 10.1016/j.bcp.2011.08.003. [ Links ]
23. Harmer D, Evans DA, Eze LC, Jolly M, Whibley EJ. The relationship between the acetylator and the sparteine hydroxylation polymorphisms. J Med Genet. 1986 Apr;23(2):155-6. [ Links ]
24. Hein DW, Doll MA, Fretland AJ, Leff MA, Webb SJ, Xiao GH, et al. Molecular genetics and epidemiology of the NAT1 and NAT2 acetylation polymorphisms. Cancer Epidemiol Biomarkers Prev. 2000 Jan;9(1):29-42. [ Links ]
25. Leff MA, Fretland AJ, Doll MA, Hein DW. Novel human N-acetyltransferase 2 alleles that differ in mechanism for slow acetylator phenotype. J Biol Chem. 1999 Dec 3;274(49):34519-22. [ Links ]
26. Fuselli S, Gilman RH, Chanock SJ, Bonatto SL, De Stefano G, Evans CA, et al. Analysis of nucleotide diversity of NAT2 coding region reveals homogeneity across Native American populations and high intra-population diversity. Pharmacogenomics J. 2007 Apr;7(2):144-52. [ Links ]
27. Kuznetsov IB, McDuffie M, Moslehi R. A web server for inferring the human N-acetyltransferase-2 (NAT2) enzymatic phenotype from NAT2 genotype. Bioinformatics. 2009 May 1;25(9):1185-6. doi: 10.1093/ bioinformatics/btp121. [ Links ]
28. Chen B, Li JH, Xu YM, Wang J, Cao XM. The influence of NAT2 genotypes on the plasma concentration of isoniazid and acetylisoniazid in Chinese pulmonary tuberculosis patients. Clin Chim Acta. 2006 Mar;365(1-2):104-8. [ Links ]
29. Taja-Chayeb L, Agúndez JA, Miguez-Muñoz C, Chavez-Blanco A, Dueñas-Gonzalez A. Arylamine N-acetyltransferase 2 genotypes in a Mexican population. Genet Mol Res. 2012 Apr 27;11(2):1082-92. doi: 10.4238/2012.April.27.7. [ Links ]
30. Salazar-González R, Gómez R, Romano-Moreno S, Medellín-Garibay S, Núñez-Ruíz A, Magaña-Aquino M, et al. Expression of NAT2 in immune system cells and the relation of NAT2 gene polymorphisms in the anti-tuberculosis therapy in Mexican mestizo population. Mol Biol Rep. 2014 Dec;41(12):7833-43. doi: 10.1007/s11033-014-3677-5. [ Links ]
31. Guaoua S, Ratbi I, Laarabi FZ, Elalaoui SC, Jaouad IC, Barkat A, et al. Distribution of allelic and genotypic frequencies of NAT2 and CYP2E1 variants in Moroccan population. BMC Genet. 2014;15:156. [ Links ]
32. Mishra S, Daschakraborty S, Shukla P, Kapoor P, Aggarwal R. N-acetyltransferase and cytochrome P450 2E1 gene polymorphisms and susceptibility to antituberculosis drug hepatotoxicity in an Indian population. Natl Med J India. 2013 Sep-Oct;26(5):260-5. [ Links ]
33. Santos NP, Callegari-Jacques SM, Ribeiro Dos Santos AK, Silva CA, Vallinoto AC, Fernandes DC, et al. N-acetyl transferase 2 and cytochrome P450 2E1 genes and isoniazidinduced hepatotoxicity in Brazilian patients. Int J Tuberc Lung Dis. 2013 Apr;17(4):499-504. doi: 10.5588/ ijtld.12.0645. [ Links ]
34. Tang K, Li X, Xing Q, Li W, Feng G, He L, et al. Genetic polymorphism analysis of cytochrome P4502E1 (CYP2E1) in Chinese Han populations from four different geographic areas of Mainland China. Genomics. 2010 Apr;95(4):224-9. doi: 10.1016/j.ygeno.2010.01.005. [ Links ]
35. Sheng YJ, Wu G, He HY, Chen W, Zou YS, Li Q, et al. The association between CYP2E1 polymorphisms and hepatotoxicity due to antituberculosis drugs: a meta-analy Infect Genet Evol. 2014 Jun;24:34-40. doi: 10.1016/j.meegid.2014.01.034. [ Links ]
36. Shimizu M, Fukami T, Kobayashi Y, Takamiya M, Aoki Y, Nakajima M, et al. A novel polymorphic allele of human arylacetamide deacetylase leads to decreased enzyme activity. Drug Metab Dispos. 2012 Jun;40(6):1183- 90. doi: 10.1124/dmd.112.044883. [ Links ]
37. Azuma J, Ohno M, Kubota R, Yokota S, Nagai T, Tsuyuguchi K, et al. NAT2 genotype guided regimen reduces isoniazid-induced liver injury and early treatment failure in the 6-month four-drug standard treatment of tuberculosis: a randomized controlled trial for pharmacogenetics-based therapy. Eur J Clin Pharmacol. 2013 May;69(5):1091-101. doi: 10.1007/ s00228-012-1429-9. [ Links ]
38. Kinzig-Schippers M, Tomalik-Scharte D, Jetter A, Scheidel B, Jakob V, Rodamer M, et al. Should we use N-acetyltransferase type 2 genotyping to personalize isoniazid doses?. Antimicrob Agents Chemother. 2005 May;49(5):1733-8. [ Links ]
Correspondencia: Heinner Guio
Dirección: Av. Defensores del Morro 2268. Chorrillos, Perú
Teléfono: 748-0000, Anexo: 424
Correo electrónico: hguio@ins.gob.pe
Recibido: 20-10-15
Aprobado: 18-11-15