Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.33 no.2 Lima Apr./Jun. 2016
http://dx.doi.org/10.17843/rpmesp.2016.332.2101
ORIGINAL BREVE
Clonación de secuencias de alfavirus y flavivirus para su uso como controles positivos en el diagnóstico molecular
Cloning alphavirus and flavivirus sequences for use as positive controls in molecular diagnostics
Daría Camacho1,a, Jesús Reyes1,b, Leticia Franco 2,c, Guillermo Comach1,d, Elizabeth Ferrer1,e
1 Instituto de Investigaciones Biomédicas (BIOMED-UC), Facultad de Ciencias de la Salud, Universidad de Carabobo. Maracay, Venezuela.
2 Centro Nacional de Microbiología. Instituto de Salud Carlos III. Majadahonda, Madrid, España.
a Master en Virología; b Licenciado en Bioanálisis; c Doctora en Bioquímica; d Master of Science; e Doctora en Biología Molecular
RESUMEN
El objetivo de la investigación fue obtener controles positivos para la validación de técnicas moleculares (RT-PCR) utilizadas en diagnóstico e investigación de infecciones virales. A partir de cepas de CHIKV, Zika, DENV-1, DENV-2, DENV-3 y DENV-4, se extrajeron ARN virales para obtener por RT-PCR los ADN complementarios (ADNc) de las secuencias nsP4 (CHIKV), NS5 (virus Zika), C/prM-M y 5´UTR-C (DENV-1, DENV-2, DENV-3, DENV-4) que fueron clonados en pGEM®-T Easy. La clonación se confirmó mediante PCR de colonias, de las cuales se extrajo el ADN plasmídico para la verificación de la clonación de los fragmentos. Se logró la clonación de ADNc correspondientes a nsP4, NS5, C/prM-M y 5´UTR-C de los distintos agentes virales. En conclusión se obtuvieron los plásmidos recombinantes con cada una de las secuencias especificadas para su posterior valoración como controles positivos en técnicas moleculares, evitando el uso de cultivos celulares que pueden resultar costosos, laboriosos y potencialmente peligrosos.
Palabras clave: Alphavirus; Flavivirus; Clonación, molecular; Diagnóstico (fuente: DeCS BIREME).
ABSTRACT
The purpose of the study was to obtain a positive control to validate molecular techniques (reverse transcription– polymerase chain reaction [RT-PCR]) used in the diagnosis and research of viral infections. From strains of Chikungunya virus (CHIKV), Zika virus, and Dengue virus (DENV-1, DENV-2, DENV- 3, and DENV-4) viral RNAs were extracted to obtain complementary DNA using RT-PCR from the nsP4 (CHIKV), NS5 (Zika virus), C/prM-M, and 5′UTR-C (DENV-1, DENV-2, DENV-3, DENV-4) sequences, which were cloned into pGEM®-T Easy. Cloning was confirmed through colony PCR, from which plasmid DNA was extracted for fragment cloning verification. Cloning of cDNA corresponding to nsP4, NS5, C/prM-M, and 5′UTR-C of the different viral agents was achieved. In conclusion, recombinant plasmids were obtained with each of the sequences specified for further assessment as positive controls in molecular techniques in an effort to avoid the use of cell cultures, which can be costly, time-consuming, and potentially dangerous.
Keywords: Alphavirus; Flavivirus; Cloning, molecular; Diagnosis, (source: MeSH NLM).
INTRODUCCIÓN
Los flavivirus y alfavirus conforman un grupo de agentes virales causantes de enfermedades en humanos (1,2). Los primeros pertenecen a la familia Flaviviridae, son transmitidos por mosquitos del género Aedes, Culex, Haemagogus, entre otros. Destacan, el virus dengue(DENV), el virus de la encefalitis japonesa (JEV), el virus de la encefalitis de San Luis (SLEV), fiebre amarilla (YFV) y el virus del Nilo Occidental (WNV) (1). En la región de las Américas las poblaciones se han visto afectadas por cualquiera de los serotipos de DENV (3,4); YFV ha cobrado importancia por su reemergencia en Sudamérica y África (5); WNV ha ampliado su distribución y focos de transmisión (6). En 2015, el virus Zika fue detectado en Brasil representando una amenaza para el resto de los países de la región (7,8).
Entre los alfavirus destacan los virus de la encefalitis equina del este y oeste (EEEV y WEEV), Semliki Forest (SFV), Chikungunya (CHIKV) y O’nyong nyong (ONNV) (2). Los alfavirus pueden causar enfermedades que pueden ser fatales [e.g. encefalitis equina del este y oeste (EEE y EEO)], Semliki Forest (SF), Chikungunya (CHIKV) y O’nyong nyong (ONN) (2). Algunos miembros de flavivirus y alfavirus, se asocian a brotes de importancia para la salud pública. DENV circula en Venezuela desde 1989, y se han identificado los cuatro serotipos del virus (9,10). En relación al virus Zika, a pesar de que no había reportes de su presencia, su identificación en Brasil (7,8) abrió la posibilidad de introducción en Venezuela. CHIKV fue reconocido en América como causa de brotes durante 2013 y 2014 (11). Desde entonces se confirmó transmisión local en más de 43 países y territorios americanos. En Venezuela, CHIKV se confirmó en 2014 (12).
Para diagnosticar flavivirus y/o alfavirus es indispensable la confirmación en los laboratorios. Se han desarrollado metodologías, entre las que destacan las técnicas moleculares como la transcripción reversa del ARN seguido de reacción en cadena de la polimerasa (RT-PCR), para amplificar secuencias nucleotídicas con una sensibilidad y especificidad elevada, demostrando su utilidad como herramienta diagnóstica en la vigilancia epidemiológica (13-15) e investigación (16). En todos los casos, se requiere del uso de controles positivos que validen los ensayos. Clásicamente, se utilizan semillas virales, pero obtenerlas requiere de técnicas laboriosas y costosas de cultivo celular, además elevados requerimientos de bioseguridad.
Una alternativa, para la obtención de controles positivos, es el uso de la tecnología de ADN recombinante para clonar secuencias específicas del genoma de diferentes agentes virales. En este reporte se muestra la clonación de secuencias del alfavirus CHIKV y los flavivirus zika, DENV-1, DENV-2, DENV-3 y DENV-4, con el fin de sugerir el uso de estos clones como controles positivos en las técnicas moleculares (13-16), lo que minimizaría el uso de sistemas de multiplicación viral, que además de ser costosos y laboriosos generan riesgo de infección en el personal.
MATERIALES Y MÉTODOS
MUESTRA BIOLÓGICA
Estuvo constituida por cepas virales. Específicamente, el alfavirus CHIKV [genotipo ECSA (East Central South África), cepa DRC 1721)] y los flavivirus zika [genotipo africano (MR766)], DENV-1 [genotipo II, (Hawaii)], DENV-2 [genotipo II NG (New Guinea)"C"], DENV-3 [genotipo II (H-87)] y DENV-4 [genotipo I (H-241)].
VECTOR DE CLONACIÓN Y CEPA BACTERIANA
Se empleó el plásmido comercial pGEM®-T Easy (Promega), diseñado para la clonación de productos de PCR. Se utilizaron células de Escherichia coli XL1-Blue MRF` (Stratagene) [genotipo: supE44, hsdR17, recA1, endA1, gyrA46, thirelA1, lacf (proAB+ lacI lacZ ∆M15,Tn10, Tetr], para la multiplicación y mantenimiento del ADN plasmídico (ADNp).
PREPARACIÓN DE CÉLULAS COMPETENTES DE E. coli XL1-BLUE MRF´
Las E. coli XL1B criopreservadas se sembraron en placas de Petri [LB sólido (triptona 10g/L, extracto de levadura 5 g/L, NaCl 5 g/L, pH 7, tetraciclina 12,5 µg/mL)] e incubadas a 37 °C durante 12 h. Luego, se realizó el pre-inóculo en 5 ml de medio LB líquido (triptona 10g/L, extracto de levadura 5 g/L, NaCl 5 g/L, pH 7). Se diluyó 1 ml de cultivo en 50 ml de medio LB e incubó a 37°C con agitación de 250 rpm. El cultivo fue centrifugado por 15 min a 3015 rpm a 4 °C y el sedimento resuspendido con CaCl2 100 mM. Esta mezcla fue centrifugada y el sedimento resuspendido en CaCl2 100 mM-Glicerol 20% para su almacenamiento a -80 °C (17).
AMPLIFICACIÓN DE FRAGMENTOS GENÓMICOS CORRESPONDIENTES A CHIKV Y A CEPAS DEL GÉNERO FLAVIVIRUS
EXTRACCIÓN DE ARN: se obtuvieron a partir de 140 μL de cultivos virales mediante el uso del estuche de extracción viral de ARN QIAamp QIAGEN® (QIAGEN Inc.; California, E.U.A), siguiendo las instrucciones del fabricante. Los productos obtenidos fueron resuspendidos en 60 μL de agua libre de nucleasas y almacenados en alícuotas de 10 μL cada una a -80 °C. Alfavirus (CHIKV): se amplificó un fragmento de 481 pb correspondiente al gen nsP4 de acuerdo a una metodología previamente descrita (13) con modificaciones. En la primera reacción se utilizó MgSO4 (2mM); y en la segunda reacción MgCl2 (2mM), dNTP´s (0,08 mM) y cebadores (8 pmoles). Las condiciones restantes fueron iguales a las descritas originalmente.
Flavivirus (Zika): en este caso también se empleó una metodología para la obtención de un fragmento de 1385 pb de la región NS5 (14) con modificaciones. En la primera reacción se empleó MgSO4 (2mM), cebadores (50 pmol); y en la segunda reacción MgCl2 (2mM) y dNTP´s (0,08 mM). El resto de las condiciones permanecieron iguales.
DENV-1, DENV-2, DENV-3 y DENV-4 (C/prM-M): para la amplificación del fragmento C/prM-M de 511 pb, común para todos los serotipos de DENV se siguieron instrucciones descritas (15).
DENV-1, DENV-2, DENV-3 y DENV-4 (5´UTR-C): estos fragmentos se obtuvieron conforme a las instrucciones descritas para obtener productos de 275 pb (DENV-1), 300 pb (DENV-2), 274 pb (DENV-3) y 339 pb (DENV-4) (16).
Los productos de las RT-PCR fueron analizados en gel de agarosa al 2 %, con bromuro de etidio 0,1 µg/mL (18). La visualización se realizó en un Gel Doc 1000 (programa Multi-Analyst Bio-Rad®). Las bandas de ADNc de las primeras reacciones (para obtener los fragmentos grandes, donde están contenidas los fragmentos pequeños de las segundas reacciones en el caso de las RT-PCR anidadas) fueron cortadas a partir del gel y purificadas empleando Wizard ® SV Gel and PCR Clean-Up System (Promega Corporation, Madison, Wisconsin, USA) de acuerdo a las indicaciones del fabricante.
CONSTRUCCIÓN DE LOS ADN RECOMBINANTES
Se empleó T4 ADN ligasa para ligar los fragmentos de ADNc amplificados y pGEM®-T Easy. La mezcla de reacción se realizó en una relación molar plásmido-ADNc (1:3). El tampón de las reacciones estuvo constituido por Tris-HCl 30 mM (pH 7,8), MgCl2 10 mM, DTT 10 mM, ATP 1 mM. La incubación se realizó a 4 °C durante 12 h.
TRANSFORMACIÓN DE CÉLULAS COMPETENTES DE E. coli XL1BLUE MRF` CON EL PLÁSMIDO RECOMBINANTE pGEM®-T Easy
Las mezclas de ligación fueron añadidas a 200 µL de células competentes para transformarlas mediante choque térmico (30 min 4 °C, 90 seg 42 °C, 2 min 4 °C). A cada tubo se agregaron 800 µL de medio LB precalentado e incubados a 37 °C, 220 rpm durante 1 hora. Con espátula de Drigalski se sembraron 200 µL de las preparaciones en placas con agar LB-Ampicilina 100 µg/mL pretratadas (X-Gal 20 mg/mL,IPTG 1 M,LB líquido). Se incubaron a 37 °C durante 12 h. Se observaron colonias azules y blancas.
IDENTIFICACIÓN DE LAS COLONIAS RECOMBINANTES PCR DE COLONIAS
Las colonias blancas seleccionadas fueron colocadas en 50 µL de mezclas de reacción para PCR [cebadores (0,2 mM), MgCl2 (1 mM), dNTP´s (0,2 mM), Taq polimerasa (GoTaq® Flexi, Promega) (1,25 U) y tampón de reacción (GoTaq® Flexi Buffer colorless)]. Los cebadores correspondieron a lo indicado (Tabla 1) de acuerdo a la región genómica a amplificar. Las condiciones de tiempo y temperatura fueron 94° C por 5 min, 30 ciclos (94 °C x 30 seg, 50° C x 30 seg, 72 °C x 2 min) y 72° C por 7 min. Se incluyó una colonia azul como control negativo. El resultado de la amplificación se verificó en gel de agarosa (2%) donde se incluyó el marcador de tamaño molecular 100 bp DNA step ladder (Axygen).
EXTRACCIÓN DEL ADN PLASMÍDICO
Verificada la recombinación de los insertos en pGEM®-T Easy se cultivaron las células transformadas en medio LB con ampicilina (100 µg/mL) para extraer el ADN plasmídico (ADNp). Los cultivos fueron centrifugados, el sedimento resuspendido en EDTA (10 mM), Tris-HCl (50 mM, pH: 8) y RNAsa (100 µg/mL) e incubadas a 4 ºC por 15 min. Se adicionó NaOH (200 mM), SDS (1%) y se mantuvieron a temperatura ambiente por 5 min, se agregó acetato de potasio (3 M, pH: 5), la suspensión se mantuvo a 4 ºC por 5 min. Posteriormente, se centrifugaron y precipitaron los ADNp con isopropanol y acetato de potasio (3 M, pH: 5,5). Finalmente, la suspensión se centrifugó durante 30 min a 14 000 rpm, al sedimento se agregó etanol 70% para su centrifugación. Cada ADNp obtenido se resuspendió en agua libre de nucleasas y conservado a -20 ºC.
VERIFICACIÓN DE CLONACIÓN A PARTIR DE LOS PLASMIDOS RECOMBINANTES
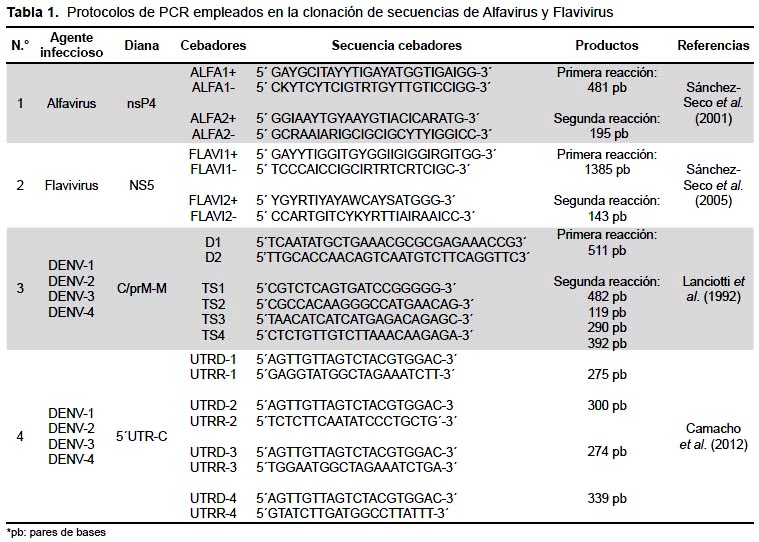
Los plásmidos recombinantes fueron analizados mediante técnicas moleculares hasta la obtención de los productos finales (Tabla 1). Los productos se analizaron en geles de agarosa.
RESULTADOS
Se obtuvieron los plásmidos recombinantes con las secuencias especificadas para flavivirus y alfavirus. Una vez amplificados, purificados y confirmada la amplificación de los productos de ADNc, se obtuvo un rendimiento entre 1,5 y 12 µg totales, con una relación A260/280 de 1,9 indicando pureza de los productos.
Luego de la recombinación y siembra de las células transformadas, se observó el crecimiento de las colonias bacterianas, sobre las cuales se realizaron los ensayos para verificar la clonación de los fragmentos de ADNc. Una vez identificadas las colonias mediante su color, se amplificaron los fragmentos de ADNc recombinados mediante una PCR de colonias donde se utilizaron los mismos cebadores empleados para la amplificación inicial de los fragmentos. Los resultados de este último ensayo evidenciaron amplificación de bandas correspondientes a los tamaños esperados para nsP4 de CHIKV (481 pb), Zika (1385 pb), C/prM-M de DENV-1, DENV-2, DENV-3 y DENV-4 (511pb) y 5´UTR-C [DENV-1 (275 pb), DENV-2 (300 pb), DENV-3 (274 pb) y DENV-4 (339 pb)] demostrando la clonación de los insertos. Adicionalmente, se extrajeron los ADNp obtenidos a partir del cultivo de las bacterias transformadas para el almacenamiento y aseguramiento de la clonación. El análisis mediante PCR de los ADNp obtenidos permitió amplificar los productos finales mediante técnicas moleculares (Tabla 1) de los genomas de los virus CHIKV, Zika, DENV-1, DENV-2, DENV-3 y DENV-4 clonados en pGEM®-T Easy (Figura 1). Los clones recombinantes serán posteriormente valorados como controles positivos, en cualquiera de las técnicas mencionadas (Tabla 1).

DISCUSIÓN
Las técnicas moleculares representan un gran avance para los laboratorios de diagnóstico e investigación. La aplicación de estas, como la PCR en sus múltiples variantes, identifica agentes infecciosos mediante amplificación de determinados fragmentos genómicos (13-16). En el estado Aragua (Venezuela), funciona el Laboratorio Regional de Diagnóstico e Investigación del dengue y otras Enfermedades Virales, donde se realiza diagnóstico molecular de alfavirus (13) y flavivirus (14); DENV (15), así como del fragmento 5´UTR-C de DENV, utilizado para síntesis de proteínas de DENV (16). Con la puesta en marcha de estas metodologías, surgió la necesidad de preparar controles positivos, comúnmente obtenidos mediante cultivos celulares, cuyas desventajas se asocian a laboriosidad, costos y riesgos de infección.
La tecnología de ADN recombinante permite solventar estas limitaciones con la clonación de secuencias diana amplificadas mediante RT-PCR (13-16). Estas, fueron insertadas en pGEM®-T Easy, representando una estrategia de clonación sencilla, comprobada mediante PCR de colonias y PCR de ADNp, inédita en el diagnóstico de estas infecciones virales, asegurando un posible banco de controles positivos permanente, evitando la dependencia tecnológica de otros países para la adquisición de controles y minimizando la ejecución de procedimientos laboriosos para obtener cepas virales como controles, mediante cultivos celulares y/o inoculación en ratones lactantes (19).
Los resultados obtenidos sugieren que el uso de técnicas de ADN recombinante, podrían favorecer la implementación de estrategias para preparar controles positivos para validación de técnicas moleculares. Estos serán posteriormente empleados en el laboratorio para valorar su aplicabilidad en el análisis de muestras obtenidas de pacientes con sospecha clínica de infección por miembros de los géneros alfavirus y flavivirus, abriendo además la oportunidad de que otros laboratorios de la región puedan preparar sus propios controles.
Agradecimientos: al Instituto "Ricardo Jorge" de Aguas de Moura (Portugal) por la gentil donación de la cepa de referencia del virus Zika MR766 (Uganda, 1947, N° acceso: AY632545) y al Instituto de Salud Carlos III (España) por la gentil donación del control de CHIKV (genotipo ECSA , cepa DRC 1721) a través de VIRORED-CYTED.
Contribuciones de autoría: DC, EF y JR participaron en la concepción y diseño del artículo, recolección, obtención de resultados, análisis e interpretación de datos, redacción del artículo, aprobación de su versión final y obtención de financiamiento. LF participó en el análisis e interpretación de datos, redacción del artículo, revisión crítica del artículo, aprobación de su versión final y aporte de pacientes o material de estudio. GC participó en el análisis e interpretación de datos, redacción del artículo, revisión crítica del artículo y la aprobación de su versión final.
Fuentes de financiamiento: Ayuda Menor CDCH-UC 0440-10, FONACIT (Misión Ciencia. Incentivo Institucional Proyecto: UC-200900434).
Conflictos de Interés: Los autores declaran no tener conflictos de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. Gubler DJ, Kuno G, Markoff L. Flaviviruses. En: Knipe DM, Howley PM, eds. Fields Virology. 5th ed. Philadelphia: Lippincott Williams and Wilkins; 2007.p. 1153–1252. [ Links ]
2. Strauss JH, Strauss EG. The alphaviruses: gene expression, replication and evolution. Microbiol Rev. 1994; 58(3):491-562. [ Links ]
3. Organización Panamericana de la Salud. Organización Mundial de la Salud. Guías para el diagnóstico, tratamiento, prevención y control. La Paz: OPS/OMS; 2009. [ Links ]
4. paho.org [internet]. Washington, D.C.: Organización Panamericana de la Salud. Organización Mundial de la Salud. [actualizado el 7 de abril de 2016; citado el 17 de marzo de 2014]. Disponible en: http://www.paho.org/hq/index.php?option=com_content&view=article&id=264&Itemid=363.
5. Monath TP. Yellow fever: an update. Lancet Infect Dis. 2001;1(1):11-20. [ Links ]
6. Briese T, Jia XY, Huang C, Grady LJ, Lipkin WI. Identification of a Kunjin/ West Nile-like flavivirus in brains of patients with New York encephalitis. Lancet. 1999; 354(9186):1261–1262. [ Links ]
7. Campos GS, Bandeira AC, Sardi SI. Zika virus outbreak, Bahia, Brazil. Emerg Infect Dis; 2015;21(10):1885-6. doi: 10.3201/eid2110.150847. [ Links ]
8. Organización Panamericana de la Salud/ Organización Mundial de la Salud [Internet]. Alerta epidemiológica. Infección por virus Zika. Washington, D.C.: OPS/OMS; 2015 [citando el 21 de mayo de 2015] Disponible en: http://www.paho.org/hq/index.php?option=com_docman&task=doc_view&Itemid=270&gid=30076&lang=es.
9. Dengue hemorrhagic fever in Venezuela. Epidemiol Bull. 1990; 11(2):7-9. [ Links ]
10. Uzcátegui NY, Comach G, Camacho D, Salcedo M, Cabello de Quintana M, Jimenez M, et al. Molecular epidemiology of dengue virus type 3 in Venezuela. J Gen Virol. 2003;84:1569-75. doi: 10.1099/vir.0.18807-0. [ Links ]
11. Van Bortel W, Dorleans F, Rosine J, Blateau A, Rousseau D, Matheus S, et al. Chikungunya outbreak in the Caribbean region, December 2013 to March 2014, and the significance for Europe. Euro Surveill. 2014;19(13). pii:20759. [ Links ]
12. Ministerio del Poder Popular para la Salud (MPPS). Información oficial de los casos de Chikungunya en Venezuela [internet]. Caracas: MPPS; 2014 [Citado 12 de noviembre de 2014] [ Links ] Disponible en: http://www.mpps.gob.ve/index.php?option=com_content&view=article&id=6645&Itemid=18.
13. Sánchez-Seco MP, Rosario D, Quiroz E, Guzmán G, Tenorio A. A generic nested-RT-PCR followed by sequencing for detection and identification of members of the alphavirus genus. J Virol Methods. 2001; 95(1-2):153-61. [ Links ]
14. Sánchez-Seco MP, Rosario D, Domingo C, Hernández L, Valdés K, Guzmán MG, et al. Generic RT-nested-PCR for detection of fllaviviruses using degenerated primers and internal control followed by sequencing for specifiic identifiication. J Virol Methods. 2005;126(1-2):101–9. doi:10.1016/j.jviromet.2005.01.025. [ Links ]
15. Lanciotti RS, Calisher CH, Gubler DJ, Chang GJ, Vorndam AV. Rapid detection and typing of dengue viruses from clinical samples by using reverse transcriptase-polymerase chain reaction. J Clin Microbiol 1992;30(3):545–51. [ Links ]
16. Camacho DE, Ferrer E, Triana-Alonso JL, Ferreras AC, Graterol H, Comach G, et al. Amplificación de la región 5 UTR-C del genoma de los cuatro serotipos de Virus Dengue. Salus. 2012;16(3):40-4. Disponible en: http://www.redalyc.org/articulo.oa?id=375939022009. [ Links ]
17. Sambrook J, Russel DW. Molecular Cloning: a laboratory manual. 3a ed. New York: Cold Spring Harbor Laboratory Press; 2001. [ Links ]
18. Ogden RC, Adams DA. Electrophoresis in agarose y acrylamide gels. Methods Enzymol 1987;152:61-87. [ Links ]
19. Guzmán MG, Kourí G. Advances in dengue diagnosis. Clin Diagn Lab Immunol. 1996;3(6):621-7. [ Links ]
Correspondencia:
Daría Elena Camacho.
Dirección: Laboratorio Regional de Diagnóstico e Investigación del Dengue y otras Enfermedades Virales,
Instituto de Investigaciones Biomédicas de la Universidad de Carabobo (LARDIDEV/ BIOMED-UC), Maracay, Venezuela.
Teléfono: +58-243-2425822.
Correo electrónico: darycamacho@yahoo.com
Recibido: 04/07/2015
Aprobado: 06/04/2016













