Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.34 no.3 Lima jul./set. 2017
http://dx.doi.org/10.17843/rpmesp.2017.343.2529
TRABAJO ORIGINAL
Caracterización molecular de bacterias patógenas de las vías respiratorias de pacientes peruanos con fibrosis quística
Molecular characterization of pathogenic bacteria of the respiratory tract in peruvian patients with cystic fibrosis
Ruth Aquino1,2,a, Emely Gonzáles1,b, Sol Samaniego3,c, Juan Rivera4,d, Virna Cedeño5,e, Yrene Urbina5,f, Benoit Diringer2,g
1 Laboratorio de Biotecnología Molecular, Universidad Nacional de Tumbes. Tumbes, Perú.
2 Empresa de Investigación y Capacitación en Biotecnología (Inca’Biotec S.A.C.). Tumbes, Perú.
3 Asociación de Padres de Niños con Fibrosis Quística (Fiqui-Perú). Lima, Perú.
4 Instituto Nacional de Salud del Niño. Lima, Perú.
5 Universidad Nacional de Tumbes. Tumbes, Perú.
a Bióloga, Maestra en Ciencias con mención en Biotecnología molecular; b bachiller en Biotecnología, Maestra en Ciencias con mención en Biotecnología molecular.; c abogada; d médico pediatra, Gastroenterólogo; e Ph.D. Genética y patología; f doctora en enfermería; g maestro en Ciencias.
RESUMEN
Objetivos. Caracterizar a nivel molecular las bacterias patógenas de las vías respiratorias de pacientes peruanos con fibrosis quística (FQ). Materiales y métodos. Se caracterizaron las comunidades bacterianas cultivables a partir de muestras de esputo de pacientes pediátricos y adultos con FQ registrados en el Hospital Nacional Edgardo Rebagliati Martins y el Instituto Nacional de Salud del Niño (INSN). Para el cultivo bacteriano se utilizaron técnicas microbiológicas estándares, y para la caracterización molecular la secuenciación del gen ARNr 16S y espectrometría de masas de tipo desorción/ionización con láser asistido por matriz con tiempo de vuelo (MALDI TOF) y MALDI TOF/TOF. Resultados. Por secuenciación del ARNr 16S se identificaron 127 cepas, encontrando las bacterias patógenas Pseudomonas aeruginosa (31,5%), Staphylococcus aureus (12,6%), Pseudomonas spp. (11,8%), Klebsiella oxytoca (3,1%), otras especies mostraron baja prevalencia. El análisis por MALDI TOF permitió obtener una serie de espectros representativos de cada especie aislada, mientras que el análisis por MALDI TOF/TOF reveló péptidos y proteínas de las especies más comunes con informaciones complementarias que revelarían datos sobre su patogenicidad o sensibilidad a antibióticos. Conclusiones. Los principales microorganismos patógenos encontrados en las vías respiratorias son similares a los reportados en otros países. Estos son los primeros hallazgos en Perú que muestran la caracterización bacteriana por secuenciación del ARNr 16S, por MALDI TOF y MALDI TOF TOF. Los hallazgos permiten la identificación bacteriana de microorganismos nativos relacionados con la FQ basada en el análisis de su proteoma.
Palabras clave: Fibrosis quística; Infecciones respiratorias; Proteoma (Fuente: DeCS, BIREME)
ABSTRACT
Objectives. To molecularly characterize the pathogenic bacteria of the respiratory tract isolated from patients with cystic fibrosis (CF) in Peru. Materials and methods. Bacterial communities cultured from sputum samples of pediatric and adult patients with CF admitted to the Edgardo Rebagliati Martins National Hospital and the National Institute of Child Health were characterized. Standard microbiological techniques were used for bacterial culture, and gene sequencing of 16S rRNA and matrix-assisted laser desorption ionization time-of-flight (MALDI-TOF) mass spectrometry and tandem MALDI-TOF mass spectrometry (MALDITOF/TOF) were used for molecular characterization. Results. Seventeen bacterial strains were characterized by 16S rRNA sequencing, and the identified pathogenic bacteria were Pseudomonas aeruginosa (31.5%), Staphylococcus aureus (12.6%), Pseudomonas spp. (11.8%), and Klebsiella oxytoca (3.1%). MALDI-TOF analysis generated a series of spectra representative of each isolated bacterial species, whereas MALDI TOF/TOF analysis identified the peptides and proteins of the most common strains and provided data on pathogenicity and sensitivity to antibiotics. Conclusions. The primary pathogenic microorganisms found in the respiratory tract of patients with CF in Peru were the same as those found in other countries. This study is the first to perform 16S rRNA sequencing as well as MALDI-TOF and MALDI-TOF/TOF analysis of the bacterial pathogens circulating in Peru. The inclusion of proteomic analysis further allowed for the identification of native microorganisms involved in CF.
Key words: Cystic fibrosis; Respiratory infections; Proteome (Source: MeSH, NLM)
INTRODUCCIÓN
La fibrosis quística (FQ) es una enfermedad autosómica recesiva potencialmente mortal, que afecta alrededor de 70 000 personas en todo el mundo. Es causada por mutaciones en el gen de la conductancia transmembrana reguladora de fibrosis quística (CFTR). Las mutaciones en el CFTR reducen el volumen del líquido de la superficie de las vías respiratorias, disminuyendo de este modo el aclaramiento mucociliar de los patógenos bacterianos, lo que causa inflamaciones crónicas (1).
Las infecciones pulmonares crónicas son la principal causa de muerte en la FQ. Las vías respiratorias son colonizadas por bacterias desde una edad temprana. Muchas de las bacterias aisladas del esputo de FQ son microorganismos ambientales comunes que pueden llegar a ser patógenos, afectan primero las vías respiratorias superiores antes de colonizar todo el sistema respiratorio. Entre los patógenos cultivables más importantes, se incluyen Pseudomonas aeruginosa, Staphylococcus aureus y Burkholderia cepacia complex (BCC). En los últimos años, también se ha reportado un número creciente de patógenos emergentes como Achromobacter y Stenotrophomonas (2).
Varios estudios muestran que la composición de la microbiota pulmonar evoluciona con la edad. Mientras que los niños pequeños presentan una importante diversidad microbiana y se infectan comúnmente con S. aureus y Haemophilus influenzae, a medida que van creciendo se contaminan con bacterias más problemáticas como P. aeruginosa y S. aureus resistentes a la meticilina (MRSA) (2). Finalmente P. aeruginosa termina dominando la microbiota pulmonar habiéndose reportado muchas cepas epidémicas específicas a FQ, por lo que se requiere una adecuada identificación molecular.
La identificación temprana de patógenos en las vías respiratorias inferiores es prioritaria para prevenir eventos de infecciones crónicas (exacerbaciones) responsables de potentes respuestas inmunitarias que conducen a una rápida degradación de los pulmones. Tradicionalmente, el estudio de la taxonomía microbiana ha estado basado en técnicas de cultivo clásico, medios selectivos, coloración de Gram y pruebas fisicoquímicas.
Si bien, en su momento estas técnicas fueron importantes, tienen limitaciones en la identificación de bacterias patógenas, que incluyen: tiempo necesario para el crecimiento bacteriano, la interferencia por los antibióticos, el crecimiento excesivo de otros microorganismos, además de la extraordinaria capacidad de modulación genética de las bacterias gracias a fenómenos de mutación, recombinación, intercambios de genes, como lo ilustra la creciente resistencia a antibióticos de numerosas cepas microbianas.

Estos complejos fenómenos hacen que dos bacterias morfológicamente pertenecientes a la misma especie demuestren comportamientos distintos en términos de virulencia y resistencia, conduciendo a relativizar la noción de especies en estos microorganismos. Las técnicas moleculares superaron algunas de estas limitaciones, con la posibilidad de detectar genes específicos, secuenciar genomas completos o detectar bacterias no cultivables. En la actualidad, el análisis de la secuencia del gen ARNr 16S se ha impuesto como la técnica de referencia o gold standard para la taxonomía microbiana (3) con el potencial de proporcionar las ventajas de la orientación temprana para la terapia con antibióticos y el tratamiento de infecciones.
Otras tecnologías de vanguardia, dirigidas a la identificación bacteriana, tales como la Espectrometría de masas desorción/ionización láser asistida por matriz con tiempo de vuelo (MALDI TOF MS), está desplazando la taxonomía clásica y la secuenciación (4). MALDI TOF es considerado como una herramienta potencial en términos de rapidez (identificación de una cepa en 3 segundos a 2 horas), disminución del costo operativo y simplicidad.
La identificación por MALDI TOF se basa en el análisis espectral de proteínas bacterianas, principalmente proteínas ribosómicas, ionizadas por irradiación láser de la célula bacteriana. La identificación se determina comparando el espectro de microorganismos investigados con una base de datos de referencia.
El MALDI TOF es una herramienta eficaz para la identificación microbiana en comparación con otras técnicas. Además, permite la caracterización de microorganismos que son difíciles de identificar usando métodos de rutina (5). Asimismo, la técnica MALDI TOF TOF permite la identificación de proteínas relacionadas a patógenos bacterianos, lo cual conduciría a tener información acerca de ciertas características moleculares que hacen a los microrganismos únicos, y permitiría la identificación de dianas involucrados en la biogénesis de biopelículas, quorum sensing, resistencia a los antibióticos, estrés oxidativo y adaptación al medio pulmonar de la FQ. Por lo tanto, esta técnica permite comprender la patogénesis bacteriana de la FQ (6).
En el Perú, la FQ es una enfermedad subdiagnosticada, debido al desconocimiento de la enfermedad y a la escasez de centros de detección y de estudios a nivel molecular, lo que conduce a diagnósticos tardíos y un tratamiento deficiente. En este contexto, el objetivo de esta investigación fue identificar y caracterizar la microbiota cultivable presente en las vías respiratorias inferiores de pacientes peruanos con FQ lo que permitiría conocer mejor la microbiota relacionada con la FQ en el Perú y orientar tratamientos adecuados. La composición de la microbiota de muestras de esputo se investigó basándose en la secuenciación del gen ARNr 16S y análisis proteómicos por espectrometría de masas MALDI TOF y MALDI TOF TOF.
MATERIALES Y MÉTODOS
DISEÑO Y POBLACIÓN DE ESTUDIO
Se realizó un estudio de tipo descriptivo. Se incluyeron pacientes con FQ registrados en el INSN y en el hospital Edgardo Rebagliati Martins del seguro social de salud (EsSalud), que han sido diagnosticados positivos para la prueba de sudor (> 60 mEq/L) o con diagnóstico molecular de sus mutaciones (7).
COLECCIÓN DE LA MUESTRA
Se colectó una muestra de esputo (2 a 4 mL) por paciente, estas se conservaron a 4 °C hasta su procesamiento (1 a 4 h). En algunos pacientes que no sabían expectorar se realizó un hisopado faríngeo. La toma de muestra se realizó en ayuno y después de un lavado bucal. La calidad de las muestras fue evaluada mediante un examen citológico y fue clasificada de acuerdo al número de células epiteliales y leucocitos polimorfonucleares. Las muestras se clasificaron en tres categorías de calidad: mala calidad, cuando las células epiteliales (células / campo) ≥ 25 y leucocitos (células / campo) ≤ 10; calidad apropiada, células epiteliales ≤ 25 y los leucocitos (células / campo) ≥ 10. Las otras combinaciones se consideraron de calidad moderada. Para este estudio se utilizaron las muestras clasificadas como de calidad apropiada y moderada (Euro Care CF y CF UK trust) (8).
CULTIVO MICROBIOLÓGICO ESTÁNDAR
Todos los protocolos de microbiología siguieron las recomendaciones de las fundaciones europeas de FQ (Euro Care CF y CF UK trust) (8). Se vertió 10 µl de la muestra sobre placas de diferentes medios selectivos; agar Sangre, agar Cetrimide, agar MacConkey y agar Burkholderia Cepacia Complex (BCC). Las placas de agar sangre se incubaron anaeróbicamente a 37 °C (5% de CO2) utilizando el sistema GasPakTM. Las placas de agar Centrimide, MacConkey y BCC se incubaron en condiciones aeróbicas a 37 ºC por 24 a 48 horas con una vigilancia de hasta tres días adicionales en caso de ausencia de crecimiento, con excepción del agar BCC que se incubó durante cinco días.
EXTRACCIÓN DE ADN Y SECUENCIACIÓN DEL GEN ARNr 16S
El ADN fue extraído a partir de cultivos puros y se siguieron las recomendaciones del proveedor (kit de extracción de ADN Concepto Azul). El ADN se amplificó mediante la reacción en cadena de la polimerasa (PCR) utilizando el kit Gold® de Taq DNA Polimerase Recombinante (Thermo Scientific), en un volumen total de 25 µLque contenía 0,6 pmol/µLde primer 27F Forward (AGAGTTTGATCMTGGCTCAG) y 0,6 pmol/µL de primer 1492R Reverse (GGYTACCTTGTTAGGACTT), 1X buffer de polimerasa, 0,2 Mm de dNTPs, 2 Mm de MgCl2, 1 U/µL de Taq polimerasa y 20 mg de ADN extraído. Las condiciones térmicas de amplificación fueron: desnaturalización inicial a 95 °C durante 5 min (1 ciclo) seguido de 35 ciclos de desnaturalización a 95 °C durante 30 s, hibridación a 58 °C durante 45 s, y extensión a 72 °C durante 90 s, con una etapa de extensión final a 72 °C durante 6 min, y se llevaron a cabo en un termociclador (Biometra-USA).
Los productos de amplificación de aproximadamente 1400 pb se separaron por electroforesis horizontal en gel de agarosa al 1,5% en una cámara electroforética (Cleaver scientific-Reino Unido) siendo observadas en un transiluminador (Vilber lourmat-Francia). Las muestras positivas fueron enviadas a la compañía Macrogen-USA, para ser secuenciadas con los primers 16SrRNAF518 (CCAGCAGCCGCGGTAATACG) y 16SrRNAR800 (TACCAGGGTATCTAATCC). Las secuencias de ARNr 16S fueron reconstruidas utilizando el programa Mega6 (9) y analizadas con BLAST (10). Basic Local Alignment Search Tool) de NCBI (National Center for Biotechnology Information). La clasificación de la especie se atribuyó únicamente a secuencias que presentaron un 99 a 100% de concordancia con la base de datos.
ESPECTROMETRÍA DE MASAS MALDI TOF
Las cepas puras fueron cultivadas en medio Trypticase Soy Broth (TSB) durante 24 horas a 37 °C. Después se centrifugó a 10000 rpm durante 10 min, se eliminó el sobrenadante, se le agregó 500 µL de agua grado cromatografía líquida de alta resolución (HPLC), se homogenizó en un vortex durante 30 s (Vortex MixerUSA) y se volvió a centrifugar 10000 rpm durante 10 min, se eliminó el sobrenadante, luego se resuspendió con 50 µL de agua HPLC. Para 1 μL de muestra, se le adicionó 1 µL de matriz ácido sinápico y se colocó en forma de microgotas a la placa MALDI. Cada muestra fue evaluada por duplicado. Las muestras fueron analizadas en un espectrómetro de masa MALDI AB SCIEX TOF TOF TM 5800, en un rango de masas de 2000 – 20000 Da. La lectura se hizo en un modo operacional linear positivo.
ESPECTROMETRÍA DE MASAS MALDI TOF TOF
EXTRACCIÓN DE PROTEÍNAS BACTERIANAS
Se realizó la extracción de proteínas totales con el kit de extracción de proteínas (Qproteome Bacterial Protein) de Qiagen®. Previamente, el cultivo bacteriano se incubó a 24 horas y se midió la densidad óptica a 600 nm con el espectrofotómetro (Eppendorf®). Se centrifugó a 10 000 rpm durante 10 min, se eliminó el sobrenadante y se incubó a -20 °C durante 30 min; se agregó 100 µL de buffer de lisis (1 mL de buffer nativo, 10 µL de lisozima, 1 µL de benzonasa/ nucleasa), se homogenizó durante 30 s y se sonicó durante 2 min con un sonicador (40 KHz) (Ultrasonic Cleaner-Korea). Luego se incubó a -20 °C durante 30 min con homogenización cada cierto tiempo, se volvió a vortexear durante 30 s y se sónico durante 5 min; finalmente, se centrifugó a 16 000 rpm durante 30 min, y se recuperó 80 µL de sobrenadante.
PREPARACIÓN DEL GEL SDS-PAGE 1D
Se preparó un gel al 12 % de separación, con un volumen final de 15 mL, como se describe en Lomonte (11) .
DIGESTIÓN DE PROTEÍNAS
Las bandas de proteínas fueron cortadas, y se colocaron en 50 µL de bicarbonato de amonio. Los siguientes pasos fueron realizados como se describe en Shevchenko et al. (12).
ANÁLISIS DE DATOS
Se utilizó el software Protein pilot 4.0 con bases de datos específicas para cada microorganismo. La identificación de proteínas se realizó con Protein blast.
CONSIDERACIONES ÉTICAS
La aprobación ética para este estudio fue otorgada por el Instituto Nacional de Salud del Niño (INSN) del Ministerio de Salud (MINSA). Una carta de consentimiento fue firmada por los padres de los pacientes después de haber sido informados del proyecto y de acuerdo con las normas éticas institucionales y nacionales.
RESULTADOS
Se evaluaron un total de 21 pacientes (42,9%) de los 49 pacientes reportados con FQ en el momento del estudio (7). La edad promedio fue de 7 años 7 meses con un rango de edades entre los 20 meses y los 20 años de edad, siendo este último paciente el único no pediátrico. La procedencia de los pacientes correspondió a siete ciudades del Perú: 14 pacientes de Lima (66,7%); 2 pacientes de Ica (9,5%) y otros 5 pacientes de Callao, Cusco, Tacna, Tumbes y Arequipa (4,7%).
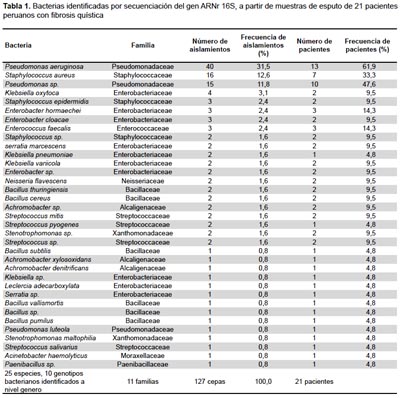
Se aislaron 127 cepas de 21 pacientes peruanos con FQ. El análisis de secuencias del gen ARNr 16S permitió identificar bacterias a diferentes niveles taxonómicos lo que incluyó 11 familias, 25 especies y 10 identificaciones a nivel de género. La familia Pseudomonadaceae presentó el mayor número de cepas (56), seguido por Enterobacteriaceae (21), Staphylococcaceae (21) y Bacillaceae (8). En términos de diversidad, la familia Enterobacteriacae presentó 10 especies, seguida por Bacillaceae (6) y Streptococcaceae (4). A nivel de cepas, las bacterias cultivables más aisladas fueron P. aeruginosa (40), S. aureus (16), Pseudomonas sp (15), Klebsiella oxytoca (4). Otros microorganismos que incluían a familias como Enterobacteriaceae y Bacillaceae se presentaron en menores cantidades (Tabla 1).
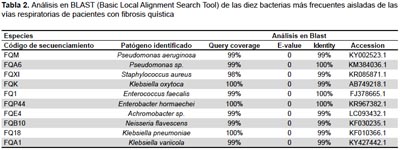
Las cepas caracterizadas en este estudio y que fueron identificadas hasta el nivel de especie (Tabla 2), tienen sus secuencias registradas y disponibles en la base de datos mundial NCBI con los números de accesión desde el MF144432 al MF144546.
La población de pacientes estaría principalmente colonizada por P. aeruginosa, seguida por Pseudomonas sp, S. aureus, E. hormaechei, E. faecalis, que fueron encontrados en 13, 10, 7 y 3 pacientes, respectivamente. Los patógenos más asociados a la degradación pulmonar fueron P. aeruginosa, S. aureus, y Stenotrophomonas maltophilia, entre otros (Tabla 1). Varios pacientes contaban con colonizaciones múltiples de tipo P. aeruginosa/P. spp o P. aeruginosa/S. spp./S. aureus P. aeruginosa/S. maltophilia (datos no presentados).
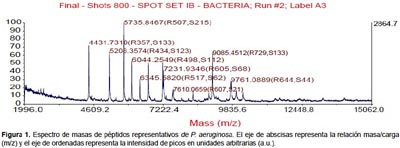
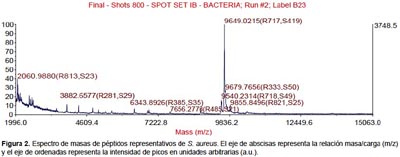
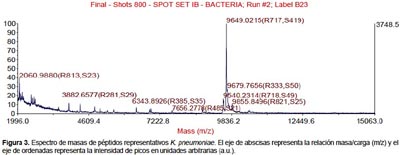
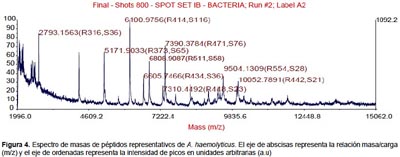
Todas las especies aisladas y caracterizadas por ARNr 16S fueron analizados por MALDI-TOF. Se obtuvieron los espectros de masas de las bacterias más representativas encontradas en pacientes peruanos con FQ (Figura 1, 2, 3, 4).
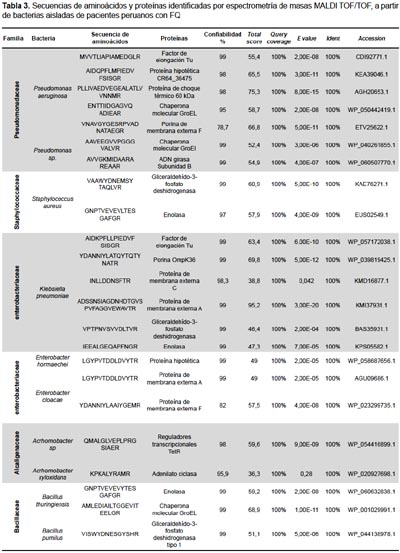
También se realizaron los análisis proteómicos por espectrometría de masas MALDI TOF/TOF obteniéndose la diversidad de proteínas de las bacterias más representativas encontradas en este estudio (Tabla 3).
DISCUSIÓN
La bacteria predominante en este estudio fue P. aeruginosa (31,5%), la cual se encontró en 61,9% de todos los pacientes analizados correspondiente al 57,1% de los pacientes pediátricos. Esta bacteria es reportada frecuentemente como uno de los patógenos oportunistas más letales en FQ, con frecuencias de hasta el 80% en pacientes pediátricos (13). Esta bacteria se caracteriza por el incremento de su frecuencia a medida que aumenta la edad (14), siendo considerada como la presunta causante de la mayoría de las exacerbaciones pulmonares y, por lo tanto, el principal objetivo del tratamiento antibiótico en los pacientes con FQ. Además, se la ha asociado con una disminución más rápida de la función pulmonar, y de la ineficiencia de las antibioterapias, especialmente cuando el fenotipo mucoide está presente (15).
S. aureus fue el segundo patógeno más frecuente (12,6% de los aislados) en la población estudiada; también es reportado como uno de los patógenos más virulentos y predominante en pacientes con FQ (16). Dentro de los S. aureus, existen varios fenotipos como los MRSA (Methicillin-resistant S. aureus) antiguamente conocidos como SCV (Small colony variant) que representan una amenaza para los pacientes infectados. El factor de virulencia se asocia a menudo con la resistencia a la meticilina, otorgada por una exoproteína llamada Panton Valentine leucocidina (PVL). Cepas de MRSA han sido asociadas con el desarrollo de infecciones pulmonares invasivas incluyendo absceso pulmonar en pacientes con FQ y capacidades de resistencias a un amplio espectro de diferentes antibióticos (17). Sin embargo, en este estudio no pudimos observar MRSA, ni fenotípicamente ni por proteómica.
Bacterias del género Achromobacter, también fueron aisladas. Varios estudios han reportado que las especies pertenecientes a este género son microorganismos ambientales cultivados a partir de una gama de hábitats naturales y son patógenos oportunistas que muestran una mayor resistencia a un amplio espectro de antibióticos, y que A. xylosoxidans es un patógeno emergente cada vez más aislado de muestras de pacientes con FQ (18).
En este estudio, una cepa de Stenotrophomonas maltophilia fue aislada a partir de la muestra de esputo de un paciente; S. maltophilia es altamente resistente a la mayoría de los antibióticos, con la excepción de trimetoprimasulfametoxazol (SXT). Se ha reportado la existencia de cepas de S. maltophilia resistentes a SXT sin poder elucidar el mecanismo molecular de la resistencia (19).
Los resultados de identificación y frecuencia obtenidos en este estudio concuerdan con trabajos reportados en poblaciones pediátricas a nivel mundial. Si bien, P. aeruginosa y S. aureus son reportadas como las principales cepas en pacientes con FQ, generalmente, las infecciones por S. aureus y H. influenzae son las que predominan en pacientes pediátricos de países del Norte (14,15) o en poblaciones más cercanas como Argentina (20) o Brasil (21). Sin embargo, en los pacientes con FQ de Perú, P. aeruginosa sería el patógeno predominante, tanto en pacientes pediátricos como en adultos. Este resultado es alarmante cuando se considera que cuanto más temprana sea la edad de adquisición de este patógeno, más se incrementan los indicadores de morbilidad y mortalidad.
La presente investigación corrobora los resultados obtenidos anteriormente en Perú por Torres (22) quien determinó que P. aeruginosa estaba presente en el 81% de muestras de esputo analizadas, seguido por S. aureus y Streptococcus pneumoniae; asimismo, Castilla (23) identificó que P. aeruginosa y S. aureus fueron los primeros patógenos que colonizaron las vías respiratorias de la FQ en 35 y 25% de casos, respectivamente. Castilla también analizó que P. aeruginosa estaba presente en el 72% de los casos de muerte. Sin embargo, estos autores trabajaron con métodos simples de cultivo donde varios tipos de bacilos Gram-negativos pueden ser ambiguamente clasificados como P. aeruginosa.
Es importante resaltar que los medios utilizados en microbiología clásica pueden inducir errores de diagnóstico. En este estudio, 16 de las 56 cepas (28,6%) de Pseudomonas aisladas a partir de medio "selectivos" no pertenecían a P. aeruginosa, lo que evidencia la utilidad de realizar una caracterización profunda. De forma similar, el análisis de las secuencias de ARNr 16S obtenidas de colonias aisladas del medio específico BCC descartó el diagnostico presuntivo de este patógeno en los pacientes analizados.
Los resultados de caracterización molecular permitieron confirmar la ausencia de S. pneumoniae y BCC que completan la lista de las principales especies patógenas clásicamente reportadas en FQ (14). La presencia de H. influenzae, considerada problemática a altas concentraciones, no fue evaluada en este estudio debido a sus requerimientos particulares de cultivo (8).
En nuestro estudio, varios pacientes presentaron colonización múltiple (datos no presentados). La presencia de múltiples patógenos en un solo paciente ha sido frecuentemente evidenciada, debido a que estos patógenos son bacterias oportunistas ubicuas presentes en abundancia en suelos, aguas, son comensales de piel o boca o animales domésticos. Estas cepas "silvestres" terminan adaptándose a las condiciones de los pulmones y se vuelven gradualmente resistentes a los antibióticos (2).
El surgimiento de las técnicas de metagenómica permitieron la caracterización de la flora de pulmones sanos y a la identificación de una "microbiota núcleo" dominada por géneros como Streptococcus, Prevotella, Veillonella, Pseudomonas, Rothia, Haemophilus, Actinomyces y Neisseria (24). Estos análisis señalan que esta microbiota núcleo se encuentra también en los pacientes más jóvenes con FQ, pero que su diversidad disminuye a medida que envejecen debido a que son desplazadas progresivamente por P. aeruginosa u otras cepas colonizadoras y multirresistentes a antibióticos.
Una característica interesante en los aislamientos en pacientes peruanos con FQ y que no ha sido reportada anteriormente, es la presencia de varias cepas de Bacillus y Klebsiella que no son géneros muy comunes en la FQ. Si bien el origen de estas cepas queda por ser investigado, un estudio reciente confirmó mediante metagenómica que la microbiota de pulmones sanos puede albergar algunas especies de Bacilli, en particular Lactobacillus spp., que se encuentran comúnmente en la vía oral y en el tracto gastrointestinal (25). Este hallazgo refuerza el concepto de conectividad del sistema digestivo con el sistema respiratorio mediante las vías respiratorias superiores (boca, faringe y senos paranasales con los pulmones).
Además de la edad, la microbiota puede ser influenciada por la geografía. En uno de los pocos estudios para evaluar las comparaciones geográficas, muestras de esputo de 19 pacientes con FQ en un centro en Reino Unido fueron comparados con las muestras obtenidas de 19 pacientes de un hospital de Estados Unidos (26). Las diferencias en las especies entre los dos grupos mostraron diferencias significativas en la composición, abundancia y diversidad. En este estudio, pudimos analizar muestras de pacientes procedentes de ciudades con ecosistemas muy diferentes como Lima, Ica, Callao, Cusco, Tacna, Tumbes o Arequipa. Sin embargo, la mayoría de pacientes (76,2%) viven en Lima, lo que impide encontrar diferencias significativas entre pacientes de diferentes orígenes.
En el presente estudio se utilizó la tecnología MALDI TOF MS con el fin de obtener espectros de masas representativos de patógenos, además de su capacidad para obtener resultados rápidos, confiable y de bajo costo (27). Varios autores afirman que la "no identificación" de algunas cepas por MALDI-TOF MS se asocia con la limitada disponibilidad de base de datos de espectros.
Algunos estudios muestran que la identificación rutinaria de aislamientos de pacientes con FQ mejora mediante el uso de una base de datos comercial (28). Sin embargo, el equipo utilizado en este estudio no tiene tales bases de datos, lo que nos conllevó a construir nuestra propia biblioteca de datos aprovechando las cepas caracterizadas por ARNr 16S y que será utilizada en estudios posteriores. Los perfiles espectrales obtenidos con esta técnica, son muy diferentes y característicos entre las especies (Figura 1, 2, 3, 4). La implementación de MALDI-TOF MS para la identificación rutinaria de las bacterias proporciona información valiosa para el tratamiento antimicrobiano (29).
Por otro lado, la proteómica, basada en espectrometría de doble masa MALDI TOF/TOF (MS/MS), se está desarrollando rápidamente en investigaciones médicas, particularmente, comparando el proteoma de individuos sanos y enfermos (26). En este estudio utilizamos esta técnica con el objetivo de obtener péptidos a partir de bacterias e identificar proteínas. Los datos obtenidos mostraron altos porcentajes de confiabilidad de identificación (Tabla 3). Algunos fragmentos de las proteínas obtenidas están relacionados con patogenicidad o exacerbaciones causadas por bacterias de las vías respiratorias de los pacientes con FQ.
El perfil proteómico obtenido en este estudio para P. aeruginosa fue variable y con una gama de secuencias peptídicas con diversas funciones metabólicas, que incluyeron factor de elongación Tu; proteína hipotética CR64 36475; proteína de choque térmico 60 KDa; chaperona molecular GroEL; porinas de membrana externa F (proteínas asociadas a membrana); proteínas de choque térmico, y proteínas hipotéticas (Tabla 3). Esto coincide con un estudio previo realizado en Australia (30) que evaluó por MALDI TOF TOF el perfil proteómico de una cepa agresiva de P. aeruginosa aislada de pacientes con FQ, y cuyas proteínas pudieron ser agrupadas en 1) proteínas asociadas a la membrana; 2) Proteínas de choque térmico/chaperonas; 3) proteínas hipotéticas, además de 4) Proteínas de estrés oxidativo que no fueron encontradas en este estudio.
La identificación por MALDI TOF TOF de algunas de las proteínas constituye una fuente de información interesante para caracterizar la patogenicidad o la sensibilidad a antibióticos y así guiar los tratamientos. Entre las secuencias encontradas por nuestro estudio, cabe resaltar la proteína GroEL que ha sido reportada en pacientes con infecciones pulmonares crónicas causadas por P. aeruginosa y que provoca una fuerte respuesta de anticuerpos (31).
En Pseudomonas sp. se identificó la proteína ADN girasa subunidad B, cuyo dominio N-terminal de unión a ATP ha sido detectado como un blanco terapéutico validado para el descubrimiento de fármacos antibacterianos(32). EnS. aureus se identificaron fragmentos de proteínas involucradas en la patogénesis de la enfermedad como la Gliceraldehído-3-fosfato deshidrogenasa (GAPDH); que tiene una actividad glicolítica y es responsable del mantenimiento del patógeno dentro del huésped (33), o la enolasa que es parte central en la producción de energía a través de la glucólisis y participa en los procesos que conducen a la infección (34).
En las especies de la familia Enterobacteraceae, como K. pneumoniae, se identificaron proteínas como el factor de elongación termoinestable (EF-Tu); la OmpK36; la proteína de membrana externa (Omp) OmpC, que están asociadas a factores de patogenicidad de Klebsiella. EF-Tu podría ser un factor potencial de patogenicidad de leucopenia causada por K. pneumoniae (35). OmpK36 se relaciona con la resistencia al antibiótico carbapenen (36) ya que su presencia indicaría sensibilidad a este antibiótico. También se encontró OmpA, cuya función es el mantenimiento de la integridad estructural celular y también contribuye a la capacidad de las bacterias Gram-negativas para invadir células de mamíferos en Enterobacter cloacae (37). Reguladores transcripcionales TetR fueron identificadas en Achromobacter sp. y están involucrados en el control transcripcional de una multitud de sistemas y vías, incluyendo resistencia a múltiples fármacos, producción de antibióticos, patogenicidad, quórum sensing, estrés osmótico, formación de biopelículas, citocinesis y diversas vías catabólicas (38).
El análisis por MALDI TOF TOF de A. xylosidans también permitió encontrar un fragmento de adenilato ciclasa (AC) que está asociada a enfermedades particulares como FQ, diabetes tipo 2 y arritmias (39). Estudios comparativos entre B. cereus, B. thuringiensis, y B. anthracis han mostrado que sus proteínas de la pared celular y del citosol son muy similares entre sí, (40) resaltando que estas especies comparten un fondo genético similar. Estos autores identificaron las proteínas citosólicas: enolasa, GroEl, entre otras. Nuestro estudio también encontró estas proteínas con una similitud de 99%, lo que sugiere preguntar sobre la inocuidad de las cepas de Bacillus encontradas en las vías respiratorias. Finalmente, varias de las proteínas descriptas en este estudio no fueron previamente relacionadas a la fisiopatología de la FQ o descritas en proteomas bacterianos. Por ello, son necesarios estudios más amplios de proteómica para lograr comprender la relación que existe entre las proteínas expresadas por las bacterias y dicha enfermedad.
En su conjunto, el proceso de caracterización microbiana por MALDI TOF TOF podría volverse una herramienta adicional de interés cuya efectividad y utilidad podría mejorarse con la aplicación de geles SDS PAGE 2D que permiten una mejor separación de las proteínas, y con la confrontación de los resultados a bases de datos de toxinas, virulencia, sensibilidad o resistencia a antibióticos globales y propios. Este último paso podría ser rápidamente optimizado con la adquisición de un software comercial capaz de optimizar el análisis de nuestros bancos de datos y de relacionar/integrar bases de datos de otros constructores. De esta manera, la capacidad de análisis sería fuertemente mejorada, en particular para las cepas raras o no encontradas.
Una limitación del presente estudio es el reducido número de muestras, principalmente porque la población de pacientes en sí es relativamente pequeña debido al subdiagnóstico de la enfermedad (7), pero también porque las muestras fueron entregadas de forma voluntaria y algunos pacientes no estaban presentes los días de muestreo. Se recomienda completar este estudio con la adición de medios de cultivos o técnicas moleculares que permitirían la detección de H. influenzae y del MRSA por ser dos patógenos asociados al deterioro de las funciones respiratorias cuando alcanzan concentraciones altas. Por otro lado, sería muy útil evaluar la presencia de hongos patógenos como Aspergillus spp., Candida spp. o virus respiratorios, que también están relacionados a exacerbaciones y hospitalizaciones.
Otro punto importante a considerar es la utilización de tecnologías de metagenómica para evaluar la diversidad microbiana completa presente en los pulmones de pacientes. Un estudio interesante sería comparar la microbiota de pacientes de edades similares que viven en los principales ecosistemas del país (selva, sierra y costa, norte y sur) entre sí y con individuos sanos, para evidenciar particularidades en el infectoma (microbiota infecciosa).
Además, sería interesante comparar las muestras de esputos de los mismos pacientes en diferentes etapas de sintomatología de las infecciones respiratorias para evaluar la evolución de la colonización de bacterias patógenas. Un próximo nivel de análisis sería relacionar las mutaciones de los pacientes con la flora microbiana ya que otros estudios han mostrado un efecto significativo del genotipo del paciente sobre el fenotipo patogénico. Este análisis sería particularmente interesante puesto que estudios anteriores de nuestro equipo evidenciaron genotipos muy raros o únicos a nivel mundial en varios pacientes con FQ de Perú (7).
La identificación y la caracterización temprana de los patógenos que colonizan las vías respiratorias inferiores son centrales para prevenir la degradación rápida de los pulmones de pacientes con FQ. En este estudio hemos combinado diferentes técnicas de identificación, a nivel genético y proteómico, de cepas bacterianas cultivables representativas de la microbiota de las vías respiratorias inferiores de pacientes peruanos con FQ. Las técnicas de genotipado del ARNr16S y de identificación de perfiles proteicos por espectrometría de masas son comúnmente utilizadas en laboratorios clínicos de países desarrollados permitiendo una identificación segura de la cepa aislada, mientras que la identificación por MALDI TOF TOF es exploratoria y fue empleada por primera vez en este estudio con fines de diagnóstico complementario. La asociación de las secuencias del ARNr 16S y de los espectros de masas permitió elaborar la primera base de datos del país a partir de cepas nativas, abriendo el camino al diagnóstico clínico por análisis MALDI TOF, mientras que los análisis por MALDI TOF TOF demostraron su utilidad para identificar caracteres relacionados a la virulencia o a respuestas a antibióticos. La disponibilidad de estas tecnologías permitirá la caracterización de otros patógenos relevantes para la salud pública a nivel nacional como Helicobacter pylori o las especies del género Mycobacterium asociados a la tuberculosis.
En conclusión, los análisis permitieron evidenciar la importante dominancia del patógeno oportunista P. aeruginosa en los pacientes peruanos con FQ que son mayormente pediátricos. Esta constatación debe relacionarse con las edades tempranas de mortalidades observadas a nivel nacional en comparación con los países desarrollados, y resaltan la necesidad de continuar los esfuerzos de prevención y de erradicación para mejorar la esperanza de vida de estos pacientes. Para ello, es imprescindible una política integral de gobierno que iniciaría con la aplicación del tamizaje neonatal para el diagnóstico temprano de la enfermedad, la disponibilidad continua de las medicinas necesarias, la capacitación de profesionales y padres para los cuidados rutinarios de fisioterapia y antibioterapia, la formación de unidades de diagnóstico genético y patología/ microbiología asociada a la FQ y, finalmente, que todos los pacientes con FQ se puedan beneficiar de servicios de salud independientemente de su situación económica.
Contribuciones de los autores: RA, EG, SS, JR, BD, VC, YU participaron en la concepción del artículo, redacción y aprobación de la versión final del manuscrito, obtención del financiamiento, recolección de datos; RA y VC, BD participaron en el análisis de datos.
Fuentes de financiamiento: esta investigación fue financiada por recursos canon y sobrecanon de la Universidad Nacional de Tumbes.
Conflictos de interés: no existen conflictos de interés en la publicación de este artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. Paganin P, Fiscarelli EV, Tuccio V, Chiancianesi M, Bacci G, Morelli P, et al. Changes in cystic fibrosis airway microbial community associated with a severe decline in lung function. PLoS ONE. 2015; 10(4): e0124348. doi:10.1371/journal.pone.0124348. [ Links ]
2. Ramsay K, Stockwell R, Bell S, Kidd T. Infection in cystic fibrosis: impact of the environment and climate. Expert Review of Respiratory Medicine 2016; 10(5): 505 – 19. doi: 10.1586/17476348.2016.1162715. [ Links ]
3. Lynch SV, Bruce KD. The cystic fibrosis airway microbiome. Cold Spring Harb Perspect Med. 2013; 3(3):a009738. doi: 10.1101/cshperspect.a009738. [ Links ]
4. Desai A, Stanley T, Atuan M, McKey J, Lipuma,J, Rogers B, Jerris R. Use of matrix assisted laser desorption ionization-time of flight mass spectrometry in a pediatric clinical laboratory for identification of bacteria commonly isolated from cystic fibrosis patients. J. Clin. Pathol. 2012; 65(9):835-838. [ Links ]
5. AbdulWahab A, Taj-Aldeen SJ, Ibrahim EB, Talaq E, Abu-Madi M, Fotedar R. Discrepancy in MALDI-TOF MS identification of uncommon Gramnegative bacteria from lower respiratory secretions in patients with cystic fibrosis. Infect Drug Resist. 2015; 8:83 -8. doi: 10.2147/IDR.S80341. [ Links ]
6. Gogichaeva NV, Williams T, Alterman MA. MALDI TOF/TOF tandem mass spectrometry as a new tool for amino acid analysis. J Am Soc Mass Spectrom. 2007; 18(2):279-84. Doi: 10.1016/j.jasms.2006.09.013. [ Links ]
7. Aquino R, Protzel A, Rivera J, Abarca H, Dueñas M, Nestarez C, Purizaga N, Diringer B. Frecuencia de las mutaciones más comunes del gen CFTR en pacientes peruanos con fibrosis quística mediante la técnica ARMS-PCR. Rev Peru Med Exp Salud Publica. 2017;34(1):62-9. doi: 10.17843/rpmesp.2017.341.276. [ Links ]
8. Coman G, Petraru E, Dahorea C, Anton DT. Current microbiological data on lower respiratory tract infection in cystic fibrosis. Part II: recommendations for microbiological diagnosis in cystic fibrosis. Rev Med.Chir.Soc.Med.Nat.Iasi. 2012; 116(3): 898 – 901. [ Links ]
9. Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. Mega 6: Molecular evolutionary genetics analysis version 6.0. Mol Biol Evol. 2013;30(12):2725-9. doi: 10.1093/molbev/mst197. [ Links ]
10. Boratyn GM, Camacho C, Cooper PS, Coulouris G, Fong A, Ma N, Madden TL, Matten WT, McGinnis SD, Merezhuk Y, Raytselis Y, Sayers EW, Tao T, Ye J, & Zaretskaya I. "BLAST: a more efficient report with usability improvements." Nucleic Acids Res. 2013; 41: W29-W33. [ Links ]
11. Lomonte B. Manual de métodos imunológicos. Universidad de Costa Rica. 2007. 138pp. Acceso libre en: http://www.icp.ucr.ac.cr/~blomonte/. [ Links ]
12. Shevchenko A, Tomas H, Havli J, Olsen J & Mann M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols 2006; 1(6): 2856 - 60. doi: 10.1038/nprot.2006.468. [ Links ]
13. Hampton T, Green D, Cutting G, Morrison H, Sogin M, Gifford A, Stanton B, O’Toole G. The microbiome in pediatric cystic fibrosis patients: the role of shared environment suggests a window of intervention. Microbioma. 2014; 2049:2618-2.
14. Sobin L, Kawai K, Irace AL, Gergin O, Cunningham M, Sawicki GS, Adil EA. Microbiology of the upper and lower airways in pediatric cystic fibrosis patients. Otolaryngol Head Neck Surg. 2017; 1:194599817702332. doi: 10.1177/0194599817702332. [ Links ]
15. Chiappini E, Taccetti G, de Martino M. Bacterial lung infections in cystic fibrosis patients: an update. Pediatr Infect Dis J. 2014;33(6):653-4. doi: 10.1097/INF.0000000000000347. [ Links ]
16. Johnson EJ, Zemanick ET, Accurso FJ, Wagner BD, Robertson CE, Harris JK. Molecular identification of Staphylococus aureus in airway samples from children with cystic fibrosis. PLoS One. 2016; e0147643. doi: 10.1371/journal.pone.0147643.
17. Davies J, Bilton D. Bugs, biofilms, and resistance in cystic fibrosis. Respir Care. 2009; 54(5):628-40. PMID: 19393107. [ Links ]
18. Wittmann J, Dreiseikelmann B, Rohde C, Rohde M, Sikorski J. Isolation and characterization of numerous novel phages targeting diverse strains of the ubiquitous and opportunistic pathogen Achromobacter xylosoxidans. PLoS One. 2014; 9 (1): e86935. doi: 10.1371/journal.pone.0086935. [ Links ]
19. Anderson S, Stapp J, Burns J, Qin X. Characterization of Small-Colony-Variant Stenotrophomonas maltophilia isolated from the sputum specimens of five patients. J Clin Microbiol. 2007; 45(2): 529 - 35. doi: 10.1128/JCM.01444-06. [ Links ]
20. Busquets NP, Baroni MR, Ochoteco MC, Zurbriggen ML, Virgolini S, Meneghetti FG. Bacterial isolates from respiratory samples of pediatric patients with cystic fibrosis and their distribution by ages. Rev Argent Microbiol. 2013; 45(1):44-9. [ Links ]
21. Rizzo LC, Fischer GB, Maróstica PJ, Mocelin HT. Profile of cystic fibrosis in two reference centers in southern Brazil. Rev Assoc Med Bras (1992). 2015; 61(2):150-5. doi: 10.1590/1806-9282.61.02.150. [ Links ]
22. Torres D. Estudio clínico epidemiológico de la fibrosis quística en el instituto de salud del niño, Lima 1991–2001. Pediátrica. 2002; 4(3):7-15. [ Links ]
23. Castilla G. Epidemiología de infecciones respiratorias en pacientes con fibrosis quística en el hospital nacional Edgardo Rebagliati Martins. Rev. Perú. pediatr. 2008; 61(2).
24. Coburn B, Wang PW, Diaz Caballero J, Clark ST, Brahma V, Donaldson S, Zhang Y, Surendra A, Gong Y, Elizabeth Tullis D, Yau YC, Waters VJ, Hwang DM, Guttman DS. Lung microbiota across age and disease stage in cystic fibrosis. Sci Rep. 2015; 14; 5:10241. doi: 10.1038/srep10241. [ Links ]
25. Lim YW, Evangelista JS, Schmieder R, Bailey B, Haynes M, Furlan M, Maughan H, Edwards R, Rohwer F, Conrad D. Clinical insights from metagenomic analysis of sputum samples from patients with cystic fibrosis. J Clin Microbiol. 2014; 52(2):425-37. doi: 10.1128/JCM.02204-13. [ Links ]
26. Stressmann F, Rogers G, Klem E, Lilley A, Donaldson S, Daniels T. Analysis of the bacterial communities present in lungs of patients with cystic fibrosis from American and British centers. J Clin Microbiol. 2011; 49: 281 – 91. doi: 10.1128/JCM.01650-10. [ Links ]
27. Hsu Y, Burnham C. MALDI-TOF MS identification of anaerobic bacteria: assessment of pre-analytical variables and specimen preparation techniques, Diag. Microbiol. Infect. Dis. 2014; 79 :144 - 148. doi: 10.1016/j.diagmicrobio.2014.02.007. [ Links ]
28. Fernández-Olmos, A., Garcia-Castillo, M., Morosini, M. I., Lamas, A., Maiz, L. & Canton, R. MALDI-TOF MS improves routine identification of non-fermenting Gram negative isolates from cystic fibrosis patients. J Cyst Fibros. 2012; 11(1), 59–62. doi: 10.1016/j.jcf.2011.09.001. [ Links ]
29. Kerem E, Conway S, Elborn S, Heijerman H. Standards of care for patients with cystic fibrosis: a european consensus. J Cyst Fibros. 2004; 4: 7 – 26. doi: 10.1016/j.jcf.2004.12.002. [ Links ]
30. Hare NJ, Solis N, Harmer C, Marzook NB, Rose B, Harbour C, Crossett B, Manos J, Cordwell SJ. Proteomic profiling of Pseudomonas aeruginosa AES1R, PAO1 and P414 reveals potential virulence determinants associated with a transmissible cytic fibrosis-associated strain. BMC Microbiol. 2012; 12:16. doi: 10.1186/1471-2180-12-16. [ Links ]
31. Westritschnig K, Hochreiter R, Wallner G, Firbas C, Schwameis M, Jilma B. A randomized, placebo-controlled phase I study assessing the safety and immunogenicity of a Pseudomonas aeruginosa hybrid outer membrane protein OprF/I vaccine (IC43) in healthy volunteers. Hum vaccin immunother. 2014; 10: 170 – 83. doi: 10.4161/hv.26565. [ Links ]
32. Li Y, Xuan Y, Ying Z, Wong Y, Yueqi M, Qi Y, et al. NMR structural characterization of the N-terminal active domain of the gyrase B subunit from Pseudomonas aeruginosa and its complex with an Inhibitor. FEBS Lett. 2015; 2683 - 9. doi: 10.1016/j.febslet.2015.07.044.
33. Mukherjee S, Dutta D, Saha B, Das A. Crystal structure of Glyceraldehyde3-Phosphate Dehydrogenase 1 from Methicillin-Resistant Staphylococcus aureus MRSA252 provides novel insights into substrate binding and catalytic mechanism. J Mol Biol. 2010; 3. 401(5): 949 - 68. doi: 10.1016/j.jmb.2010.07.002.
34. Wu Y, Wang C, Lin S, Wu M, Han L, Tian C, et al. Octameric structure of Staphylococcus aureus enolase in complex with phosphoenolpyruvate. Acta Crystallogr D Biol Crystallogr. 2015; 1-71(12): 2457 - 70. doi: 10.1107/S1399004715018830.
35. Liu H, Cheng Z, Song W, Wu W, Zhou Z. Immunoproteomic to analysis the pathogenicity factors in leukopenia caused by Klebsiella pneumonia bacteremia. PLoS One. 2014; 16-9(10): e110011. doi: 10.1371/jornal.pone.0110011.
36. Doménech A, Martínez L, Hernández S, del Carmen M, Pascual A, Tomás J, et al. Role of Klebsiella pneumoniae OmpK35 porin in antimicrobial resistance. Antimicrob agents chemother. 2003; 47(10): 3332 - 5. doi: 10.1128/AAC.47.10.3332-3335.2003. [ Links ]
37. Jeannin P, Magistrelli G, Goetsch L, Haeuw J, Thieblemont N, Bonnefoy J, et al. Outer membrane protein A (OmpA): a new pathogen-associated molecular pattern that interacts with antigen presenting cells-impact on vaccine strategies. Vaccine. 2002; 20: A23 - A27. doi: 10.1016/S0264410X(02)00383-3. [ Links ]
38. Deng W, Li C, Xie J. The underlying mechanism of bacterial TetR/ AcrR family transcriptional repressors. Cell Signal. 2013; 25. 1608 – 13. doi: 10.1016/j.cellsig.2013.04.003. [ Links ]
39. Cooper D, Tabbasum V. Adenylate cyclasecentred microdomains. Department of Pharmacology, University of Cambridge. 2014; 462. 199 – 213. doi: 10.1042/BJ20140560. [ Links ]
40. Gohar M, Gilois N, Graveline R, Garreau C, Sanchis S, Lereclus D. A comparative study of Bacillus cereus, Bacillus thuringiensis and Bacillus anthracis extracellular proteomes. Proteomics. 2005; 5. 3696 – 3711. doi: 10.1002/pmic.200401225. [ Links ]
Correspondencia: Ruth Aquino.
Dirección: Laboratorio de Biotecnología molecular – Universidad Nacional de Tumbes Av. Universitaria S/N, Tumbes-Perú.
Teléfono: +51-73-948359340.
Correo electrónico: ruthaquinord@gmail.com.
Recibido: 03/12/2016
Aprobado: 12/07/2017
En línea: 29/09/2017