Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.35 no.4 Lima oct./dic 2018
http://dx.doi.org/10.17843/rpmesp.2018.354.3101
ARTÍCULO ORIGINAL
Anticuerpos de cadena única de alpaca para la detección de antígenos de Fasciola hepatica
Single-chain antibodies from alpaca for the detection of Fasciola hepatica antigens
Teresa Barreto1,a, Yenisey Alfonso1,b, Pierre Lafaye2,c, María del Pilar García Lazaro1,d, L. Agueda Perez1,e, Patricia Herrera-Velit1,f, Jose R. Espinoza1,g
1 Unidad de Biotecnología Molecular, Laboratorios de Investigación y Desarrollo, Universidad Peruana Cayetano Heredia. Lima, Perú.
2 Plateforme d’Ingénierie des Anticorps, Institut Pasteur. París, Francia.
a Bióloga, magíster en Bioquímica; b bioquímica, doctor en Ciencias; c farmacéutico, PhD en Bioquímica; d bióloga; e bióloga; f bióloga, PhD en Inmunología; g biólogo, PhD en Genética molecular
RESUMEN
Objetivo. Producir anticuerpos recombinantes de cadena única de alpaca que se unan con alta afinidad y especificidad al antígeno excretado-secretado (ES) de Fasciola hepatica para el desarrollo de tecnologías nuevas de diagnóstico de fascioliasis humana y animal. Materiales y métodos. Se ha construido una genoteca de cADNde los dominios variables de anticuerpos de cadena única pesada, conocidos como VHH, a partir de células mononucleares de sangre periférica de una alpaca inmunizada con el antígeno ES de F. hepatica. La genoteca fue tamizada con el antígeno ES por despliegue diferencial de fagos (phage display), seleccionando diez VHH que se unen específicamente a ES. El VHH anti ES fue clonado en un vector de expresión, la proteína recombinante (VHH-ES1) de 15,3 kDa fue producida por fermentación en E. coli y purificada a homogeneidad por cromatografía de afinidad. La unión del VHH-ES1 al antígeno ES fue evaluada por ELISA usando VHH-ES1 como anticuerpo de captura, antisuero policlonal anti-ES de conejo y conjugado anti IgG de conejo con peróxidasa de rábano. Resultados. Se ha identificado y producido un VHH-ES1 recombinante que se une al antígeno ES (VHH-ES1) que correspondía a un anticuerpo de la subclase IgG2 de bisagra larga. La unión del anticuerpo VHH-ES1 al antígeno muestra linealidad respecto a la concentración de ES en el rango de 50-5000 ng/mL y el valor límite de detección del antígeno está en el rango de 30-170 ng/mL de ES (R2=0,99). Conclusión. El VHH-ES1 se une con afinidad y especificidad al antígeno ES de F. hepatica y es un anticuerpo promisorio a evaluar para el desarrollo de nuevas tecnologías de diagnóstico de fascioliasis.
Palabras clave: Anticuerpos de Dominio Único; Fascioliasis; Anticuerpos; Camélidos del Nuevo Mundo; Perú (fuente DeCS BIREME).
ABSTRACT
Objectives. To produce recombinant single-chain antibodies from alpaca that will bind to the excreted-secreted (ES) Fasciola hepatica antigen with high affinity and specificity, so as to develop new diagnostic technologies of human and animal fascioliasis. Materials and Methods. A gene bank of DNA of the variable dominions of heavy single-chain antibodies (VHH) has been created, based on mononuclear cells of peripheral blood of an alpaca immunized with the ES antigen of F. hepatica. The gene bank was screened with the ES antigen by differential phage display, selecting ten VHH that bind specifically to ES. The anti-ES VHH was cloned in an expression vector, the recombinant protein (VHH-ES1) of 15.3 kDa was produced by fermentation in E. coli and purified to homogeneity by affinity chromatography. The binding of VHH-ES1 to the ES antigen was evaluated by ELISA using VHH-ES1 as capture antibody, policlonal anti-ES serum of rabbit and conjugated rabbit anti IgG with radish peroxidase. Results. A VHH that binds to the ES antigen (VHH-ES1) has been identified through differential phage display and produced by fermentation in E. coli; this corresponds to an antibody of the long-hinge IgG2 subclass. The binding of the VHH-ES1 antibody to the antigen shows linearity with respect to the concentration of ES in the 50-5,000 ng/mL range and the limit of detection value of the antigen is in the 30-170 ng/mL range of ES (R2=0.99). Conclusions. The VHH-ES1 binds with affinity and specificity to the ES antigen of F. hepatica and is a promissory antibody to be assessed for the development of new fascioliasis diagnostic technologies.
Keywords: Single-Domain Antibodies; Fascioliasis; Antibodies; Camelids, New World; Peru (source MeSH NLM).
INTRODUCCIÓN
A nivel mundial, se estima que existen entre 5-17 millones de personas con fascioliasis; la infección es endémica en las zonas rurales de países como Perú, Bolivia, Ecuador, Irán, Georgia, Vietnam, Egipto, entre otros (1); donde, debido a las condiciones inadecuadas de provisión de agua, salubridad, educación y pobreza, el parásito no es contenido en su ciclo natural en animales herbívoros, logrando infectar a los humanos (2).
A pesar que la fascioliasis es un problema de salud pública y de sanidad animal (3), las innovaciones de nuevos agentes para el control y tratamiento de la infección son limitadas, lo cual contribuye a que la fascioliasis sea una enfermedad desatendida (2). El desarrollo de nuevas tecnologías de diagnóstico de la fascioliasis humana y animal requiere de la identificación de nuevas moléculas que se unan a antígenos marcadores de la infección para el desarrollo de tecnologías de diagnóstico sencillas de bajo costo y aplicables en las zonas rurales pobres que son endémicas para la infección.
Hasta el momento las moléculas usadas para la detección de antígenos de Fasciola hepatica en diferentes formatos de diagnóstico son los anticuerpos monoclonales de ratón; no obstante, los dominios variables de los anticuerpos de cadena única de la alpaca (VHH) tienen la capacidad de unirse a los antígenos de los patógenos (4,5) , y son un recurso biotecnológico promisorio para la generación de nuevas moléculas para el diagnóstico de la fascioliasis (6); ya que pueden ser clonados y expresados en E. coli conservando la capacidad de unirse a los antígenos con alta afinidad, especificidad, estabilidad y rendimiento (6), características ventajosas para el desarrollo de tecnologías de diagnóstico de bajo costo y realizables en el campo.
Este estudio tiene como objetivo identificar y producir VHHs que se unan al antígeno de excretado-secretado (ES) de F. hepatica con alta afinidad y especificidad para el desarrollo de tecnologías nuevas de diagnóstico de la fascioliasis humana y animal.
MATERIALES Y MÉTODOS
ES-ELISA DE ALPACAS
Se añadió 300 µl de ES, preparado (7), en buffer carbonato-bicarbonato pH 9,6 por pozo de microplacas (IMMULON 4 HBX Dynex Technologies, INC, USA) e incubó a 4 °C toda la noche. Las placas fueron lavadas cinco veces por cinco minutos con PBS-T (PBS 1X, 0,05% Tween 20), bloqueadas con 2% BSA por una hora a 37° C y lavadas. Se añadió 100 µL de suero diluido en PBS-T, 2% BSA, y se incubó a 37 °C por una hora. Luego de cinco lavados se añadió 100 µL de proteína A peroxidasa en PBS-T, 2% BSA, y se incubó una hora a 37 °C. Se añadió 100 µL de tetrametilbenzidina (TMB. KPL, USA) y se incubó por diez minutos a temperatura ambiente. La reacción fue detenida con 50 µL de 2M H2SO4. Se determinó A450 en un lector de ELISA (Biotek, USA). El punto de corte de la prueba es A450 = 0,165 (7).

INMUNIZACIÓN CON ES DE LA ALPACA
Una alpaca fue inmunizada con 400µg de ES con adyuvante completo de Freund en la primera inmunización y refuerzos con 500 µg y 700 µg de ES con adyuvante incompleto de Freund (Sigma-Aldrich, USA).
PREPARACIÓN DE ARN TOTAL DE CÉLULAS BLANCAS DE ALPACA
Se colectaron muestras de sangre de la alpaca inmunizada con ES en tubos Vacutainer con EDTA. Las células mononucleares de sangre periférica (PBMC, sigla del inglés peripheral blood mononuclear cell) fueron separadas por centrifugación en Histopaque-1077 (Sigma-Aldrich, USA) y lavadas con solución Hanks (Sigma-Aldrich, USA). El conteo celular se realizó en una cámara Neubauer. Las células (3 x 106) fueron suspendidas en 0,75 ml Trizol LS (Invitrogen) y almacenadas a -80 °C. El ARN fue purificado con el kit Qiamp RNA blood, cuantificado por A260 en NanoDrop 100 (ThermoScientific), y visualizado en electroforesis en geles 1% agarosa, 0,5 X TBE con bromuro de etidio.
CONSTRUCCIÓN DE LA BIBLIOTECA DE cADN
El ADN fue sintetizado a partir del ARN extraído de PBMC usando oligo(dT) como cebador de poly(A)+ ARN. La biblioteca de cADN de VHH fue construida amplificando los VHH a partir del cADN con el cebador común ALVHHBgl (5’-GAGCCTAGCCGGCKCAGKTGCAGCTCGTGGAGTCNGG-3’), específico para el FR1-bisagra corta; ALVHHR1Not (5’-TTGCGGCCGCTGGGGTCTTCGCTGTGGTGCG-3’) y específico para FR1-bisagra larga ALVHHR2Not (5’-TTGCGGCCGCTTGTGGTTTTGGTGTCTTGGG-3’) (8). 1,2 µg de pHEN2 fue digerido con 10 U de NotI (Fermentas) a 37 °C y con 10 U de SfiI (Fermentas) a 50 °C. El vector pHEN2/NotI/SfiI y los VHH amplificados fueron purificados en geles de 1,5% agarosa con el kit Qiaquick (Gel Extraction kit, Qiagen). Se digirió 400 ng de los productos VHH amplificado con 20 U de NotI y 20 U de BglI (Fermentas) a 37 °C O/N y purificado en geles de 1,5% agarosa. La ligación procedió con 80 ng de inserto y 40 ng pHEN2/NotI/SfiI en 20 μl buffer y 400 U de T4 ADN Ligasa a 16 °C durante 12 horas. Se electroporó E. coli TG1 en cubetas de 0,1 cm, pulso de 1,8 kV, 25 μF, 200 Ω, se suspendió en 250 μl 2xYT a 37 °C por una hora y fueron plaqueadas en 2xYTAG (ampicilina 100 μg/ml, 2% Glucosa) a 37 °C toda la noche.
PHAGE DISPLAY DE LA BIBLIOTECA DE ADN VHH ANTI ES
La biblioteca de cADN VHH anti-ES en pHEN2 (1x107ufc/ml) (9) fue expandida fermentando 3x108 bacterias en 100 mL de 2YTAG (100 μg/mL ampicilina, 1% glucosa) a 37 °C en agitación hasta alcanzar 1x1010 bacterias. El cultivo fue infectado con 100uL de fago helper M13KO7 (New England Biolabs, 1x1013 fagos/mL) incubada 30 minutos en reposo y 30 minutos con agitación a 150 rpm a 37 °C; luego el cultivo fue centrifugado a 4500 rpm por diez minutos. El precipitado solubilizado en 100 mL de 2YTA, Kan (50µg/mL), 1 mM IPTG y fermentó a 30 °C en agitación 150 rpm por 16 horas. El cultivo fue centrifugado a 8000 rpm a 4 °C, el sobrenadante disuelto en 20 mL 20% PEG 2,5M NaCl y mantenido en hielo por dos horas. Luego se centrifugó a 8000 rpm por diez minutos a 4 °C y el precipitado disuelto en 40 mL H2O, se añadió 8 mL 20% PEG 2,5M NaCl y se centrifugó a 8000 rpm por diez minutos a 4 °C. Los fagos fueron suspendidos en 2 mL 1X PBS, centrifugados a 15 000 rpm por un minuto a 4 °C y el sobrenadante almacenado a 4 °C.
SELECCIÓN DE FAGOS VHH ANTI ES
Se inoculó una colonia en 25 mL de 2YT y se incubó a 150 rpm, 37 °C hasta A600 0,5. Se agregó 40 μg de ES en inmunotubo (Nunc Maxisorp, ThermoFisher Scientific, USA) e incubó a 4 °C por 16 horas. El inmunotubo se lavó seis veces con PBS-T, tres veces con PBS y se bloqueó con 2% leche descremada por 30 minutos a temperatura ambiente. La segunda ronda de selección se usó Odissey-Licor (1/2 en PBS) y la tercera ronda se utilizó BSA 2%, descartando la solución bloqueadora. Se añadió 1 mL de fagos con agitación durante una hora a temperatura ambiente. La segunda selección se incubó 45 minutos y la tercera por 30 minutos. Los fagos se eluyeron con 1 mL de 100 mM trietanolamina por cinco minutos a temperatura ambiente y se añadió 0,5 mL 1M Tris-HCl pH 7,5. Se añadió 10 mL de E. coli TG1 a 37 °C sin agitación y 30 minutos a 150 rpm. Se hicieron diluciones en 100 μL del cultivo, desde 10-1 hasta 10-5 y se sembró en 2YTGA. El resto se centrifugó, se disolvió en 2 mL de medio 2YT, se sembró en una placa 24x24 con 2YTGA e incubó a 37 °C toda la noche. Las bacterias fueron cosechadas y colocadas en 5 mL 2YTA. Se colocó 30 μL en 100 mL de medio 2YTAG para proseguir con el rescate y el resto se criopreservó en DMSO al 8% a -80 °C.
EXPRESIÓN DE FAGOS VHH ANTI ES
Se picaron colonias al azar y se sembraron en placas de 96 pozos (Costar 3799) en 200 μL de 2YTAG, y se incubó a 37 oC toda la noche en agitación. Se tomó 3 μl de cada cultivo para sembrar en 200 μL 2YTAG, placas de 96 pozos de 2,2 mL (Abgene AB-0661). Las placas fueron incubadas a 37 oC hasta alcanzar la fase logarítmica. Se realizó la infección con 100 μl de fago helper M13KO7 (1x1011ufp/mL). Las células centrifugadas a 4500 rpm por cinco minutos y suspendidas en 500 μl de 2YTAK+IPTG 1mM por pozo. Las placas fueron incubadas a 30 oC a 150 rpm toda la noche, centrifugadas a 4500 rpm por cinco minutos a 4 °C y guardadas a 4 °C.
TAMIZAJE DE FAGOS VHH POR ELISA
Se fijaron placas Maxisorp con 100 μL ES (5 μg/mL) en PBS a 4 °C toda la noche. Las placas fueron lavadas cinco veces con PBS-T y bloqueadas con 100 μL de 0,5% gelatina en PBS-T (PBSTG) por una hora a 37 °C. Las placas fueron lavadas y se colocó 20 μL de sobrenadante de cultivo en 80 μL PBSTG, las placas fueron incubadas 1,5 h a 37 °C. Luego de cinco lavados con PBS-T, se adicionó 100 μL del anti MSIgG-HRP (Abcam) 1/1000 en PBSTG e incubadas a 37 °C por una hora. Luego de lavados se adicionó 100 μl de anti M13 g8p 1/1000 en PBSTG e incubó a 37 °C por una hora; se volvió a lavar y se añadió 100 μL de TMB e incubó por diez minutos a temperatura ambiente. La reacción fue detenida con 50 μL de 3N H2SO4. Se realizó la lectura a A450nm en el espectrofotómetro (Biotek, USA). Los clones reactivos a ES y poco reactivos al antígeno no relacionado fueron amplificados por PCR con los cebadores M13-40 y Myc-TAG y las secuencias fueron analizadas con Clustal W.
EXPRESIÓN DE VHH ANTI ES RECOMBINANTE EN pET22b+
1,2 μg del producto del VHH anti ES amplificada por PCR con cebadores VHHNcoI-NotI de los fagémidos P3BC3 y P3BC12 y 2 μg plásmido pET22b+ fueron digeridos con NcoI y NotI (New England Biolabs) a 37 °C y purificado (Wizard SV PCR purification kit). La ligación procedió con 200 ng pET22b+, 100 ng de inserto y 400 U de T4 ADN ligasa (NEB) a 16 °C por 17 horas. La reacción fue precipitada con etanol y solubilizada en 5 μL de H2O. Se electroporó E. coli BL21 a 1.8KV en cubeta de 0,1 cm. Las transformantes seleccionadas en placas LB con ampicilina (100 μg/mL) y las colonias positivas cultivadas en 3 mL LB amp a 37 °C toda la noche. Luego centrifugadas a 5000 rpm por diez minutos, el precipitado suspendido en 1 mL de LB amp. 30 μL de cultivo se añadió a 3 mL de LB amp por dos horas a 37 °C e inducida con 1mM IPTG por dos horas a 37 °C. Las células fueron centrifugadas a 7650 g por 30 minutos a 4 °C. El sobrenadante fue analizado por SDS-15% PAGE y los VHH anti ES recombinantes purificados por cromatografía de afinidad Ni-Agarosa.
VHH ANTI-ES ELISA INDIRECTO
500 ng ES en PBS fue fijado en placas Maxisorp a 4 °C toda la noche, luego lavadas cinco veces con PBS-T y bloqueadas con 2% BSA en PBS-T por una hora a 37 °C. Se realizó cinco lavados con PBS-T. Se añadió 1-2 μg de VHH-ES1 en 100 μL PBS 2% BSA e incubadas una hora a 37 °C. Luego de lavados con PBS-T, se añadió 100 μL (1/2000) Penta His HRP conjugate (Qiagen, USA), 2% BSA e incubadas una hora a 37 °C, lavadas con PBS-T. Se añadió 100 μL de TMB e incubó por diez minutos a temperatura ambiente en oscuridad. La reacción fue detenida con 50 μL de 2M H2SO4. A450 se determinaron en un lector de ELISA (Biotek, USA).
VHH ELISA CAPTURA DE ANTÍGENO ES
Se añadió 100 μL del anticuerpo de captura VHH-ES1 (10 ug/mL) a microplacas Maxisorp e incubó toda la noche a 4 °C en buffer carbonato-bicarbonato. Se realizó cinco lavados con 200 μL del buffer de lavado (PBS-Tween 0,05%) y cinco minutos de incubación en cada lavado. Se añadió 200 μL del buffer de bloqueo (PBS-Tween 0.05%-BSA 5%), e incubó a 37 °C por una hora y se lavó como fue descrito. Se agregó 100 μL de ES en concentraciones de 0, 50, 200, 1000 y 5000 ng/mL por pozo, se incubó a 37 °C por una hora, y se lavó cinco veces con 30 segundos de incubación cada paso. Se añadió 100 μL del anticuerpo de policlonal de conejo anti-ES (10 μg/mL) incubando por una hora a 37 °C y luego se lavó cinco veces con 30 segundos de incubación. Se añadió 100 μL (1/30 000) del conjugado anti-rabbit-HRP (KPL, USA) e incubó a 37 °C por una hora. Se lavó cinco veces con 30 segundos de incubación. Se añadió 100 μL de TMB e incubó cinco minutos en oscuridad, Se agregó 50 μL de 3N H2SO4 para detener la reacción. Los valores de A450 se estimaron en un lector de ELISA (Biotek, USA).
ANÁLISIS DE DATOS
Microsoft Excel® se usó para analizar datos de ELISA, las secuencias de ADN fueron alineadas con el software Clustal W, analizadas con el programa NCBI/Blast y la estructura secundaria de VHH modelada con SwissModel.
CONSIDERACIONES ÉTICAS
El presente estudio (código 61578) fue aprobado por el Comité Institucional de Ética de la Universidad Peruana Cayetano Heredia. Las alpacas estuvieron bajo seguimiento de un médico veterinario.
RESULTADOS
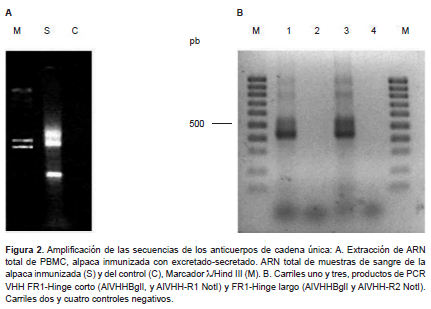
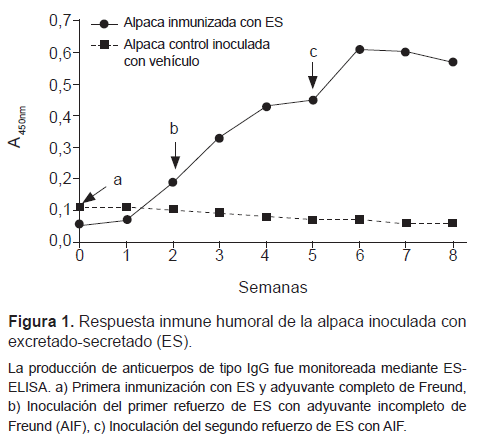
Los niveles de IgG circulantes anti-ES basales fueron de A450 de 0,112 y 0,063 para la alpaca control e inmunizada respectivamente. Niveles crecientes de IgG anti-ES se observaron a partir de la segunda semana posinmunización, elevándose A450 0,455 después del segundo refuerzo. En la alpaca control se mantuvieron negativos durante el periodo de inmunización (Figura 1). Después del segundo refuerzo se colectaron 5x107 células blancas, de las que se obtuvo 41 µg de ARN total (ratio A260/280 de 2); el ARN no presentó degradación y mostraba las bandas características ARN total (Figura 2A). A partir del ARN se sintetizó ADN resultando en productos de amplificación de 400 bp característico de los VHH (Figura 2B).
El ADN de los VHH fue amplificado y clonado en el vector pHEN2 y trasformados en E. coli TG1 obteniéndose 1,2x106 colonias transformantes, los auto-ligados representaban el 0,06% de los transformantes. Se realizó transformaciones seriadas para escalar la biblioteca de ADN a 5x107 transformantes.
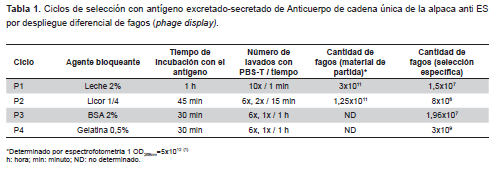
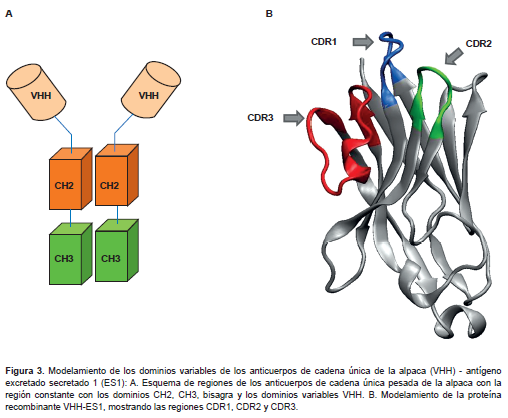
La selección de VHH anti ES de la biblioteca se realizó por despliegue diferencial de fagos con ES (Tabla 1). Los fagos obtenidos en cada ciclo de selección disminuyeron ligeramente en el P2 y luego aumentó en P3 y P4. Completados los ciclos P2, P3 se analizaron 384 clones por ES-ELISA indirecto, seleccionando 37 positivos que unen ES, los cuales fueron nuevamente analizados con ES-ELISA, obteniéndose diez clones positivos que no mostraban reacción cruzada con un antígeno no relacionado: P3AC2, P3AF7, P3AH7, P3AH10, P3BA12, P3BC3, P3BC12, P3BE7, P3BE9, P3BF, los cuales fueron amplificados por PCR y secuenciados, resultando que los diez clones eran VHH idénticos, derivados de un anticuerpo de las subclase IgG2 de bisagra larga y mostraban las regiones FR1, FR2 FR3, CDR1, CDR2 y CDR3 características de los VHHs de camélidos (Figura 3A). La regiones de reconocimiento al epítope del antígenos CDR1 tiene ocho aminoácidos, CDR2 ocho aminoácidos y CDR3 20 aminoácidos en una estructura secundaria con la correspondiente tres horquillas de las regiones de reconocimiento del antígeno y las regiones andamio FR1, FR2, FR3 (Figura 3B).
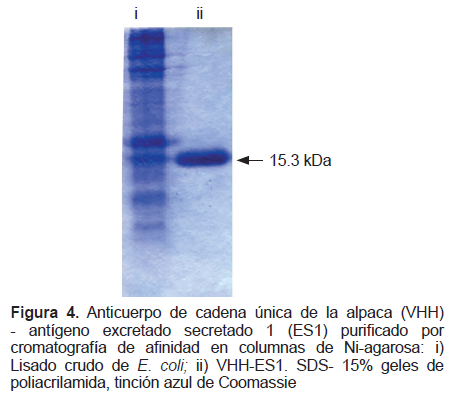
El ADN del VHH anti-ES fue subclonado en el vector de expresión pET22b+ y la proteína recombinante VHH-ES1 (PM 15,3 kDa) fue producida en E. coli BL21 y purificada por cromatografía de afinidad (Figura 4). Ensayos de ELISA indirecto mostraron que la unión del VHH-ES1 a ES es de alta afinidad detectándose los valores de A450 de 0,311 ±0,08 y 0,726 ±0,027 para uno y dos µg de ES respectivamente, mientras que el control A450 de 0,175 ±0,08; y específica ya que era desplazada por competencia con un policlonal anti-ES de conejo (datos no mostrados).
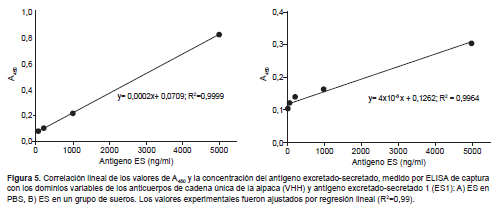
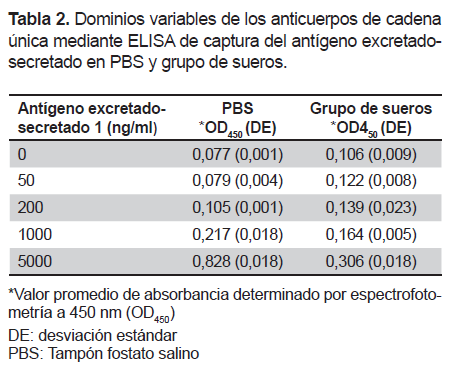
Los ensayos de ELISA de captura se realizaron usando VHH-ES1 como anticuerpo de captura, un policlonal anti-ES de conejo como antígeno secundario y con diferentes concentraciones de ES (0, 50, 200, 1000 y 5000 ng/mL) en PBS y en un grupo de sueros humanos control. La inmunoreactividad determinada por A450 se ajustaba a linealidad en el rango de concentraciones de 50-5000 ng/mL de ES (R2=0,99). El límite de detección de ES en PBS fue de 31,11 ng/mL y en el grupo de sueros el valor fue 171,92 ng/mL (Tabla 2, Figura 5A, Figura 5B).
DISCUSIÓN
Con el objetivo de contar con nuevas moléculas para el desarrollo de pruebas de diagnóstico de fascioliasis usando anticuerpos de cadena única de alpacas, se han sintetizado genotecas de cADN de VHH (1x107 clones) para seleccionar VHH que se unen específicamente al antígeno ES de F. hepatica por despliegue diferencial de fagos. Esta estrategia se ha aplicado en otros agentes infecciosos (10,11,12). La genoteca de cADN de VHH tiene una alta representación de los anticuerpos de cadena única de los grupos IgG2 e IgG3 (5x107 clones) derivados del ARN de células blancas de una alpaca inmunizada con ES. Sin embargo, el tamizaje por despliegue diferencial de fagos con ES rescató 10 VHH de secuencias idénticas de la clase de anticuerpos IgG2 de bisagra larga. Lo que sugiere que el VHH reconoce un epítope de una proteína antigénica componente de ES. La estructura secundaria modelada de la secuencia VHH-ES1 (Figura 3) revela que tiene un alto grado de homología con los VHH de alpaca (13).
El principal hallazgo del estudio es el anticuerpo VHH-ES1 que se une al antígeno ES con alta afinidad y especificidad. Los resultados del ELISA indirecto muestran que VHH-ES1 se une al antígeno en relación lineal con la concentración de ES. Ensayos de ELISA de captura con VHH-ES1 muestra que los valores de límite de detección están en el rango de 30-170 ng/ml de ES, lo que sugiere su uso potencial en la detección del antígeno en muestras biológicas. El ELISA de captura es el formato idóneo para el desarrollo y validación de tecnologías de detección de coproantígenos o antígenos circulantes en pacientes infectados con F. hepatica como se aplican los monoclonales ES70 (14) y MM3 (15) en la detección de coproantígenos para el diagnóstico de fascioliasis.
Los valores límites de detección de VHH-ES1 son menores a los reportados para los mAbs anti-ES ES70 (14,16) y MM3 (15,16) (5-15 ng/mL) (14,15), que podría considerarse una limitación a ser resuelta en el desarrollo de la tecnología de diagnóstico. Hay que tener en cuenta que ES es un antígeno de composición compleja que tiene al menos 60 diferentes polipéptidos (18,19) del parásito y del hospedero que perturban los ensayos de la determinación de la sensibilidad y especificidad de unión al antígeno; no se conoce el antígeno al que se une VHH-ES1, pero se puede identificar con técnicas de purificación por afinidad y secuenciamiento del antígeno purificado.
Los VHH son más estables y tienen menores costos de producción que los mAbs (17), constituyen una alternativa ventajosa para el desarrollo de métodos de diagnóstico (6) de aplicación en zonas endémicas de fascioliasis, ya que las técnicas de diagnóstico que usan mAbs son costosas y tienen baja estabilidad por ser muy lábiles (17).
En conclusión, se ha generado un fragmento recombinante de un anticuerpo de cadena única de alpaca IgG2 denominado VHH-ES1, que es una proteína recombinante de 15,3 kDA que se une con alta afinidad al antígeno ES de F. hepatica. VHH-ES1 es una nueva biomolécula promisoria para el desarrollo de nuevos métodos de diagnóstico de fascioliasis basado en la detección de coproantígenos o antígenos circulantes en humanos y animales infectados con F. hepatica.
Agradecimientos: Dr. J. Rodriguez, E. Gushiken por el modelamiento del VHH-ES1. El vector pHEN2 fue donado por el Dr. D. Baty y Dr. D. Kastelic (Protein Biotechnology Group, Babraham Bioscience Technologies, UK). TB realizó una estadía en el Instituto Pasteur, Francia bajo la supervisión del Dr. P. Lafaye.
Contribuciones de los autores: JRE ha concebido el presente estudio, ha participado en el diseño experimental, el análisis, la interpretación de datos, ha preparado el manuscrito, realizado la revisión crítica del manuscrito y la aprobación de su versión final. TB, YA, MPGL, PL, LAP realizaron los experimentos, interpretaron los resultados, participaron en la revisión crítica del manuscrito y aprobación de la versión final. PHV ha realizado la revisión crítica de resultados, participado en la redacción del manuscrito y aprobación de la versión final.
Fuentes de financiamiento: El trabajo fue financiado por el Programa Nacional de Innovación para la Competitividad y Productividad (Innóvate Perú), contrato 123-FINCyT-IA-2013, «Generación de anticuerpos de dominio-único de alpacas para el diagnóstico y tratamiento de enfermedades hepáticas». YA recibió financiamiento por el proyecto Postdoctoral FONDECYT «Generación de Anticuerpos de dominio único de alpacas para el diagnóstico y tratamiento de enfermedades hepáticas», EF-015-2013
Conflictos de interés: Los autores declaran no tener conflicto de interés en la publicación del presente trabajo.
REFERENCIAS BIBLIOGRÁFICAS
1. Mas Coma S, Valero MA, Bargues MD. Fasciola, lymnaeids and human fascioliasis, with a global overview on disease transmission, epidemiology, evolutionary genetics, molecular epidemiology and control. Adv Parasitol. 2009;69:41-146. doi: 10.1016/S0065-308X(09)69002-3. [ Links ]
2. Hotez PJ, Bottazzi ME, Franco-Paredes C, Ault SK, Periago MR. The Neglected Tropical Diseases of Latin America and the Caribbean: A Review of Disease Burden and Distribution and a Roadmap for Control and Elimination. PLoS Negl Trop Dis. 2008;2(9):e300. doi: 10.1371/journal.pntd.0000300. [ Links ]
3. Espinoza JR, Terashima A, Herrera-Velit P, Marcos LA. Fasciolosis humana y animal en el Perú: impacto en la economía de las zonas endémicas. Rev Peru Med Exp Salud Publica. 2010;27(4):604-12. doi: http://dx.doi.org/10.17843/rpmesp.2010.274.1535. [ Links ]
4. Hamers-Casterman C, Atarhouch T, Mulydermans S, Robinson G, Hammers C, Bajyana E, et al. Naturally occurring antibodies devoid of light chains. Nature. 1993;363(6428):446-8. [ Links ]
5. Nguyen VK, Desmyter A, Muyldermans S. Functional heavy-chain antibodies in Camelidae. Adv Immunol. 2001;79:261-96. [ Links ]
6. Muyldermans S. Nanobodies: natural single-domain antibodies. Annu Rev Biochem. 2013;82:775-97. doi: 10.1146/annurev-biochem-063011-092449. [ Links ]
7. Neyra V, Chavarry, Espinoza JR. Cysteine proteinases Fas1 and Fas2 are diagnostic markers for Fasciola hepatica infection in alpacas (Lama pacos). Vet Parasitol. 2002;105(1):21-32. [ Links ]
8. Maass DR, Sepulveda J, Pernthaner A, Shoemaker CB. Alpaca (Lama pacos) as a convenient source of recombinant camelid heavy chain antibodies (VHHs). J Immunol Methods. 2007;324(1-2):13-25. doi: 10.1016/j.jim.2007.04.008. [ Links ]
9. Kastelic D, Frkovic-Grazio S, Baty D, Truan G, Komel R, Pompon D. A single-step procedure of recombinant library construction for the selection of efficiently produced llama VH binders directed against cancer markers. J Immunol Methods. 2009;350(1-2):54-62. doi: 10.1016/j.jim.2009.08.016. [ Links ]
10. Deckers N, Saerens D, Kanobana K, Conrath K, Victor B, Wernery U et al. Nanobodies, a promising tool for species-specific diagnosis of Taenia solium cysticercosis. International journal for parasitology. 2009;39(5):625-33. [ Links ]
11. Sallam MA. Anti-Schistosoma single-domain antibody-nanoparticles conjugate: A novel tool for diagnostic and therapeutic applications [Tesis Doctoral]. El Cairo: School of Sciences and Engineering, The American University in Cairo; 2012. [ Links ]
12. Fernandes CF, Pereira SDS, Luiz MB, Zuliani JP, Furtado GP, Stabeli RG. Camelid Single-Domain Antibodies as an Alternative to Overcome Challenges Related to the Prevention, Detection, and Control of Neglected Tropical Diseases. Front Immunol. 2017;8:653. doi: 10.3389/fimmu.2017.00653. [ Links ]
13. Vu KB, Ghahroudi MA, Wyns L, Muyldermans S. Comparison of llama VH sequences from conventional and heavy chain antibodies. Mol Immunol. 1997;34(16-17):1121-31. [ Links ]
14. Espino AM, Finlay CM. Sandwich enzyme-linked immunosorbent assay for detection of excretory secretory antigens in humans with fascioliasis. J Clin Microbiol. 1994;32(1):190-3. [ Links ]
15. Ubeira FM, Muiño L, Valero MA, Periago MV, Pérez-Crespo I, Mezo M, et al. MM3-ELISA detection of Fasciola hepatica coproantigens in preserved human stool samples. Am J Trop Med Hyg. 2009;81(1):156-62. [ Links ]
16. Mas-Coma S, Bargues MD, Valero MA. Diagnosis of human fascioliasis by stool and blood techniques: update for the present global scenario. Parasitology. 2014;141(14):1918-46. doi: 10.1017/S0031182014000869. [ Links ]
17. Van der Linden RHJ, Frenken LGJ, De Geus B, Harmsen MM, Ruuls RC, Stok W, et al. Comparison of physical chemical properties of llama VHH antibody fragments and mouse monoclonal antibodies. Biochim Biophys Acta. 1999;1431(1):37-46. [ Links ]
18. Jefferies JR, Campbell AM, van Rossum AJ, Barrett J, Brophy PM. Proteomic analysis of Fasciola hepatica excretory-secretory products. Proteomics. 2001;1(9):1128-32. [ Links ]
19. Cordova M, Herrera P, Guerra H, Espinoza JR. Fasciola hepatica cysteine proteinases: immunodominant antigens in human fasciolosis. Am J Trop Med Hyg. 1997;57(6):660-6. [ Links ]
Correspondencia: Dr. Jose R. Espinoza
Laboratorios de Investigación y Desarrollo, Universidad Peruana Cayetano Heredia.
Dirección: Av. Honorio Delgado 430, San Martin de Porres, Lima 31, Perú.
Email: jose.espinoza@upch.pe
Recibido: 04/09/2018
Aprobado: 28/11/2018
En línea: 21/12/2018