Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.35 no.4 Lima oct./dic 2018
http://dx.doi.org/10.17843/rpmesp.2018.354.3646
ORIGINAL BREVE
Especies rickettsiales en casos humanos con síndrome febril agudo inespecífico en Perú
Rickettsial species in human cases with non-specific acute febrile syndrome in Peru
Rosa Palacios-Salvatierra 1,a, Omar Cáceres-Rey 1,b, Andrés Vásquez-Domínguez 1,c, Patricia Mosquera-Visaloth 1,d, Elizabeth Anaya-Ramírez 1,a
1 Centro Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
a Bióloga, Magister en Salud Pública; b Biólogo, Magister en Biología Molecular; c Licenciado en Biología; d Técnica de Laboratorio.
RESUMEN
Con el objetivo de caracterizar molecularmente aislamientos rickettsiales procedentes de humanos con síndrome febril agudo inespecífico se realizó un estudio descriptivo transversal, con aislamientos propagados en cultivos celulares Vero ATCC y líneas alternativas, verificando viabilidad mediante Inmunofluoresencia Indirecta (IFI). Previa extracción del ADN, se amplificó el gen gltA mediante PCR convencional, y se analizó su secuencia. Doce aislamientos fueron amplificados, cinco con suficiente ADN para secuenciarlos, evidenciando compatibilidad con R. asembonensis en cuatro, y estrecha identidad con Coxiella burnetti en uno. Al menos tres de siete líneas celulares alternativas mostraron rendimiento significativo en sub cultivos. Se identificó R. asembonensis en cuatro aislamientos de humanos con síndrome febril agudo inespecífico, procedentes de las regiones de Ayacucho, Cajamarca y Madre de Dios en Perú, y Coxiella burnetti en uno procedente de la región Loreto.
Palabras clave: Rickettsia; Reacción en Cadena de la Polimerasa; Perú (fuente: DeCS BIREME).
ABSTRACT
With the objective of molecularly characterizing rickettsial isolates from humans with non-specific acute febrile syndrome, a cross-sectional descriptive study was conducted, with isolates propagated in Vero ATCC cellular cultures and alternative lines, verifying the viability by means of Indirect Immunofluorescence. Prior to DNA extraction, the gltA gene was amplified by means of conventional PCR, and its sequence was analyzed. Twelve isolates were amplified, five with sufficient DNA so as to sequence them, exhibiting compatibility with R. asembonensis in four, and a close identity with Coxiella burnetti in one. At least three of seven alternative cellular lines showed significant yield in sub-cultures. R. asembonensis was identified in four isolates of humans with non-specific acute febrile syndrome, coming from the regions of Ayacucho, Cajamarca, and Madre de Dios in Peru, and Coxiella burnetti in one coming from the Loreto region.
Keywords: Rickettsia; Polymerase Chain Reaction; Peru (source: MeSH NLM).
INTRODUCCIÓN
El síndrome febril agudo inespecífico (SFAI) ocurre frecuentemente en Perú. Un grupo involucrado, aunque con relativamente baja prevalencia, pero significativa trascendencia, son las Rickettsias, una de las zoonosis más antiguas conocidas y un paradigma ideal para entender las infecciones emergentes y re emergentes, en especial las asociadas a transmisión por vectores. Por mucho tiempo, la mayor parte de especies rickettsiales eran consideradas no patogénicas, pero recientemente a raíz del empleo de técnicas moleculares y procedimientos de cultivo celular para aislamiento y caracterización, se detectan en todo el mundo Rickettsias asociadas con infecciones humanas y también especies nuevas de patogenicidad desconocida (1).
El género Rickettsia pertenece a la Clase α-Proteobacteria, Orden Rickettsiales; Familia Rickettsiaceae. Son pequeñas bacterias cocobacilares gram negativas, estrictamente intracelulares. Son transmitidas a través de ectoparásitos como pulgas, piojos, garrapatas o ácaros. Se ha propuesto su clasificación en base al análisis filogenético detallado de características genéticas y antigénicas de aproximadamente 20 especies, en cuatro grupos: Grupo Ancestral (Rickettsia bellii y Rickettsia canadensis; asociado con garrapatas); Grupo Tifus (R. prowazeckii y R.typhi; asociado con piojos y pulgas respectivamente); Grupo Fiebres Manchadas (R.rickettsii, Rickettsia parkeri, Rickettsia rhipicephali, Rickettsia honei, entre otras, siendo el más numeroso, todas asociadas con garrapatas) y Grupo Transicional (Rickettsia akari, Rickettsia australis y Rickettsia felis; asociado con garrapatas, pulgas y ácaros) (2).
Las principales manifestaciones clínicas del síndrome rickettsial en humanos incluyen fiebre, rash y presencia de escara, y pueden producirse combinaciones; no obstante, no son patognomónicas (3). En Perú, por evidencia serológica (4) se reconoce a la enfermedad causada por Rickettsias similar a SFAI, mayormente sin rash ni escara. Las especies patogénicas circulantes en la actualidad son prácticamente desconocidas por la escasez de estudios para caracterizar molecularmente aislamientos. Resulta importante dilucidar la participación de especies patogénicas antiguas y nuevas de Rickettsias durante casos o brotes de SFAI, para el diagnóstico diferencial con otras zoonosis virales o bacterianas prevalentes, asociadas a transmisión por vectores, tales como dengue, leptospirosis entre otras. El objetivo del presente estudio es caracterizar molecularmente los aislamientos rickettsiales procedentes de humanos en Perú.
EL ESTUDIO
Se realizó un estudio descriptivo, transversal que incluyó aislados rickettsiales crio preservados obtenidos de muestras sanguíneas humanas con diagnóstico de SFAI, que llegaron al Laboratorio de Metaxénicas Bacterianas del Instituto Nacional de Salud durante 2009 al 2012. Fueron incluidos aislados positivos por IFI, crio preservados a -80° C, no contaminados y con ADN exitosamente amplificados.
Los aislados fueron propagados en cultivo celular Vero (riñón de Cercopithecus aethiops ATCC CCL-81), de bajo pasaje y también empleando los siguientes cultivos celulares alternativos: DH-82 (macrófagos de Canis familiaris ATCC CRL-10389), C6/36 (estadio larval de Aedes albopictus ATCC CRL-1660), J774A-1 (macrófagos de sarcoma de células reticulares de Mus musculus), THP-1 (monocitos de leucemia aguda de Homo sapiens). Además de cultivos secundarios de piel humana (queratinocitos y fibroblastos) a fin de evaluar sensibilidad a desarrollo de Rickettsias in vitro. La viabilidad de los subcultivos fue verificada por IFI (5). Cada aislado fue propagado en al menos una de siete líneas celulares, ejecutando máximo siete sub cultivos en condiciones de bioseguridad nivel II (6).

Se extrajo en pool ADN bacteriano de cada aislamiento cosechado, luego fue cuantificado en espectrofotómetro y amplificado mediante PCR convencional empleando oligonucleótidos específicos para región objetivo del gen gltA (7). Los productos de PCR fueron purificados y secuenciados en ambas hebras ADN con Kit BigDye Terminator V 3.1 Cycle Sequencing (Applied Biosystems, Foster City, CA, EUA) en termociclador modelo Veriti (Applied Biosystems) siguiendo protocolos estandarizados para tiempo, temperatura de incubación y ciclos (8). Los productos del secuenciamiento fueron purificados utilizando columnas Centrisep; eluidos, secados al vacío y sus sedimentos suspendidos en formamida ultra pura, denaturando a 90° C durante 3 min y colocando inmediatamente en hielo. Los productos finales fueron colocados en secuenciador automático de capilares 3500XL (Applied Biosystems). Los datos fueron procesados mediante Blastn 2.0.
El estudio fue aprobado por el Comité Institucional de Ética en Investigación del Instituto Nacional de Salud.
RESULTADOS
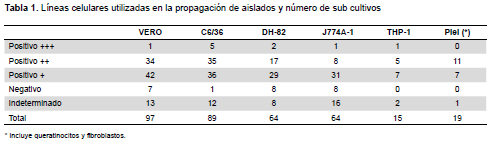
Fueron procesadas 4395 muestras de sangre humana de diferentes regiones del territorio peruano. De las que se extrajo leucocitos para aislar Rickettsias en cultivos celulares, varias de ellas de áreas endémicas como Loreto, Cusco, Cajamarca, Piura, Madre de Dios, Ayacucho, entre otras. Después de cultivarlos, la muestra de positivos fue 117 aislamientos crio preservados. Tomando en cuenta pérdidas por contaminación y no propagación exitosa, 97 fueron la muestra final. En la Tabla 1 se presentan los resultados de la reactivación de aislamientos realizados en líneas celulares y en líneas secundarias según categorías positivos fuertes, débiles y negativos, exhibiendo una distribución normal de frecuencias.
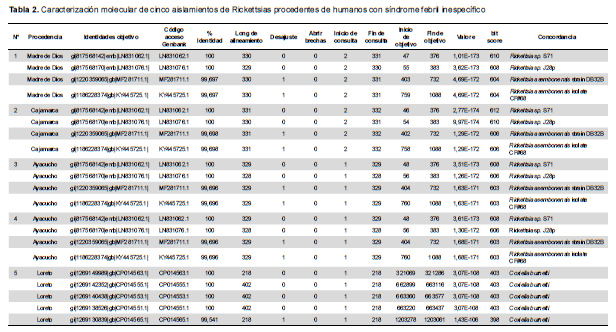
Producto de la corrida de la prueba PCR para gen gltA, resultaron positivos doce aislados, sólo cinco de ellos con suficiente ADN para ser secuenciados. Del análisis molecular de estos cinco aislados secuenciados provenientes de Ayacucho, Cajamarca y Madre de Dios, se obtuvo 100% y 99,7% de identidad en secuencias con Rickettsia asembonensis. Un aislado procedente de Loreto mostró 100% de identidad con Coxiella burnetti (Tabla 2).
DISCUSIÓN
Se reporta el hallazgo de Rickettsia asembonensis en casos de humanos con SFAI en Perú. Estudios previos habían demostrado su alta prevalencia en pulgas, a diferencia de lo que sucede con Rickettsia felis, especie probadamente patogénica para humanos, pero con baja prevalencia en pulgas (9).
Muchas Rickettsias actualmente reconocidas como patógenas para humanos, fueron primero identificadas en artrópodos ectoparásitos, generalmente garrapatas. Años o décadas después se demostró de manera concluyente la asociación con enfermedad humana. Ocho de trece especies o sub especies de Rickettsias del Grupo Fiebres Manchadas fueron confirmadas como patógenas recién desde 1984 (10).
Poder obtener un aislamiento de Rickettsias puede demorar bastante tiempo, porque es reconocido que son microorganismos dificultosos de ser cultivados y propagados (11), especialmente cuando proceden de muestras clínicas primarias de sangre de pacientes con SFAI, con mínima carga microbiana, y más aún cuando son obtenidas fuera del periodo óptimo para aislamiento o durante tratamiento antibiótico (6). Esto es lo que ha constituido una limitante para nuestro estudio, tener que hacer múltiples sub cultivos de reactivación y propagación para cada uno de los aislamientos.
Para diagnóstico rápido de infecciones de este tipo, en que el aislamiento en cultivo se demora y dificulta, han resultado muy útiles las pruebas moleculares con iniciadores específicos para identificar productos amplificados. Sin embargo, es necesario interpretar con precaución los resultados obtenidos (12).
El aislamiento de Candidatus Rickettsia asemboensis ha sido previamente reportado, únicamente en ectoparásitos (9,13-15). Fue encontrada en Carolina del Sur (EE. UU.) en 2009, Egipto en 2006 y 2007, Hungría en 2010, Senegal en 2012, Himalaya en 2015 y Kenia en 2016, siendo referida con dicha denominación al haber sido encontrada infectando pulgas C. felis colectadas en Asembo (9). Igual que R. felis, también R. asembonensis tiene gen ompA, codificando proteínas auto transportadoras rOmpA en superficie, preservadas en el Grupo Fiebre Manchada. Entonces, las células infectadas exhiben antígenos en superficie, reaccionando por IFI con Suero Anti Grupo Fiebre manchada, alto contra R. parkeri y en menor grado con anti-Grupo Tifus, alto contra R. typhi.
Mediante caracterización genotípica de cinco secuencias de genes rickettsiales (rrs, gltA, ompA, ompB, y sca4) la cepa NMRCiiT fue identificada como Rickettsia y clasificada como especie novel con estrecha relación filogenética con Rickettsia felis. Es bacteria intracelular obligada de crecimiento aeróbico en línea celular de Aedes albopictus (C6/36) a 25° C en medio suplementado con 5% de suero bovino fetal inactivado por calor. El tamaño del genoma completo es de 1,46 Mb y 32,28 moles % de guanina y citosina (G+C) (15).
En Brasil se le ha descrito infectando garrapatas R. sanguineus, y ha sido denominada Rickettsia asembonensis cepa KX196267 (16), relacionada con pulgas C. felis infectadas. Existen reportes de resultados PCR positivos para los genes gltA, ompB y 16S rRNA en pulgas en Colombia, donde las secuencias presentan alta identidad (98,5%, 97,4% y 99,2%, respectivamente para cada gen) con R. felis, y demuestran 99.7% y 100% de identidad con gltA y ompB de Candidatus Rickettsia asemboensis (17). Por otro lado, alta homología de secuencias de C. felis con Ca. R. asemboensis, concuerda con recientes hallazgos en pulgas colectadas de perros en Ecuador (13), siendo necesario complementar la caracterización de especies dentro del grupo Rickettsias de Fiebres Manchadas.
R. asembonensis y R. felis son especies muy relacionadas, pero genéticamente diferentes para considerarlas como especies separadas. Probablemente también R. asembonensis pertenezca al Grupo Transicional como R. felis, que ha sido reclasificada. Se requieren estudios filogenéticos para clasificar el grupo de estas nuevas especies; en consecuencia, R. asembonensis debería estar incluida en futuros estudios filogenéticos de Rickettsia spp. para evaluar su distribución en diferentes regiones geográficas (1).
Con relación al otro hallazgo, la Coxiella burnetti pertenece a las Gamma Proteobacterias con alto potencial infeccioso para el ganado y animales silvestres. Exhibe características diferentes a Rickettsiae y puede sobrevivir en diversos medioambientes; como en EE. UU. en el 2010 en ambientes con actividad humana, con prevalencias entre 6 hasta 44% sugiriendo que la exposición humana a Coxiella burnetti puede ser más común de lo que indican los reportes de fiebre Q. Además, en la naturaleza existen muchas especies de mamíferos, aves y garrapatas reservorios de C. burnetti (18). Se diferencian de las rickettsias «típicas» en tamaño, naturaleza pleomórfica, resistencia a altas temperaturas y pH bajo, contenido genómico guanina + citosina, forma de proliferación intracelular, vector transmisor y sensibilidad a antibióticos. Puede infectar a humanos vía inhalación de aerosoles, ingestión de carne y leche, contacto con sangre infectada y vectores. La infección es con frecuencia latente en animales, con dispersión persistente.
Nuestro hallazgo requiere investigación adicional tomando en consideración los factores epidemiológicos involucrados, por ser Loreto área de encuentro de diversas poblaciones. Cabe mencionar que fiebre Q puede manifestarse en humanos como enfermedad febril aguda generalmente auto limitada, con neumonía o hepatitis o como enfermedad crónica en pacientes inmunocomprometidos y gestantes. Coxiella burnetii ha sido detectada en garrapatas en la región americana; un reporte de Argentina lo demuestra (19).
En conclusión, los hallazgos confirman la presencia de especies no reportadas de Rickettsias en humanos durante SFAI en cuatro regiones del territorio peruano durante el periodo 2009 al 2012. Similares estudios en la región confirman la participación rickettsial en la etiología del SFAI (20). La especie Rickettsia asembonensis ha sido previamente encontrada en Sudamérica, infectando ectoparásitos (21-23), esta sería la primera vez que es aislada de muestras humanas en el mismo periodo de estudio en el que fueron encontradas habitando en ectoparásitos, pero en regiones no reportadas, lo cual amerita vigilancia epidemiológica efectiva y estudios adicionales para caracterización completa de especies circulantes.
Contribuciones de los autores: RPS conceptuó, diseñó y redactó el manuscrito, OCR analizó e interpretó los datos en aspectos moleculares e hizo revisión crítica, AVD desarrollo los procedimientos moleculares, PMV realizó los procedimientos de cultivos y propagación de aislamientos. EAR aportó en la revisión crítica. Todos aprobaron la versión final.
Fuentes de financiamiento: El estudio fue financiado por el Instituto Nacional de Salud de Perú, a través del Centro Nacional de Salud Pública.
Conflictos de interés: Los autores declaran no tener conflictos de intereses en la publicación de este artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. Parola P, Paddock CD, Socolovschi C, Labruna MB, Mediannikov O, Kernif T, et al. Update on tick-borne rickettsioses around the world: a geographic approach. Clin Microbiol Rev. 2013;26(4):657-702. doi: 10.1128/CMR.00032-13. [ Links ]
2. Sahni SK, Narra HP, Sahni A, Walker DH. Recent molecular insights into rickettsial pathogenesis and immunity. Future Microbiol. 2013;8(10):1265-88. doi: 10.2217/fmb.13.102. [ Links ]
3. Faccini-Martínez ÁA, García-Álvarez L, Hidalgo M, Oteo JA. Syndromic classification of rickettsioses: An approach for clinical practice. Int J Infect Dis. 2014;28:126-39. doi: 10.1016/j.ijid.2014.05.025. [ Links ]
4. Anaya-Ramirez E, Palacios-Salvatierra R, Mosquera P, Álvarez C, Peralta C, Gonzales R, et al. Prevalencia de anticuerpos a Rickettsias y Ehrlichias en cuatro departamentos fronterizos del Perú. Rev Peru Med Exp Salud Pública. 2017;34(2):268-272. doi: 10.17843/rpmesp.2017.342.1812. [ Links ]
5. Oteo JA, Nava S, Sousa R, Mattar S, Venzal JM, Abarca K, et al. Latinamerican guidelines of RIICER for diagnosis of tick-borne rickettsioses. Rev Chilena Infectol. 2014;31(1):54-65. doi: 10.4067/S0716-10182014000100009. [ Links ]
6. La Scola B, Raoult D. Laboratory diagnosis of rickettsioses: current approaches to diagnosis of old and new rickettsial diseases. J Clin Microbiol. 1997;35(11):2715-27. [ Links ]
7. Santibañez S, Portillo A, Santibáñez P, Palomar AM, Oteo JA. Usefulness of rickettsial PCR assays for the molecular diagnosis of human rickettsioses. Enferm Infecc y Microbiol Clin. 2013;31(5):283-288. [ Links ]
8. Blair PJ, Jiang J, Schoeler GB, Moron C, Anaya E, Cespedes M, et al. Characterization of spotted fever group rickettsiae in flea and tick specimens from northern Peru. J Clin Microbiol. 2004;42(11):4961-4967. [ Links ]
9. Jiang J, Maina AN, Knobel DL, Cleaveland S, Laudisoit A, Wamburu K, et al.Molecular detection of Rickettsia felis and Candidatus Rickettsia Asemboensis in Fleas from Human Habitats, Asembo, Kenya. Vector Borne Zoonotic Dis. 2013;13(8):550-8. doi: 10.1089/vbz.2012.1123. [ Links ]
10. Raoult Didier, Parola Philippe. Rickettsial Diseases, 2007. Infectious Disease and Therapy Book 43. CRC Press; 1st ed, Ed. Kindle; 379 pags.
11. Richards AL. Worldwide detection and identification of new and old rickettsiae and rickettsial diseases. FEMS Immunol Med Microbiol. 2012;64(1):107-10. doi: 10.1111/j.1574-695X.2011.00875.x. [ Links ]
12. Fénollar F, Raoult D. Molecular genetic methods for the diagnosis of fastidious microorganisms. APMIS. 2004;112(11-12):785-807. [ Links ]
13. Oteo JA, Portillo A, Portero F, Zavala-Castro J, Venzal JM, Labruna MB. “Candidatus Rickettsia asemboensis” and Wolbachia spp. in Ctenocephalides felis and Pulex irritans fleas removed from dogs in Ecuador. Parasit Vectors 2014;7:455. https://doi.org/10.1186/s13071-014-0455-0.
14. Jima DD, Luce-fedrow A, Yang Y, Maina AN, Snesrud EC, Otiang E, et al. Strain NMRCii , Isolated from Fleas of Western Kenya. Genome Announc. 2015;3(2). pii: e00018-15. doi: 10.1128/genomeA.00018-15. [ Links ]
15. Maina AN, Luce-Fedrow A, Omulo S, Hang J, Chan TC, Ade F, et al. Isolation and characterization of a novel Rickettsia species (Rickettsia asembonensis sp. nov.) obtained from cat fleas (Ctenocephalides felis). Int J Syst Evol Microbiol. 2016;66(11):4512-4517. doi: 10.1099/ijsem.0.001382. [ Links ]
16. Silva AB, Vizzoni VF, Costa AP, Costa FB, Moraes-Filho J, Labruna MB, et al. First report of a Rickettsia asembonensis related infecting fleas in Brazil. Acta Tropica. 2017;172:44-49. doi: 10.1016/j.actatropica.2017.04.004. [ Links ]
17. Faccini-Martínez AA, Ramírez-Hernandez A, Forero-Becerra E, Cortés-Vecino JA, Escandón P, Rodas JD, et al. Molecular evidence of different Rickettsia species in Villeta, Colombia. Vector Borne Zoonotic Dis. 2016;16(2):85-7. doi: 10.1089/vbz.2015.1841. [ Links ]
18. Kersh GJ, Wolfe TM, Fitzpatrick KA, Candee AJ, Oliver LD, Patterson NE, et al. Presence of Coxiella burnetii DNA in the environment of the United States, 2006 to 2008. Appl Environ Microbiol. 2010; 76(13):4469-75. doi: 10.1128/AEM.00042-10. [ Links ]
19. Pacheco RC, Echaide IE, Alves RN, Beletti ME, Nava S, Labruna MB. Coxiella burnetii in ticks, Argentina. Emerg Infect Dis. 2013; 19(2):344-6. [ Links ]
20. Faccini-Martinez, Ramirez-Hernandez, Barreto C, et al. Epidemiology of Spotted Fever Group Rickettsioses and Acute Undifferentiated Febrile Illness in Villeta, Colombia. Am J Trop Med Hyg. 2017;97(3):782-788. doi: 10.4269/ajtmh.16-0442. [ Links ]
21. Kocher C, Morrison AC, Leguia M, Loyola S, Castillo RM, Galvez HA, et al. Rickettsial disease in the Peruvian Amazon Basin. PLoS Negl Trop Dis, 2016; ;10(7):e0004843. doi: 10.1371/journal.pntd.0004843. [ Links ]
22. Palacios-Salvatierra R, Anaya-Ramírez E, Juscamayta-López J, Cáceres-Rey O, Mendoza-Uribe L, Mosquera-Visaloth P. Perfil epidemiológico y molecular de Rickettsiosis en localidades de frontera peruana. Rev Peru Med Exp Salud Publica. 2017;34(1):76-84. doi: 10.17843/rpmesp.2017.341.2769. [ Links ]
23. Loyola S, Flores-Mendoza C, Torre A, Kocher C, Melendrez M, Luce-Fedrow A, et al. Rickettsia asembonensis Characterization by Multilocus Sequence Typing of complete genes, Peru. Emerg Infect Dis. 2018;24(5):931-933. doi: 10.3201/eid2405.170323. [ Links ]
Correspondencia: Rosa Palacios Salvatierra
Dirección: Laboratorio de Metaxénicas Bacterianas – Instituto Nacional de Salud - Cápac Yupanqui 1400 - Jesús María, Lima 11, Perú.
Teléfono: (511) 748 1111 – 2103
Correo electrónico: rpalaciossalvatierra@gmail.com
Recibido: 30/04/2018
Aprobado: 05/12/2018
En línea: 21/12/2018