Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.35 no.4 Lima Oct./Dec. 2018
http://dx.doi.org/10.17843/rpmesp.2018.354.3755
ORIGINAL BREVE
Caracterización de metalo-β-lactamasas en aislados clínicos de Pseudomonas aeruginosa recuperados de pacientes hospitalizados en el Hospital Militar Central
Characterization of metallo-β-lactamase in clinical isolates of Pseudomonas aeruginosa retrieved from patients hospitalized in the Central Military Hospital
Gina Salvador-Luján1,2,a, Ruth García-de-la-Guarda3,b, Edgar Gonzales-Escalante4,c
1 Laboratorio de Microbiología del Hospital Militar Central. Lima, Perú.
2 Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos. Lima, Perú.
3 Laboratorio de Microbiología Molecular y Biotecnología, Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos. Lima, Perú.
4 Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales (CITBM), Universidad Nacional Mayor de San Marcos. Lima, Perú.
a Bióloga con mención en Microbiología y Parasitología, magíster en Microbiología; b bióloga microbióloga, magíster en Bioquímica; c tecnólogo médico, magíster en Microbiología.
Tesis de la autora Gina Salvador-Luján para optar al grado académico de magíster en Microbiología por la Universidad Nacional Mayor de San Marcos; 2017.
RESUMEN
Con el objetivo de caracterizar y determinar la frecuencia de genes que codifican metalo-β-lactamasas (MBL) en aislados clínicos de Pseudomonas aeruginosa (P. aeruginosa), se realizó un estudio transversal en el Hospital Militar Central (HMC) de Lima, Perú, entre enero y setiembre del 2016. Se analizaron 76 aislados de P. aeruginosa con sensibilidad disminuida a carbapenémicos y resistentes a ceftazidima. La detección fenotípica de MBL se realizó por el método de sinergia de doble disco entre imipenem y meropenem frente al ácido etilendiaminotetraacético (EDTA), y la identificación de los genes por reacción en cadena de la polimerasa. De los 76 aislados, 24 (31,6 %) fueron positivos para MBL por el método fenotípico, confirmándose genéticamente todos. Se encontró el gen blaIMP en 23/24 (95,8 %) y blaVIM en 1/24 (4,2 %). Ningún aislado presentó blaNDM. El gen blaIMP resultó ser el más frecuente entre los aislados clínicos de P. aeruginosa no sensibles a carbapenémicos en el HMC.
Palabras clave: Pseudomonas aeruginosa; Carbapenémicos; Beta-Lactamasas; Resistencia betalactámica; Perú (fuente: DeCS BIREME).
ABSTRACT
hat codify metallo-β-lactamases (MBL) in clinical isolates of Pseudomona aeruginosa (P. aeruginosa), a cross-sectional study was conducted in the Central Military Hospital (HMC) of Lima, Peru, between January and September, 2016. Seventy-six (76) isolates of P. aeruginosa with diminished sensitivity to carbapenemes and resistant to ceftazidime were analyzed. The phenotype detection of MBL was made by double disc synergy between imipenem and meropenem versus ethylenediaminetetraacetic acid (EDTA), and the identification of the genes was performed by polymerase chain reaction. Of the 76 isolates, 24 (31.6%) were positive for MBL by the phenotype method, genetically confirming all. The blaIMP gene was found in 23/24 (95.8%) and blaVIM in 1/24 (4.2%). No isolate had blaNDM. The blaIMP gene turned out to be the most frequent among clinical isolates of P. aeruginosa not sensitive to carbapenemics at this Hospital.
Keywords: Pseudomonas aeruginosa; Carbapenems; Beta-Lactamases; Beta-Lactam resistance, Peru (source: MeSH NLM).
INTRODUCCIÓN
Pseudomonas aeruginosa (P. aeruginosa) es un patógeno oportunista de gran importancia clínica en el ambiente hospitalario. Fue el agente infeccioso más frecuente (16 %) hallado en un hospital nivel IV en Lima (1). Asimismo, es el primer causante de neumonía asociada a la ventilación mecánica en las unidades de cuidados intensivos (UCI) causando elevadas tasas de mortalidad (2,3). Su capacidad de adaptación, diseminación, resistencia intrínseca a los antimicrobianos y su capacidad para adquirir nuevos mecanismos de resistencia hacen difícil el tratamiento de las infecciones causadas por esta bacteria (2,3).
La resistencia intrínseca a los antimicrobianos en P. aeruginosa, se debe principalmente a la presencia de una membrana externa poco permeable, a una enzima β-lactamasa tipo Amp C inducible y a un sistema de bombas de expulsión activa de antimicrobianos (4). Además, este microorganismo tiene una elevada capacidad para adquirir resistencia a los antibióticos denominados antipseudomónicos, incluyendo carbapenémicos, que son antibióticos betalactámicos de amplio espectro y que frecuentemente se usan en el tratamiento de infecciones causadas por P. aeruginosa multirresistentes (MDR).
Actualmente se reporta un incremento de cepas de P. aeruginosa resistentes a carbapenémicos, siendo los principales mecanismos: mutaciones o pérdida de porina OprD, sobreexpresión de bombas de eflujo, la hiperproducción de betalactamasa cromosómica AmpC y la producción de enzimas carbapenemasas (4,6). En P. aeruginosa se han descrito carbapenemasas de clase A, B (metalo-β-lactamasas) y D (oxacilinasas), siendo las metalo-β-lactamasas (MBL) las de mayor prevalencia a nivel mundial. Una de las características de las MBL es que no hidrolizan el aztreonam, por lo cual, no hacen resistencia a este antibiótico, pudiendo ser indicativo de la presencia de estas enzimas (5,6).
Se han descrito varios grupos de MBL y las variantes tipo imipenemasas (IMP), Verona Integron-encoded Metallo-β-lactamase (VIM) y New Delhi Metallo-β-lactamase (NDM) son las de mayor frecuencia e importancia clínica (7,8). Las MBL son transferibles en genes cassettes de integrones y en plásmidos o transposones, además de estar asociados a otros genes de resistencia ubicados en los mismos elementos genéticos móviles, lo cual confiere a las cepas portadoras, resistencia a múltiples antibióticos (2). De allí que las MBL de transmisión plasmídica tienen gran importancia, debido a su difusión mundial entre Pseudomonas spp., Acinetobacter spp., Klebsiella spp. y E. coli (2).

En el Hospital Militar Central (HMC) no se han realizado estudios de resistencia a los carbapenémicos en P. aeruginosa. La producción de MBL es uno de los mecanismos más importantes de resistencia a estas drogas, siendo su caracterización y detección temprana fundamental para mejorar las medidas de control orientadas a evitar posibles brotes de bacterias multirresistentes.
El objetivo del presente estudio es caracterizar y determinar la frecuencia de los genes de MBL en P. aeruginosa recuperadas de pacientes hospitalizados en el HMC.
EL ESTUDIO
Se realizó un estudio transversal. Se analizaron 76 aislados de P. aeruginosa, tomando sólo el primer aislamiento por cada paciente que cumplieron con el criterio de sensibilidad disminuida (halos menores de 23 mm) a los carbapenémicos imipenen o meropenem y resistentes a ceftazidima, recuperados de muestras clínicas de secreción respiratoria (58 %), heridas (21 %), orina (16 %) y sangre (5 %) de los servicios de hospitalización del HMC, entre enero y septiembre del 2016.
La susceptibilidad antimicrobiana se determinó por el método de disco difusión, siguiendo las recomendaciones del Clinical and Laboratory Standars Institute (CLSI) (9). Se incluyeron los discos de antibióticos conteniendo: amikacina 30µg (AMK), aztreonam 30µg (ATM), cefepime 30µg (FEP), ceftazidima 30µg (CAZ), ciprofloxacina 5µg (CIP), gentamicina 10µg (GEN), imipenem 10µg (IPM), meropenem 10µg (MEM) y piperacilina/tazobactam 100/10µg (TZP) (Bioanalyse®). La interpretación se realizó según los puntos de corte del CLSI (9).
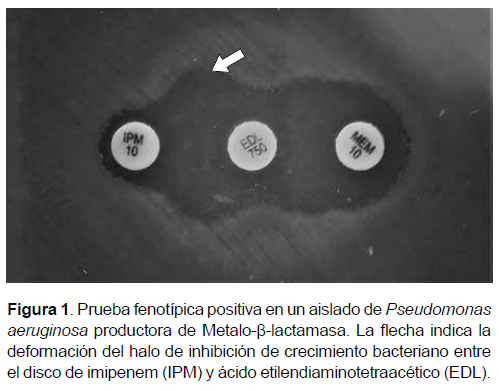
Para la determinación fenotípica se empleó la prueba de sinergia de doble disco descrito por Arakawa et al. (10), que se basa en la propiedad de inhibición de las MBL por agentes quelantes de metales como el ácido etilendiaminotetraacético (EDTA) (7,10). Las placas se inocularon según las recomendaciones del CLSI (9) y los discos de EDTA (Bioanalyse®), IPM y MEM se colocaron a una distancia de 15 mm, de centro a centro, entre los discos con antibiótico y el inhibidor (2,11). Las placas se incubaron por 18 h a 35 °C. Luego se realizó la lectura y se interpretó como posible presencia de MBL cuando existía un agrandamiento o distorsión entre los halos de inhibición de los discos de IPM o MEM hacia el disco de EDTA (10,11).
La detección genotípica de MBL se ejecutó en el Laboratorio de Epidemiología Molecular y Genética del Instituto de Medicina Tropical «Daniel A. Carrión» de la Universidad Nacional Mayor de San Marcos. Se utilizó ADN total como molde. Se amplificaron los genes codificantes de las MBL blaIMP y blaVIM por el método de reacción en cadena de la polimerasa (PCR) múltiple según Ellington et al. (12) y el gen blaNDM por PCR simple según Pasteran et al. (13) (Tabla 1).
Se reportan las frecuencias y porcentajes con sus respectivos intervalos de confianza al 95% (IC 95%). Se utilizaron los programas estadísticos SPSS versión 21 y Excel de Windows.
El protocolo del estudio fue aprobado por el Comité de Ética del HMC. Todos los procedimientos del presente estudio tratan de preservar la integridad y los derechos fundamentales de los pacientes sujetos a investigación, de acuerdo con los lineamientos de las buenas prácticas clínicas y de ética en investigación biomédica.
HALLAZGOS
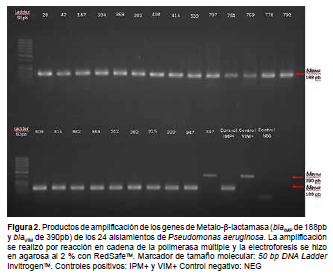
De los 76 aislados de P. aeruginosa, 24 (31,6 %) (IC 95%: 26,2 % - 36,9 %) fueron productores de MBL según la prueba fenotípica, estos mostraron sinergia (Figura 1) con al menos uno de dos carbapenémicos (IPM, MEM). La detección genotípica confirmó que los 24 aislados portaban genes codificantes de MBL, de los cuales 23 (95,8 %, IC 95%: 91,7 % - 99,9 %) presentaron el gen blaIMP y uno (4,2 %, IC 95%: 0,1 % - 8,3 %) presentó el gen blaVIM. No se detectó el gen blaNDM (Figura 2).

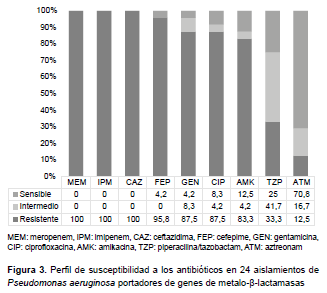
El perfil de susceptibilidad antimicrobiana de los 24 aislados de P. aeruginosa portadores de genes de MBL se muestran en la Figura 3.
La frecuencia de aislados de P. aeruginosa MBL positivas según las unidades de procedencia fueron: 75,0 % (IC 95%: 70,0 % - 80,0 %) de medicina, 12,5 % (IC 95%: 8,7 % - 16,3%) de cuidados intensivos y 4,2 % (IC 95%: 1,9 % - 6,5 %) de cirugía, neumología y ortopedia-traumatología.
La frecuencia de aislados con P. aeruginosa MBL positivas según tipo de muestra fueron: 54,2 % (IC 95%: 48,4 % - 60,0 %) de secreción respiratoria, 29,2 % (IC 95%: 24,0 % - 34,4 %) de herida, 12,5 % (IC 95%: 8,7 % - 16,3 %) de orina y 4,2 % (IC 95%: 1,9 % - 6,5 %) de sangre.
DISCUSIÓN
De las 76 cepas seleccionadas se encontró que 24 (31,6 %) de aislamientos, fueron positivos para la detección fenotípica de carbapenemasas tipo MBL, las cuales se confirmaron molecularmente como portadoras de genes codificantes de MBL, siendo el gen blaIMP el más frecuente (95,8 %), seguido del gen blaVIM (4,2 %), no detectándose el gen blaNDM.
La frecuencia de genes MBL hallada supera el 21,7 % reportado en el Instituto Nacional de Salud del Niño (14), de los cuales el 92 % fueron genes blaIMP y 8 % genes blaVIM. Además, supera el 18,8 % reportado por Ríos (15) en hospitales de Lima, quien sólo detectó genes blaIMP, no encontrando blaVIM, ni blaNDM. Estos datos sugieren que en los hospitales de Lima circula con mayor frecuencia P. aeruginosa con genes blaIMP y con menor frecuencia genes blaVIM.
En Latinoamérica (16,17), los reportes publicados entre el 2004 y el 2012 indican que los genes de MBL más detectados en P. aeruginosa varían según países, así, en Brasil fueron los genes blaSPM, seguido de blaIMP y blaVIM, en tanto que en Argentina fueron blaIMP seguido de blaVIM, en Chile y Colombia sólo blaVIM y en México blaVIM y blaIMP. En un hospital de Guadalajara (18) se aislaron P. aeruginosa MBL positivas en un 34 %, de las cuales el 85,7 % portaban genes blaIMP.
En Perú, existen pocos estudios sobre la frecuencia de los mecanismos de resistencia, a pesar que Hong et al. (19) y Labarca et al. (17) señalan que, según los reportes de la Organización Panamericana de la Salud del 2010 el Perú muestra una tasa de resistencia al IPM y al MEM en P. aeruginosa de 66 % y 57 %, respectivamente, siendo esta, la más alta de Latinoamérica y una las mayores del mundo.
Según Hong et al. (19), en la mayoría de países, las tasas de P. aeruginosa resistentes a carbapenémicos están en el rango de 10 % a 50 %, encontrando en Canadá y República Dominicana las tasas más bajas (menos de 10 %), en tanto que en Brasil, Perú, Costa Rica, Rusia, Grecia, Polonia, Irán y Arabia Saudita se encuentran las tasas más altas (mas de 50 %). En la India, se aisla con mayor frecuencia P. aeruginosa con genes blaVIM (20).
En el HMC, la frecuencia de MBL en P. aeruginosa pudo haberse incrementado por una presion de selección debido al uso de carbapenémicos, descrito previamente como un factor principal para la aparición de bacterias con nuevos mecanismos de resistencia (18). De los 24 aislamientos de P. aeruginosa MBL positivas, la mayoría procedían de la unidad de medicina (75,0 %), a diferencia de lo hallado en el Instituto Nacional de Salud del Niño (14), en un hospital de Buenos Aires (6) y en un hospital de Zaragoza (4), donde la mayor frecuencia fue reportada en salas de UCI con 46 %, 78 % y 68 %, respectivamente, y en un hospital de Logroño en España donde procedían principalmente del área de cirugía (18 %) (4).
Nuestros resultados indican que el tipo de muestra con mayor frecuencia de P. aeruginosa MBL positiva fue de secreción respiratoria (54,2 %), seguida de herida (29,2 %), orina (12,5 %) y sangre (4,2 %), demostrando que, en la unidad de medicina, las afecciones más frecuentes son las respiratorias, debido probablemente, al uso prolongado de ventilación mecánica. Esto coincide con lo reportado por Hidalgo et al. (1), donde la infección intrahospitalaria más común fue neumonía, asociada principalmente a ventilación mecánica; asimismo, Ríos (15) y Gonzáles Escalante (14) reportaron P. aeruginosa MBL positivas en muestras respiratorias. En general, la frecuencia de P. aeruginosa MBL positiva varía según el tipo de muestra y las distintas unidades hospitalarias (1,4,6,14,15,20).
Todos los aislados productores de MBL fueron MDR, por presentar resistencia a tres familias de antibióticos diferentes. Varios estudios señalan que P. aeruginosa MDR porta genes bla que codifican MBL, localizados en casettes génicos dentro de integrones, transposones o plásmidos, junto con genes que codifican resistencia a otras familias de antibióticos (2,4,7).
Como limitaciones de nuestro estudio podemos mencionar que no se analizaron otros mecanismos de resistencia frente a carbapenémicos como la alteración o pérdida de porina OprD, sobreexpresión de bombas de eflujo o hiperproducción de AmpC. Además, es necesario el secuenciamiento para conocer las variantes alélicas de estas enzimas y su codificación genética, que nos permita saber si existen otros genes de resistencia relacionados.
De los resultados obtenidos, se concluye la existencia de enzimas MBL tipo IMP en cepas de P. aeruginosa aisladas de pacientes hospitalizados en el HMC, siendo más frecuentes en la unidad de medicina y en las muestras de secreciones respiratorias. Además, la frecuencia reportada de MBL supera a otros estudios en hospitales de Lima. Este es el primer reporte de MBL en el HMC, que es relevante porque advierte el hallazgo del gen blaIMP, de fácil transferencia entre bacilos gramnegativos. Esperamos que los hallazgos del presente estudio permitirán mejorar los protocolos del programa de control de infecciones nosocomiales, así como fortalecer la vigilancia epidemiológica de bacterias multirresistentes y prevenir su diseminación.
Contribuciones de los autores: GSL y RGG: han participado en la concepción, diseño, recolección de muestras, análisis e interpretación de datos, redacción y revisión crítica del artículo. EGE participó en el análisis molecular y revisión crítica del artículo. Todos los autores revisaron y aprobaron la versión final del manuscrito.
Fuentes de financiamiento: Autofinanciado
Conflictos de interés: Los autores declaran no tener ningún conflicto de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. Hidalgo LF, Marroquín JE, Antigoni J, Samalvide F. Prevalencia de infecciones hospitalarias en un hospital peruano de nivel IV, en el año 2008. Rev Med Hered. 2011;22(2):76-81. doi: 10.20453/rmh.v22i2.1106. [ Links ]
2. Nicolau CJ, Oliver A. Carbapenemasas en especies del género Pseudomonas. Enferm Infecc Microbiol Clin. 2010;28(Supl 1):19-28. [ Links ]
3. Vilar-Compte D, Jacquemin B, Díaz-González A, Velásquez C, Volkow P. Brote por Pseudomonas aeruginosa, en el área de atención ambulatoria de heridas quirúrgicas, en pacientes posmastectomizadas. Salud pública Méx. 2003;45(5):371-78. [ Links ]
4. Estepa V. Caracterización de mecanismos de resistencia a carbapenémicos, integrones y tipificación molecular en cepas de Pseudomonas aeruginosa de diferentes orígenes. [Tesis Doctoral]. Logroña: Universidad de la Rioja; 2014. [ Links ]
5. Gutkind GO, Di Conza J, Power P, Radice M. β-lactamase-mediated resistance: a biochemical, epidemiological and genetic overview. Curr Pharm Des. 2013;19(2):164-208. [ Links ]
6. Cejas D, Almuzara M, Santella G, Tuduri A, Palombarani S, Figueroa S, et al. Caracterización fenotípica y genotípica de la resistencia a imipenem en Pseudomonas aeruginosa aisladas en un hospital de Buenos Aires. Rev argent microbiol. 2008;40(4):238-45. [ Links ]
7. Radice M, Marin M, Giovanakis M, Vay C, Almuzara M, Limansky A. et al. Criterios de ensayo, interpretación e informe de las pruebas de sensibilidad a los antibióticos en los bacilos gram negativos no fermentadores de importancia clínica: recomendaciones de la subcomisión de antimicrobianos de la SADEBAC- AAM. Rev argent microbiol. 2011;43:136-56. [ Links ]
8. Togneri A, Gómez A, Podestá L, Pérez M, Faccone D, Ríos L, et al. Diseminación de blaIMP-8 en enterobacterias aisladas en un hospital de Buenos Aires. Rev Argent Microbiol. 2013;45(2):104-09. [ Links ]
9. CLSI. Performance standards for antimicrobial susceptibility testing. 26th ed. CLSI supplement. M100. Wayne, PA: Clinical and Laboratory Standards Institute; 2016. [ Links ]
10. Arakawa Y, Shibata N, Shibayama K, Kurokawa H, Yagi T, Fujiwara H, et al. Convenient Test for screening Metallo-β-lactamase-producing gram-negative bacteria by using thiol compounds. J Clin Microbiol. 2000;38(1):40-43. [ Links ]
11. Instituto Nacional de Enfermedades Infecciosas. Pseudomonas spp. Colocación estratégica de discos. Buenos Aires: INEI-ANLIS 2013. Disponible en: http://antimicrobianos.com.ar/ATB/wp-content/uploads/2013/01/PAE.pdf. [ Links ]
12. Ellington M, Kistler J, Livermore D, Woodford N. Multiplex PCR for rapid detection of genes encoding acquired metallo-β-lactamases. J Antimicrob Chemother. 2007;59(2):321-2. doi: 10.1093/jac/dkl481. [ Links ]
13. Pasteran F, Albornoz E, Faccone D, Gomez S, Valenzuela C, Morales M, et al. Emergence of NDM-1-producing Klebsiella pneumoniae in Guatemala. J Antimicrob Chemother. 2012;67(7):1795-7. doi: 10.1093/jac/dks101. [ Links ]
14. Gonzales Escalante E. Detección y caracterización molecular de metalo-β-lactamasas en aislamientos de Pseudomonas aeruginosa recuperados en el Instituto Nacional de Salud del Niño Lima-Perú [Internet]. Lima: Instituto Nacional de Salud del Niño; 2013. Disponible en: http://www.insn.gob.pe/sites/default/files/investigaciones/desarrollo/informes/2018/Informe%20Final%20TO-02-2011.pdf. [ Links ]
15. Ríos P. Frecuencia de los genes blaIMP, blaVIM y blaNDM productores de metalo-b-Lactamasas en aislamientos de Pseudomonas aeruginosa no sensibles a Carbapenems en Lima-Perú [Tesis para optar licenciatura en Tecnología Médica]. Lima: Universidad Nacional Mayor de San Marcos. 2013. Disponible en: http://cybertesis.unmsm.edu.pe/bitstream/handle/cybertesis/5425/Rios_sp.pdf?sequence=1&isAllowed=y.
16. Saavedra S, Duarte C, Gonzales M, Realpe M. Caracterización de aislamientos de Pseudomonas aeruginosa productores de carbapenemasas de siete departamentos de Colombia. Biomédica. 2014; 34(Supl1):217-23. doi:10.7705/biomedica.v34i0.1685. [ Links ]
17. Labarca JA, Salles MJ, Seas C, Guzmán-Blanco M. Carbapenem resistance in Pseudomonas aeruginosa and Acinetobacter baumannii in the nosocomial setting in Latin America. Crit Rev Microbiol. 2016;42(2):276-92. doi: 10.3109/1040841X.2014.940494. [ Links ]
18. Hernandez E. Identificación de metalobetalactamasas (MBL) en aislamientos clínicos de Pseudomonas aeruginosa resistentes a carbapenemes en el Hospital Civil de Guadalajara, Jalisco. [Tesis para obtener el título de licenciado en química clínica]. Xalapa: Universidad Veracruzana. 2005. Disponible en: http://www.remeri.org.mx/tesis/INDIXE-TESIS.jsp?id=oai:cdigital.uv.mx:123456789/35584. [ Links ]
19. Hong DJ, Bae IK, Jang IH, Jeong SH, Kang HK, Lee K. Epidemiology and Characteristics of Metallo-β-Lactamase-Producing Pseudomonas aeruginosa. Infect Chemother. 2015;47(2):81-97. doi: 10.3947/ic.2015.47.2.81. [ Links ]
20. Arunagiri K, Sekar B, Sangeetha G, John J. Detection and characterization of metallo-β-lactamases in Pseudomonas aeruginosa by phenotypic and molecular methods from clinical samples in a tertiary care hospital. West Indian Med J. 2012;61(8):778-83. [ Links ]
Correspondencia: Gina Salvador-Luján.
Dirección: Laboratorio de Microbiología del Hospital Militar Central. Av. Faustino Sánchez Carrión s/n Jesús María. Lima, Perú.
Teléfono: (511) 997395057
Correo electrónico: gsalvador3@hotmail.com
Recibido: 23/06/2018
Aprobado: 28/11/2018
En línea: 21/12/2018