Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.36 no.2 Lima jun./set. 2019
http://dx.doi.org/10.17843/rpmesp.2019.362.3960
ORIGINAL BREVE
Marcadores de resistencia plasmídica a quinolonas qnr en aislamientos clínicos de enterobacterias productoras de betalactamasas CTX-M en Lima, Perú
Markers of plasmid resistance to qnr quinolones in clinical isolates of CTX-M beta-lactamases-producing enterobacteria in Lima, Peru
Lesdyth Jessica Toribio Arias1,a, Carlos Raúl Sevilla Andrade1,2,b, Edgar Gonzales-Escalante2,3,a,b
1 Escuela de Tecnología Médica, Universidad Nacional Mayor de San Marcos. Lima, Perú.
2 Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales - CITBM, Universidad Nacional Mayor de San Marcos. Lima, Perú.
3 Instituto Nacional de Salud del Niño. Lima, Perú.
a Tecnólogo médico, b magíster en Microbiología.
El presente estudio forma parte de la tesis de Lesdyth Jessica Toribio Arias para obtener la licenciatura en Tecnología Médica con mención en Laboratorio Clínico y Anatomía Patológica por la Universidad Nacional Mayor de San Marcos.
RESUMEN
Con el objetivo de reportar marcadores de resistencia plasmídica a quinolonas qnr en aislamientos clínicos de enterobacterias productoras de betalactamasas CTX-M, se realizó un estudio descriptivo, con aislamientos del cepario del proyecto TO-06/09 del Instituto Nacional de Salud del Niño. Se recuperaron 138 aislamientos. La susceptibilidad antimicrobiana se determinó por el método de disco difusión y la identificación de genes por reacción en cadena de la polimerasa. De los 138 aislados, 67 (48,5%) fueron positivos para proteínas qnr por el método genotípico. De los cuales 38 (56,7%) presentaron determinantes qnrB y 48 (71,6%) determinantes qnrS. Ningún aislado presentó determinantes qnrA. Se detectó determinantes qnr en aislamientos que presentaban betalactamasas CTX-M en una población no expuesta.
Palabras clave: Farmacorresistencia bacteriana; Quinolonas; Betalactamasas; Enterobacteriaceae (fuente: DeCS BIREME).
ABSTRACT
Aimed at reporting markers of plasmid resistance to qnr quinolones in clinical isolates of CTX-M beta-lactamase-producing enterobacteria, a descriptive study was conducted with isolates from the strain repository of TO-06/09 project of the National Children´s Health Institute. 138 isolates were recovered. Antimicrobial susceptibility was determined by the diffusion disk method, and gene identification by polymerase chain reaction. Of the 138 isolates, 67 (48.5%) were genotypically positive for qnr proteins; of these, 38 (56.7%) had qnrB determinants and 48 (71.6%) had qnrS determinants. No isolate presented qnrA determinants. qnr determinants were detected in isolates containing CTX-M beta-lactamases in a non-exposed population.
Keywords: Bacterial drug resistance; Quinolones; Beta-lactamases; Enterobacteriaceae (source: MeSH NLM).
INTRODUCCIÓN
La diseminación mundial de microorganismos resistentes a los antimicrobianos es un problema en la atención en salud que disminuye las opciones disponibles para el tratamiento apropiado. No es sorprendente que la propagación mundial de la resistencia a los medicamentos sea reconocida como una gran amenaza para la salud humana, siendo los betalactámicos y las quinolonas, dos de los grupos de antimicrobianos más utilizados en la terapia frente a infecciones por bacterias gram negativas (1).
La producción de β-lactamasas de espectro extendido (BLEE) es el mecanismo de resistencia más importante frente a cefalosporinas de tercera generación, siendo las enzimas de tipo CTX-M predominantes a nivel mundial (2).
Por el lado de las quinolonas, la resistencia se presenta típicamente como resultado de alteraciones en las enzimas diana (ADN girasa y topoisomerasa IV), transferencia de plásmidos, mutaciones en las porinas y sistemas de expulsión. El tipo de resistencia por mutaciones de carácter cromosómico permite la transferencia vertical de resistencia a la descendencia del microorganismo, como un principio de carácter hereditario establecido por mutaciones en las regiones QRDR (quinolone-resistance determinig region) (3,4).
Entre los determinantes de resistencia a quinolonas mediada por plásmidos (PMQR) y que disminuyen su efecto terapéutico, tenemos a las proteínas qnr (quinolone resistance: qnrA, qnrB, qnrC, qnrD, qnrS y qnrVC), enzima aac (6’)-Ib-cr, y bombas de expulsión QepA y OqxAB, que brindan resistencia de transmisión horizontal a través de plásmidos favoreciendo una mayor diseminación a otras bacterias receptoras que lo carecen. Aunque estos marcadores sólo proporcionan resistencia a quinolonas de bajo nivel por sí mismos, se ha demostrado in vitro que facilitan la selección de mutaciones cromosómicas en QRDR (5).
Por consenso se define al gen qnr como un alelo de origen natural que codifica un pentapéptido repetido que confiere susceptibilidad reducida a quinolonas; in vitro ha demostrado proteger la ADN girasa y la topoisomerasa IV por reducción del número de sitios blanco (para quinolonas) al complejo enzima-ADN (bacteriano) (6). Asimismo, la existencia de un vínculo reconocido entre la resistencia a quinolonas y las BLEE en enterobacterias, se relaciona a vehículos genéticos denominados integrones (7).
El objetivo de este estudio fue reportar marcadores de resistencia plasmídica a quinolonas qnr en aislamientos clínicos de enterobacterias productoras de betalactamasas CTX-M.
EL ESTUDIO
Se realizó un estudio descriptivo donde se estudiaron enterobacterias productoras de betalactamasas CTX-M (recolectadas entre agosto de 2012 y enero de 2013) del cepario obtenido del proyecto TO-06/09 «Detección y caracterización molecular β-lactamasas de expectro extendido en E. coli y K. pneumoniae aisladas en el Instituto Nacional de Salud del Niño». Se recuperaron 138 aislamientos consecutivos no repetidos, obtenidos de muestras de orina, secreciones y sangre, provenientes de pacientes pediátricos de los servicios de hospitalización y consultorio externo.
La identificación se realizó mediante pruebas bioquímicas convencionales, mientras que la susceptibilidad antimicrobiana se determinó por el método de disco difusión, siguiendo las recomendaciones del Clinical and Laboratory Standars Institute (CLSI) (8).

La detección genotípica se efectuó en el Laboratorio de Epidemiología Molecular y Genética del Instituto de Medicina Tropical «Daniel A. Carrión» de la Universidad Nacional Mayor de San Marcos (UNMSM). Se utilizó ADN total como molde. Se amplificaron los genes codificantes de los genes qnrA y qnrB por el método de reacción en cadena de la polimerasa (PCR) según Youn et al. (9) y el gen qnrS por PCR según Chen et al. (10).
Se reportan las frecuencias y porcentajes para las variables de interés. Se utilizó el programa Microsoft Excel 2010 y el programa WHONET (Word Health Organization Net Versión 5.6).
El protocolo del estudio fue aprobado por la Escuela de Tecnología Médica de la UNMSM. Todos los procedimientos del presente estudio siguen los lineamientos de las buenas prácticas clínicas y de ética en investigación biomédica.
RESULTADOS
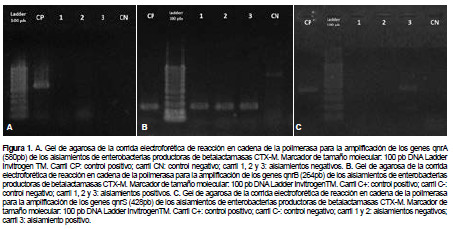
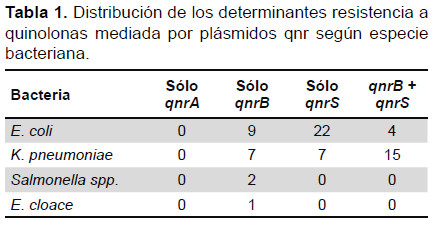
De los 138 aislamientos de enterobacterias productoras de betalactamasas CTX-M, 67 (48,5%) fueron productores de proteínas qnr, de los cuales 38 (56,7%) presentaron determinantes qnrB, distribuidos entre Escherichia coli (13/38), Klebsiella pneumoniae (22/38), Salmonella spp. (2/38) y Enterobacter cloacae (1/38); y 48 (71,6%) presentaron determinantes qnrS, presente en Escherichia coli (26/48) y Klebsiella pneumoniae (22/48); no se encontraron determinantes qnrA. Además, se evidenciaron dos tipos de proteínas qnr en Escherichia coli (4) y Klebsiella pneumoniae (15) (Figura 1) (Tabla 1).
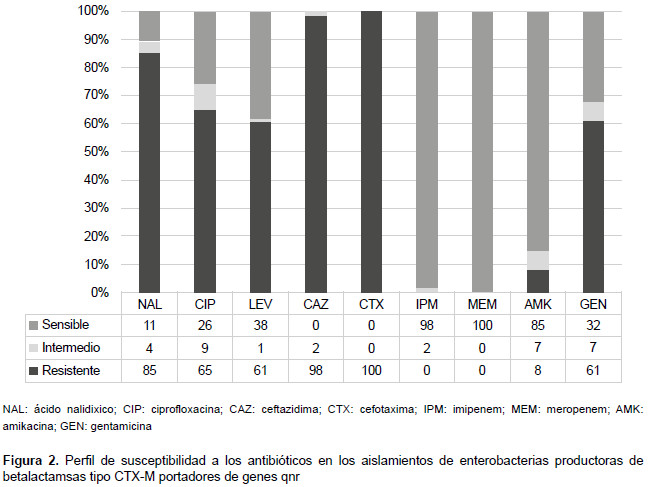
El perfil de susceptibilidad a los antibióticos en los aislamientos de enterobacterias productoras de betalactamsas CTX-M portadores de genes qnr se muestra en la Figura 2.
La frecuencia de aislados productores de genes qnr según procedencia fueron: 37,3% hospitalario y 62,7% comunitario. La frecuencia de aislados productores de genes qnr según tipo de muestra fueron: 77,6% de orina y 22,4% de secreciones.
DISCUSIÓN
A pesar de haber sido demostrada la seguridad del uso de quinolonas en los niños (11), su uso en esta población no es habitual y está limitado según las patologías. Nuestro país no es la excepción, y el uso de quinolonas en niños es poco común. Sin embargo, ya se han informado niveles altos de resistencia a las quinolonas en microorganismos aislados de niños (12).
En nuestro estudio, las enterobacterias productoras de betalactamsas CTX-M presentaron un alto porcentaje de resistencia a quinolonas; similar a lo reportado en Irán en 2011, con una población semejante, donde se encontró un 64% de resistencia a ciprofloxacino (13), y el mismo año en un estudio realizado en México se obtuvo un 26% de resistencia a ciprofloxacino (14).
De los 138 aislamientos seleccionados se encontró 67 (48,5%) de genes qnr, se evidencia un aumento si lo comparamos con el estudio realizado también en Perú el 2012 donde obtuvieron un 14% de genes qnr en enterobacterias productoras de BLEE de diferentes hospitales de Lima (15). Otro estudio también realizado en Perú sobre resistencia a quinolonas en Escherichia coli diarrogénicas y comensales recuperadas de niños (16), reporta la presencia de PMQR, entre ellos qnrB en 8,2% (8/96), a diferencia de nuestro estudio donde observamos qnrB en 27,5% (38/138) de los casos; tenemos que mencionar que los aislamientos del estudio eran portadores de betalactamasas CTX-M, lo que podría indicar una relación directa entre los mecanismos de resistencia frente a quinolonas y betalactámicos mediada por elementos genéticos móviles como integrones o plásmidos (17).
Aunque este estudio se centró en los PMQR, la resistencia a quinolonas se produce principalmente como resultado de mutaciones en los genes cromosómicos que codifican los blancos de quinolonas. Por lo tanto, mutaciones en el gen gyrA en la posición 83 (que conduce a Leu, Val o Ala) son los mecanismos de resistencia a quinolonas más frecuentemente observados (18).
Los genes PMQR confieren niveles bajos de resistencia a quinolonas, estas proteínas cumplen su función de manera proporcional a su concentración y se ha sugerido que facilitan la selección de niveles más altos de resistencia. Además, se ha demostrado que qnrA, qnrB y qnrS pueden jugar un papel importante en la adquisición de resistencia clínica a las quinolonas y, por lo tanto, al fracaso terapéutico en el tratamiento (19).
Asimismo, se encontraron aislamientos que portaban el gen qnrS (5/48) y el gen qnrB, (2/38), que no presentaban resistencia a ninguna quinolona; sin embargo, la sensibilidad fenotípica en estos casos se encontraba disminuida y en el border line. La presencia de un único PMQR generalmente no confiere resistencia de alto nivel a estos antimicrobianos, sino que aumenta el riesgo de desarrollar resistencia a quinolonas. Sin embargo, la diversidad y el número de PMQR en las presentes muestras, junto con la presencia de mutaciones en las regiones QRDR, pueden explicar los altos niveles de resistencia a quinolonas (16).
Adicionalmente, se halló el gen qnrB en dos aislamientos de Salmonella spp., debido a que las quinolonas también son empleadas en uso veterinario, la aparición de genes en bacterias de transmisión zoonótica como la salmonella indicarían un aumento en el uso de este tipo de antimicrobianos en ese campo (20).
Existen limitaciones del estudio que deben ser reconocidas. La epidemiología en otras poblaciones e instituciones podría ser diferente con la de nuestro estudio, ya que analizamos un solo centro; además, no se buscaron otros determinantes PMQR, como otras variantes de los genes qnr, enzimas modificadoras o bombas de flujo. También sería importante el secuenciamiento, para conocer las variantes alélicas, así como evaluar el entorno genético de los plásmidos que portan los genes de resistencia de betalactamasas CTX-M y genes qnr.
En conclusión, se ha demostrado la presencia de determinantes de PMQR involucrados en la resistencia a quinolonas en aislamientos que presentaban betalactamasas CTX-M en una población no expuesta. Lo que indicaría una relación entre las betalactamasas y las PMQR, por lo menos a nivel de las proteínas qnr; esto nos ayuda a conocer la situación de la epidemióloga de la resistencia bacteriana en nuestro medio.
Contribuciones de autoría: Todos los autores han participado en la idea de la investigación, concepción del artículo, la recolección de datos, material de estudio y redacción del artículo. Todos los autores aprobaron la versión final del manuscrito.
Fuentes de financiamiento: Fondo de Promoción de Trabajos de Tesis financiado por la Facultad de Medicina de la Universidad Nacional Mayor de San Marcos
Conflictos de interés: Los autores declaran no tener conflicto de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. Organización Panamericana de la Salud. Informe Anual de la Red de Monitoreo/Vigilancia de la Resistencia a los Antibióticos. Rev de patolog tropica. 2011;40(Supl. 1):1-114. doi: 10.5216/rpt.v40i1.17413. [ Links ]
2. Cantón R, González-Alba J, Galán J. CTX-M enzymes: origin and diffusion. Front Microbiol. 2012;3:110. doi: 10.3389/fmicb.2012.00110 [ Links ]
3. Cattoir V, Nordmann P. Plasmid –mediated quinolone resistance in Gram- negative bacterial species: An update. Curr med Chem. 2009;16(8):1028-1046. doi : 10.2174/092986709787581879. [ Links ]
4. Chen F, Lauderdale T, Ho M. The roles of mutations in gyrA, parC, and OmpK35 in fluoroquinolone resistance in Klebsiella pneumoniae. Microb Drug Resist. 2003;9(3):265-271. doi:10.1089/107662903322286472. [ Links ]
5. Rodríguez-Martínez JM, Cano ME, Velasco C, Martínez-Martínez L, Pascual A. Plasmid-mediated quinolone resistance: an update. J Infect Chemother. 2011;17(2):149-82. doi: 10.1007/s10156-010-0120-2. [ Links ]
6. Kim H, Park C, Kim C, Kim E, Jacoby G, Hooper D. Prevalence of plasmid – mediated quinolone resistance determinants over a 9-year period. Antimicrob Agents Chemother. 2009;53(2):639-645. doi: 10.1128/AAC.01051-08. [ Links ]
7. Martínez-Martínez L. Asociación de BLEE con otros mecanismos de resistencia. Enferm Infecc Microbiol Clin. 2007;25(2):38-47. [ Links ]
8. Clinical and Laboratory Standard Institute. Performance Standards for Antimicrobial Susceptibility Testing. Twenty-eight Informational Supplement M100-S24 [Internet]. Wayne, Pennsylvania: Clinical and Laboratory Standard Institute; 2014 [citado el 1 de octubre de 2018] [ Links ].
9. Youn S, Chul K, Woo J, Hoon J, Hyun Y. Characteristics of aac(6')-Ib-cr Gene in Extended-Spectrum b-Lactamase Producing Escherichia coli and Klebsiella pneumoniae Isolated from Chungnam Area. Korean J Lab Med. 2009;29(6):541- 50. doi: 10.3343/kjlm.2009.29.6.541 [ Links ]
10. Chen X, Zhang W, Pan W, Yin J, Pan Z, Gao S, Jiao X. Prevalence of qnr, aac(6=)-Ib-cr, qepA, and oqxAB in Escherichia coli Isolates from Humans, Animals, and the Environment. Antimicrob Agents Chemother. 2012;56(6):3423-342. doi: 10.1128/AAC.06191-11 [ Links ]
11. Murray TS, Baltimore RS. Pediatric uses of fluoroquinolone antibiotics. Pediatr Ann 2007;36(6):336–42. [ Links ]
12. Ochoa TJ, Ruiz J, Molina M et al. High frequency of antimicrobial drug resistance of diarrheagenic Escherichia coli in infants in Peru. Am J Trop Med Hyg. 2009;81(2):296–301. [ Links ]
13. Pakzad I, Ghafourian S, Sadeghifard N, Abtahi H, Rahbar M, Mansory Jamshidi N. qnr Prevalence in Extended Spectrum Beta-lactamases (ESBLs) and None- ESBLs Producing Escherichia coli Isolated from Urinary Tract Infections in Central of Iran. Iran J Basic Med Sci. 2011;14(5):458-464. [ Links ]
14. Pavón S, Zalazar M, Morales M, Rojas M. Presencia de β-lactamasas de espectro extendido en enterobacterias aisladas de casos de infección nosocomial. Ciencia ergo sum. 2011;18(2):164-170. [ Links ]
15. Sevilla A, Puray C, Alarcón V, Cabezas S, Guevara D, Valencia B, et al. Caracterización de los mecanismos de resistencia en enterobacterias productoras de BLEE aisladas. Libro de resúmenes: XI congreso de la Sociedad Argentina de Infectología. 2011. Lima, Perú [ Links ].
16. Pons MJ, Mosquito S, Gomes C, Del Valle LJ, Ochoa TJ, Ruiz J. Analysis of quinolone-resistance in commensal and diarrheagenic Escherichia coli isolates from infants in Lima, Peru. Trans R Soc Trop Med Hyg. 2014;108(1):22-8. doi: 10.1093/trstmh/trt106. [ Links ]
17. Escobar A, Porto A, Joris R, Sansevich M, Gutkind G, Di Conza J, Truppia L. Detección de genes qnr en aislamientos de enterobacterias con resistencia simultánea a fluorquinolonas y oximinocefalosporinas. Revista FABICIB. 2010;14(1):39-45. doi: 10.14409/fabicib.v14i1.850. [ Links ]
18. Ruiz J. Mechanisms of resistance to quinolones: Target alterations, decreased accumulation and DNA gyrase protection. J Antimicrob Chemother. 2003;51(5):1109-17. doi: 10.1093/jac/dkg222. [ Links ]
19. Ruiz J, Pons MJ, Gomes C. Transferable mechanisms of quinolone resistance. Int J Antimicrob Agents. 2012;40(3):196–203. doi: 10.1016/j.ijantimicag.2012.02.011. [ Links ]
20. Orden J, De la Fuente R. Repercusiones en la salud pública de la resistencia a quinolonas en bacterias de origen animal. Rev Esp Salud Pública. 2001;75:313-320. [ Links ]
Correspondencia: Edgar Gonzales Escalante
Dirección: Calle José Santos Chocano 199, Ciudad Universitaria. Bellavista. Callao, Perú
Correo electrónico: egones_5@hotmail.com
Recibido: 30/09/2018
Aprobado: 22/05/2019
En línea: 28/06/2019