Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Medicina Experimental y Salud Publica
Print version ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.36 no.2 Lima June/Sep. 2019
http://dx.doi.org/10.17843/rpmesp.2019.362.3901
ORIGINAL BREVE
Susceptibilidad antimicrobiana y mutaciones en el gen ARNr 23S de Helicobacter pylori en pacientes dispépticos
Antimicrobial susceptibility and mutations in the 23S rRNA gen of Helicobacter pylori in dyspeptic patients
Jesús Guzmán1,a, Denis Castillo1,b, Manuel Ojeda2,c, Michel Sauvain3,d
1 Laboratorio Mixto Internacional Andino Amazónico de Química de la Vida, Universidad Peruana Cayetano Heredia. Lima, Perú.
2 Clínica Médica Cayetano Heredia. Lima, Perú
3 UMR 152 PharmaDev, Université de Toulouse. Toulouse, Francia.
a Químico farmacéutico, maestro en Bioquímica y Biología Molecular; b biólogo, maestro en Bioquímica y Biología Molecular; c médico cirujano, especialista en Gastroenterología; d químico farmacéutico, PhD en Farmacoquímica.
RESUMEN
Con el objetivo de evaluar la susceptibilidad antimicrobiana y detectar mutaciones puntuales en el gen ARNr 23S en cepas de Helicobacter pylori se realizó un estudio transversal que incluyó a 95 pacientes con dispepsia atendidos en una clínica privada de Lima. Mediante endoscopía se colectaron biopsias de antro para el aislamiento de cepas de Helicobacter pylori para la evaluación de la susceptibilidad antimicrobiana empleando la técnica de microdilución en caldo. La detección de mutaciones puntuales se desarrolló mediante PCR-RFLP. El porcentaje de infección por Helicobacter pylori fue de 46,3%, se observaron valores de resistencia de 52,3% a claritromicina, 29,6% a metronidazol, 45,5% a levofloxacino y 4,6% a amoxicilina. El porcentaje de mutaciones puntuales A2142G y A2143G asociados a resistencia a claritromicina fue 43,5%. En conclusión, encontramos que las tasas de resistencia antimicrobiana y el porcentaje de cepas de Helicobacter pylori circulantes en una clínica privada de Lima fueron elevadas.
Palabras clave: Infecciones por Helicobacter; Claritromicina; Pruebas de sensibilidad microbiana; Farmacorresistencia microbiana; Perú (fuente: DeCS BIREME).
ABSTRACT
In order to evaluate antimicrobial susceptibility and detect specific mutations in the 23S rRNA gene in Helicobacter pylori strains, a cross-sectional study was performed on 95 patients with dyspepsia treated in a private clinic in Lima. Antrum biopsies were collected by endoscopy for isolation and evaluation of antimicrobial susceptibility using the broth microdilution method. The detection of specific mutations was developed by PCR-RFLP. The percentage of infection by Helicobacter pylori was 46.3%. Resistance values of 52.3% to clarithromycin, 29.6% to metronidazole, 45.5% to levofloxacin, and 4.6% to amoxicillin were observed. The percentage of specific A2142G and A2143G mutations associated with clarithromycin resistance was 43.5%. In conclusion, we found that antimicrobial resistance rates and the percentage of Helicobacter pylori strains circulating in a private clinic in Lima were high.
Keywords: Helicobacter infections; Clarithromycin; Microbial sensitivity tests; Microbial drug resistance; Peru (source: MeSH NLM).
INTRODUCCIÓN
Helicobacter pylori (H. pylori) es la principal especie del género Helicobacter, el cual es un patógeno común en humanos y agente causal de gastritis crónica, úlcera péptica, linfoma tipo MALT y cáncer gástrico. Se estima que la mitad de la población mundial está infectada por H. pylori. Esta prevalencia varia ampliamente entre países y se considera que en los países en desarrollo es superior al 70% mientras que en países desarrollados oscila entre 20% y 50% (1).
Durante la infección crónica con H. pylori, la bacteria no puede ser eliminada por el sistema inmune del hospedero, por lo que para erradicarla se requiere del uso de agentes antimicrobianos. Esta terapia idealmente debe tener una tasa de erradicación superior al 90%, se utiliza una terapia triple o cuádruple que incluye un preparado de bismuto, un inhibidor de la bomba de protones y antimicrobianos, principalmente claritromicina, metronidazol, levofloxacino y/o amoxicilina. Sin embargo, la resistencia antimicrobiana en la actualidad es un importante problema de salud global que conlleva al fallo terapéutico (2).
La eficacia de la erradicación de H. pylori depende de la selección de esquemas de tratamiento idóneos basados en los patrones de resistencia locales. La claritromicina, que es un componente importante en la terapia de erradicación, es también uno de los factores principales que conlleva a fallo en el régimen terapéutico de la infección por H. pylori (3).
El gen de ARNr 23S en H. pylori juega un rol importante en la resistencia a macrólidos. Desde el reporte de Versalovic et al. diversos estudios muestran que el principal mecanismo de resistencia son las mutaciones puntuales en las posiciones A2142G, A2142C y A2143G del gen, correspondiente a la región de la peptidil transferasa en el dominio V del ARNr 23S, principal componente de la subunidad 50S (4,5).
El objetivo del presente estudio es evaluar la susceptibilidad antimicrobiana en cepas de H. pylori aisladas de pacientes peruanos con síntomas de dispepsia que se atendieron en una clínica privada de Lima. Además de realizar la detección de mutaciones puntuales en la región del dominio V del gen de ARNr 23S responsable de conferir resistencia a claritromicina en cepas de H. pylori.
EL ESTUDIO
Se realizó un estudio transversal, que incluyó a 95 pacientes con síntomas de dispepsia, sin diagnóstico previo de H. pylori o que no recibieron tratamiento de erradicación de H. pylori tres meses previos a su reclutamiento en el servicio de gastroenterología de la Clínica Médica Cayetano Heredia de Lima, Perú, entre abril y noviembre del 2015. Los pacientes firmaron de manera voluntaria el consentimiento informado. El estudio fue aprobado por el Comité Institucional de Ética para Humanos de la Universidad Peruana Cayetano Heredia, cada muestra y cepa aislada fue codificada a fin de mantener la confidencialidad.
El aislamiento de cepas de H. pylori se inició con la colecta de biopsias de mucosa gástrica (antro) mediante procedimiento endoscópico. Las biopsias fueron homogenizadas y cultivadas en Agar sangre (agar BHI (BD®) suplementado con 10% de sangre desfibrinada de cordero, anfotericina B (Sigma-Aldrich®) y suplemento selectivo Skirrow (Oxoid®)) a 37 °C en una atmósfera de 5% de O2 y 10% de CO2 durante tres a cinco días. Las colonias de H. pylori fueron identificadas en base a la apariencia de sus colonias, tinción Gram y las pruebas bioquímicas de oxidasa, catalasa y ureasa (6). El análisis histológico se realizó mediante la tinción de las biopsias con hematoxilina-eosina y observación de las bacterias de H. pylori a 1000X en un microscopio óptico. La estrategia de diagnóstico por cultivo fue comparada con el reporte de diagnóstico de histología y la tasa de aislamiento fue calculada según la concordancia observada entre ambos diagnósticos.

En el ensayo de susceptibilidad, la concentración mínima inhibitoria (CMI) fue determinada mediante el método de microdilución en caldo. El cultivo de bacterias fue preparado a 2.0 McFarland (1x107 a 1x108 UFC/ml) en caldo BHI (BD®) suplementado con 10% de suero fetal bovino (Sigma-Aldrich®) y 1% de IsoVitalex®. Claritromicina, levofloxacino, metronidazol y amoxicilina (Sigma-Aldrich®) fueron agregadas al medio en diluciones seriadas a partir de 2 µg/ml, 2 µg/ml, 16 µg/ml y 0,24 µg/ml, respectivamente. Posteriormente se incubó a 37 °C en una atmósfera de 5% de O2 y 10% de CO2, durante 72 horas. La CMI se definió como la menor concentración de antimicrobiano que muestra una completa inhibición de crecimiento visible en el cultivo. Las cepas fueron consideradas resistentes en base a los puntos de corte recomendados por el European Committee on Antimicrobial Susceptibility Testing (EUCAST). Los ensayos fueron realizados por triplicado y se empleó como control de calidad la cepa de H. pylori ATCC 43504 (7-9).
El ensayo de PCR-RFLP fue llevado a cabo a partir de un cultivo fresco de H. pylori equivalente a 2.0 McFarland en caldo BHI (BD®) y la extracción de ADN genómico fue desarrollada con un kit de purificación Thermo Scientific®, según protocolo del fabricante. Se desarrolló una PCR convencional siguiendo protocolos estandarizados para temperatura, tiempo y ciclos, empleando los primers Hp23-1 [5′-CCACAGCGATGTGGTCTCAG-3′] y Hp23-2 [5′-CTCCATAAGAGCCAAAGCCC-3´] que permitió la amplificación de un fragmento de 425 pb del gen de ARNr 23S (4,10). Los fragmentos de amplificación fueron analizados mediante electroforesis en gel de agarosa 1% mediante fotodocumentador (Bio-Rad®).
Posteriormente se desarrolló un RFLP (Restriction Fragment Length Polymorphism) empleando las enzimas de restricción BbsI y BsaI (Thermo Scientific®) para detectar mutaciones puntuales en la posición A2142G y A2143G, respectivamente. El ADN digerido se analizó mediante electroforesis en gel de agarosa al 2,5% (10,11). La restricción fue validada mediante secuenciamiento de los amplicones (Mclab, USA).
Los datos fueron incorporados en una tabla en Excel 2013 y fueron analizados con el paquete estadístico Stata/SE 15.1. Se realizó un análisis descriptivo para todas las variables, la tasa de aislamiento fue calculada considerando como referencia a la prueba histológica y se determino mediante el análisis de concordancia entre ambas estrategias diagnósticas empleando el índice Kappa. Los datos de evaluación de susceptibilidad antimicrobiana fueron expresados en frecuencias y porcentajes. La concentración mínima que inhibe al 50% (CMI50) de las cepas de H. pylori fue calculada mediante un análisis de regresión logística Probit de los porcentajes de inhibición acumulados frente a la distribución de las CMI observadas en las cepas aisladas de H. pylori para cada antimicrobiano. Los contrastes se realizaron a un nivel de confianza del 95% y un nivel de significancia del 5%.
RESULTADOS
La muestra estuvo conformada por 95 casos con síntomas de dispepsia que posteriormente fueron diagnosticados con infección por H. pylori mediante métodos de cultivo e histología. La frecuencia de infección por H. pylori fue de 44 (46,3%) por cultivo y de 47 (49,5%) por histología. La concordancia entre el cultivo microbiológico y la histología determinada por el índice Kappa (K) fue de 0,937 (p=0,001).
La evaluación de susceptibilidad se realizó mediante la técnica de microdilución en caldo, la cual mostró tasas de resistencia de 52,3% a claritromicina, 29,6% a metronidazol, 45,5% a levofloxacino y 4,6% a amoxicilina. El análisis de regresión logística Probit permitió determinar la concentración mínima inhibitoria de antimicrobiano que inhibe el total crecimiento del 50% de cepas de H. pylori (Tabla 1).
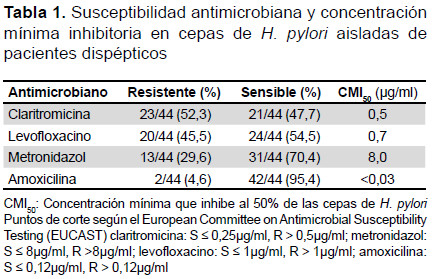
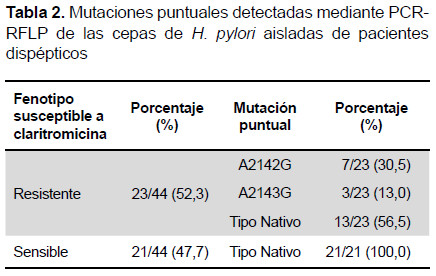
Posteriormente, se realizó un análisis dirigido al gen de ARNr 23S mediante la técnica de PCR-RFLP. Esta técnica permitió la detección de las mutaciones puntuales A2142G y A2143G correspondiente al dominio V del gen de ARNr 23s en el 43,5% de cepas resistentes a claritromicina (Tabla 2) (Figura 1).
DISCUSIÓN
Nuestra metodología de cultivo permitió determinar un porcentaje de infección por H. pylori de 46,3%, diagnóstico que fue contrastado mediante la prueba histológica. Esta observación es comparable con el último valor de prevalencia reportado en el 2002 en otra clínica privada de Lima, donde se menciona una prevalencia de 45% (12). Esto sugiere que el riesgo de infección entre poblaciones con características socioeconómicas similares podría mantenerse estable.
El cultivo microbiológico es un factor importante para el efectivo manejo de la infección por H. pylori; no obstante, su aislamiento se ve limitado debido a su característica como microorganismo «fastidioso» (9). En nuestro caso la concordancia entre el cultivo y la histología fue alta; además, la tasa de aislamiento (93,7%) fue superior a lo reportado en diversos estudios (75% a 90%) (13). El proceso de aislamiento fue realizado considerando aquellas variables que puedan limitar su recuperación, cultivo y aislamiento, tales como el lugar de toma de muestra en la mucosa gástrica, el transporte de las muestras, el medio de cultivo y las condiciones de incubación (14).
La evaluación de la susceptibilidad antimicrobiana mostró un aumento en las tasas de resistencia a claritromicina (52,3%) y levofloxacino (45,5%), respecto a lo reportado en 1983 y 2013 para claritromicina (6,7% y 35,5%, respectivamente) y lo reportado en 2011 para levofloxacino (36.9%), en Perú (12,15,16). Según Megraud et al. este aumento de las tasas de resistencia podría estar influenciado por el incremento del consumo de estos fármacos en los últimos años, llegando al 70% respecto a otros antimicrobianos (17). Además, estudios realizados a nivel local muestran que el H. pylori podría colonizar incluso a menores de tres meses de edad, lo que podría promover un mayor tiempo de exposición a los antimicrobianos (18), propiciando la presencia de cepas resistentes tanto a macrólidos como a quinolonas por mecanismos biológicos de selección.
Se observó que la tasa de resistencia a metronidazol en los pacientes dispépticos evaluados ha disminuido. A diferencia de claritromicina y levofloxacino, este hecho se podría relacionar a la disminución de la utilización de este fármaco como tratamiento contra H. pylori a causa de las altas tasas de resistencia reportadas en años anteriores (>60%) (12). Esto sugiere que se habría disminuido la presión de selección ejercida por el antimicrobiano, permitiendo una ganancia de sensibilidad en los últimos años, esto debido a fenómenos genéticos relacionados con la activación enzimática de este profármaco, lo que afecta el metabolismo de las variantes resistentes y reduce sus clonas en ausencia de la exposición al fármaco (19).
Posteriormente, se realizó un análisis dirigido al gen de ARNr 23S mediante la técnica de PCR-RFLP. Se observó que el 43,5% de los fenotipos resistentes a claritromicina presentaron cambios en las posiciones A2142G y A2143G. Además, se observó que la transición A2142G fue la mutación puntual de mayor frecuencia. Estos cambios en el genoma del H. pylori limitan la interacción de macrólidos al ribosoma, y se reporta que están presentes en más del 90% en cepas de H. pylori fenotípicamente resistentes a claritromicina. Sin embargo, estudios actuales reportan una reducción en la frecuencia de estas mutaciones puntuales sobre fenotipos resistentes (54,8%) (20). Esto sugiere la importancia de realizar una evaluación en regiones más extensas del gen de ARNr 23S que brinde información de nuevas mutaciones involucradas en la resistencia a claritromicina.
Se requieren investigaciones adicionales tomando en consideración los factores clínicos relacionados a la estrategia de erradicación de H. pylori, debido a que en la práctica los esquemas de tratamiento utilizan de forma simultánea dos o más antimicrobianos. Se sugiere realizar estudios a mayor escala y que el análisis genotípico involucre regiones más amplias de los sitios activos asociados a resistencia antimicrobiana. Así se refuerza la monitorización de las infecciones asociadas al patógeno H. pylori.
En conclusión, en el presente estudio se observa que los porcentajes de infección por H. pylori son elevadas. El método de cultivo es una estrategia diagnóstica comparable a la prueba de histología, además de facilitar la evaluación de la resistencia a los antimicrobianos, la cual ha aumentado respecto a años anteriores, afectando a los fármacos claritromicina y levofloxacino. Además, nuestros hallazgos sugieren la participación de mecanismos de resistencia a claritromicina no asociados a los cambios en el gen de ARNr 23S de H. pylori.
Agradecimientos: Los autores agradecen la valiosa colaboración del Dr. Cesar Salinas Cerquín por la realización del análisis histopatológico.
Contribución de los autores: JG, DC, y MS han participado en la concepción, diseño del estudio; JG y MO participaron en la recopilación de datos; JG y DC participaron en el procesamiento, análisis e interpretación de los datos; JG participó en la redacción del artículo; DC, MO y MS revisaron críticamente el artículo. Todos los autores aprobaron la versión final.
Fuentes de financiamiento: el estudio fue financiado por el Consejo Nacional de Ciencia, Tecnología e Innovación Tecnológica (CONCYTEC), mediante la Resolución 207-2013-CONCYTEC-P que otorga becas para estudiantes de la Maestría de Bioquímica y Biología Molecular de la Universidad Peruana Cayetano Heredia.
Conflicto de interés: los autores declaramos no tener conflicto de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. Eusebi LH, Zagari RM, Bazzoli F. Epidemiology of Helicobacter pylori infection. Helicobacter. 2014;19 Suppl 1:1-5. doi: 10.1111/hel.12403. [ Links ]
2. Malfertheiner P, Megraud F, O’Morain CA, Atherton J, Axon AT, Bazzoli F, et al. Management of Helicobacter pylori infection-the Maastricht IV/ Florence Consensus Report. Gut. 2012;61(5):646-64. doi: 10.1136/gutjnl-2016-312288.
3. Fischbach L, Evans EL. Meta-analysis: the effect of antibiotic resistance status on the efficacy of triple and quadruple first-line therapies for Helicobacter pylori. Aliment Pharmacol Ther. 2007;26(3):343-57. doi: 10.1111/j.1365-2036.2007.03386.x [ Links ]
4. Versalovic J, Shortridge D, Kibler K, Griffy M, Beyer J, Flamm R, et al. Mutations in 23S rRNA are associated with clarithromycin resistance in Helicobacter pylori. Antimicrob Agents Chemother. 1996;40(2):477-80. [ Links ]
5. Szczebara F DL, Vincent P, Husson MO. Evaluation of rapid molecular methods for detection of clarithromycin resistance in Helicobacter pylori. Eur J Clin Microbiol Infect Dis. 1997;16(2):162-4. [ Links ]
6. Ndip RN, MacKay WG, Farthing MJ, Weaver LT. Culturing Helicobacter pylori from clinical specimens: review of microbiologic methods. J Pediatr Gastroenterol Nutr. 2003;36(5):616-22. [ Links ]
7. European Committee on Antimicrobial Susceptibility Testing. Breakpoint tables for interpretation of MICs and zone diameters.Version 9.0, 2019 [Internet]. Basilea: EUCAST; 2019 [Citado el 20 de octubre de 2018] Disponible en: http://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_9.0_Breakpoint_Tables.pdf. [ Links ]
8. Piccolomini R DBG, Catamo G, Carbone F, Neri M. Comparative evaluation of the E test, agar dilution, and broth microdilution for testing susceptibilities of Helicobacter pylori strains to 20 antimicrobial agents. J Clin Microbiol. 1997;35(7):1842–6. [ Links ]
9. Clinical and Laboratory Standars Institute. Methods for antimicrobial dilution and disk susceptibility testing of infrequently isolated or fastidious bacteria; Approved guideline-Second edition. Wayne, PA: CLSI; 2010. [Citado 20 octubre de 2018] Disponible en: https://clsi.org/media/1450/m45ed3_sample.pdf. [ Links ]
10. Umegaki N, Shimoyama T, Nishiya D, Suto T, Fukuda S, Munakata A. Clarithromycin-resistance and point mutations in the 23S rRNA gene in Helicobacter pylori isolates from Japan. J Gastroenterol Hepatol. 2000;15(8):906-9. [ Links ]
11. Ho SL, Tan EL, Sam CK, Goh KL. Clarithromycin resistance and point mutations in the 23S rRNA gene in Helicobacter pylori isolates from Malaysia. J Dig Dis. 2010;11(2):101-5. doi: 10.1111/j.1751-2980.2010.00423.x. [ Links ]
12. Ramirez-Ramos A, Sanchez S. Helicobacter pylori 25 años después (1983 -2008): Epidemiología, Microbiología, Patogenia, Diagnóstico y Tratamiento. Rev Gastroenterol Perú. 2009;29(2):158-70. [ Links ]
13. Fresnadillo Martínez MJ, Rodríguez Rincón M, Blázquez de Castro AM, García Sánchez E, García Sánchez JE, Trujillano Martín I, et al. Comparative evaluation of selective and nonselective media for primary isolation of Helicobacter pylori from gastric biopsies. Helicobacter. 1997;2(1):36-9. [ Links ]
14. Morton D, Bardhan KD. Effect of transport medium and transportation time on culture of Helicobacter pylori from gastric biopsy specimens. J Clin Pathol. 1995;48(1):91. [ Links ]
15. Boehnke KF, Valdivieso M, Bussalleu A, Sexton R, Thompson KC, Osorio S, et al. Antibiotic resistance among Helicobacter pylori clinical isolates in Lima, Peru. Infect Drug Resist. 2017;10:85-90. doi: 10.2147/IDR.S123798. [ Links ]
16. Mochizuki H, Noriega AP. Determinación de la susceptibilidad de cepas de Helicobacter pylori a Levofloxacino en formato pequeño y método de difusión en disco usando Agar yema de huevo. Rev Gastroenterol Perú. 2011;31(3):224-9. [ Links ]
17. Megraud F, Coenen S, Versporten A, Kist M, Lopez-Brea M, Hirschl AM, et al. Helicobacter pylori resistance to antibiotics in Europe and its relationship to antibiotic consumption. Gut. 2013;62(1):34-42. doi: 10.1136/gutjnl-2012-302254. [ Links ]
18. Ramirez-Ramos A. Helicobacter pylori en el Perú: Cambios en el tiempo en su prevalencia y su relación con la Patología Gastroduodenal. Rev Gastroenterol Perú. 2003;23(1):11-5. [ Links ]
19. Kwon DH, Osato MS, Graham DY, El-Zaatari FA. Quantitative RT-PCR analysis of multiple genes encoding putative metronidazole nitroreductases from Helicobacter pylori. Int J Antimicrob Agents. 2000;15(1):31-6. doi: 10.1016/S0924-8579(00)00122-9 [ Links ]
20. De Francesco V, Zullo A, Giorgio F, Saracino I, Zaccaro C, Hassan C, et al. Change of point mutations in Helicobacter pylori rRNA associated with clarithromycin resistance in Italy. J Med Microbiol. 2014;63(Pt 3):453-7. doi: 10.1099/jmm.0.067942-0. [ Links ]
Correspondencia: Michel Sauvain
Dirección: Avenida Honorio Delgado 430 San Martin de Porres. Lima, Perú
Teléfono: (511) 481 17 94
Correo electrónico: michel.sauvain@ird.fr
Recibido: 03/09/2018
Aprobado: 13/03/2019
En línea: 28/06/2019















