INTRODUCCIÓN
El síndrome de Guillain-Barré (SGB) es una polineuropatía aguda. Se manifiesta como una parálisis flácida aguda ascendente; su diagnóstico es clínico, basado en la anamnesis y los hallazgos del examen físico, complementados con resultados electromiográficos y cambios característicos en el líquido cefalorraquídeo (LCR) 1. Su fisiopatología tiene base en un fenómeno inmunológico, generalmente desencadenado por un proceso infeccioso. Se postula que esta respuesta inmune la inicia una variedad muy amplia de antígenos, como infecciones por virus o bacterias, particularmente Campylobacter jejuni 2. Mediante una revisión sistemática se ha estimado que entre el 40 y 70% de los casos de SGB están precedidos por una infección respiratoria alta o por una infección del tracto gastrointestinal, una de las más frecuentes es la debida a C. jejuni 3 que puede ser la causa del SGB hasta en un 31% de los casos 2. En un metaanálisis, se reportó que la vacuna monovalente inactivada contra virus de la influenza A (H1N1) incrementa el riesgo de SGB en 1,6 casos de exceso por millón de dosis aplicadas, un incremento pequeño 4.
Entre los virus reportados como posibles agentes etiológicos del SGB está el de Zika. Se han reportado brotes de zika asociado a SGB en la Polinesia Francesa 5, Brasil y Colombia 6, República Dominicana, El Salvador, Honduras, Surinam y Venezuela 7. Un sesgo en todos estos estudios es que no se han realizado pruebas para descartar otros agentes etiológicos diferentes al zika, dengue y chikungunya.
El género Enterovirus es otro de los virus asociados a la parálisis flácida aguda (PFA), con un cuadro clínico similar al SGB. Este género tiene 12 especies que incluyen los enterovirus A hasta la J y los rinovirus A, B y C. La vía de trasmisión depende del serotipo, algunos se trasmiten por vía fecal-oral y otros por las vías respiratorias. Muchos enterovirus son neurotrópicos y pueden afectar el sistema nervioso central o periférico. Por ejemplo, los virus de Coxsackie y echovirus pueden causar meningitis viral; los virus de Coxsackie, enterovirus A71 y echovirus 9 pueden producir meningoencefalitis ( 8.
Los poliovirus y los enterovirus D68 y A71 pueden producir PFA. En 2014 en EE. UU. ocurrió un incremento de infección respiratoria aguda producidas por EV-D68 y se observó un brote de PFA que fue asociado a EV-68 9. También se han reportado que otros virus, como los de Coxsackie y echovirus, son causantes de PFA en Corea, pero su incidencia es baja 10. La incidencia del SGB varía con la edad, la estacionalidad y el lugar donde se realizan las mediciones. Considerando todos los grupos de edad, la incidencia varía de 0,16 a 3 × 105 personas-año, es mayor en hombres y se incrementa con la edad 3. El Perú ha presentado un incremento inusual de casos de SGB desde 2018, en el que se reportaron 1378 casos, con una incidencia de 4,59 × 105 habitantes, en comparación a los años previos, que fue de aproximadamente 0,91 × 105 habitantes 11. En 2019, hasta la semana epidemiológica 47, se habían notificado 1341 casos 12. Un alto porcentaje de los casos se presentaron en departamentos del norte y centro del país (Lambayeque, La Libertad, Piura y Lima). No obstante, casi todos los departamentos del Perú fueron afectados por el brote. Se han planteado varias hipótesis sobre los posibles agentes infecciosos asociados al SGB 13, sin embargo, no se ha sistematizado la información acerca de los pacientes afectados y los resultados de las pruebas realizadas.
El objetivo de este estudio es describir los resultados de los exámenes de laboratorio realizados en muestras biológicas de pacientes con diagnóstico de SGB recibidas en el Instituto Nacional de Salud entre 2018 y 2019.
MENSAJES CLAVE
Motivación para realizar el estudio: Debido al inusual incremento de casos del síndrome Guillain-Barré (SGB) entre los años 2018 y 2019, se realizó la búsqueda del posible agente etiológico que desencadena el proceso autoinmune que causa este síndrome neurológico.
Principales hallazgos: Entre los agentes patógenos identificados en las muestras tomadas de pacientes con SGB, se reportó la presencia de una variante de Campylobacter jejuni asociado a brotes de SGB en otros países, el cual proponemos como probable agente causal de los casos en Perú.
Implicancias: Estos hallazgos abren la oportunidad para identificar la fuente de infección desencadenante de SGB e implementar las medidas de prevención y control para cortar la cadena de trasmisión de Campylobacter jejuni.
MATERIALES Y MÉTODOS
Tipo de estudio y población
Se realizó un estudio observacional y descriptivo de los pacientes con SGB atendidos en la red hospitalaria del Ministerio de Salud (MINSA), el Seguro Social (EsSalud) y la Sanidad de las Fuerzas Armadas y Policiales, de los cuales se obtuvieron muestras biológicas para ser analizadas en el Instituto Nacional de Salud (INS) o que fueron procesadas en los Laboratorios de Referencia Regionales. A fin de contextualizar el brote, se consultó en los reportes de la página web del Centro Nacional de Epidemiología, Prevención y Control de Enfermedades (CDC-Perú) el número de casos de SGB reportados por semana epidemiológica desde el inicio de 2018.
El INS es el organismo estatal encargado de la investigación en salud pública y diagnóstico especializado. El INS y el CDC-Perú trabajan en la investigación de ocurrencias sanitarias inusuales y brotes epidémicos. Como parte de la investigación de un brote, se realizan exámenes de laboratorio inmunológicos, químicos, fenotípicos y moleculares, entre otros, para explorar la asociación con agentes infecciosos, químicos o ambos.
Se incluyó a los pacientes notificados en el sistema de vigilancia epidemiológica y que, además, enviaron muestras biológicas a los Laboratorios de Referencia Regional de Lima, Piura, Lambayeque, La Libertad, Cajamarca, Junín o al INS para descarte de enfermedades infecciosas. Se describe, además, el reporte de un laboratorio privado que procesó muestras biológicas de ocho pacientes y cuyos resultados se remitieron al INS.
En el Perú, desde 2016 el SGB es de notificación obligatoria en el Sistema de Vigilancia Epidemiológica, en todos los establecimientos de salud públicos y privados, y según el protocolo sanitario de urgencia para la vigilancia del SGB 14. Esta norma define como caso clínicamente confirmado de SGB (nivel 3 de certeza diagnóstica, según los criterios de Brighton) cuando se cumplen los siguientes criterios: debilidad muscular bilateral flácida en las extremidades; reflejos osteotendinosos profundos disminuidos o ausentes en las extremidades con debilidad; enfermedad monofásica, con un intervalo entre el inicio y el nadir de la debilidad entre 12 horas y 28 días, seguida de una meseta clínica; y ausencia de diagnóstico alternativo que explique la debilidad. Puede o no tener hallazgos electrofisiológicos con SGB y LCR con disociación albúmino-citológica. En junio de 2019 el CDC-Perú, ante el incremento de casos de SGB en el norte del Perú, emitió una alerta epidemiológica en la que estableció la notificación de todos los casos clínicos de SGB y la colecta de las siguientes muestras biológicas: suero, heces, hisopado nasofaríngeo, LCR y orina 15.
Procedimiento
Las pruebas realizadas en suero fueron ELISA IgM e IgG para los virus del dengue, zika y chikungunya; y reacción en cadena de la polimerasa cuantitativa (qPCR) para los virus del dengue y zika. En LCR se procesaron ELISA IgM y reacción en cadena de la polimerasa (PCR) para los virus del dengue y zika; PCR para enterovirus, panel meningoencefalitis/encefalitis FilmArray®. En el hisopado nasofaríngeo se hizo reacción en cadena de la polimerasa con transcriptasa reversa en tiempo real (qRT-PCR) para virus de la influenza A y B, rinovirus, virus respiratorio sincicial y adenovirus; inmunofluorescencia directa para virus de la influenza A, B, virus respiratorio sincicial, adenovirus, virus parainfluenza 1, virus parainfluenza 2, virus parainfluenza 3 y metaneumovirus. En los hisopados rectales y muestras de heces se realizó cultivo para bacterias enteropatógenas y enterovirus, así como, PCR para enterovirus, Campylobacter spp., Escherichia coli diarreogénica y Shigella spp.
Se consideró positivo a cualquier resultado de PCR que mostró la presencia de material genético del microorganismo, así como a los resultados de ELISA en valores predeterminados como diagnósticos para cada agente etiológico 16. En el caso de resultados remitidos al INS por otros laboratorios, se indicó el método reportado en el informe de resultados.
Se seleccionaron los agentes infecciosos de acuerdo con su frecuencia para ser sometidos a secuenciación del genoma completo. En el caso de los virus, el secuenciación se realizó con el método Sanger 17. El resultado de la secuenciación se obtuvo en formato ABI, y las secuencias fueron ensambladas y analizadas utilizando el software Geneious V8, asimismo, se utilizaron los algoritmos Blast y Kallisto para detectar los transcritos más similares 18.
Cuatro muestras de LCR y tres muestras de heces colectadas en 2018 y cuyo resultado a la prueba PCR dio positivo por enterovirus en el INS, se enviaron al Center of Diseases Control de Estados Unidos de Norteamérica (CDC-EUA), en las que se realizaron PCR para enterovirus y parechovirus humano.
Análisis estadístico
Se obtuvieron los resultados de laboratorio del aplicativo NetLab, el sistema de reporte de resultados de laboratorio del INS y lo exportamos a una base de datos en Excel (Microsoft Office®). Se describieron las variables categóricas mediante frecuencias relativas y absolutas. Para las variables numéricas, se determinó su distribución mediante la prueba de Shapiro-Wilk y se describieron en medias y desviaciones estándares o medianas y rango intercuartílico, respectivamente, si cumplían o no con el criterio de normalidad. El análisis se realizó en Stata versión 9 (StataCorp, College Station, Texas, EUA).
Aspectos éticos
El presente estudio es parte de la investigación del brote de SGB en Perú. Se utilizaron códigos del registro del NetLab sin datos que permitan la identificación de los pacientes, y únicamente se tomaron sus resultados de laboratorio en el contexto de esta situación epidemiológica, por lo que no fue necesario su sometimiento a un comité de ética institucional para su aprobación.
RESULTADOS
El INS recibió un total de 2051 muestras biológicas que incluyeron suero (738), LCR (247), hisopado nasofaríngeo (475), orina (278), hisopado rectal y/o heces (313). Estas muestras se obtuvieron de un total de 906 pacientes con diagnóstico clínico de SGB desde el 27 de abril de 2018 al 5 de noviembre de 2019, de las cuales, 26 (2,9%) muestras se obtuvieron en 2018 y 880 (97,1%) de las muestras en 2019. La Figura 1 muestra el número de pacientes con SGB notificados por el CDC-Perú y el número de pacientes cuyas muestras biológicas se han enviado al INS (incluidos en el presente estudio) en el periodo mencionado.
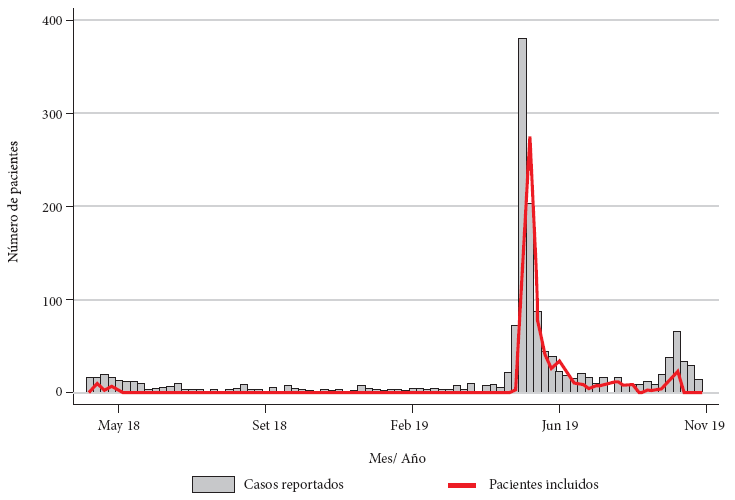
Figura 1 Casos de síndrome de Guillain-Barré notificados por el Centro Nacional de Epidemiología, Prevención y Control de Enfermedades y pacientes con muestras biológicas enviadas al Instituto Nacional de Salud, Perú, 2018-2019.
La mayoría de pacientes procedían de los departamentos de Lima, Piura, La Libertad y Junín (Tabla 1). Cincuenta y dos (5,9%) pacientes procedían del Hospital de Apoyo Santa Rosa de Piura; 49 (5,6%) del Hospital Nacional Almanzor Aguinaga Asenjo de Chiclayo; 48 (5,5%) del Hospital Edgardo Rebagliati Martins; y 43 (4,9%) del Hospital Nacional Daniel Alcides Carrión de Junín, que fueron los hospitales con mayor número de pacientes incluidos.
Tabla 1 Características demográficas y departamento de procedencia de casos de síndrome de Guillain-Barré con muestras biológicas enviadas al Instituto Nacional de Salud, 2018-2019 (n = 906).
| Características generales | n (%) |
|---|---|
| Mediana de la edad (RIC) | 40 (24-56) |
| 0-14 | 112 (12,7) |
| 15-24 | 113 (12,8) |
| 25-40 | 207 (23,4) |
| 41-60 | 271 (30,7) |
| >60 | 181 (20,5) |
| Sin dato | 22 (2,4) |
| Sexo masculino | 539 (59,5) |
| Departamento de procedencia | |
| Lima | 358 (39,5) |
| Piura | 199 (22,0) |
| La Libertad | 98 (10,8) |
| Junín | 82 (9,1) |
| Lambayeque | 61 (6,7) |
| Cajamarca | 48 (5,3) |
| Otros | 60 (6,6) |
RIC: Rango intercuartílico
De las 738 muestras de suero, 735 tuvieron resultados de serología o PCR negativos para arbovirus; tres pacientes dieron positivo por dengue y procedían de los departamentos de Lambayeque, La Libertad y Lima, respectivamente. Asimismo, tres pacientes tuvieron PCR positivo en orina para virus de Zika. De las muestras de LCR, 12 fueron positivas por PCR para enterovirus, sin embargo, todas fueron en 2018 y todos los cultivos fueron negativos. Cinco de estos pacientes también presentaron PCR positivo por enterovirus en heces. De las 220 muestras de LCR correspondientes al 2019 analizadas por PCR y cultivo para enterovirus, no hubo ninguna positiva; las 90 muestras procesadas por la técnica de FilmArray fueron negativas para los 14 patógenos que identifica la prueba. Respecto a los virus respiratorios, una muestra fue positiva para virus de la influenza A, dos para virus de la influenza B, dos para adenovirus, cinco para virus respiratorio sincitial y diez para rinovirus. Por último, en 24 pacientes, el cultivo en heces/hisopado rectal fue positivo para Campylobacter spp., y 69 para E. coli diarrogénicas (Tabla 2).
Tabla 2 Resultados de laboratorio de las muestras biológicas enviadas al Instituto Nacional de Salud, de pacientes con síndrome de Guillain-Barré, Perú, 2018-2019.
| Tipo de muestra | Prueba diagnóstica | Positivas n (%) | Total procesadas |
|---|---|---|---|
| Suero | ELISA Dengue IgM | 2 (0,5) | 369 |
| qRT-PCR Dengue | 1 (1,9) | 54 | |
| qRT-PCR Zika | 0 (0) | 372 | |
| ELISA Zika IgM | 0 (0) | 597 | |
| ELISA Chikungunya IgM | 2 (0,3) | 581 | |
| qRT-PCR Chikungunya | 0 (0) | 15 | |
| Orina | qRT-PCR Zika | 3 (1,1) | 278 |
| LCR | qRT-PCR Zika | 0 (0) | 82 |
| ELISA IgM Zika | 0 (0) | 64 | |
| qRT-PCR Dengue | 0 (0) | 5 | |
| ELISA IgM Dengue | 0 (0) | 1 | |
| RT-PCR Enterovirus | 12 (5,1) | 234 | |
| Cultivo Enterovirus | 0 (0) | 222 | |
| FilmArray® panel Meningitis/ Encefalitis | 0 (0) | 90 | |
| Hisopado nasofaríngeo | IFD Virus de la influenza A | 0 (0) | 293 |
| qRT-PCR Virus de la influenza A | 1 (0,2) | 475 | |
| IFD Virus de la influenza B | 1 (0,3) | 304 | |
| qRT-PCR Virus de la influenza B | 1 (0,2) | 464 | |
| qRT-PCR Rinovirus | 10 (5,6) | 179 | |
| qRT-PCR VSR | 5 (2,8) | 177 | |
| IFD VSR | 0 (0) | 171 | |
| qRT- PCR Adenovirus | 2 (1,1) | 176 | |
| IFD Adenovirus | 0 (0) | 171 | |
| IFD Parainfluenza 1 | 0 (0) | 171 | |
| IFD Parainfluenza 2 | 0 (0) | 171 | |
| IFD Parainfluenza 3 | 0 (0) | 171 | |
| IFD Metaneumovirus | 0 (0) | 171 | |
| Hisopado rectal y/o heces | RT-PCR Enterovirus | 5 (2,0) | 250 |
| Campylobacter spp (por PCR y Cultivo) | 24 (6,5) | 367 | |
| E. coli diarreogénico | 69 (22,9) | 302 |
LCR: líquido cefalorraquídeo, qRT-PCR: reacción en cadena de la polimerasa con transcriptasa reversa en tiempo real; RT-PCR: reacción en cadena de la polimerasa con transcriptasa reversa en tiempo real; PCR, reacción en cadena de la polimerasa; IFD: inmunofluorescencia directa.
Un laboratorio particular en la ciudad de Lima reportó al INS los resultados obtenidos mediante panel gastrointestinal FilmArray®, de ocho pacientes con SGB notificados en 2018. Hubo cinco resultados positivos para E. coli enteropatogénica, cuatro para Campylobacter spp., y tres para rinovirus/enterovirus humano, Shigella spp., E. coli enteroinvasiva, y E. coli enteroagregativa; respectivamente.
Se procedió al estudio molecular de las muestras de LCR y heces recibidas por el INS con resultados PCR positiva por enterovirus. En primer lugar, se amplificó la región 5’ UTR y se identificó como enterovirus. Luego se procedió a la secuenciación del genoma completo; sin embargo, la cantidad de fragmentos detectados fue menor al 10% del genoma del virus, por lo cual, no fue posible identificar la especie. De cuatro muestras de LCR y tres de heces con resultado PCR positivo a enterovirus se enviaron al CDC-EUA; solo una muestra de heces fue positiva para echovirus 1.
De igual manera, se procedió a la secuenciación del genoma completo de las diez cepas aisladas de Campylobacter spp.: seis muestras biológicas recibidas en el INS (dos de Piura y cuatro de Junín) y cuatro cepas aisladas por el Naval Medical Research Unit 6 (NAMRU-6). Todos los especímenes fueron de 2019. Se procedió de acuerdo con los métodos descritos previamente y el aislamiento correspondió a la especie C. jejuni. El análisis filogenético de la secuencia genómica de esta cepa se identificó como el genotipo ST2993, nunca antes reportado en Perú. Los diez aislamientos correspondieron al mismo clon, es decir que comparten la misma información genética. Como parte del análisis genómico, también se lograron identificar en estas cepas, la presencia de los genes cst-II y cst-III, que codifican los lipooligosacáridos asociados con la patología de SGB. Asimismo, se observó la presencia de una mutación puntual específica en la región determinante de la resistencia a quinolonas (QRDR) del gen gyrA (Thr86Ile), relacionada con resistencia a las fluoroquinolonas.
DISCUSIÓN
Se logró sistematizar los resultados de laboratorio de pacientes con diagnóstico de SGB en el Perú cuyas muestras biológicas se analizaron en el INS entre 2018 y 2019, y de manera adicional de aquellos cuyos resultados se remitieron al INS. La literatura médica reporta que hasta un 75% de los casos de SGB tienen el antecedente de haber presentado una infección respiratoria o digestiva previa en las últimas seis semanas; sin embargo, en muchos de ellos, al momento de la búsqueda del agente etiológico, dicha infección ya ha sido controlada y no se logra aislar en los exámenes respectivos 19. Dentro de los agentes infecciosos aislados o identificados en este estudio, resalta la presencia de C. jejuni (6,5%). Otros agentes identificados incluyen arbovirus (1,1%), enterovirus (2%), E. coli diarreogénica (22,9%) y virus respiratorios como rinovirus en 5,6% y VSR en 2,8%.
Este es el primer reporte de aislamiento de C. jejuni ST2993 en Perú y América. Una cepa de C. jejuni correspondiente al mismo genotipo ST2993 se reportó en 2007 en China, la cual se asoció a un brote de SGB 20. Los reportes previos de secuenciación genética de aislamientos peruanos de Campylobacter muestran cepas genéticamente muy alejadas a esta nueva cepa 21. La secuenciación genética realizada en diez cepas aisladas fue indistinguible entre ellas, por lo que concluimos que se trata del mismo clon con amplia distribución ya que se identificó en las regiones de Piura, Junín y Lima. Existen múltiples reportes que asocian C. jejuni con SGB 2 , 20 , 21. La fisiopatología del SGB está relacionada con la presencia de macrófagos activados, linfocitos T y anticuerpos séricos contra los gangliósidos humanos y se cree que los lipooligosacáridos de C. jejuni mimetizan antigénicamente a estos gangliósidos, desencadenando la reacción inmune 22. Por lo descrito resulta relevante el presente hallazgo y urge investigar la cadena de trasmisión de C. jejuni asociado usualmente con las aves de corral, consumo de alimentos lácteos, agua, carne, entre otros.
Como parte de los hallazgos, se identificó la presencia de E. coli diarreogénica en muestras de heces/hisopados rectales de 69 pacientes. Sin embargo, estas cepas que mostraron alta heterogeneidad correspondieron a diferentes patotipos. Existe poca información sobre la asociación entre esta bacteria y el desarrollo del SGB, y los reportes documentados se refieren predominantemente a infecciones urinarias más que a infecciones entéricas 23. No hemos encontrado reportes de brotes del SGB causados por esta etiología, por lo que parece poco probable que exista una asociación con este brote, más aún considerando que la E. coli es un germen que forma parte del microbioma del tubo digestivo humano.
Respecto a los arbovirus, identificamos infección por el virus del dengue en tres pacientes (dos por anticuerpos IgM y uno por PCR en suero) y tres con infección por virus de Zika (PCR positivo en orina). Adicionalmente, dos pacientes tuvieron anticuerpos IgM positivos contra el virus chikungunya; uno procedía de Piura y el otro de Cajamarca (provincia de Jaén). Existen numerosos reportes en la literatura sobre asociación entre SGB con infección por el virus de Zika 24. Sin embargo, en nuestro estudio fueron solo tres casos lo que refleja probablemente la trasmisión usual en áreas endémicas de este virus. Lo mismo ocurre para el chikungunya, habiéndose reportado un incremento de casos de SGB durante un brote de chikungunya en la Polinesia Francesa y otros reportes con casos esporádicos 25. Sin embargo, hay que considerar que este brote de SGB ha afectado incluso zonas de la sierra central y norte del Perú, donde no existe trasmisión de estos virus. Es más, recientemente se viene cuestionando la contribución del virus de Zika en estos brotes debido a que los estudios de los que provienen dichos reportes son en su mayoría sin grupo control y no permiten determinar asociación 26.
Pocos pacientes tienen resultados positivos a virus respiratorios. A pesar de que la asociación entre el SGB y el virus de la influenza se encuentra documentada 27, consideramos que debido a la poca frecuencia observada en este grupo de pacientes es poco probable que estos virus puedan considerarse de relevancia en el desarrollo de SGB en este brote.
El hallazgo de enterovirus en LCR en pacientes diagnosticados con SGB en 2018 fue identificado por los laboratorios del CDC-EUA (echovirus 1) y un laboratorio particular, los que fueron concordantes con los hallazgos del INS. Sin embargo, todos los cultivos virales para enterovirus fueron negativos, posiblemente por una muestra inadecuada o algún problema que pudiera haber afectado la viabilidad del virus. Además, este hallazgo se limitó al 2018. Durante el brote de 2019 no se logró aislar ni identificar material genético de enterovirus en las muestras de heces y LCR de ningún paciente. Existen reportes de la literatura que asocian enterovirus con síndrome neurológico agudo, específicamente del enterovirus D68 9. Sin embargo, esos reportes se refieren a cuadros de parálisis flácida aguda con un cuadro neurológico que afecta la motoneurona del asta anterior de la medula espinal, un cuadro clínico y electrofisiológicamente distinto al SGB en la que se afectan los nervios periféricos en sus distintas formas de presentación 22, que es lo que se ha venido reportando en el Perú. Debido a estos hallazgos, se descartó la vinculación epidemiológica del enterovirus con el presente brote de SGB.
Son escasas las publicaciones sobre el estudio de la filogenia de agentes patógenos digestivos en el Perú y las pocas que existen se han realizado en niños menores de cinco años, lo cual limita nuestro conocimiento sobre los microorganismos que circulan en nuestro país. En una época en la que los avances tecnológicos permiten el acceso a la secuenciación genómica en la salud pública, esta brecha en el conocimiento debe ser rápidamente resuelta con investigaciones o, idealmente, con la implementación de una vigilancia centinela específica.
Nuestro estudio tiene algunas limitaciones. Al ser un diseño descriptivo y no tener un grupo control, la asociación entre SGB y un potencial agente etiológico no queda demostrada; sin embargo, nuestra hipótesis es que C. jejuni es el potencial agente etiológico, lo que debe demostrarse con posteriores estudios analíticos. Las pruebas han sido realizadas por laboratorios regionales y por el INS, bajo los parámetros usuales de control de calidad, sin estudios adicionales de concordancia entre laboratorios. Se ha colectado una sola muestra de heces o hisopado rectal para realizar los coprocultivos y aislar Campylobacter spp., por lo que la sensibilidad de esta prueba puede estar disminuida. Un estudio reportó una sensibilidad de hasta 100% con la colecta de tres muestras consecutivas de heces 28. En el Perú, la automedicación con antibióticos es relativamente frecuente, puede ir desde el 34% en población general 29 hasta el 70% en estudiantes universitarios 30; al no tener esta información sobre los pacientes, la sensibilidad de los coprocultivos podría estar disminuida, el tiempo entre la colecta de las muestras de heces y su procesamiento para realizar el coprocultivo en muchos casos ha sido mayor de 24 horas lo que reduce la sensibilidad para aislar C. jejuni. La colecta de muestras biológicas no se realizó de acuerdo con lo establecido en el sistema de vigilancia; muestras como LCR se colectaron solo en el 25,8%, orina en el 30,7% y heces o hisopado rectal en el 40,5% de los casos. Por último, las muestras de suero se han colectado usualmente después de iniciar el tratamiento lo que podría alterar los exámenes serológicos con resultados falsos positivos en los pacientes que se les administró inmunoglobulinas, o falsos negativos si se realizó plasmaféresis.
En conclusión, se han identificado diversos agentes patógenos en muestras biológicas de pacientes con SGB en los brotes ocurridos en el Perú entre 2018 y 2019. En 2018, el hallazgo más resaltante es la identificación de enterovirus por PCR en el INS; sin embargo, este hallazgo no pudo corroborarse mediante la secuenciación del genoma completo y, además, de las cuatro muestras enviadas al CDC-EUA solo una fue positiva a echovirus; todas las muestras de LCR y de heces fueron negativas para enterovirus por cultivo y PCR en 2019. Por otro lado, se logró aislar C. jejuni genotipo ST2993 en 2019, un patógeno ampliamente relacionado al SGB en varios continentes. Este hallazgo abre nuevas líneas de investigación para realizar los estudios analíticos correspondientes, identificar el reservorio y la fuente de infección para implementar las medidas de prevención más efectivas y cortar la cadena de trasmisión. Asimismo, se abre todo un nuevo campo de investigación clínica, como los factores genéticos asociados a la susceptibilidad a este síndrome, los factores pronósticos para presentar complicaciones o para morir, la efectividad de los distintos abordajes de tratamiento, entre otros.











 texto en
texto en 



