INTRODUCCIÓN
Las infecciones causadas por bacterias multirresistentes a antibióticos son un problema de salud pública mundial debido al gran impacto que tienen sobre la morbimortalidad, estas generaron cerca de 250 000 muertes en el 2017 1. Frente a esto la Organización Mundial de la Salud (OMS) publicó una relación de bacterias prioritarias a vigilar por su mayor riesgo para la salud humana y el ambiente hospitalario, entre las cuales están especies como Acinetobacter baumannii, Pseudomonas spp. y la familia Enterobacteriaceae resistentes a carbapenémicos 2.
Estas especies y en especial las gram negativas formadoras de enzimas carbapenemasas 3 han sido reportadas desde la década del 90 4 y actualmente son categorizadas en familias enzimáticas de diferentes clases (A, B, C y D). Algunas de estas clases, como la Metalo-β-lactamasas (MBLs) representaron inicialmente una amenaza en escasas áreas geográficas, pero luego se expandieron a zonas de Europa, Asia y América, considerándose esta situación como una epidemia que genera resistencia a múltiples fármacos 5.
En Latinoamérica y el Caribe se identificaron las clases A, B y D descritas por Ambler 6 entre cuyos elementos genéticos móviles e integrones predomina el tipo KPC aislado por primera vez en el 2001 en Estados Unidos 7. Esto sugiere que cepas específicas se han propagado con éxito, adquiriendo la categoría de endémicas en algunos países como Brasil, Colombia, Argentina y México 8. Frente a esta realidad, el objetivo del presente estudio es describir el estado actual de las carbapenemasas circulantes en el Perú con la finalidad de fortalecer la vigilancia epidemiológica de infecciones asociadas a la atención en salud (IAAS) y bacterias multiresistentes, como también al programa de uso racional de antimicrobianos (PROA) en los hospitales públicos y privados.
MENSAJES CLAVE
Motivación para realizar el estudio: En Perú, la resistencia a los carbapenémicos es un problema de salud pública, existe una creciente diseminación de bacterias multirresistentes codificados en su mayoría por la presencia de plásmidos que pueden pasar de un género bacteriano a otro, es necesario conocer los tipos de genes circulantes en el país.
Principales hallazgos: Se identificaron 185 cepas con presencia de enzimas carbapenemasas de clase A, B y D en cepas de 30 instituciones prestadoras de servicios de salud del Perú durante el 2019. Su prevalencia fue del 59,7%, la clase B fue la más frecuente, y los genes más frecuentemente detectados fueron blaNDM, blaIMP, blaOXA24-like, blaKPC y blaOXA23-like.
Implicancias: El impacto en salud de la resistencia a los carbapenémicos debe fortalecer los programas de vigilancia epidemiológica.
EL ESTUDIO
Estudio observacional descriptivo de corte transversal realizado a partir de cepas provenientes de 30 instituciones prestadoras de servicios en salud (IPRESS) de 12 regiones del Perú de enero a diciembre del 2019. La población estuvo constituida por 331 cepas enviadas para confirmación diagnóstica al Laboratorio de Referencia Nacional de Infecciones Intrahospitalarias (LRNIIH) del Instituto Nacional de Salud (INS).
Para la identificación de las cepas se utilizaron medios de cultivo selectivos de tipo agar MacConkey y medios bioquímicos (Citrato, TSI, LIA, MIO, Urea) 9, marca (Becton y Dickinson). La sensibilidad antimicrobiana se determinó por el método de Kirby Bauer y la concentración inhibitoria mínima (CIM) por el método de Epsilon Test (E-Test, marca Liofilchem) siguiendo las pautas de interpretación para halos propuestas por el CLSI (Clinical and Laboratory Standards Institute) 10. Para la confirmación fenotípica de la producción de carbapenemasas se utilizó la prueba de Blue CARBA 11 y en la identificación, la prueba de sinergia con discos de ácido fenilborónico (APB, marca Liofilchem) y ácido etilendiaminotetraacético/ácido mercaptoacético de sodio (EDTA/SMA, marca Bioanalyse).Los controles de calidad internos para discos de sensibilidad y medios de cultivo se realizaron con cepas procedentes del Servicio de Antimicrobianos del Laboratorio Nacional de Referencia en resistencia a los antimicrobianos INEI-ANLIS Dr. Carlos G. Malbrán en Argentina.
La extracción del ADN bacteriano se obtuvo de cepas previamente identificadas con resistencia a carbapenémicos utilizando el sobrenadante para amplificación por reacción de cadena de polimerasa (PCR, en inglés) utilizando iniciadores específicos diseñados para blaKPC, blaNDM, blaIMP, blaVIM, blaOXA-23, blaOXA-24, blaOXA-51, blaOXA-58, blaOXA-48, y blaOXA-143 12. Finalmente, los productos fueron observados en gel de agarosa al 1,5% en transiluminador de luz ultravioleta (UV) (Figura 1) utilizando controles positivos y negativos obtenidos del cepario del Programa de Control de Calidad Latinoamericano del INEI-ANLIS.
Figura 1 Productos de la reacción en cadena de polimerasa observados en gel agarosa al 1,5% en transiluminador de luz ultravioleta.
Con relación a las consideraciones éticas, se guardó la estricta confidencialidad de los datos provenientes de las instituciones de las cuales procedieron las cepas, siguiendo las normativas de la Declaración de Helsinki. Esta investigación se elaboró en el marco del proceso de control de calidad para confirmación de diagnóstico de muestras enviadas al INS a partir de IPRESS a nivel nacional. Finalmente, no se requirió la aprobación de un Comité de Ética por las características previamente descritas.
HALLAZGOS
Se obtuvieron 331 cepas procedentes de 12 regiones del país distribuidas el 70%(n=21) en IPRESS del MINSA y 23%(n=7) de EsSalud. La mayoría de IPRESS estaban localizadas en Lima y Callao (56,7%; n=17). Del total de estas cepas se excluyeron inicialmente 21 por no ser viables o estar contaminadas teniendo como muestra final 310 aislamientos viables. De éstas el 87,4% (n=271) fueron bacterias gramnegativas. La prevalencia de carbapenemasas fue del 59,7% (n=185), de las cuales el 42,2% (n=78) corresponden a Enterobacteriaceae; 32,9% (n=61) a P. aeruginosa y 24,9% (n=46) a Acinetobacter spp. Adicionalmente el 27,7% (n=86) fueron cepas sensibles a carbapenémicos.
Con relación a la distribución geográfica de las carbapenemasas, en la Figura 2 puede observarse su distribución por región y en el caso de Lima según distrito a partir de la locación de la IPRESS. Se presentó un mayor número de aislamientos de carbapenemasas en instituciones del MINSA (18,4%) y EsSalud (81,6%), y en las regiones de Cusco (12,4%), La Libertad (6,4%), Callao (6,4%) y Lambayeque (4,9%).
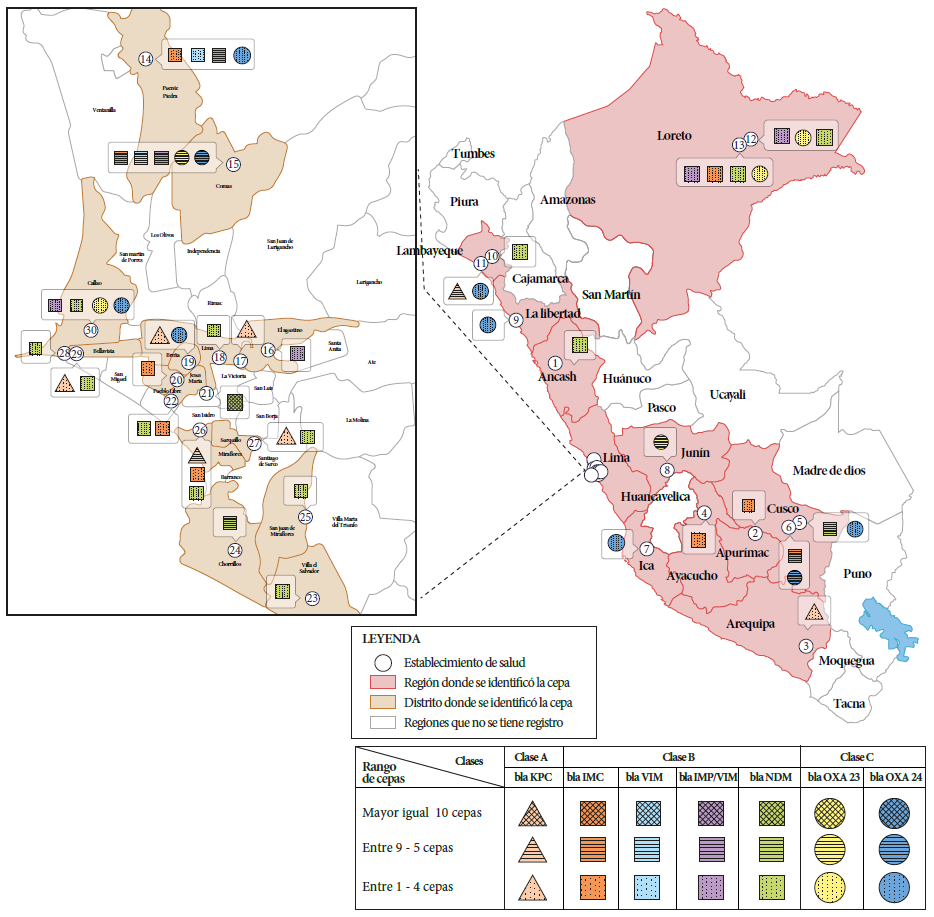
Figura 2: Distribución de Carbapenemasas Clase A, B y D en el Perú durante el 2019, por área geográfica.
Según el tipo de carbapenemasas, se reportó la presencia de clase A en el 12,4% (n=23), siendo del tipo blaKPC las encontradas en Klebsiella pneumoniae y Escherichia coli. La clase B se reportó en el 62,7% (n=116), hallándose el tipo blaNDM (n=57) en K. pneumoniae, E. coli, Providencia rettgeri; en P. aeruginosa se encontró el tipo blaIMP (n=32) y el tipo blaVIM (n=13) así como la coproducción de tipo bla IMP/VIM (n=14). La clase D fue reportada en un 24,9% (n=46) encontrándose únicamente los tipos blaOXA23-like y blaOXA24-like en Acinetobacter spp. El gen blaOXA51-like hidroliza débilmente penicilinas y carbapenémicos y es usado como marcador de especie para A. baumannii, no siendo reportado en la actual serie (Tabla 1).
Tabla 1 Tipos de carbapenemasa según cepa en instituciones prestadoras de servicios de salud del Perú en el 2019.
| Tipo de Carbapenemasa | n (185) | % |
|---|---|---|
| Clase A (n=23; 12,4 %) | ||
| KPC (Klebsiella pneumoniae) | 22 | 11,9 |
| KPC ( Escherichia coli) | 1 | 0,5 |
| Clase B (n=116; 62,7%) | ||
| NDM (Klebsiella pneumoniae) | 44 | 23,8 |
| NDM (Escherichia coli) | 9 | 4,9 |
| NDM (Providencia rettgeri) | 2 | 1,1 |
| NDM (Pseudomonas aeruginosa) | 2 | 1,1 |
| VIM (Pseudomonas aeruginosa) | 13 | 7,0 |
| IMP (Pseudomonas aeruginosa) | 32 | 17,3 |
| IMP/VIM (Pseudomonas aeruginosa) | 14 | 7,6 |
| Clase D (n=46; 24,9%) | ||
| OXA-23-like(Acinetobacter spp. ) | 19 | 10,3 |
| OXA-24-like (Acinetobacter spp.) | 27 | 14,6 |
| OXA-48-like | 0 | 0,0 |
| OXA-51-like | 0 | 0,0 |
| OXA-58-like | 0 | 0,0 |
| OXA-143-like | 0 | 0,0 |
KPC: Klebsiella pneumoniae productora de carbapenemasa, NDM: metalobetalactamasa de Nueva Delhi, IMP: metalobetalactamasa que hidroliza imipenem, VIM: metalobetalactamasa codificada por integrón de Verona.
DISCUSIÓN
En 1988 se reportó la primera carbapenemasa en Japón, y cinco años después se reportó por primera vez en un país de Latinoamérica (Argentina), iniciando una cadena de reportes hasta la actualidad 8 , 13. En el 2013 en Perú se reportó el primer caso de carbapenemasas blaKPC-2 en una cepa de K. pneumoniae derivado de un hemocultivo en el Hospital Nacional Arzobispo Loayza 14. Desde aquel primer reporte los casos han aumentado inclusive demostrando la presencia de genes de resistencia 15.
En relación con la clase A de carbapenemasas, el tipo blaKPC identificado en este estudio es el tercer más frecuente reportado (12,4%) y presente en 23 cepas de Enterobacteriaceae (22 en K. pneumoniae y 1 en E. coli) en cuatro regiones del país (Arequipa, Lima, Lambayeque y Callao). Este gen fue reportado por primera vez en el 2005 en Colombia 8 y fue el primer blaKPC reportado en el Perú (Lima) 14 - 16. Su presencia aumentada y distribuida en IPRESS de regiones altamente pobladas es un hallazgo importante, ya que su presencia en la sangre está asociada a elevadas tasas de letalidad en pacientes hospitalizados y que reciben tratamiento antibiótico efectivo 17.
La clase B fue la más frecuente carbapenemasa reportada en este estudio (62,7%), identificándose 53 cepas de gen blaNDM (44 en K. pneumoniae; 9 en E. coli) presentes en seis regiones del país (Ancash, Cusco, Lambayeque, Loreto, Lima y el Callao). El primer reporte a nivel mundial de estas cepas fue en Suecia en el 2008 en un paciente procedente de la India 8 y posteriormente en el 2011 el primer caso en Latinoamérica se identificó en Guatemala 8 , 18. En el Perú, esta cepa se reportó por primera vez en el 2013 en el Hospital Nacional Edgardo Rebagliati Martins y posteriormente se diseminó por todo el territorio nacional 15 , 18. El gen blaVIM se presentó en el 7,0% (n=13) del total de carbapenemasas analizadas en este estudio y presentes en cepas de P. aeruginosa de tres IPRESS de Lima y Loreto.
En el caso del gen blaIMP en nuestro estudio se presentaron en el 17,3% del total, encontrándose en cepas de P. aeruginosa de diez IPRESS en seis regiones (Apurímac, Ayacucho, Cusco, Loreto, Lima y el Callao). Los genes previamente mencionados están entre los más diseminados por el mundo. En el Perú, su primer reporte fue en el 2013 19. Estas cepas utilizan un mecanismo de difusión mediante plásmidos lo que facilita su replicación en proporciones aceleradas y con escaso consumo de energía, lo cual permite la transferencia de genes, como los relacionados a la resistencia antimicrobiana, favoreciendo su diseminación en nosocomios y regiones más extensas.
Este estudio reporta por primera vez la coproducción de carbapenemasas blaIMP/VIM en cepas de P. aeruginosa, presente en el 7,6% (n=14) del total de estas enzimas en cinco IPRESS en Lima, Loreto y Callao. Su existencia puede explicarse mediante la transmisión por plásmidos ya que ambos son codificados por integrones de clase 1 que han sido descritos desde el 2006 en Polonia 20. Su importancia en la salud pública radica nuevamente en su fácil transmisión y el consecuente efecto multiplicador y diseminador relacionado con la resistencia antibacteriana.
Las carbapenemasas de clase D se presentaron en el segundo lugar de frecuencia (24,9%) encontrándose 27 con el gen blaOXA24-like en Acinetobacter spp. en nueve IPRESS de Cusco, Ica, La Libertad, Lambayeque, Lima y Callao; y 19 con el gen blaOXA23-like en cinco IPRESS de Junín, Loreto, Lima y Callao. No se encontraron cepas con genes blaOXA 48-like, blaOXA58-like y blaOXA143-like a pesar de que se crearon iniciadores específicos para ello. En la literatura científica el gen blaOXA-24 fue identificado por primera vez en 1997 en España y el blaOXA-23 en 1997 por primera vez en Escocia ( 8. En el Perú estos genes fueron aislados por primera vez en el 2014 en IPRESS en Lima 19, y poseen un espectro de actividad amplificado sobre cefalosporinas y carbapenémicos. Un ejemplo de ello es el Acinetobacter baumannii que ha adquirido mecanismos de resistencia a antibióticos a través de la producción de Oxacilinasas, además de la resistencia contra carbapenémicos, aminoglicósidos, quinolonas y polimixinas, lo cual desde el punto de vista clínico complica su manejo no solo considerando el amplio espectro de resistencia sino también las limitaciones en el diagnóstico y carencia de métodos fenotípicos estandarizados. Esto pone en evidencia que la resistencia antimicrobiana es frecuente en nuestro país y limita el uso de terapias efectivas contra las enfermedades, favoreciendo el crecimiento y diseminación de patógenos resistentes, con la consecuente prolongación de la estancia hospitalaria, aumento de costos en salud y mayores tasas de mortalidad y complicaciones a largo plazo.
Entre las limitaciones del estudio se debe mencionar un muestreo no probabilístico, ya que se reciben cepas de diferentes IPRESS como parte de los procesos de control diagnóstico, cuya selección depende del envió de la cepa. Por esta razón, los resultados del estudio no pueden extrapolarse a todo el Perú, pero son importantes porque reportan la presencia de cepas y genes específicos de vigilancia mundial y que están presentes en el país.
En conclusión, se identificaron 185 cepas con presencia de enzimas carbapenemasas de clase A, B y D en cepas de 30 IPRESS del Perú, durante el 2019. Su prevalencia fue del 59,7%, la clase B fue la más frecuente, y los genes más detectados fueron blaNDM, blaIMP, blaOXA24-like, blaKPC y blaOXA23-like.











 text in
text in 



