INTRODUCCIÓN
El Streptococcus agalactiae o estreptococo del grupo B (EGB), es una bacteria grampositiva, betahemolítica, anaerobia facultativa, catalasa negativa, positiva a prueba de Christie, Atkins y Munch-Petersen (CAMP) y resistente a la bacitracina 0,04U (BA) 1. Es un microorganismo comensal de la microbiota gastrointestinal que suele colonizar el tracto genitourinario. Es causa importante de infecciones graves en recién nacidos, embarazadas y adultos mayores de 60 años con enfermedades preexistentes 2. Además, posee una amplia variedad de factores de virulencia que son importantes para la adherencia e invasión celular; y la evasión de la inmunidad del huésped, lo que facilita su capacidad de causar enfermedad 3.
Entre los principales factores de virulencia de EGB se encuentran las adhesinas (FbsA, Lmb) ( 4, invasinas (hylB, SodA, cfb, Bca y Rib) 3 y las evasinas (Bac, ScpB y CPS) 4.
La penicilina es el tratamiento de elección frente a la infección causada por EGB o para la profilaxis antibiótica intraparto 2. En el caso de pacientes alérgicos a este antibiótico, se administra comúnmente agentes antimicrobianos alternativos como clindamicina, eritromicina o vancomicina 2.
La resistencia a macrólidos, lincosamidas y estreptograminas B se conoce como MLSb y puede estar mediada por varios mecanismos, como la metilación de la diana ARNr 23S, codificada por genes erm (ermB, ermTR, etc.), que puede presentar un fenotipo constitutivo (cMLSb) y se expresa tanto en presencia como en ausencia de macrólidos o un fenotipo inducible (iMLSb) en presencia de eritromicina, y presenta únicamente resistencia a los macrólidos de 14 y 15 átomos y sensibilidad in vitro a macrólidos de 16 átomos, lincosamidas y estreptograminas B 5.
Otro mecanismo de resistencia es la bomba de eflujo mediada por genes mefA/E que expresa resistencia de bajo nivel a eritromicina y sensibilidad a lincosamidas (fenotipo M) 5. Además, la resistencia a las lincosamidas (fenotipo L) es menos frecuente, resulta de la modificación e inactivación por las enzimas lincosamida nucleotidiltransferasa codificadas por genes lnu 5.
En el Perú, las investigaciones relacionadas con EGB están centradas en la colonización vaginal de gestantes 6. Tamariz et al. en 2004 7 ) describieron la presencia de resistencia a eritromicina. Sin embargo, existen limitados estudios sobre los genes de virulencia y resistencia asociados con este tipo de infecciones.
Las infecciones invasivas por EGB son una causa importante de morbilidad y mortalidad en los recién nacidos, gestantes y adultos mayores. Además, el incremento de EGB resistentes a eritromicina y clindamicina representa una preocupación importante en el tratamiento de infecciones. Por tal motivo, el objetivo de este estudio fue identificar molecularmente los genes de virulencia y de resistencia a macrólidos en aislamientos clínicos de EGB.
MENSAJES CLAVE
Motivación para realizar el estudio: el Streptococcus agalactiae (EGB) es causa de infecciones y posee varios factores de virulencia. El tratamiento de elección es la penicilina; sin embargo, en pacientes alérgicos, se administra clindamicina o eritromicina, pero se ha observado un aumento en la resistencia.
Principales hallazgos: el fenotipo cMLSb, los genes ermB y lmb fueron los más frecuentes. Todos fueron sensibles a penicilina y la resistencia a eritromicina y clindamicina fue cercana al 50%.
Implicancias: los resultados muestran la presencia de genes de virulencia y resistencia a macrólidos. Los estudios de epidemiología molecular permitirán conocer la realidad de EGB en el Perú.
EL ESTUDIO
Se realizó un estudio descriptivo transversal en el que se incluyeron todos los aislamientos clínicos (n=31) de EGB entre enero y abril de 2019, provenientes del Hospital San Bartolomé (n=19) y la Clínica Centenario Peruano Japonesa (n=12), recuperados de muestras de secreciones vaginales (SV) (n=9) y orina (n=22). Todos procedentes de pacientes de sexo femenino.
La identificación y susceptibilidad antimicrobiana se realizó con el sistema automatizado Vitek® 2 (BioMérieux, Francia). En el Laboratorio de Bacteriología del Instituto de Medicina Tropical «Daniel A. Carrión» de la Universidad Nacional Mayor de San Marcos (UNMSM), se confirmó la identificación evaluando la hemólisis, tinción de Gram, la prueba de catalasa, CAMP y la resistencia a BA. Además, se realizó el método fenotípico D-test y la interpretación según el Clinical and Laboratory Standards Institute (CLSI) para determinar los fenotipos cMLSb, iMLSb, M y L 8.
La detección molecular se llevó a cabo en el Laboratorio de Epidemiología Molecular y Genética del Instituto de Medicina Tropical «Daniel A. Carrión» del Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales (CITBM) de la UNMSM. Para la identificación de los genes de virulencia (lmb, bca y rib) y de resistencia a macrólidos (ermB, ermTR y mefA) se utilizó la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) convencional empleando como molde ADN total, tal como se ha descrito anteriormente 9.
Los reactivos fueron de Invitrogen (EE. UU.). Se empleó un termociclador VERITI Applied Byosistem (EE. UU.) y la siguiente reacción de amplificación: desnaturalización 95 °C, 5 min; seguida de 30 ciclos (lmb y rib), 32 ciclos (bca, mefA y ermTR) y 40 ciclos (mefA) (desnaturalización 95 °C, 30 s; hibridación 55 °C, 30 s; amplificación 72 °C, 45 s) y un período final de elongación 72 °C, 5 min. Los fragmentos esperados fueron bca (184pb), lmb (300pb), rib (369pb), mefA (345pb), ermTR (385pb) y ermB (639pb).
Se elaboró una base de datos en Excel de Windows XP. El análisis descriptivo se realizó con frecuencias y porcentajes del perfil de susceptibilidad antimicrobiana y la presencia de genes de virulencia y resistencia a macrólidos.
Este estudio se encuentra en el marco de las tesis de investigación de segunda especialidad y maestría en Microbiología que fue aprobado por la Unidad de Posgrado de la Facultad de Tecnología Médica de la Universidad Nacional Federico Villarreal y la Facultad de Farmacia de la UNMSM, respectivamente. El estudio sigue los lineamientos de las buenas prácticas y de ética en investigación biomédica, se trabajó con aislamientos almacenados en el banco de cepas de los dos establecimientos de salud, por lo que la información personal de las pacientes estuvo protegida.
HALLAZGOS
Los 31 aislamientos fueron confirmados como EGB, todos fueron betahemolíticos, cocos Gram positivos, catalasa negativa; además, resistentes a BA y positivos a la prueba de CAMP. El perfil de susceptibilidad mostró que fueron sensibles a penicilina, ampicilina y vancomicina, la resistencia a eritromicina, clindamicina y levofloxacino fue de 48,4%, 54,8% y 19,4%, respectivamente (Figura 1). La prueba fenotípica D-test mostró la presencia del fenotipo sensible (13/31), cMLSb (12/31), iMLSb 2/31, L (3/31) y M (1/31) (Tabla 1) (Figura 2). De los 18 aislamientos resistentes a eritromicina y/o clindamicina, el análisis por PCR mostró que 11/18 presentaban el gen ermB, 6/18 el gen ermTR y 2/18 el gen mefA. Cuatro aislamientos fueron positivos para más de un gen; tres para ermB - ermTR y uno para ermTR - mefA (Tabla 1).
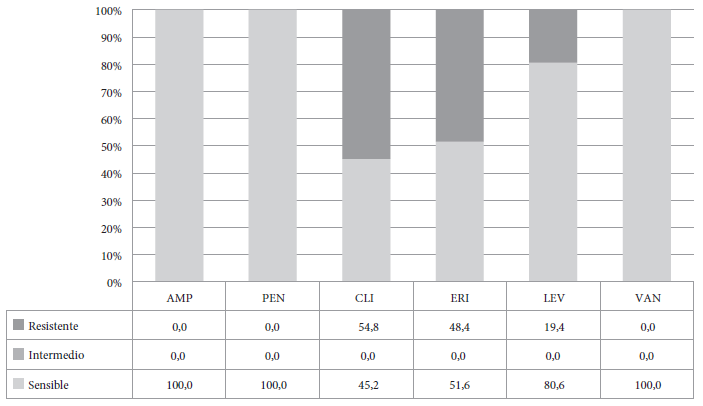
Figura 1 Perfil de susceptibilidad a los antimicrobianos de los aislamientos de Streptococcus agalactiae (n = 31).

Figura 2 Imagen de D- test positivo en aislamiento de Streptococcus agalactiae. Se observa un achatamiento del halo de inhibición de clindamicina (DA: 2μg) por acción de la inducción de eritromicina (E: 15 μg), esto es indicativo de un fenotipo iMLSb.
Tabla 1 Distribución de los fenotipos y genotipos de resistencia a macrólidos y lincosamidas en los aislamientos de Streptococcus agalactiae (n=31).
| Fenotipo | ERI | CLI | D-test | n | Genotipo | n |
|---|---|---|---|---|---|---|
| Sensible | S | S | - | 13 | Ninguno | 13 |
| cMLSb (constitutivo) | R | R | - | 12 | Solo ermB | 8 |
| Solo ermTR | 1 | |||||
| ermB-ermTR | 3 | |||||
| iMLSb (inducible) | R | S | + | 2 | Solo ermTR | 1 |
| ermTR-mefA | 1 | |||||
| M | R | S | - | 1 | Solo mefA | 1 |
| L | S | R | - | 3 | Ninguno | 3 |
ERI: eritromicina, CLI: clindamicina, R: resistente, S: sensible, (+): positivo; (-): negativo.
El análisis molecular reveló que el gen de virulencia lmb fue el más frecuente (23/31), seguido del gen bca (19/31) y el gen rib (8/31). En dos de los aislamientos no se evidenció la presencia de ninguno de los genes estudiados. Se detectó que 21/31 de los aislamientos tuvieron más de un gen de virulencia: 14/31 presentaron los genes lmb-bca y 7/31 los genes rib-lmb (Tabla 2).
Tabla 2 Presencia de genes de virulencia en los aislamientos de Streptococcus agalactiae.
| Genes de virulencia | Orina (n= 22) | Secreción vaginal (n=9) | Total (n=31) |
|---|---|---|---|
| n (%) | n (%) | n (%) | |
| No detectado | 2 (9,1) | 0 (0,0) | 2 (6,5) |
| Solo lmb | 1 (4,5) | 1 (11,1) | 2 (6,5) |
| Solo rib | 1 (4,5) | 0 (0,0) | 1 (3,2) |
| Solo bca | 4 (18,2) | 1 (11,1) | 5 (16,1) |
| lmb-bca | 10 (45,5) | 4 (44,4) | 14 (45,1) |
| rib-lmb | 4 (18,2) | 3 (33,3) | 7 (22,6) |
DISCUSIÓN
Los resultados del presente estudio muestran, que el fenotipo y genotipo de resistencia a macrólidos predominante fue cMLSb (38,7%) y el gen ermB (33,3%), respectivamente. El gen de virulencia lmb (74,2%) fue detectado con más frecuencia entre los EGB. Todos los aislamientos fueron sensibles a penicilina y la resistencia a eritromicina y clindamicina fue de casi el 50%. Estas observaciones refuerzan la importancia del EGB como un problema de salud pública y la necesidad de estrategias terapéuticas y de prevención alternativas contra estas infecciones, esto se logrará con una oportuna identificación de los marcadores moleculares y una mejor comprensión de los mecanismos de la patogénesis y resistencia 2.
Con relación al perfil de susceptibilidad, todos los aislamientos fueron sensibles a penicilina como es común encontrar en EGB 6 , 10 , 11. En cuanto a la resistencia a macrólidos y lincosamidas en EGB, esta fue cercana al 50% (eritromicina:15/31; clindamicina:17/31). Estos resultados fueron mayores que los observados en otros países de Latinoamérica como Chile (eritromicina: 17% y clindamicina:13%) 11; Colombia (eritromicina y clindamicina: 28%) 12; Brasil (eritromicina: 4,1% y clindamicina: 3%) 13 y Argentina donde se vio un aumento con los años (2013, eritromicina: 11,6% y clindamicina: 1,8%; 2019, eritromicina: 25% y clindamicina: 13,2%) 10 , 14. En Estados Unidos 15 se reportaron valores de resistencia a eritromicina de 50,7% y de clindamicina de 38,4%, resultados más cercanos a estos hallazgos. En Perú, un estudio publicado en 2004 mostró una resistencia a eritromicina de 11,5% 7 lo que indica una evolución en la resistencia a este fármaco a través de los años.
El fenotipo predominante fue cMLSb (12/15; 80%), que es también el fenotipo descrito con más frecuencia entre los aislamientos resistentes a macrólidos y lincosamidas reportados en España, Colombia, Brasil y Estados Unidos 5 , 12 , 13 , 15.
El análisis genotípico mostró al gen ermB como el más habitual (11/15; 73,3%), estos resultados son similares a lo reportado en Francia (76%) 16 y España (82%) 5. Sin embargo, un estudio en Brasil muestra al gen ermA como el más frecuente seguido del gen ermB, aunque con una frecuencia menor, 39 y 28%, respectivamente 13.
Se detectaron tres aislamientos con el fenotipo L (de resistencia solo a lincosamidas) que no presentaron ninguno de los genes de resistencia estudiados. Sin embargo, este fenotipo es altamente asociado al gen lnu que no fue investigado en este trabajo debido a su baja frecuencia 5.
Dentro de los genes de virulencia, el gen lmb fue el más frecuente (74,2%; 23/31), especialmente en aislamientos de SV (88,9%; 8/9), lo que sugiere que este factor es crucial para la colonización del tracto genital inferior 4 , 9. Estos hallazgos son similares a los reportados por Oviedo et al., Udo et al., Dutra et al., Eskandarian et al., Wu et al. y Bobadilla et al., con frecuencias de 94,3% en aislamientos de SV de gestantes, 88,3% en SV y orina, 100% y 97,1% en colonizados; y 94,9 y 94% en adultos asintomáticos, respectivamente 4 , 10 , 13 , 17 - 19.
Con relación al gen bca, que se asocia a la adherencia a las células epiteliales y diseminación intracelular, se encontró en el 61,3% (19/31) de los aislamientos, siendo menor a lo reportado por Oviedo et al. y Bobadilla et al. con 88,6 y 87,8% en aislamientos de SV de gestantes, respectivamente 10 , 19. Sin embargo, fue mayor que lo informado por Dutra et al., Jalalifar et al., Eskandarian et al. y Wu et al. con 54,4%, 22%, 14,6% y 6% en aislamientos de infecciones y colonizaciones, respectivamente 13 , 17 , 18 , 20.
La frecuencia del gen rib fue de 25,8% (8/31), similar a los reportado por Eskandarian et al. (29,1%) 17. Sin embargo, fue menor a lo que informaron Bobadilla et al. 19 con 82,5%, Laczeski et al. 9 con 54,5% y Jalalifar et al. 20 con 40% en aislamientos de infecciones y colonizaciones. Probablemente estas diferencias observadas en la frecuencia de los genes bca y rib podrían atribuirse a diversos factores como variaciones geográficas, diferencias temporales en la distribución de las cepas de EGB, origen de los aislamientos, tiempo de investigación y población, como ha sido manifestado por otros investigadores 10 , 13. Solo en dos de los aislamientos analizados no se detectó ningún gen de virulencia, probablemente podrían portar otros genes no evaluados, ya que la virulencia del EGB es atribuible a múltiples genes 4.
Este trabajo presenta algunas limitaciones que se deben mencionar; los aislamientos incluidos en el estudio se obtuvieron de dos establecimientos de salud durante un periodo específico, por lo que no pueden extrapolarse a todos los establecimientos de salud; asimismo, solo se evaluaron algunos genes de virulencia y de resistencia a macrólidos, falta conocer la frecuencia de otros genes.
En conclusión, se observó la presencia de, por lo menos, un gen de virulencia en casi todos los aislamientos y una resistencia discretamente elevada a macrólidos de 48,4%, siendo el mecanismo de resistencia más frecuente la producción de metilasa, mayoritariamente por el gen ermB, hecho que constituye el primer reporte de genes de virulencia y resistencia a macrólidos en el Perú. Estos hallazgos muestran la necesidad de implementar estudios de epidemiología molecular con un mayor número de aislamientos en establecimientos de salud para poder conocer la realidad de EGB en el Perú.















