Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Horizonte Médico (Lima)
Print version ISSN 1727-558X
Horiz. Med. vol.14 no.4 Lima Oct./Dec. 2014
ARTÍCULOS ORIGINALES
Análisis de asociación genética entre el SNP RS914458 del gen proteína tirosina fosfatasa no receptor tipo 1 (ptpn1) y diabetes tipo 2 en población peruana
Genetic association analysis between SNP rs914458 of protein tyrosine phosphatase, non-receptor type 1(ptpn1) gene and Type 2 Diabetes in Peruvian Population
Mónica Paredes Anaya1, Wilser Andrés García-Quispes2, Frank Lizaraso Soto3, Carlos Padilla Rojas4, Dina TorresGonzales5, Jorge Calderón Ticona6, Helard Manrique Hurtado6, José Solís Villanueva7
1 PhD, Escuela de Genética y Biotecnología, Facultad de Ciencias Biológicas, UNMSM.
2 PhD, Escuela de Postgrado, Facultad de Ciencias Biológicas, UNMSM.
3 PhD MD, Escuela de Postgrado, Facultad de Medicina Humana, USMP.
4 Doctorando Escuela de Postgrado, Facultad de Ciencias Biológicas, UNMSM.
5 Doctorando Escuela de Postgrado, Facultad de Ciencias Biológicas, UNMSM; Lab. de Biomedicina, INS.
6 MD, Servicio de Endocrinología, Hospital Arzobispo Loayza.
7 PhD MD, Jefe del Servicio de Endocrinología, Hospital Arzobispo Loayza.
RESUMEN
Objetivo: Evaluar la posible asociación entre el SNP rs914458 (C>G) del gen PTPN1con la DM2 en una población de la zona urbana de Lima – Perú.
Material y Métodos: El estudio incluyó un total de 216 personas de la zona urbana de Lima correspondientes a un grupo control (n = 123) y un grupo de pacientes diagnosticados con diabetes tipo 2 provenientes del Hospital A. Loayza (n = 93). La genotipificación del SNP se llevó a cabo mediante PCR y con un secuenciador ABI PRISM 310. El análisis de asociación se llevó a cabo con el uso de la herramienta web SNPStats para realizar cinco modelos de regresión logística. El efecto de la asociación genética se estableció con el valor de OR.
Resultados: La frecuencia del MAF (alelo G) fue de 0.22 en el grupo de controles y en el grupo de pacientes. Ninguno de los modelos de regresión muestra valores de OR (considerando los CI) por encima o por debajo del valor de referencia.
Conclusión: No se encontró asociación genética significativa entre el SNP rs914458 del gen PTPN1 y la DM2 para la población de la zona urbana de Lima – Perú.
Palabras clave: Gen PTPN1, polimorfismo de nucleótido simple (SNP), frecuencia alélica, asociación genética, diabetes tipo 2. (Fuente: DeCS BIREME).
ABSTRACT
Objective: Evaluate the association between SNP rs914458 (C>G) of PTPN1 gene with T2DM in a population from the urban area of Lima – Peru.
Material and Methods: This study included a total of 216 subjects from the urban area of Lima. The number of subjects in control group was 123, and 93 patients diagnosed with type 2 diabetes from Hospital A. Loayza. SNP genotyping was performed by PCR and sequencing using ABI PRISM 310 DNA sequencer. The association analysis was carried out using the web tool SNPStats and five logistic regression models were performed. The effect of genetic association was established with the OR.
Results: The frequency of MAF (allele G) was 0.22 in both control and patient groups. The OR values were not different from the reference values considering the respective confidence interval.
Conclusion: No significant association was found between the SNP rs914458 and the PTPN1 gene with T2DM for the urban population of Lima – Peru.
Key words: PTPN1 Gene, sample nucleothic polymorphism (SNP), alelic frequency, genetic association, type 2 diabetes. (Source: MeSH NLM).
INTRODUCCIÓN
Las proteínas tirosina fosfatasas son una familia de enzimas que catalizan la defosforilación de proteínas tirosina fosforiladas y participan tanto en el inicio como en la terminación de diferentes señales celulares. La proteína tirosina fosfatasa 1B (PTP1B), codificada por el gen PTPN1, es un miembro de esta familia y ha sido ampliamente estudiada por su gran interés farmacéutico principalmente por su participación en el metabolismo de la insulina aunque también ha sido vinculada, en interacción con otros factores, sobre el desarrollo de procesos inflamatorios, enfermedades cardiacas, obesidad, cáncer de mama, cáncer de próstata y diabetes tipo 2 (DM2) (1, 2). PTP1B está encargada de la defosforilación de residuos de fosfotirosina, que es la forma activada del receptor de insulina, interrumpiendo la vía de señalización de esta hormona (3, 4). En estudios in vivo, se ha demostrado que ratones deficientes en PTP1B presentan un aumento en la sensibilidad a insulina y resistencia a la obesidad inducida por la dieta (5, 6).
Se han realizado numerosos estudios para encontrar genes candidatos de DM2 y se ha podido determinar que en el cromosoma 20, se concentran varios genes con un efecto sobre la modulación del riesgo para la aparición de la enfermedad, siendo el gen PTPN1 un miembro importante de este grupo (7). Uno de los estudios más prometedores, encontró evidencias de la asociación y ligamiento del gen PTPN1 con DM2 (8). En contraste, otro estudio describe que la distribución de alelos y genotipos para cinco marcadores polimórficos del gen PTPN1 no fue diferente entre un grupo control y un grupo de pacientes con DM2 concluyendo que no había asociación entre el gen y DM2 (9). Resultados similares, de no asociación con DM2, fueron descritos al estudiar dos variantes del promotor PTPN1 en individuos polacos e iraníes (10). Polimorfismos en regiones no codificantes (como intrones o regiones cercanas al gen) también han sido evaluados, así, algunos SNPs no muestran asociación genética como ocurre con el SNP rs16989673, localizado en la región 3-UTR, para una población polaca (11), mientras que otros SNPs también localizados en regiones no codificantes del gen PTPN1 sí han mostrado asociación con DM2 (12, 13), por ejemplo, el SNP rs941798 del intrón 1 (14) o los SNPs 7077 G/C y rs914458 localizados a 10kb en el extremo 3 del gen PTPN1 (15). El gen PTPN1 también ha mostrado asociación con la sensibilidad a la insulina, la dislipidemia, el índice de masa corporal y los niveles de colesterol (16, 17, 18). Sin embargo, las asociaciones genéticas encontradas deben ser explicadas por los procesos biológicos que lleven al desarrollo de la enfermedad, así, una red de interacción basada en la expresión génica (mediante microarrays) muestra que la expresión incrementada de EGFR (factor de crecimiento epidermal) lleva a una nefropatía diabética mediante las interacciones de EGFR con PTPN1 y con CAV1 (caveolin 1) que también es considerado un gen candidato de DM2 por lo que esta interacción tendría un rol importante en el desarrollo de esta enfermedad (19).
Un estudio llevado a cabo con una población iraní muestra que el polimorfismo 1484insG del gen PTPN1 está asociado con la resistencia a la insulina en varones no diabéticos aunque sugiere la replicación del estudio con una población de mayor tamaño (20). Como ocurre con otras enfermedades complejas, se observan resultados contradictorios para los estudios de asociación y son varios los motivos que pueden explicar estas diferencias. Entre ellos se encuentra la posible interacción entre el gen evaluado y otros factores moduladores que son parte del perfil genético de las diferentes poblaciones o grupos étnicos, los diferentes factores ambientales considerados en cada estudio, el establecimiento del grupo control o el poder estadístico que está influenciado por el tamaño de las muestras y por la frecuencia de los alelos en la población evaluada (21, 22, 23).
El objetivo de nuestro estudio fue evaluar la posible asociación entre el SNP rs914458 (C>G) del gen PTPN1 con la diabetes tipo 2 en una población de la zona urbana de Lima – Perú.
MATERIAL Y MÉTODOS
Población en estudio
Incluyó un total de 216 personas de la zona urbana de Lima y de origen mestizo: grupo control (n = 123) y un grupo de pacientes (n = 93) diagnosticadas con DM2. De este grupo, 24 eran solo diabéticas mientras que 69 eran diabéticas obesas. El grupo control estuvo conformado por individuos sanos que no presentaban: DM2, obesidad, hipertensión, problemas cardiovasculares e hipercolesterolemia, con edad promedio de 30 años, provenientes del Hospital A. Loayza.
Extracción de DNA
Se extrajo el DNA genómico, utilizando el kit de extracción PureLink Genomic DNA Kits (INVITROGEN), obteniéndose a partir de 200µL de linfocitos, 50µL de DNA que fue almacenado a -20º C.
Genotipificación
Información general sobre el SNP rs914458 del gen PTPN1 se obtuvo mediante la revisión de la base del datos del National Center for Biotechnology Information (NCBI). Las mezclas de PCR se realizaron en un volumen de 20 µL, con buffer PCR 1X, 200 µmol de dNTP, 1.5 mM de MgCl2, 0.25 U AmpliTaq Gold, 40 ng de DNA genómico y 0.5 µL de cada primer. Las condiciones de amplificación para el SNP fueron: 95ºC por 2 min, 30 ciclos de 95ºC por 30 s, 64ºC por 30 s y 72ºC por 10 s, y finalmente 72ºC por 10 min. La visualización de cada fragmento se realizó por electroforesis en gel de agarosa al 1.5%. Los fragmentos amplificados fueron purificados con el kit High Pure PCR cleanup micro. Se realizó una segunda amplificación con el kit SnaPshot Multiplex (INVITROGEN) a las siguientes condiciones: 25 ciclos de 96°C por 10 s, 50°C por 5 min, 60°C por 30 s y 4°C 30 s. Para la remoción de los grupos fosforilados se realizó la digestión con la enzima Calf Intestinal Alkaline Phosphatase (CIAP) añadiéndose 1µL de CIAP a cada producto de amplificado y se llevó al termociclador a 37°C por 1 h, 75°C por 15 min y 4°C por 30 min. Posteriormente, los SNPs fueron visualizados en el secuenciador ABIPRISM 310 del Laboratorio de Biotecnología y Biomedicina del INS.
Análisis estadístico
El cálculo de las frecuencias alélicas, genotípicas así como el análisis de asociación se llevaron a cabo con el uso de la herramienta web SNPStats (24). La evaluación del equilibrio Hardy-Weinberg (H-W) para los alelos del SNP se llevó a cabo con un test exacto. Se realizaron cinco modelos de regresión logística correspondientes a los modelos codominante, dominante, sobredominante, recesivo y log-aditivo. El efecto de la asociación genética se estableció con el valor de odds ratio (OR).
RESULTADOS
Los resultados generales de los grupos evaluados se muestran en las tablas 1 y 2. La frecuencia alélica para el alelo menos frecuente (alelo G) es de 0.22 tanto en el grupo de controles como en el de pacientes, no se observó diferencias para las frecuencias alélicas del polimorfismo entre los grupos control.
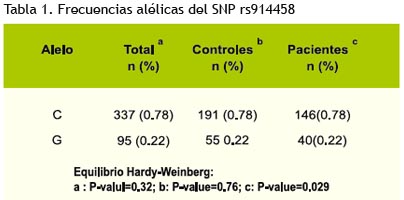
Antes de realizar el análisis de asociación, se determinó si los alelos se encontraban en equilibrio de Hardy-Weinberg (test exacto) para el grupo control y para el grupo de pacientes. En el grupo de controles no se encontró una desviación significativa del equilibrio H-W (P=0.79) por lo que este grupo puede ser usado como referencia. En el grupo de pacientes se observó una desviación del equilibrio H-W (P=0.029) y que se muestra en la tabla 1.
La desviación del equilibrio H-W en el grupo de pacientes no afecta el análisis de asociación que se describe en la tabla 2, sin embargo puede indicar, entre otras cosas, una estratificación de este grupo.
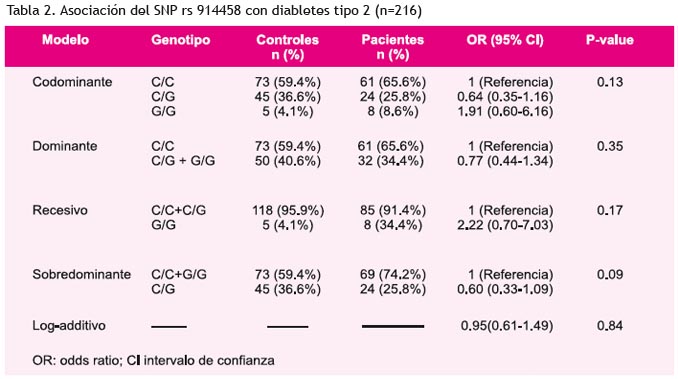
El análisis de asociación se llevó a cabo considerando cinco modelos de herencia: codominante, dominante, sobredominante, recesivo y el logaditivo. El modelo codominante corresponde a la comparación de los genotipos heterocigoto y homocigoto (portadores del alelo menos frecuente) con el genotipo homocigoto del alelo más frecuente.
El modelo dominante considera la sumatoria de los genotipos heterocigoto y homocigoto (portadores del alelo menos frecuente) comparado con el genotipo homocigoto del alelo más frecuente. De manera similar se realizan los modelos sobredominante y recesivo. En el caso del modelo log-aditivo, se considera la variación sobre el riesgo cuando existe una o dos copias del alelo menos frecuente, G y GG, respectivamente. Las frecuencias genotípicas encontradas pueden ser observadas en el modelo codominante para el grupo de controles y el de pacientes.
Los valores de OR (odds ratio) indican la asociación de los genotipos con la enfermedad, así, un valor mayor a 1 corresponde a un incremento de riesgo, mientras que un valor menor a 1 indica una reducción en el riesgo. Sin embargo, los valores de OR deben ser considerados en conjunto con los intervalos de confianza. Considerando todo esto, no se encuentra ninguna asociación estadística entre el polimorfismo evaluado y la enfermedad.
Adicionalmente, se realizó un análisis de asociación estratificando el grupo de pacientes en un subgrupo de 24 personas diabéticas y otro subgrupo de 69 personas diabéticas obesas. Los resultados obtenidos para el análisis asociación no muestran diferencias con respecto a los resultados globales mostrados en la tabla 2.
DISCUSIÓN
El polimorfismo rs914458 (C>G) se encuentra localizado a 10 kb downstream del gen PTPN1 que codifica una proteína tirosina fosfatasa 1B (PTP1B). Esta proteína interviene en la regulación negativa de la producción de insulina por lo que la evaluación de los polimorfismos en este gen o en su cercanía tiene un carácter relevante en la investigación sobre el desarrollo de DM2 mediante estudios de asociación genética. Por este motivo nuestro grupo ha evaluado la posible asociación entre el polimorfismo rs914458 del gen PTPN1 sobre el desarrollo de diabetes tipo 2 en la población peruana (123 controles y 93 pacientes).
En la población peruana poco se conoce sobre la frecuencia de éste y otros SNPs que han sido previamente investigados para conocer su papel en el desarrollo de la DM2. En este estudio, reportamos una frecuencia de 0.22 para el alelo menos frecuente (G) en la población peruana. Esta frecuencia es la misma en ambos grupos, controles y pacientes por lo que se puede intuir que no existe una asociación de este alelo con la enfermedad.
Al realizar el análisis de asociación genética se han tenido en cuenta factores como el tamaño de los grupos, así como el equilibrio H-W de los alelos. Una desviación del equilibrio H-W se encontró en el grupo de pacientes. Son varios los motivos que pueden explicar esta desviación, entre ellos, una posible asociación con la enfermedad (aunque esto no se ve reflejado al realizar los análisis de asociación en nuestro estudio). Otro motivo para la desviación del equilibrio H-W es que el grupo en cuestión está formado por varios subgrupos (como se ha descrito en resultados).
Los polimorfismos del gen PTPN1 han sido estudiados previamente en diferentes poblaciones a nivel mundial. Algunos polimorfismos de este gen sí muestran asociaciones con el desarrollo de DM2, enfermedades relacionadas a DM2 y con la capacidad de respuesta frente a la glucosa en la sangre (respuesta aguda a la insulina o con el índice de sensibilidad a la insulina) (14, 15, 25, 26). Otros estudios, por el contrario, reportan una falta de asociación de los SNPs del gen PTPN1 con la DM2 (27). Estas diferencias pueden ser explicadas por perfil genético del grupo poblacional evaluado así como por otros factores ambientales implicados en el desarrollo de la enfermedad y que no son siempre considerados en los estudios de asociación caso-control. También se debe tener en consideración el tamaño de los grupos controles y pacientes. Aunque existe evidencia de la implicación de los polimorfismos del gen PTPN1 con la DM2, el SNP rs914458 no ha mostrado asociación genética en diferentes poblaciones (14, 25, 26). Sin embargo, un estudio para la población francesa, llevado a cabo con 1227 pacientes y 1047 controles muestra que el SNP rs914458 está asociado estadísticamente con el riesgo de DM2 (bajo un modelo dominante, OR = 1.43 [1.06–1.94]) y con el fenotipo obesidad moderada (OR = 1.20 [1.01–1.43]) (15).
Nuestro grupo, viene estudiando este polimorfismo en análisis de asociación genéticos basados en familias, la obtención y análisis de esta información, permitirá aclarar el papel de este polimorfismo en el desarrollo de la enfermedad.
Abreviaciones
H-W: Hardy-Weinberg DM2: Diabetes tipo 2 OR: odds ratio CI: Intervalo de confianza PTPN1: gen de la proteína tirosina fosfatasa no receptor tipo 1. MAF: alelo menos frecuente
Agradecimiento
Este proyecto ha sido financiado por la Facultad de Medicina Humana de la Universidad de San Martin de Porres y la Fundación Hipólito Unanue.
REFERENCIAS BIBLIOGRÁFICAS
1. Cho H. Protein tyrosine phosphatase 1B (PTP1B) and obesity. Vitam Horm. 2013;91:405-424.
2. Feldhammer M, Uetani N, Miranda-Saavedra D et al. PTP1B: a simple enzyme for a complex world. Crit Rev Biochem Mol Biol. 2013;48(5):430-445.
3. Goldstein BJ, Bittner-Kowalczyk A, White MF et al. Possible facilitation by the formation of a ternary complex with the Grb2 adaptor protein. J Biol Chem. 2000;275(6):4283-4289.
4. Seely BL, Staubs PA, Reichart DR et al. Protein tyrosine phosphatase 1B interacts with the activated insulin receptor. Diabetes. 1996;45(10):1379-1385.
5. Elchebly M, Payette P, Michaliszyn E et al. Increased insulin sensitivity and obesity resistance in mice lacking the protein tyrosine phosphatase-1B gene. Science. 1999;283(5407):15441548.
6. Klaman LD, Boss O, Peroni OD et al. Increased energy expenditure, decreased adiposity, and tissue-specific insulin sensitivity in protein-tyrosine phosphatase 1B-deficient mice. Mol Cell Biol. 2000;20(15):5479-5489.
7. Hale PJ, López-Yunez AM, Chen JY. Genome-wide meta-analysis of genetic susceptible genes for Type 2 Diabetes. BMC Syst Biol. 2012;6Suppl 3:S16.
8. Ghosh S, Watanabe RM, Valle TT et al. The Finland-United States investigation of non-insulin-dependent diabetes mellitus genetics (FUSION) study. I. An autosomal genome scan for genes that predispose to type 2 diabetes. Am J Hum Genet. 2000;67(5):11741185.
9. Wanic K, Malecki MT, Klupa T et al. Lack of association between polymorphisms in the gene encoding protein tyrosine phosphatase 1B (PTPN1) and risk of Type 2 diabetes. Diabet Med. 2007;24(6):650-655.
10. Meshkani R, Taghikhani M, Al-Kateb H et al. Polymorphisms within the protein tyrosine phosphatase 1B (PTPN1) gene promoter: functional characterization and association with type 2 diabetes and related metabolic traits. Clin Chem. 2007;53(9):1585-1592.
11. Malodobra M, Pilecka A, Gworys B et al. Single nucleotide polymorphisms within functional regions of genes implicated in insulin action and association with the insulin resistant phenotype. Mol Cell Biochem. 2011;349(1-2):187-193.
12. Echwald SM, Bach H, Vestergaard H et al. A P387L variant in protein tyrosine phosphatase-1B (PTP-1B) is associated with type 2 diabetes and impaired serine phosphorylation of PTP-1B in vitro. Diabetes. 2002;51(1):1-6.
13. Mok A, Cao H, Zinman B et al. A single nucleotide polymorphism in protein tyrosine phosphatase PTP-1B is associated with protection from diabetes or impaired glucose tolerance in Oji-Cree. J Clin Endocrinol Metab. 2002;87(2):724-727.
14. Bento JL, Palmer ND, Mychaleckyj JC et al. Association of protein tyrosine phosphatase 1B gene polymorphisms with type 2 diabetes. Diabetes. 2004;53(11):3007-3012.
15. Cheyssac C, Lecoeur C, Dechaume A et al. Analysis of common PTPN1 gene variants in type 2 diabetes, obesity and associated phenotypes in the French population. BMC Med Genet. 2006;5;7:44.
16. Spencer-Jones NJ, Wang X, Snieder H, et al. Protein tyrosine phosphatase-1B gene PTPN1: selection of tagging single nucleotide polymorphisms and association with body fat, insulin sensitivity, and the metabolic syndrome in a normal female population. Diabetes. 2005;54(11):3296-3304.
17. Kipfer-Coudreau S, Eberle D, Sahbatou M, et al. Single nucleotide polymorphisms of protein tyrosine phosphatase 1B gene are associated with obesity in morbidly obese French subjects. Diabetologia 2004;47:1278–1284.
18. Olivier M, Hsiung CA, Chuang LM et al. Single nucleotide polymorphisms in protein tyrosine phosphatase 1beta (PTPN1) are associated with essential hypertension and obesity. Hum Mol Genet. 2004;13(17):1885-1892.
19. Sengupta U, Ukil S, Dimitrova N et al. Expression-based network biology identifies alteration in key regulatory pathways of type 2 diabetes and associated risk/complications. PLoS One. 2009;4(12):e8100.
20. Meshkani R, Taghikhani M, Mosapour A et al. 1484insG polymorphism of the PTPN1 gene is associated with insulin resistance in an Iranian population. Arch Med Res. 2007;38(5):556-562.
21. Balding DJ. A tutorial on statistical methods for population association studies.Nat Rev Genet. 2006;7(10):781-791.
22. Clarke GM, Anderson CA, Pettersson FH et al. Basic statistical analysis in genetic case-control studies. Nat Protoc. 2011;6(2):121-133.
23. Lewis CM, Knight J. Introduction to genetic association studies. Cold Spring Harb Protoc. 2012;3:297-306.
24. Solé X, Guinó E, Valls J et al. SNPStats: a web tool for the analysis of association studies. Bioinformatics. 2006;22(15):1928-1929.
25. Florez JC, Agapakis CM, Burtt NP et al. Association testing of the protein tyrosine phosphatase 1B gene (PTPN1) with type 2 diabetes in 7,883 people. Diabetes. 2005;54(6):1884-1891.
26. Palmer ND, Bento JL, Mychaleckyj JC et al. Association of protein tyrosine phosphatase 1B gene polymorphisms with measures of glucose homeostasis in Hispanic Americans: the insulin resistance atherosclerosis study (IRAS) family study. Diabetes. 2004;53(11):3013-3019.
27. Bodhini D, Radha V, Ghosh S et al. Lack of association of PTPN1 gene polymorphisms with type 2 diabetes in south Indians. J Genet. 2011;90(2):323-326.
Fuentes de financiamiento
Las fuentes de financiación no han tenido participación en el diseño del estudio, la colección de datos, el análisis o la interpretación de estos, en la redacción del manuscrito o en la decisión de enviarlo para publicación.
Conflicto de interés
Los autores expresan que no hay conflicto de interés al redactar el manuscrito.
Correspondencia:
Mónica Paredes Anaya.
Dirección: Facultad de Ciencias Biológicas. Universidad Nacional Mayor de San Marcos. Lima (Perú).
Teléfono: (01) 6197000
Correo electrónico: monica.paredes.anaya@gmail.com
Recibido: 29 de Agosto de 2014
Aprobado: 06 de Octubre de 2014













