Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Horizonte Médico (Lima)
Print version ISSN 1727-558X
Horiz. Med. vol.15 no.1 Lima Jan./Mar. 2015
ARTÍCULOS ORIGINALES
Asociación entre los Microsatelites D4S2912, D4S230 y D4S3001 (4P15.1) y Diabetes Tipo 2 en la población peruana
Association of Microsatellites D4S2912, D4S230 and D4S3001 (4P15.1) to type 2 diabetes in peruvian population
Mónica Paredes Anaya1, Dina Torres Gonzales2, Frank Lizaraso Soto3, Carlos Padilla Rojas4, Jorge Calderón Ticona5, Jorge Medina3.1, Giovanna Rodríguez Lay6
1 PhD, Escuela de Genética y Biotecnología, Facultad de Ciencias Biológicas, UNMSM, Lima - Perú.
2 Doctorando Escuela de Postgrado, Facultad de Ciencias Biológicas, UNMSM, Lima - Perú.
3 PhD, Escuela de Postgrado, Facultad de Medicina Humana, USMP, Lima - Perú.
3.1 MG. Facultad de Medicina Humana, Universidad de San Martín de Porres, Lima - Perú.
4 Blgo. Laboratorio de Biotecnología y Biología Molecular, Instituto Nacional de Salud (INS), Lima - Perú.
5 MD, Servicio de Endocrinología, Hospital Arzobispo Loayza, Lima - Perú.
6 Dr. MD, Servicio de Endocrinología, Hospital Arzobispo Loayza., Lima - Perú.
RESUMEN
Objetivo: Determinar la asociación entre los microsatélites D4S2912, D4S230 y D4S3001 y diabetes tipo 2 (DM2) en la población peruana.
Material y Métodos: En 99 diabéticos se analizaron los marcadores de los microsatélites D4S2912, D4S230 y D4S3001 y como controles: individuos sanos procedentes de Lima, 120 para D4S2912, 129 para D4S230 y 133 para D4S3001. Se procesó el ADN extraído de sangre endovenosa, amplificándose por la técnica de la reacción en cadena de la polimerasa (PCR) a los tres marcadores de la región 4p15.1. Finalmente, se analizó las frecuencias alélicas y genotípicas de los marcadores y determinó la homogeneidad genética de la población mediante la prueba de Hardy-Weinberg (H-W), continuándose con el estudio de asociación a DM2 en casos versus controles.
Resultados: Se encontró en D4S2912 y D4S3001 ausencia de un alelo en el grupo control al compararlo con la población diabética. Al realizar el análisis de asociación utilizando el modelo de regresión logística condicional y la prueba de permutación de Montecarlo, se observó asociación entre D4S3001 y población peruana con DM2.
Conclusión: Esta investigación muestra al microsatélite D4S3001, localizado en la región cromosómica 4p15.1 como un marcador asociado a DM2 en la población peruana y que se encuentra en una región cromosómica que contendría un gen(es) que jugaría(n) un rol en la etiología de DM2.
Palabras clave: Frecuencia alélica, frecuencia genotípica, equilibrio de Hardy-Weinberg, asociación, microsatélite. (Fuente: DeSC BIREME).
ABSTRACT
Objective: To determine the association of microsatellite markers D4S2912, D4S230 and D4S3001 with diabetes type 2 (DM2) in the Peruvian population.
Material and Methods: We analyzed the microsatellite markers D4S2912, D4S230 and D4S3001 in 99 diabetics and 120 healthy individuals to D4S2912, D4S230 and 129 to 133 for D0453001 as controls from Lima. We processed the 0NA extracted from blood intravenously, amplified by the technique of polymerase chain reaction (PCR) for the three markers in the region 4p15.1. Finally, we analyzed the polymorphisms of the markers and determined the genetic homogeneity of the population through proof of Hardy-Weinberg (HW), and continued in the study of association of 0M2 in patients versus controls.
Results: In D4S2912 and D4S3001 there was absence of an allele in the control group when compared with the diabetic population. When performing association analysis using conditional logistic regression model and the Monte Carlo permutation test, they showed an association between D4S3001 and Peruvian population with DM2.
Conclusion: This research shows the microsatellite D4S3001, located on chromosome 4p15.1 region as a marker associated with DM2 in the Peruvian population and is located in a chromosomal region that contain a gene(s) which would play a role in the etiology DM2.
Key words: allelic frequency, genotypic frequency, Hardy-Weinberg, association, microsatellite. (Source: MeSH NLM).
INTRODUCCIÓN
La diabetes mellitus tipo 2 (DM2), es un trastorno metabólico que incapacita al paciente en producir suficiente insulina o usarla adecuadamente (1), sin la cantidad suficiente de insulina el organismo no es capaz de metabolizar la glucosa en las células.
Desde el punto de vista genético, se considera que algunas personas presentan susceptibilidad genética a desarrollar DM2, y esto sería influenciado a su vez por otros factores de riesgo como obesidad, hipertensión, hipercolesterolemia e hipertrigliceridemia (2,3).
Esta enfermedad crónica presenta una etiología genética que viene siendo dilucidado a través de la búsqueda de genes candidatos (4).
A la fecha, se han encontrado genes candidatos asociados a DM2 involucrados en distintos grupos étnicos (3,5,6). 5eclen et al. (7) realizaron estudios de prevalencia de DM2 en población peruana y observaron que esta enfermedad es más frecuente en la costa (7.6%) que en la sierra (1.3%).
Estos resultados confirman otros estudios que relacionan prevalencia de DM2 y el componente genético de la población, observando 20% en México- americanos, 15% en personas de origen africano y 11.9% en las poblaciones de origen caucásico (8), considerando que poblaciones con un origen étnico común como las poblaciones hispanoamericano comparten variaciones (mutaciones) en genes comunes causales de DM2.
Los marcadores moleculares como los microsatélites han permitido descubrir el componente genético que subyace en las enfermedades crónicas de alta prevalencia como DM2 (9).
Estos marcadores tienen una alta tasa de mutación y un mayor número de alelos por locus a diferencia de otros marcadores como los polimórfismos de nucleótido simple (SNP's) (10).
Además los pocos antecedentes que existen de esta región cromosómica (4p15.1) han sido realizados en microsatélites, postulándose genes de susceptibilidad asociada a obesidad en población mexicana (11,12), observando al microsatélite D4S2912 con desequilibrio de ligamiento de 4.5, considerado un valor alto para enfermedades genéticas complejas (4,11) Fig. 1.
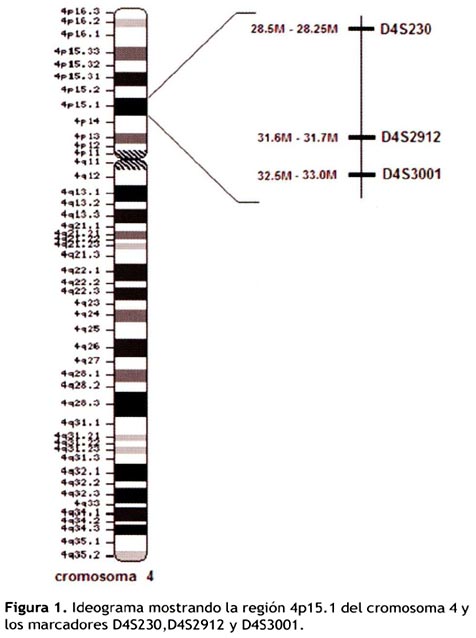
En un estudio familiar (tríos caso-progenitores), el microsatélite D4S230 (4p15.1) presentaba trasmisión diferencial de alelos de padres a hijos, encontrándose que los estudios familiares reducían la probabilidad de falsos positivos a diferencia de los problemas de estratificación poblacional observados en los estudios caso-control (13).
Considerando estos antecedentes, se propuso evaluar la posible asociación entre los microsatélites D4S230, D4S2912 y D4S3001 (4p15.1) y diabetes tipo 2 en la población peruana.
MATERIAL Y MÉTODOS
Población en estudio
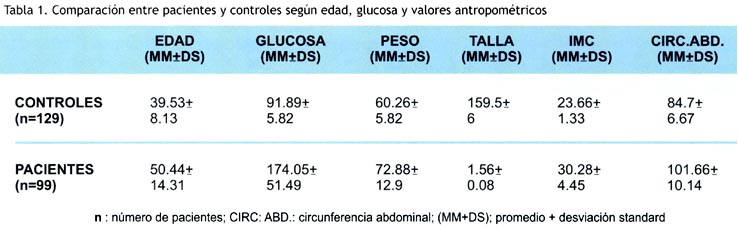
Se analizaron dentro de la población urbana de Lima, mayoritariamente de origen mestizo a 99 pacientes diagnosticados con DM2 y como controles 120 individuos sanos para 0452912, 129 para D4S230 y 133 para D4S3001 que no presentan DM2, sobrepeso u obesidad, (Tabla 1), provenientes de los Servicios de Endocrinología del Hospital Edgardo Rebagliati Martins y Hospital Nacional Arzobispo Loayza (Lima- Perú).
Extracción de ADN
Se realizó la extracción de ADN a partir de 3 ml de sangre obtenida de cada paciente utilizando kit de extracción de ADN de Invitrogen. Los pacientes firmaron el consentimiento informado aprobado por el Comité de Ética de la Facultad de Medicina Humana de la Universidad de San Martín de Porres.
Análisis molecular
A partir del ADN extraído, se amplificaron los fragmentos correspondientes a los 3 microsatélites de la región 4p15.1 utilizando los partidores (primers) D4S2912, D4S230 y D4S3001 con el siguiente protocolo: se realizó la mezcla para PCR en un volumen de 10µl conteniendo buffer de PCR 1x, 0.4µl de dNTP, 1.75mmol de MgCl2 (D4S2912), 1.5mmol de MgCl2 (D4S3001) y 1.25 mmol de MgCl2 (D4S230), 0.4µl de cada primer, 0.25U de Amplitaq Gold (Applied biosystem) y 1µl de ADN genómico. Las condiciones del protocolo de PCR fueron 94ºC por 10min., 94ºC por 30 seg., 55ºC por 30 seg., 72ºC por 30 seg. Por 40 ciclos y 72ºC por 10 min. Los productos de PCR se analizaron en el secuenciador automático ABIPRISM-310 del Laboratorio de Biotecnología y Biomédicina del Instituto Nacional de Salud.
Análisis estadístico:
Las frecuencias alélicas y genotípicas para cada microsatélite fueron estimadas por el programa STATA 8.0 (Statacorp, 2003), evaluándose el equilibrio de Hardy Weinberg (H-W) (16), este análisis permite determinar homogeneidad genética de las poblaciones en estudio y es necesario para continuar los estudios de asociación, los resultados fueron validados con un Chi cuadrado de Pearson y de razón de verosimilitud, respectivamente.
Al no observarse, equilibrio de H-W, fue necesario realizar dos pruebas de asociación; permutación de Montecarlo y regresión logística condicional, para validar los resultados obtenidos en este estudio.
RESULTADOS
La población en estudio estuvo conformada por 99 pacientes DM2 y 120 controles individuos sanos para D4S2912, 129 para D4S230 y 133 para D4S3001.
El primer microsatélite en estudio, D4S2912, presentó 9 alelos en el grupo de diabéticos y 8 alelos en el grupo control de la población peruana (238pb), observándose diferencias estadísticamente significativas en el alelo 230pb(p< 0.05) al comparar casos vs controles (Tabla 2).No se registró equilibrio de H-W en D4S2912 tanto en casos como en controles.
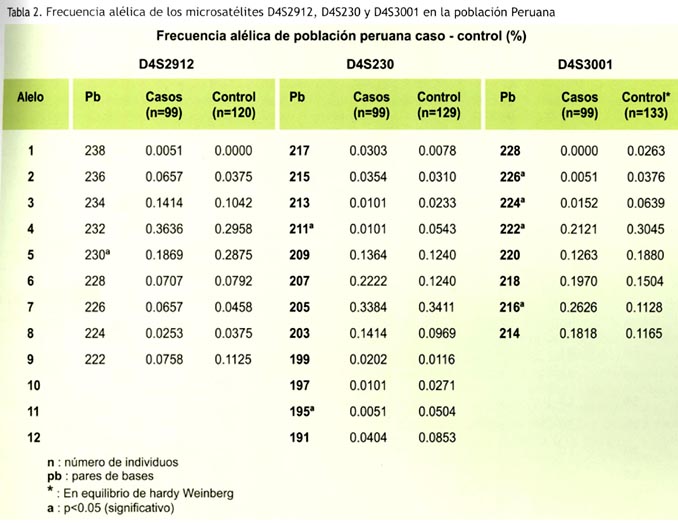
El segundo microsatélite en estudio, D4S230, registró la presencia de 12 alelos para ambos grupos de estudio, observándose diferencias estadísticamente significativas en los alelos 211pb y 195pb al comparar casos versus controles (p< 0.05).
El alelo menos frecuente fue 195pb en diabéticos y 217pb en el grupo control (Tabla 2). No se registró equilibrio de H-W en D4S230 tanto en casos como en controles.
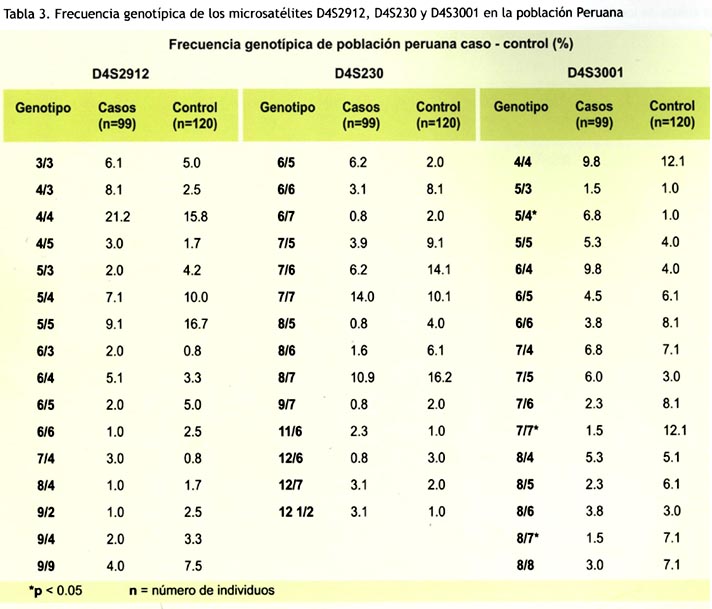
El tercer microsatélite en estudio, D4S3001, presentó 7 alelos en el grupo afectado y 8 alelos en el grupo control (nuevo alelo 238pb). Al comparar ambos grupos de estudio, se registró diferencia estadísticamente significativa encontrando en los alelos de 224pb, 222pb mayor frecuencia en los controles y, el alelo 216pb se observó con mayor frecuencia en la población afectada. El microsatélite D4S3001 se encuentra homogéneamente distribuida al encontrarse en equilibrio de Hardy Weinberg (Pearson x2 = 0.1786) en el grupo control pero no en el grupo afectado. La Tabla 3, presenta el análisis de frecuencias genotípicas de los 3 microsatélites en estudio, observándose asociación estadísticamente significativa en los genotipos 54, 77 y 87 del microsatélite 0453001 a DM2 (p<0.05) en población peruana, estos resultados tienen importancia biológica porque previamente se han observado asociaciones alélicas del marcador a DM2.
Al observar que los microsatélites no están en equilibrio de H-W en la población afectada y que solo D4S3001 está en equilibrio de H-W en la población control, se consideró adecuado realizar el análisis de asociación con dos métodos estadísticos diferentes: el modelo de regresión logística condicional y la prueba de permutación de Montecarlo, que permitieron validar los resultados y confirmar la asociación entre el microsatélite D4S3001 y DM2 en la población peruana (p=0.00001 y p=0.032) (Tabla 4).
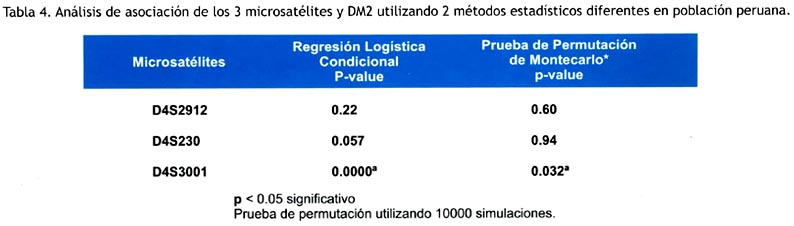
DISCUSIÓN
El equilibrio de H-W es una pieza clave al estudiar asociaciones entre mutaciones genéticas y enfermedades. La prueba de H-W es un paso inicial para determinar si la población es genéticamente homogénea, y esto a su vez, es necesario para continuar con el estudio de asociación entre casos versus controles, validando el estudio de asociación (14,15).
En un estudio realizado en la población de Sami (Escandinavia), se observó que en la región cromosómica 4p15.1, se encuentran los microsatélites D4S230 y D4S2912 que poseen una alta tasa de mutaciones, un gran número de alelos por locus y desequilibrio de ligamiento .: 3, sustentándose que en 4p15.1 existiría una mayor concentración de genes comparado con otras regiones del genoma humano (16). En tanto que, el microsatélite D4S2912 presentaba un desequilibrio de ligamiento de 4.5, valor considerado alto para obesidad en población mexicana (11) y en otro estudio familiar se observó asociación en población inglesa (13), sugiriendo que entre los microsatélites D4S230 y D4S2912 están localizados genes asociados a la etiología de la obesidad y DM2.
A diferencia de los antecedentes expuestos, D4S2912 y D4S230 no se encuentran en equilibrio de H-W tanto en diabéticos como en los controles de la población peruana, esto indicaría que la población en estudio no presenta una distribución genéticamente homogénea para dichos marcadores, esto podría explicarse por la presencia de alelos en común en controles y diabéticos, obteniéndose valores inferiores a lo previsto en el equilibrio de H-W (16). Por ello, Blanco et al. (17) proponen que las asociaciones genotipo-enfermedad con marcadores en desequilibrio de H-W se consideren como provisionales y deberán ser confirmados por poblaciones independientes o estudios basados en familias.
Sin embargo, los resultados obtenidos en la presente investigación nos demuestran la asociación del microsatélite D4S3001 a DM2, utilizando para ello dos pruebas estadísticas diferentes, como son la prueba de permutación de Montecarlo y el análisis mediante el modelo de regresión logística condicional, obteniéndose por ambos métodos la asociación de D4S3001 a DM2 y se sustentan estos resultados con las asociaciones alélicas y genotípicas de este microsatélite a DM2, siendo esta la base biológica que respalda los resultados estadísticos.
Según otras investigaciones (14), una población en desequilibrio de H-W indicaría error de genotipificación (evento poco probable porque se utilizó un método automatizado en el análisis molecular) o un riesgo competitivo de muerte asociado al gen mutante (14). Estos investigadores sugieren que en enfermedades de aparición tardía como por ejemplo la enfermedad de Alzheimer, el genotipo estaría fuertemente asociado a un incremento en la mortalidad, debido a ello, algunos genotipos serían menos frecuentes al morir los portadores, esto ocasionaría que la población esté en desequilibrio de H-W(18). La DM2 al expresarse en poblaciones adultas y ancianas, genéticamente se comportaría de forma similar a la enfermedad de Alzheimer, sugiriendo que el desequilibrio de H-W en los microsatélites D4S2912 y D4S230 en la población peruana se debería a la falta de algunos genotipos por mayor mortalidad de los mismos.
Considerando los antecedentes que relacionan D4S230 y D4S2912 a DM2 y obesidad, la información encontrada en esa región cromosómica con un mayor número de genes que en otras regiones cromosómicas, los resultados obtenidos en la presente investigación de la asociación de 0453001 y DM2, y a la gran distancia observada entre los microsatélites (>1 Mb), es posible sustentar que se encontrarían potenciales genes candidatos involucrados en la etiología de DM2 en este segmento del genoma. Esto se sustenta en antecedentes de asociación entre 4p15.1 y obesidad en población mexicana (11) y entre 4p15.1 y DM2 en otras poblaciones (12,13), considerando para este efecto que las enfermedades metabólicas comparten vías de transducción de señales comunes y por ende genes comunes entre DM2 y obesidad.
Es importante señalar que estos resultados deberían ser considerados como exploratorios debido a que encontramos que dos microsatélites no están en equilibrio de H-W. Por lo anteriormente expuesto, se complementará esta investigación, captando familias nucleares (estudio caso-progenitores) con antecedentes familiares de esta enfermedad.
En conclusión, este estudio muestra al microsatélite D4S3001, localizado en la región cromosómica 4p15.1 como un marcador asociado a DM2 en la población peruana y que se encuentra en una región cromosómica que contendría un gen(es) que jugaría(n) un rol en la etiología de diabetes tipo 2.
REFERENCIAS BIBLIOGRÁFICAS
1. Franceschini N, Almasy L, MacCluer JW, Goring HH, Cole SA, Diego VP, et al. Diabetes-specific genetic effects on obesity traits in American Indian populations: the Strong Heart Family Study. BMC Med Genet. 2008 Oct 14;9:90.
2. Anderson JW, Kendall CW, Jenkins 0J. Importance of weight management in type 2 diabetes: review with meta-analysis of clinical studies. J Am Coll Nutr. 2003 Oct;22(5):331-9.
3. Ghosh S, Watanabe RM, Valle TT, Hauser ER, Magnuson VL, Langefeld C0, et al. The Finland-United States investigation of non-insulin-dependent diabetes mellitus genetics (FUSION) study. I. An autosomal genome scan for genes that predispose to type 2 diabetes. Am J Hum Genet. 2000 Nov; 67(5):1174- 85.
4. Carlson CS, Eberle MA, Kruglyak L, Nickerson DA. Mapping complex disease loci in whole-genome association studies. Nature. 2004 May 27;429(6990):446-52.
5. Wiltshire S, Hattersley AT, Hitman GA, Walker M, Levy JC, Sampson M, et al. A genomewide scan for loci predisposing to type 2 diabetes in a U.K. population (the Diabetes UK Warren 2 Repository): analysis of 573 pedigrees provides independent replication of a susceptibility locus on chromosome 1q. Am J Hum Genet. 2001 5ep;69(3):553-69.
6. Mori Y, Otabe S. 0ina C, Yasuda K, Populaire C, Lecoeur C, et al. Genome-wide search for type 2 diabetes in Japanese affected sib-pairs confirms susceptibility genes on 3q, 15q, and 20q and identifies two new candidate Loci on 7p and 11p. 0iabetes. 2002 Apr;51(4):1247-55.
7. Seclen S, Leey J, Villena A, Herrera B, Menacho J, Carrasco A, et al. Prevalencia de Diabetes Mellitus, hipertensión arterial, hipercolesterolemia, y obesidad como factor de riesgo coronario y cerebrovascular en población adulta de la costa, sierra y selva del Perú. Premio Rousell 1997. Lima; Hoechst Marion Roussel; 1997, 32 p.
8. Ford ES, Giles WH, Dietz WH. Prevalence of the metabolic syndrome among US adults: findings from the third National Health and Nutrition Examination Survey. JAMA. 2002 Jan 16;287(3):356-9.
9. Kirov G, Williams N, Sham P, Craddock N, Owen MJ. Pooled genotyping of microsatellite markers in parent-offspring trios. Genome Res. 2000 Jan;10(1):105-15.
10. Schaid 0J, Jacobsen SJ. Biased tests of association: comparisons of allele frequencies when departing from Hardy-Weinberg proportions. Am J Epidemiol. 1999 Apr 15;149(8):706-11.
11. Arya R, 0uggirala R, Jenkinson CP, Almasy L, Blangero J, O'Connell P, et al. Evidence of a novel quantitative-trait locus for obesity on chromosome 4p in Mexican Americans. Am J Hum Genet. 2004 Feb;74(2):272-82.
12. Blackett PR, Li S, Mulvihill JJ. Ring chromosome 4 in a patient with early onset type 2 diabetes, deafness, and developmental delay. Am J Med Genet A. 2005 Aug 30;137(2):213-6.
13. Stone S, Abkevich V, Hunt SC, Gutin A, Russell DL, Neff C0, et al. A major predisposition locus for severe obesity, at 4p15-p14. Am J Hum Genet. 2002 Jun;70(6):1459-68.
14. Xu J, Turner A, Little J, Bleecker ER, Meyers DA. Positive results in association studies are associated with departure from Hardy-Weinberg equilibrium: hint for genotyping error? Hum Genet. 2002 0ec;111(6):573-4.
15. Ryckman K, Williams SM. Calculation and use of the Hardy- Weinberg model in association studies. Curr Protoc Hum Genet. 2008 Apr;Chapter 1:Unit 1.18.
16. Johansson A, Vavruch-Nilsson V, Edin-Liljegren A, Sjolander P, Gyllensten U. Linkage disequilibrium between microsatellite markers in the Swedish Sami relative to a worldwide selection of populations. Hum Genet. 2005 Jan;116(1-2):105-13.
17. Blanco R, Suazo J, Santos JL, Paredes M, Sung H, Carreño H, et al. Association between 10 microsatellite markers and nonsyndromic cleft lip palate in the Chilean population. Cleft Palate Craniofac J. 2004 Mar;41(2):163-7.
18. Llorca J, Prieto-Salceda D, Combarros O, 0ierssen-Sotos T, Berciano J. Riesgos competitivos de muerte y equilibrio de Hardy-Weinberg en estudios de casos y controles sobre asociación entre genes y enfermedades. Gac Sanit. 2005 Jul- Aug;19(4):321-4.
Fuentes de financiamiento
Las fuentes de financiación no han tenido participación en el diseño del estudio, la colección de datos, el análisis o la interpretación de estos, en la redacción del manuscrito o en la decisión de enviarlo para publicación.
Proyecto Financiado por la Facultad de Medicina Humana, Universidad de San Martín de Porres y el CON5EJO NACIONAL 0E CIENCIA Y TECNOLOGIA (CONCYTEC). PROCYT 155-2006
Conflictos de interés
Los autores expresan que no hay conflicto de interés al redactar el manuscrito.
Correspondencia:
Mónica Paredes Anaya.
0irección: Facultad de Ciencias Biológicas. Universidad Nacional Mayor de 5an Marcos. Lima- Perú. Teléfono: 619 7000
Correo electrónico: monica.paredes.anaya@gmail.com
Recibido: 21 de Octubre de 2014
Aprobado: 06 de Enero de 2015














