Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Horizonte Médico (Lima)
versión impresa ISSN 1727-558X
Horiz. Med. vol.16 no.1 Lima ene./mar. 2016
ARTÍCULO ORIGINAL
Frecuencia del polimorfismo 282 C>T del gen N-Acetiltransferasa (NAT2) en poblaciones peruanas e implicancias en la salud
Prevalence of the N-Acetyltransferase (NAT2) gene polymorphism 282C>T in Peruvian population and health implications
Salazar-Granara Alberto1, Youn-Ho Kim1, Figueroa-Tataje Javier1, Quijano Zapata Fernando1, Ore-Chávez Daniel2, Sandoval- Sandoval José3
1 Universidad de San Martín de Porres. Facultad de Medicina Humana. Centro de Investigación de Medicina Tradicional y Farmacología.
2 Laboratorio de Ecología Molecular. Instituto Antonio Raimondi. Facultad de Ciencias Biológicas. Universidad Nacional Mayor de San Marcos.
3 Universidad de San Martín de Porres. Facultad de Medicina Humana. Centro de Investigación de Genética y Biología Molecular.
RESUMEN
Objetivo: Determinar la frecuencia del polimorfismo C282T del gen NAT2 (N-Acetiltransferasa) en poblaciones peruanas. Trabajo de campo enfocado en explorar un factor de riesgo genético en poblaciones peruanas, el cual presenta influencia en la respuesta a fármacos y en la génesis de neoplasias.
Material y Métodos: Estudio descriptivo transversal. Participaron voluntariamente 116 individuos, procedentes de Lima, Lambayeque, Apurímac, Puno, San Martín, Amazonas y Loreto. Se realizó un muestreo por conveniencia y se empleó la técnica convencional de RFLP-PCR.
Resultados: Las frecuencias alélicas globales de 54 % (n=126) para C282, 46 % (n=106) para T282. Por procedencia destacan las frecuencias del alelo T de 42 % (n=25) en Lima, 47 % (n=16) en Amazonas, 74% (n=28) en San Martín, y 50 % (n=13) en Apurímac (X2, p>0.05). Las frecuencias genotípicas globales fueron 26.7 % (n=31) para C282/C282, 56.0 % (n=65) para C282/T282 y 17.2 % (n=20) para T282/T282 (Prueba de Hardy Weinberg p>0.05). Puno presentó desequilibrio alélico (Prueba de Hardy Weinberg p<0.05), las demás poblaciones se presentaron en equilibrio (Prueba de Hardy Weinberg p>0.05).
Conclusión: Se presentó una frecuencia global de 46 % del alelo NAT2 T282; San Martín tuvo la más alta prevalencia (74%). El alelo T282 presenta asociación a neoplasias y reacciones adversas por fármacos antituberculosos, estos resultados servirán para la aplicación de la farmacogenética en el Perú.
Palabras clave: Gen, NAT2, mutación, farmacogenética, acetilador lento, acetilador rápido, población peruana, Perú.
ABSTRACT
Objective: To determine the frequency of the C282T polymorphism of the NAT2 gene (N acetyltransferase) in Peruvian populations. Field work, focused on exploring genetic risk factor in Peruvian populations, which has influence in the response to drugs and malignancies aetiology.
Material and Methods: Cross-sectional study. 166 voluntaries from Lima, Lambayeque, Apurimac, Puno, San Martin, Amazonas and Loreto were enrolled. The sampling was done by convenience and it was use the RFLP-PCR conventional technique was used.
Results: The allele frequency were 54% (n=126) for C282 and 46% (n=106) for T282. For the T allele, by its orign, stand out those which origins were Lima 42% (n=25), Amazonas 47% (n=16), San Martin 74% (n=28) and Apurimac 50% (n=13) (X2, p>0.05). A global genotype frequency were 26.7% (n=31) for C282/C282, 56.0% (n=65) for C282/T282 and 17.2% (n=20) for T282/T282 (Hardy Weinberg Test p>0.05). By origin, Puno presented allelic imbalance (Hardy Weinberg test p<0.05) and the others populations presented allelic balance (Hardy Weinberg test p>0.05).
Conclusion: The overall frequency of NAT2 allele T282 was 46%; San Martin had the highest prevalence (74%). The T282 allele is linked to neoplastic diseases and adverse reactions to anti-TB drugs, these results will be used for the application of pharmacogenetics in Peru.
Keywords: Gene, NAT2, mutation, pharmacogenetic, slow acetylator, rapid acetylator, peruvian population, Perú.
INTRODUCCIÓN
La Medicina Genómica es la aplicación del conocimiento sobre el genoma humano en la salud humana; el genoma humano contiene el código genético, el cual expresa proteínas que tienen implicancia sobre diversas cascadas fisiológicas y fisiopatológicas en los individuos. El código genético es idéntico al 99,9% en todos los individuos, pero en el restante 0,1 % radica las diferencias en: susceptibilidad a enfermedades, eficacia y respuesta adversa a fármacos, etc., lo cual hace que no haya dos pacientes iguales (1).
Así, el estudio del genoma permitiría predecir y prevenir diversas enfermedades, incluyendo las más comunes como el cáncer, la hipertensión arterial, la diabetes, etc.; pero también es importante su influencia en la respuesta a fármacos ampliamente utilizados como los analgésicos, los antiretrovirales, los antibacterianos, entre otros; esto conlleva a un modelo de medicina personalizada, en la que a partir de las características genéticas de cada paciente, se individualizaría el diagnóstico y el tratamiento (2).
Los términos farmacogenómica y farmacogenética, usualmente se emplean como sinónimos, sin embargo una definición de farmacogenómica radicaría en el estudio de las variantes genéticas de más de un gen y la interacción de estos en la respuesta a un fármaco, en contraste, la farmacogenética, estudia las mutaciones de un gen y su influencia sobre la respuesta a un fármaco (3).
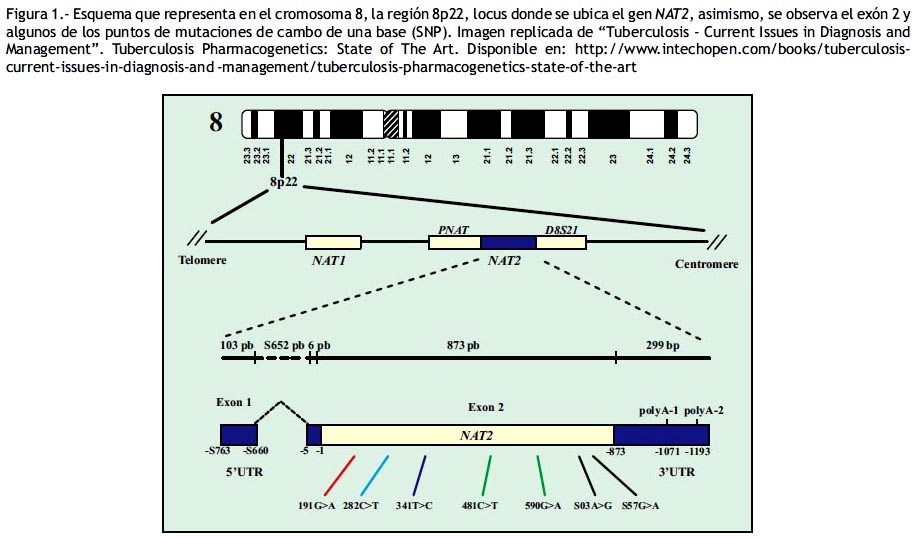
En este marco de estudio, hace más de 40 años que se descubrió el gen NAT2 (N-Acetiltransferasa), y hoy se sabe que presenta polimorfismos o variantes (referido a presencia de mutaciones), este gen está ubicado en la región 8p22 del brazo corto del cromosoma 8, está conformado por el exón 1 no codificante, y el exón 2 codificante (4).
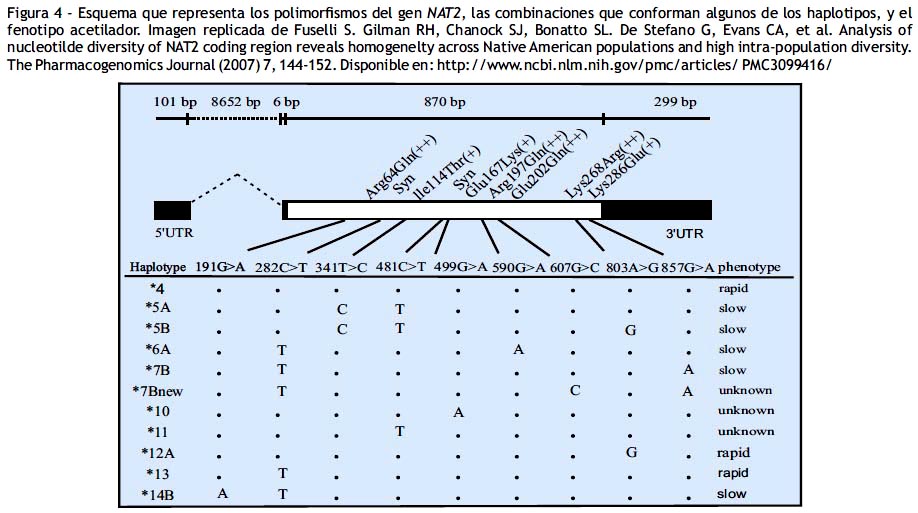
La región exónica 2 está constituida por 870 pares de bases, y en su secuencia ha sido observado 9 sustituciones frecuentes (mutaciones o polimorfismos), siendo estas: 191G>A (Arg64Gln), 341T>C (Ile114Thr), 590G>A (Arg197Gln), 803A>G (Lys268Arg), 857G>A (Lys286Glu), 282C>T y 481C>T. (Figura 1)
Estas mutaciones afectan la expresión de la enzima N–Acetiltransferasa, alterando la velocidad con que acetila (biotransforma o metaboliza) sus diferentes sustratos (xenobióticos). De esta forma se presentan tres fenotipos acetiladores: rápidos, intermedios y lentos, se acepta considerar a los individuos acetiladores rápidos e intermedios, como fenotipo acetilador rápido (5).
En el hombre, la función principal de la enzima N–Acetiltransferasa, radica en la biotransformación de xenobióticos en el hígado (medicamentos, alimentos u otros), transfiriendo desde la Acetil Coenzima A (Acetil-CoA) grupos acetilos al nitrógeno terminal del xenobiótico, generando así nuevos metabolítos, los cuales pueden ser moléculas inactivas (moléculas hidrosolubles), o activas (con efecto biológico) (6).
Esta es una vía principal de biotransformación para muchas arilaminas, hidracinas y drogas, así como de una serie de toxinas y agentes carcinógenos conocidos, que se encuentran presentes en la dieta occidental, en el humo del cigarrillo y en algunos gases presentes en ambientes laborales específicos (6).
Así, cuando un individuo es un acetilador lento y se expone a algún xenobiótico, va acumular metabolítos en sangre, a su vez estos pueden generar efectos adversos, tóxicos, cancerígenos, y hasta letales. Al contrario, si un individuo es un acetilador rápido, transformará el xenobiótico a un metabolito inactivo (hidrosoluble), disminuyendo o anulando su eficacia, pero en ocasiones, se convertirá en un metabolito con acción biológica (7).
La frecuencia de mutaciones o polimorfismos acetiladores del gen NAT2 en las poblaciones humanas presenta gran diversidad (Figura 2), en tal sentido, entre los caucásicos predominan los polimorfismos del gen NAT2 que determinan un fenotipo acetilador lento, estando entre 50 – 63%; sin embargo, en los japoneses y chinos predominan los polimorfismos de acetilador rápido, con una frecuencia de 92% y 80% respectivamente (8).
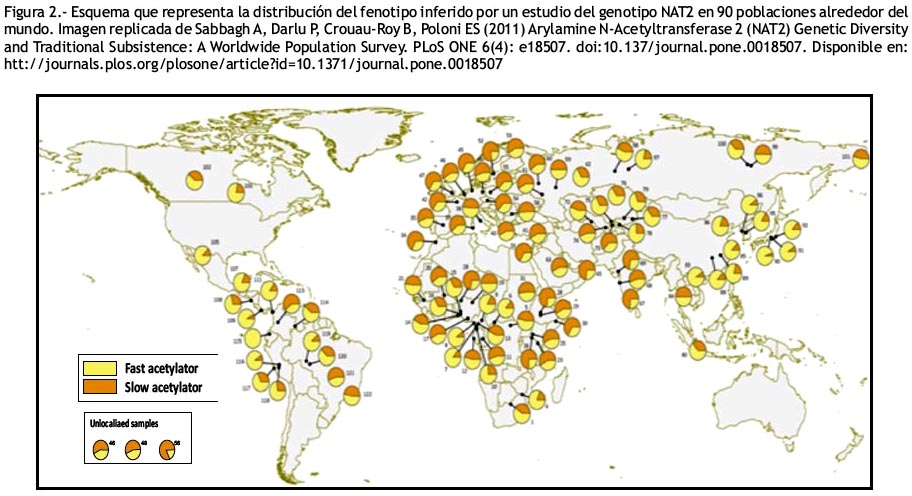
En referencia a los acetiladores lentos y procedencia étnica encontramos frecuencias entre el 5% al 25% en pobladores del este asiático y 11,5% en chinos; 40% a 60% en caucásicos, 74% en pobladores del sur de India, 90% en los árabes, 90% en pobladores del norte de África y 60% en afroamericanos (9).
En Latinoamérica existen pocos estudios, y gran parte de estos provienen de poblaciones brasileñas, argentinas y paraguayas, estos demuestran una distribución de polimorfismos NAT2 de 18%, 56% y 25% para acetilador rápido, intermedio y lento respectivamente, pero también se revelan polimorfismos nuevos (10).
Existen datos recientes de los polimorfismos NAT2 en poblaciones peruanas, estos proceden de estudios desarrollados en Brasil, con muestras poblacionales pequeñas, sin inclusión de la región sur, norte y la amazonia; así, en base a resultados de poblaciones tales como San Martín (n=11), Tayacaja (n=11), Pampas de Lima (n=33), y Lima (n=15), se observó una distribución similar de los polimorfismos NAT2 a las poblaciones sudamericanas antes descritas (11).
La importancia de conocer las frecuencias de estas mutaciones o polimorfismos del gen NAT2, radica en las implicancias que vienen demostrando en los procesos de salud y enfermedad, así, la evidencia científica muestra una relación con la patogénesis de diversos tipos de cáncer y a la variabilidad de respuestas frente a medicamentos comunes.
Estudios han demostrado la asociación del genotipo NAT2 acetilador lento, como un factor de riesgo para el cáncer de vejiga, hígado, colon, estómago, y médula ósea, asimismo, se le asocia a reacciones adversas de medicamentos como la Isoniacida, el Clonazepam, las Sulfas y la Nicotina (12).
Por ejemplo, se ha demostrado en individuos tuberculosos procedentes del Japón, India y China en tratamiento con isoniacida, y con genotipo NAT2 de acetilador lento, la asociación a reacciones adversas hepáticas y neurológicas, pero cuando el individuo es un acetilador rápido, afecta la actividad bactericida temprana de esta droga, debido a que no se alcanza los niveles séricos adecuados para una concentración inhibitoria mínima (13).
Basado en la evidencia científica, instituciones americanas como la Food and Drug Administration, y europeas como la European Medicines Agency, recomiendan realizar diagnóstico del genotipo de los individuos, para genes relevantes como el NAT2, CYP2D6, CYP2C9, CYP2C19, CYP3A4, ABCB1, entre otros, cuya utilidad radicaría en disminuir los riesgos e incrementar la eficacia terapéutica (14).
Por ello, en la actualidad, existe en el mercado internacional la oferta de análisis genético de enzimas del metabolismo de los fármacos, los cuales son de alto costo, y consideran variantes genéticas (mutaciones) de etnias europeas, africanas, asiáticas y norteamericanas, denotando un importante sesgo al no considerar variantes de poblaciones de Latinoamérica.
No contar con centros diagnósticos para estos genes relevantes, como no conocer nuestra realidad acerca de sus variantes (mutaciones), nos pone en desventaja, perdiendo la posibilidad de contar con más alternativas para el uso de medicamentos como la isoniacida frente a la tuberculosis en Perú, o advertir a pacientes con predisposición a cáncer de cólon o vejiga a que no se expongan a xenobióticos como el tabaco y alimentos ricos en compuestos aromáticos.
El objetivo principal de la presente investigación se centró en la exploración de la frecuencia del polimorfismo de cambio de una base del gen NAT2, C282T (SNP or Single Nucleotide Polimorphism), en una muestra de pobladores peruanos procedentes de Lima (Lima), Lambayeque (San José), Apurímac (Andahuaylas), Puno (Lago Titicaca), San Martín (Lamas), Amazonas (Chachapoyas) y Loreto (Andoas).
Con esta investigación se motiva a los estudios de farmacogenética en el Perú, y se pone a disposición una base de datos para un futuro servicio de diagnóstico molecular aplicado a la salud.
MATERIAL Y MÉTODOS
Tipo de diseño, población y muestra
Estudio observacional, comparativo, descriptivo y transversal. Las muestras poblacionales correspondieron a individuos peruanos de las regiones de Lima (Lima), Lambayeque (San José), Apurímac (Andahuaylas), Puno (Lago Titicaca), San Martín (Lamas), Amazonas (Chachapoyas) y Loreto (Andoas), todos con acreditación voluntaria de su participación por medio de un consentimiento informado, y con por lo menos tres generaciones en el lugar de origen.
La estrategia de selección de la muestra se realizó por muestreo de tipo no probabilístico y por conveniencia, de tal forma que el tamaño muestral fue de ciento dieciséis (n=116).
La distribución por procedencia fue la siguiente: 30 individuos de Lima, 10 individuos de Lambayeque, 13 individuos de Apurímac, 11 individuos de Puno, 19 individuos de San Martín, 17 individuos de Amazonas y 16 individuos de Loreto.
Muestra biológica y extracción del Ácido Desoxirribonucleico (ADN)
Se colectó 5 ml de sangre por punción venosa, esta se almacenó en tubos con anticoagulante Ácido etilendiaminotetraacético (EDTA). Asimismo, se obtuvo muestras de células bucales, esto consistió en el frotamiento de la cavidad bucal con hisopo por un minuto, luego se almacenó en un recipiente con amortiguador de lisis.
Ambos tipos de muestras fueron conservados en refrigeración hasta su traslado a la Facultad de Medicina Humana de la Universidad de San Martín de Porres, donde se realizó la extracción del ADN y tipificación molecular.
La extracción de ADN de linfocitos y/o células bucales se efectuó por una técnica simplificada descrita por Salazar-Granara Alberto et al. (15), que consistió en lo siguiente: a) Lavado de células con Tris-EDTA (TE). b) Lisado con N-Laurylsarcosina. c) Digestión proteica con Proteinasa K. d) Precipitación, y extracción con alcohol, y sales, para la obtención del ADN. e) Seguidamente, el ADN se colocó en amortiguador TE 10X, y almacenó en refrigeradora a -4°C. f) Para el control de calidad del DNA extraído, se vertió 10ul del ADN por cada individuo en geles de agarosa al 1%, y se llevó a cabo una electroforesis en amortiguador Tris- Borato-EDTA (TBE) 1X a 100V por 1 hora. g) Luego, el gel de agarosa es suspendido en bromuro de etídio por periodo de 40 minutos, con fines de tinción del ADN. h) Finalmente, se sometió el gel de agarosa a radiación ultravioleta, para observar el ADN y comprobar que este se encontrará en buen estado o degradado, las muestras de ADN degradadas fueron reemplazadas por muestras de ADN en estado óptimo, siguiendo el mismo procedimiento de validación.
Reacción en cadena de la polimerasa (PCR)
La amplificación del exón 2 del gen NAT2, se hizo por reacción en cadena de la polimerasa, y se utilizaron los cebadores reportados por Patín et al. 2006 (16).
Se preparó, una mezcla con un volumen de reacción final de 12ul, con 1X de amortiguador PCR (Fermentas), 2mM de MgCl2 (Fermentas), 1,5U de Taq Polimerasa (Fermentas), 200uM de cada dNTPs (dinucleotidos trifosfato) y 4 pmol de cada primer.
Esta mezcla fue llevada a un termociclador, con un programa de amplificación que consistió de un paso inicial de denaturación a 95 ºC por 5 minutos, seguido de 35 ciclos, que comprendió una denaturación a 95 ºC por 30 segundos, un paso de hibridación a 55 ºC por 45 segundos, y un paso de extensión a 72 ºC por 2 minutos, finalmente una extensión de 72 ºC por 10 minutos.
Los productos de PCR, fueron sometidos a electroforesis en gel de agarosa al 1%, a 100V en amortiguador TBE 1X; posteriormente fue teñido en bromuro de etídio y visualiza dos en un transiluminador ultravioleta, registrándose fotográficamente la banda correspondiente a un producto de PCR de 1211 pb (pares de bases) fue el indicador de un amplificado óptimo.
Reacción de restricción de polimorfismo de longitud de fragmentos (RFLP)
Los productos de PCR del exón 2 del gen NAT2, fueron sometidos a la reacción RFLP, esto consistió en la aplicación de una enzima de restricción o de reconocimiento de una base específica, está según sea el caso determinó la presencia o ausencia del polimorfismo, según Patín et al. 2006 (16).
Se utilizó la enzima FokI (Fermentas) para la restricción del alelo C282T; esto consistió en la preparación de una mezcla con 10 ul del producto de PCR, 1 ul de amortiguador Tango (Fermentas), 0.3 ul de enzima FokI (Fermentas) y 3.7 ul de agua, luego se incubó por 16 horas a 55°C.
El producto de la digestión, se sometió a electroforesis en gel de agarosa al 2.5 %, con voltaje de 100 m ven amortiguador TBE por aproximadamente 1 hora; posteriormente fue teñido en bromuro de etídio y visualizados en un transiluminador ultravioleta, registrándose fotográficamente las bandas correspondientes a lo siguiente:
-
Bandas con longitud de 429, 337, 288, 122 y 35 pares de bases, correspondió a dos copias del alelo salvaje C282, y al genotipo homocigoto salvaje C282/C282.
-
Bandas con longitud de 766, 429, 337, 288, 122 y 35 pares de bases, correspondió a una copia del alelo salvaje C282 y a una copia del alelo polimórfico o mutante T282, y al genotipo heterocigoto C282/T282.
-
Bandas con longitud de 766, 288, 122 y 35 pares de bases, correspondió a dos copias del alelo polimórfico o mutante T282, y al genotipo homocigoto mutante T282/T282.
Análisis de los datos
Los datos se organizaron de forma tabular y se muestran en tablas de frecuencias simples y cruzadas. Para el análisis de significancia estadística, se realizó la prueba del Chi cuadrado de Pearson, que considero un valor p<0.05 como significativo. Asimismo, se aplicó la prueba de equilibrio alélico de Hardy-Weinberg. Se empleó el Software estadístico SPSS versión 16.0 y Arlequín 5.0.
RESULTADOS
Frecuencia de la mutación C282T del gen NAT2 en pobladores peruanos
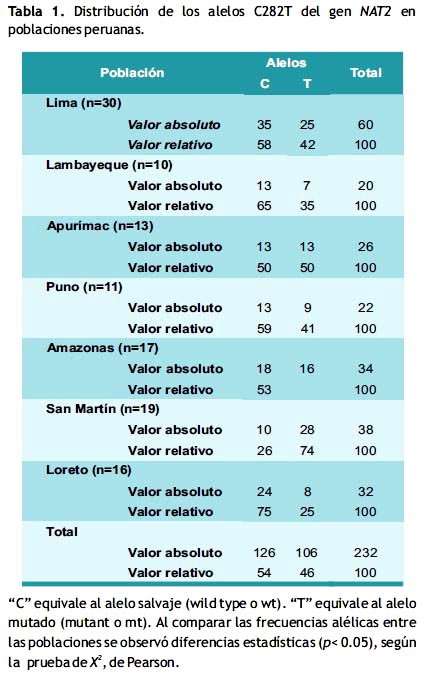
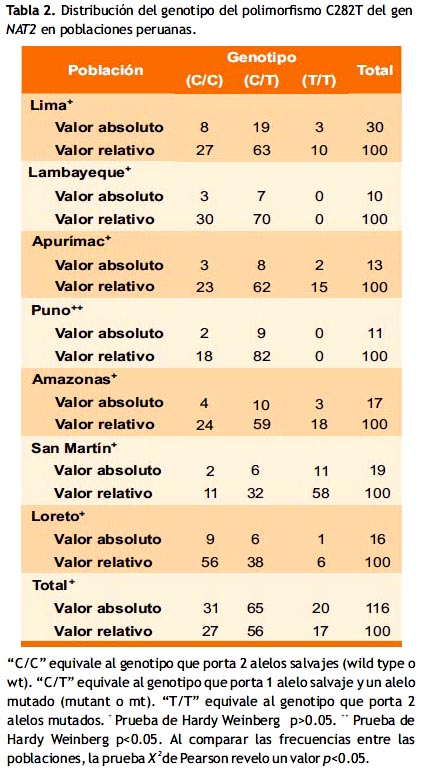
A partir de una muestra de 116 pobladores peruanos, en la tabla 1 se observa la presencia del alelo salvaje C282 en un 54% (n=126), frente a 46% (n=106) del alelo mutante T282. (Figura 3)
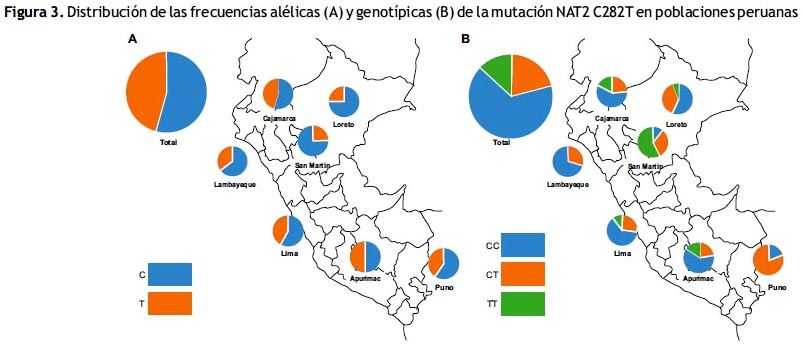
Según lugar de procedencia las frecuencias fueron: Lima 58% (n=35) versus 42% (n=25), Lambayeque 65% (n=13) versus 35% (n=7), Amazonas 53% (n=18) frente a 47% (n=16), San Martin 26% (n=10) frente a 74% (n=28), Loreto 75% (n=24) versus 23% (n=8), Apurímac 50% (n=13) versus 50% (n=13), y Puno 59% (n=13) frente a 41% (n=9), del alelo salvaje versus el mutante respectivamente. (Tabla 1)
Frecuencia del genotipo C282T del gen NAT2 en pobladores peruanos
La frecuencia global fue (tabla 2): 26.7% (n=31) con genotipo C282/C282 (homocigoto salvaje), 56.0% (n=65) con genotipo C282/T282 (heterocigoto) y 17.2% (n=20) T282/T282 (homocigoto mutante). (Figura 3)
Según lugar de procedencia, respectivamente se presentaron las siguientes frecuencias: Lima 27.0% (n=8), 63.0% (n=19), y 10.0% (n=3). Lambayeque 30% (n=3), y 70.0% (n=7). Apurímac 23.0% (n=3), 62.0% (n=8), y 15.0% (n=2). Puno 18% (n=2), y 82.0% (n=9). San Martín 11.0% (n=2), 32.0% (n=6), y 58.0% (n=11). Amazonas 24.0% (n=4), 59.0% (n=10), y 18.0% (n=3). Loreto 56% (n=9), 38.0% (n=6), y 6.0 (n=1). (Tabla 2)
DISCUSIÓN
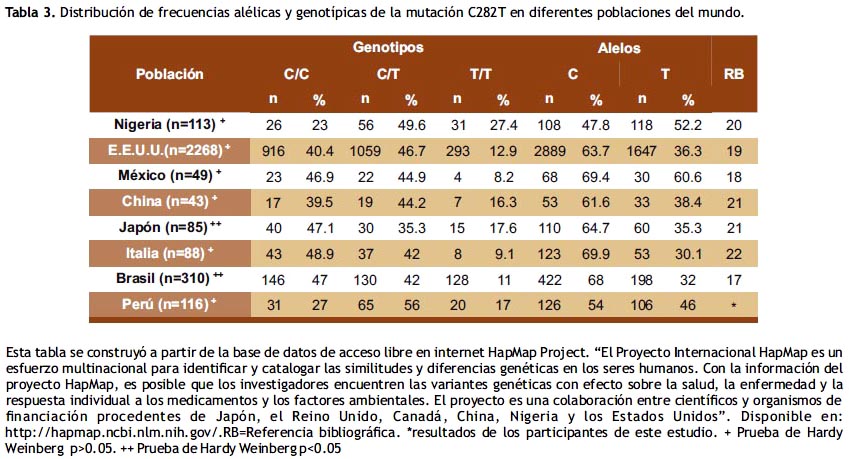
La frecuencia del alelo T del polimorfismo C282T del gen NAT2 en la población peruana se presentó en el 46% de la muestra estudiada, en comparación con otras poblaciones como la brasileña con 32% (17), mexicana con 61% (18), estadounidense con 36.3% (19), Nigeriana con 52.2% (20), japonesa con 35.3% (21), e Italiana con 30.1% (22). (Tabla 3)
Estas comparaciones denota la variabilidad genética entre las poblaciones en el mundo, factor a considerar para los análisis diagnósticos de los polimorfismos del gen NAT2 que actualmente se ofertan en el mercado internacional (23).
Por otra parte, los hallazgos de este estudio podrían ser considerados para el desarrollo de análisis diagnósticos en la población peruana, así, posiblemente se evitaría análisis con resultados de falsos positivos.
Lo antes dicho, por ejemplo debería ser enfatizado en la población peruana de San Martín, donde la frecuencia del aleo T se presentó en un 74%, por tanto, en un posible análisis diagnóstico que se estructure para esta población, tendría que considerar el alelo T como un marcador importante. Por otra parte, se abre un camino de investigación promisorio acerca de la asociación de este alelo y su acción en la génesis de ciertas patologías o de la respuesta a fármacos, lo cual podría extenderse a la población de Apurímac, donde se presentó una frecuencia del alelo T del 50% y a la de Amazonas que presentó una frecuencia de 47%.
En contraste, acorde a la ecuación de Hardy Weinberg, la presencia del genotipo T/T (homocigoto mutante) y C/T (heterocigoto portador), se presenta en equilibrio, esto significa que lo que observamos se está presentando de generación en generación como una estructura poblacional estable (24).
Además, en la tabla 3 se observa que la población de Japón y Brasil, según la ecuación de Hardy Weinberg, no se encuentran en equilibrio, similarmente se observó en la población peruana de Puno.
Lo antes descrito podría deberse a factores externos que influyen en el genoma, por ejemplo, en poblaciones de familias endogámicas, por inter-ocurrencia de exposición a xenobióticos, migraciones, condiciones geológicas, entre otros (25), es posible que el desequilibrio que se observa en Puno, recaiga en características como condiciones climáticas con frio extremo y de elevada altitud, aspectos que podrían ser relevantes para los fines de aplicación en salud pública.
Por tanto, en base a este marcador genético, la población puneña tendría una estructura genotípica inestable, lo cual podría influir en el perfil del fenotipo acetilador de su población, aspecto a considerar para futuras investigaciones clínicas, en especial los ensayos clínicos.
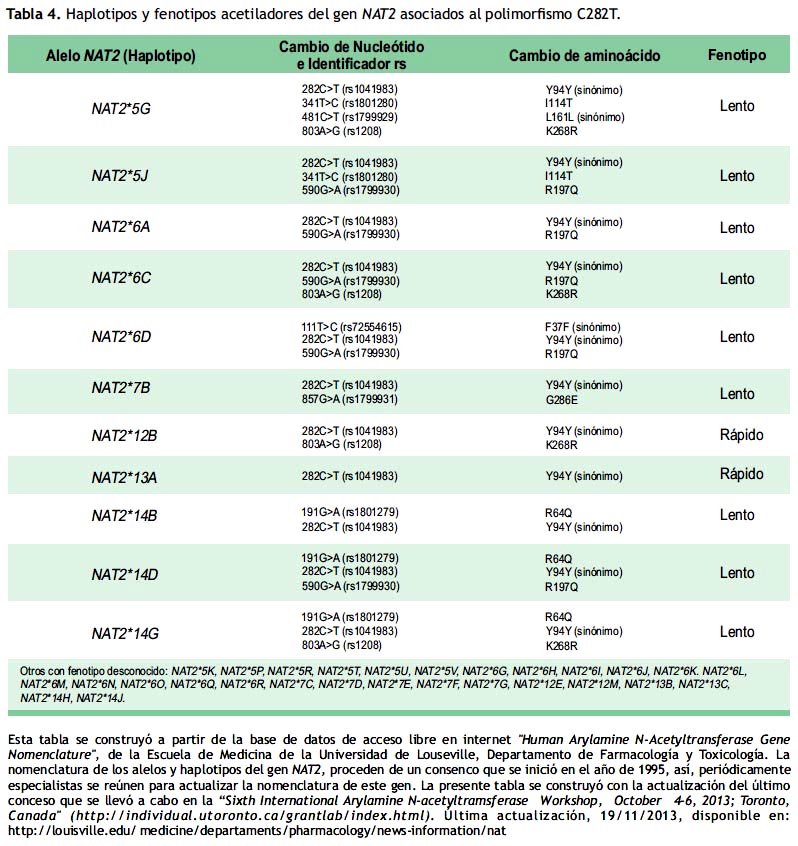
El genotipo acetilador del gen NAT2, es el conjunto de las combinaciones de los polimorfismos presentes en el exón 2, lo cual se denomina haplotipos (figura 3), en tal sentido el alelo T del polimorfismo C282T del gen NAT2, se presentará en combinación con otros polimorfismos (4,5), en la tabla 4 se muestra los haplotipos y fenotipos del gen NAT2 asociados al alelo T del polimorfismo C282T.
Es necesario considerar que la principal limitación de este estudio radicaría en la reducida muestra poblacional, y en que solo se ha explorado un polimorfismo de este gen, lo cual imposibilita determinar con certeza el tipo de acetilador para las poblaciones estudiadas, esto deberá lograrse con futuras investigaciones.
Sin embargo, exponemos poblaciones peruanas que hasta la actualidad no fueron estudiadas mediante este marcador biológico, tales como Amazonas, Apurímac, Loreto, Puno y Lambayeque; asimismo, ampliamos resultados para las poblaciones que cuentan con estudios previos como Lima y San Martín. (Tabla 2)
En tal sentido, considerando los fenotipos a los que se liga el polimorfismo C282T del gen NAT2 (Tabla 4), se observa que principalmente se asocia al fenotipo acetilador lento.
La relevancia clínica es que el acetilador lento está asociado con la génesis de diversos tipos de neoplasias de vías gastrointestinales, respiratorias y genitourinarias, esto en concomitancia con la ingesta de dieta rica en compuestos aromáticos, nitrogenados, y/o exposición al humo del cigarro.
Por ejemplo, en individuos alemanes con tabaquismo y cáncer de pulmón, se observó una frecuencia de 56.1% del acetilador lento, por otra parte, en individuos norteamericanos con tabaquismo y cáncer de vejiga, la frecuencia del acetilador lento fue de 68.3% (8).
Por otra parte, al acetilador lento clínicamente se le ha asociado con reacciones adversas y/o tóxicas a drogas, especialmente a medicamentos utilizados en trastornos bipolares, infecciones de tracto urinario y tuberculosis (4,7).
Así, en pacientes coreanos con infección por Mycobacterium tuberculosis en tratamiento con isoniacida, se ha demostrado la relación de hepatotoxicidad y el genotipo acetilador lento (OR, 5.41; IC95%, 1.76–16.59, p<0.005), similarmente, se observó, en pacientes tuberculosos de Taiwán, Japón, e India (13).
Por otra parte, aunque hasta la actualidad no es lo más frecuente, el polimorfismo C282T, también se asocia al fenotipo acetilador rápido (tabla 4), en tal sentido, aunque los estudios aun no son concluyentes, se observa que los individuos con genotipo acetilador rápido, presentan una reducción en la actividad bactericida temprana (EBA) de la Isoniacida (26,28).
Por ejemplo, en pacientes europeos acetiladores rápidos y con tuberculosis, recibieron dosis escalonadas de isoniacida desde 0.2 a 12mg/kg (5mg/kg es la dosis estándar mundial); con un corte de AUC0-∞ ≥ 10.52 ug/ml (área bajo la curva) para una EBA90 efectiva; se demostró que la dosis que alcanzó el punto de corte del AUC fue de 10mg/kg, resultados semejantes se observaron en pacientes tuberculosos procedentes de Norteamérica, África, y Asia (26,28).
Por lo tanto, es de mucho interés considerar la potencial aplicación del conocimiento de la genómica y en especial del genotipo acetilador en la población peruana, donde la tuberculosis presenta un panorama de alta morbimortalidad, incremento de la resistencia bacteriana, multi-drogo-resistencia y de resistencia extensiva a medicamentos antituberculosos (29,30).
Asimismo, sería útil para advertir y educar a los individuos acetiladores lentos, para que eviten la exposición a tabaco, a dietas occidentales, o a ciertos ambientes laborales, ligados a neoplasias como cáncer de estómago y colon, que se presentan en alta prevalencia en el Perú.
Para ambos casos, sea en tuberculosis o en neoplasias, se deberá en el futuro volcar mayores esfuerzos para emprender investigaciones acerca de la medicina genómica y la implicancia del genotipo acetilador en estas enfermedades y en pacientes peruanos.
En conclusión, en la población peruana estudiada, se presentó una frecuencia global del 46% del alelo T del polimorfismo C282T del gen NAT2, es de resaltar que la población que presento la más alta prevalencia fue San Martín con 74%.
Asimismo, el genotipo más prevalente en la población peruana estudiada fue el heterocigoto (C282/T282) con 56 %, asimismo, Puno presentó la más alta frecuencia con 82%.
Agradecimientos
Al Dr. Frank Lizaraso Caparó, Decano de la Facultad de Medicina Humana de la Universidad de San Martín de Porres, por su invaluable ayuda y colaboración.
REFERENCIAS BIBLIOGRÁFICAS
1. Geoffrey S. Ginsburg, Huntington F. Willard. Genomic and Personalized Medicine Second edition. United Kingdom, 2013 Elsevier Inc.
2. W. Gregory Feero, Alan E. Guttmacher, Francis S. Collins. Genomic Medicine — An Updated Primer. N Engl J Med 2010;362:2001-11. [ Links ]
3. Lee J.W., Aminkeng F., Bhavsar A.P., Shaw K., Carleton B.C., Hayden M.R., Ross C.J.D. The emerging era of pharmacogenomics: current successes, future potential, and challenges. Clin Genet 2014: 86: 21–28. [ Links ]
4. Online Mendelian Inheritance in Man [database on the Internet]. N-ACETYLTRANSFERASE 2; NAT2. National Library of Medicine (US); Copyright © 1966-2014- [cited 2014 Jun 10]. Available from: http://www.omim.org/entry/612182. [ Links ]
5. Sabbagh A, Darlu P, Crouau-Roy B, Poloni ES (2011) Arylamine N-Acetyltransferase 2 (NAT2) Genetic Diversity and Traditional Subsistence: A Worldwide Population Survey. PLoS One. 2011 Apr6; 6(4 ): e18507. doi: 10.1371/journal.pone.0018507.
6. Jason M. Walraven, Yu Zang, John O. Trent, David W. Hein. Structure/Function Evaluations of Single Nucleotide Polymorphisms in Human N-Acetyltransferase 2. Curr Drug Metab. 2008 Jul; 9(6): 471–486. [ Links ]
7. Olivera, M.; Martinez, C.; Gervasini, G.; Carrillo, J. A.; Carrillo, J. A.; Ramos, S.; et al. Effect of Common NAT2 Variant Alleles in the Acetylation of the Major Clonazepam Metabolite, 7-minoclonazepam. Drug Metabolism Letters, Volume 1, Number 1, January 2007, pp. 3-5(3)
8. Hannu Norppa. Cytogenetic biomarkers and genetic polymorphisms. Toxicology Letters 2004; 149: 309–334. [ Links ]
9. Samar I. Hamdy, Masahiro Hiratsuka, Kaori Narahara, Naomi Endo, Mervat El-Enany, Nadia Moursi, Mohammed S-E. Ahmed, Michinao Mizugaki. Genotype and allele frequencies of TPMT, NAT2, GST, SULT1A1 and MDR-1 In the Egyptian population. Br J Clin Pharmacol 2003; 55: 560–569. [ Links ]
10. G. Bailliet, M.R. Santos, E.L. Alfaro, J.E. Dipierri, D.A. Demarchi, F.R. Carnese, N.O. Bianchi. Allele and genotype frequencies of metabolic genes in Native Americans from Argentina and Paraguay. Mutation Research 2007; 627: 171–177. [ Links ]
11. S Fuselli, R H Gilman, S J Chanock, S L Bonatto, G De Stefano, C A Evans, et al. Analysis of nucleotide diversity of NAT2 coding region reveals homogeneity across Native American populations and high intra-population diversity. The Pharmaco genomics Journal (2007)7, 144–152. doi:10.1038/sj.tpj.6500407. [ Links ]
12. Eckart Schnakenberg, Karl-Rainer Fabig, Martin Stanulla, Nils Strobl, Michael Lustig, Nathalie Fabig, Werner Schloot. A cross-sectional study of self-reported chemical-related sensitivity is associated with gene variants of drug- metabolizing enzymes. Environmental Health 2007; 6:6. [ Links ]
13. Norihide Higuchi, Naoko Tahara, Katsunori Yanagihara, Kiyoyasu Fukushima, Naofumi Suyama, Yuichi Inoue, et al. NAT2*6A, a haplotype of the N-acetyltransferase 2 gene, is an important biomarker for risk of anti-tuberculosis drug-induced hepatotoxicity in Japanese patients with tuberculosis. World J Gastroenterol 2007; 13(45): 6003-6008. [ Links ]
14. U.S. Food and Drug Administration. Drugs. Table of Pharmacogenomic Biomarkers in Drug Labeling. Page Last Updated: 05/20/2015. Disponible en: http://www.fda.gov/drugs/scienceresearch/researchareas/pharmacogenetics/ucm083378.htm. [ Links ]
15. Salazar-Granara Alberto, Sandoval-Sandoval José, Mendizábal-Arbocco Rafael, Kikushima-Tukiuda Francisco, Madsen Hans O., Garred Peter, et al. Variantes del gen Mannose binding lectin (mbl) en pobladores amazónicos de Andoas-Loreto y su posible implicancia en la salud. Revista Horizonte Médico 2006; 6: 1. [ Links ]
16. Etienne Patin, Luis B. Barreiro, Pardis C. Sabeti, Frederic Austerlitz, Francesca Luca, Antti Sajantila, et al. Deciphering the Ancient and Complex Evolutionary History of Human Arylamine N-Acetyltransferase Genes. Am. J. Hum. Genet. 2006; 78: 423–436. [ Links ]
17. Database of Single Nucleotide Polymorphisms (dbSNP). Bethesda (MD): National Center for Biotechnology Information, National Library of Medicine. (dbSNP Build ID: rs1041983). Disponible en: http://www.ncbi.nlm.nih.gov/projects/SNP/snp_viewTable.cgi?pop=6488. [ Links ]
18. Database of Single Nucleotide Polymorphisms (dbSNP). Bethesda (MD): National Center for Biotechnology Information, National Library of Medicine. (dbSNP Build ID: rs1041983). Disponible en: http://www.ncbi.nlm.nih.gov/SNP/snp_viewTable.cgi?pop=12161. [ Links ]
19. Database of Single Nucleotide Polymorphisms (dbSNP). Bethesda (MD): National Center for Biotechnology Information, National Library of Medicine. (dbSNP Build ID: rs1041983). Disponible en: http://www.ncbi.nlm.nih.gov/SNP/snp_ss.cgi?ss=ss342253601. [ Links ]
20. Database of Single Nucleotide Polymorphisms (dbSNP). Bethesda (MD): National Center for Biotechnology Information, National Library of Medicine. (dbSNP Build ID: rs1041983). Disponible en: http://www.ncbi.nlm.nih.gov/SNP/snp_viewTable.cgi?pop=1412. [ Links ]
21. Database of Single Nucleotide Polymorphisms (dbSNP). Bethesda (MD): National Center for Biotechnology Information, National Library of Medicine. (dbSNP Build ID: rs1041983). Disponible en: http://www.ncbi.nlm.nih.gov/SNP/snp_viewTable.cgi?pop=1410. [ Links ]
22. Database of Single Nucleotide Polymorphisms (dbSNP). Bethesda (MD): National Center for Biotechnology Information, National Library of Medicine. (dbSNP Build ID: rs1041983). Disponible en: http://www.ncbi.nlm.nih.gov/SNP/snp_viewTable.cgi?pop=12163. [ Links ]
23. S B Haga,N M A LaPointe. The potential impact of pharmacogenetic testing on medication adherence. The Pharmacogenomics Journal (2013) 13, 481–483.
24. Wang J, Shete S (2011) Testing Hardy-Weinberg Proportions in a Frequency-Matched Case-Control Genetic Association Study. PLoS ONE 6 (11): e27642. doi: 10.1371/journal.pone.0027642.
25. Chen AQ, Zang J, Feng YX, Wang KJ. Influential factors of Hardy-Weinberg equilibrium on the study of association between gene polymorphism and disease. Zhonghua liuxingbingxue zazhi. 2009 Nov;30(11):1203-6. [ Links ]
26. Tawanda Gumbo, Arnold Louie, Weiguo Liu, David Brown, Paul G. Ambrose, Sujata M. Bhavnani, George L. Drusano. Isoniazid Bactericidal Activity and Resistance Emergence: Integrating Pharmacodynamics and Pharmacogenomics To Predict Efficacy in Different Ethnic Populations. ANTIMICROBIAL AGENTS AND CHEMOTHERAPY 2007; 51:7. [ Links ]
27. Ryuji Kubota, Masako Ohno, Tomoko Hasunuma, Hajime Iijima, Junichi Azuma. Dose-escalation study of isoniazid in healthy volunteers with the rapid acetylator genotype of arylamine N-acetyltransferase 2. Eur J Clin Pharmacol 2007;63:927–933. [ Links ]
28. R. Donald, P. Parkin, I. Seifart, S. Schaaf, D. van Helden, J. Werely, A. Sirgel, A. Venter, S. Maritz. The influence of dose and N-acetyltransferase-2 (NAT2) genotype and phenotype on the pharmacokinetics and pharmacodynamics of isoniazid. Eur J Clin Pharmacol 2007; 63:633–639. [ Links ]
29. Sala Situacional de Tuberculosis. ESN PCT/DGSP/MINSA/PERU, Febrero 2010. Disponible en: http://www.tuberculosis-peru.org/Docs/SalaSituTB2010.pdf. [ Links ]
30. Informe Operacional De Estadísticas De Tuberculosis En El Perú; Periodo 2001,2002.2003 Y 2004. Elaborado Por El Programa Nacional Para El Control De La Tuberculosis Del Ministerio De Salud Del Perú. Disponible en: http://www.minsa.gob.pe/portal/Especiales/PareTB/Archivos/TBCMINSA%202001-2003.pdf. [ Links ]
Fuentes de Financiamiento
Este artículo ha sido financiado por los autores.
Conflicto de interés
Los autores declaran no tener ningún conflicto de interés.
Correspondencia:
Alberto Salazar Granara
Dirección: Av. El Corregidor N° 1531, Urbanización Los Sirius, Las Viñas, La Molina, Lima 12, Perú.
Teléfono: 3652300 anexo 151.
Correo electrónico: alberto.salazar@gmail.com, asalazarg@usmp.pe
Recibido: 29 de enero de 2016
Aprobado: 16 de febrero de 2016