Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Horizonte Médico (Lima)
versión impresa ISSN 1727-558X
Horiz. Med. vol.17 no.2 Lima abr./jun. 2017
http://dx.doi.org/https://doi.org/10.24265/horizmed.2017.v17n2.04
ARTÍCULO ORIGINAL
Distribución del polimorfismo Q192R del gen de la paraoxonasa 1 en una población del distrito de Junín (Región Junín, Perú)
Distribution of the Q192R polymorphism of the paraoxonase 1 gene in a population of the district of Junín (Junín region, Peru)
Elizabeth Carranza Alva1,a, Carmen Peña Suasnábar2,a, Alejandro Florentini Carranza3,a
1. Instituto Nacional de Biología Andina, Facultad de Medicina.
2. Facultad de Farmacia y Bioquímica.
3. Facultad de Ciencias Biológicas.
a. Universidad Nacional Mayor de San Marcos.
RESUMEN
Objetivo: La paraoxonasa 1 (PON1), una esterasa asociada a las lipoproteínas de alta densidad (HDL), presenta diversos polimorfismos: el polimorfismo PON1 Q192R (región codificante) es el responsable de las variaciones de la actividad paraoxonasa de la enzima. El alelo PON1 192R es considerado un factor de riesgo para desarrollar trastornos cardiometabólicos en algunas poblaciones. El objetivo del presente estudio es determinar la distribución de los polimorfismos PON1 Q192R, y su asociación con el perfil lipidíco y APO A1 en una población andina.
Materiales y métodos: Estudio descriptivo, transversal, en el que participaron 79 personas adultas (26 hombres y 53 mujeres), clínicamente sanas, residentes del distrito de Junín a 4105 msnm. Se empleó la técnica PCR/RFLP para el análisis del sitio polimórfico Q192R del gen PON1, y se determinaron los valores séricos del perfil lipídico y APO A1, y las actividades paraoxonasa y arilesterasa de la enzima.
Resultados: La distribución de los genotipos para PON1192 fue: QQ 13,9%, QR 45,6% y RR 40,5%, y el alelo más frecuente fue 192R 63%. Las actividades paraoxonasa basal y estimulada fueron diferentes según sus genotipos, mientras que la actividad esterasa de la enzima no estuvo influenciada por el polimorfismo. Por otra parte, no se encontró asociación entre el polimorfismo PON1 Q192R y el perfil lipídico y APO A1.
Conclusiones: La alta prevalencia del alelo PON1 192R en la población estudiada probablemente indique un papel importante en el desarrollo de enfermedades cardiometabólica.
Palabras clave: Paraoxonasa; polimorfismo; factores de riesgo cardiovascular; altitud.
ABSTRACT
Objective: Paraoxonase 1 (PON1), an esterase associated with high-density lipoproteins (HDL), has several polymorphisms: the PON1 Q192R polymorphism (coding region) is responsible for variations in the paraoxonase activity of the enzyme. The PON1 192R allele is considered a risk factor for developing cardiometabolic disorders in some populations. The aim of the present study is to determine the distribution of the PON1 Q192R polymorphism and its association with the lipid profile and APO A1 in an Andean population.
Materials and methods: A descriptive, cross-sectional study involving 79 healthy adults (26 males and 53 females), residents of the district of Junín at 4105 m.a.s.l. The PCR/RFLP technique was used for the analysis of the PON1 Q192R polymorphism. The serum values of the lipid profile and APO A1, and the paraoxonase and arylesterase activities of the enzyme were determined.
Results: The distribution of the genotypes for PON1192 was QQ 13.9%, QR 45.6% and RR 40.5%, and the most frequent allele was 192R 63%. The baseline and stimulated paraoxonase activities were different based on their genotypes, while the esterase activity of the enzyme was not influenced by the polymorphism. On the other hand, no association was found between the PON1 Q192R polymorphism and the lipid profile and APO A1.
Conclusions: The high prevalence of the PON1 192R allele among the studied population probably indicates an important role in the development of cardiometabolic diseases.
Keywords: Paraoxonase; polymorphism; cardiovascular risk factor; altitude.
INTRODUCCIÓN
La paraoxonasa 1 sérica humana (PON1) (CE 3.1.8.1) es una lactonasa asociada a las lipoproteínas de alta densidad (HDL) y dependiente de Ca2+, que es capaz de hidrolizar una amplia variedad de sustratos: lactonas y tiolactonas, ésteres arílicos y pesticidas organofosforados (1).
Esta enzima es una glicoproteína de 354 aminoácidos y de aproximadamente 43 KDa de masa molecular; se sintetiza en el hígado y la proteína madura, secretada a la circulación, conserva su secuencia de señal hidrofóbica en la región N-terminal (con la excepción de la metionina inicial) que le permite asociarse con la HDL, circula por la sangre unida a las APO A1 y apo J o clusterina de las HDL. Por ultra centrifugación se ha detectado su presencia en las HDL3 y en las lipoproteínas de muy alta densidad (VHDL) (2).
Mackness et al. (3) demostraron por primera vez, que la PON1 purificada podía impedir la acumulación de lipoperóxidos en lipoproteínas de baja densidad (LDL) in vitro y que la PON1 asociada a las HDL era la principal responsable de la función antioxidante de las HDL, por lo tanto se podía vincular a PON1 con el metabolismo de los lípidos y la aterogénesis. Durante las últimas dos décadas PON1 ha sido ampliamente investigada en el campo de la aterosclerosis, debido a que se demostró que la proteína puede proteger a las LDL de la oxidación; también puede revertir los efectos biológicos de la LDL oxidada, pues puede despojarle de sus propiedades aterogénicas hidrolizando los lipoperóxidos formados y revirtiendo a su condición de LDL nativa, por lo cual ya no son captados por los receptores "scavenger" de los macrófagos y de esta manera se evita la formación de células espumosas; la PON 1 también puede preservar las funciones de las HDL inhibiendo su oxidación (4,5), posiblemente porque puede hidrolizar peróxido de hidrógeno (H2O2), que se produce bajo estrés oxidativo durante la aterogénesis, y de esta manera, la enzima puede proteger la membrana celular (6).
Diversos estudios clínicos encontraron una menor actividad sérica de la paraoxonasa en pacientes con enfermedad cardiovascular y con enfermedades que presentan elevado estrés oxidativo, como la diabetes, síndrome metabólico (SM), procesos inflamatorios, entre otras (7).
Se ha observado una amplia variación en los niveles y actividad sérica de la PON1 en las diferentes poblaciones estudiadas y que son originadas por presencia de polimorfismos de la región promotora y codificante del gen. El polimorfismo PON1192 (región codificante) es el responsable de las variaciones de la actividad paraoxonasa de la enzima. El alelo con arginina en la posición 192 (PON1R192) hidroliza paraoxon a una tasa más alta que el alelo con glutamina en esta posición (PON1Q192). La actividad arilesterasa no está afectada por el polimorfismo (8).
Asimismo, algunos estudios de casos y controles sobre el polimorfismo del gen PON1192 sugieren una correlación con enfermedad de las arterias coronarias, infarto, hipercolesterolemia familiar, diabetes tipo 2, y la enfermedad de Parkinson (7). Serrato et al. (9) mostraron que los pacientes con enfermedad coronaria arterial presentaban una mayor frecuencia del alelo R del polimorfismo PON1 192 que la población general. Otros grupos de investigadores encontraron resultados similares, y formularon la hipótesis de que esta variante alélica podría estar asociada a un mayor riesgo de ECV. También se ha sugerido que el genotipo PON1192QQ juega un papel importante en la prevención de la formación de lesiones ateroscleróticas (10). Resultados similares se obtuvieron en estudios realizados en pacientes que sufren accidentes cerebrovasculares isquémicos, en los cuales el genotipo PON1192RR se correlaciona con el riesgo de accidente cerebrovascular (7). Por otro lado estudios de metaanálisis indican una débil asociación entre el alelo R del polimorfismo PON1-192 y el riesgo de ECV (11).
Los factores de riesgo cardiometabólico pueden variar entre las poblaciones evaluadas, según del área geográfica, costumbres, nivel socioeconómico, estilo de vida, cultura, etnia, altitud, etc. Con respecto a la altitud, hasta ahora no hay información disponible acerca del polimorfismo de esta enzima en poblaciones andinas. El presente estudio tiene por objetivo establecer la distribución del polimorfismo Q192 R de la paraoxonasa 1 y la relación con su actividad, perfil lipídico y APO A1 en una población de Distrito de Junín que habita a 4105 msnm.
MATERIALES Y MÉTODOS
Participantes del estudio
En el presente estudio participaron 79 personas adultas (26 hombres/53 mujeres) clínicamente sanas, a quienes previamente se les dio una charla concerniente a los alcances del estudio y que firmaron el respectivo consentimiento informado.
Recolección de la muestra
Los participantes del estudio se presentaron a primera hora de la mañana para la toma de muestra respectiva en condiciones de ayuno absoluto (12 horas previas) y sin haber realizado actividad física. Las muestras sanguíneas de 10 mL fueron obtenidas mediante punción venosa del antebrazo previa asepsia, una parte de las cuales se recibió con anticoagulante EDTA para los análisis de biología molecular. El resto de la sangre extraída se recibió en tubos de ensayo sin anticoagulante para separar el suero. Las muestras biológicas se colocaron en un recipiente hermético para conservarlas refrigeradas, y fueron transportadas hacia la ciudad de Lima. La extracción del ADN se realizó antes de los tres días de recolectada la muestra sanguínea. Tanto el suero como el ADN extraído se mantuvieron en congelación a -20 oC hasta su posterior análisis.
Recolección de la muestra
La medición de la presión arterial en el brazo izquierdo la efectuó un profesional de la salud con la persona sentada. Se pesaron y tallaron a los individuos sin calzado y con ropa ligera. Asimismo, se les midieron los perímetros de cintura y cadera. Según normas internacionales, el índice de masa corporal (IMC) es igual al peso en kg dividido entre el cuadrado de la estatura en metros. El síndrome metabólico se definió, de acuerdo a los criterios del National Cholesterol Education Program (NCEP) Adult Treatment Panel III (ATP III) (12). Se consideró SM la presencia de tres o más de los siguientes criterios: la presión sistólica y diastólica ≥135/85 mmHg, circunferencia de la cintura >102 cm para hombres y >88 cm para mujeres, glucosa en ayuno ≥110 mg/dL, HDL-col ≤40 mg/dL en hombres y ≤50 mg/dL en mujeres y triglicéridos ≥150 mg/dL.
Determinaciones
Las determinaciones cuantitativas de glucosa, colesterol total, HDL-colesterol, y triglicéridos, fueron realizadas por métodos enzimáticos convencionales, utilizando kits comerciales (Valtek Lab.). La concentración de LDL- colesterol se determinó aplicando la fórmula de Friedewald (12). APO A1 se determinó por inmunoturbidimetría mediante kit de RANDOX (Reino Unido).
La determinación de la actividad de la paraoxonasa se midió sin (basal) y con (estimulada) NaCl 1M según el método descrito por Hernández (13). Se utilizó paraoxon (dietil 4-nitrofenil fosfato) como sustrato. El aumento en la absorbancia debido a la liberación del 4-nitro-fenol de color amarillo, se monitorizó a 405 nm durante tres minutos. La mezcla del ensayo basal estaba formada por 1 mM de paraoxon, 1 mM de CaCl2 en buffer glicina 50 mM, pH 10, y 0,020 mL de suero. Para la actividad POasa estimulada por NaCl, la mezcla de ensayo anterior incluyó además 1 M de NaCl y 0,010 mL de suero. Para los cálculos se utilizó el coeficiente de extinción molar (ε405) del p-nitrofenol: 180500 M–1 cm–1. La actividad POasa se expresó como µmol de p-nitrofenol formado por min por L de suero (U/L). La actividad arilesterasa se determinó utilizando fenilacetato como sustrato (14). La tasa inicial de la hidrólisis se determinó por espectrofotometría a 270 nm durante 3 minutos. La mezcla para el ensayo estaba compuesta por 1 mM de fenilacetato, CaCl2 0,9 mM en buffer Tris-HCl 20 mM, pH 8,0 y 0,025 mL de muestra (suero diluido 1:40). Se calculó la actividad de la enzima utilizando el coeficiente de extinción molar del fenol (1310 M–1cm–1). La actividad arilesterasa se expresó como mmol de fenilacetato hidrolizados por min por L de suero (kU/L).
Los coeficientes de variación intra e inter ensayo para paraoxonasa basal fueron 2,2% y 7,0%, respectivamente, y para arilesterasa 3,3 % y 8,3%.
Genotipificación
Para la extracción del ADN se utilizó un protocolo basado en la aplicación del método de solventes orgánicos fenol – cloroformo. Las muestras se guardó a –20 ºC hasta su análisis.
El DNA obtenido de cada individuo se amplificó por el método de reacción en cadena de la polimerasa (PCR) según protocolo estándar descrito por Humbert et al (15). Para amplificar la región polimórfica 192 Q/R del gen PON1 en las muestras se utilizó: 100 ηg de ADN, 1 U Taq ADN – polimerasa y cebadores específicos (F) 5’-ATTGTTGCTGTGGGACCTGA-3’ y (R) 5’-CACGCTAAACCCAAATACATCTC-3’. La amplificación se efectuó en un termociclador (Perkin Elmer 2400), donde se realizó una primera desnaturalización a 94 ºC por 4 min y luego 35 ciclos 30 segundos a 94°C, 60 segundos a 60°C y 1 minuto a 72°C y finalmente una extensión por 7 min a 72 ºC.
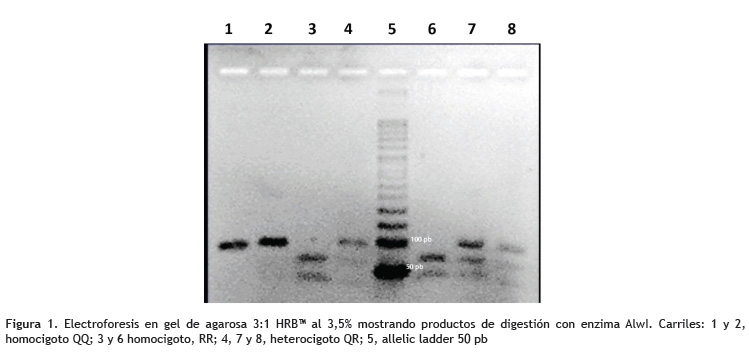
A 1 μg de cada producto amplificado se añadió 2,5 U de AlwI (enzima de restricción), las condiciones de reacción fueron realizadas según las especificaciones del fabricante, a excepción del tiempo de incubación que fue de 12 horas a 37 °C. Luego de este período los productos de la digestión se separaron por electroforesis en geles de agarosa 3:1 HRBTM (Amresco) al 3,5%. Finalmente, los geles de agarosa conteniendo el ADN se colorearon con bromuro de etidio. El alelo PON1192R tiene un sitio de restricción, por lo que se observa dos fragmentos de 63 y 36 bp; el alelo PON1192Q no presenta punto de corte, dado lo cual se obtiene un fragmento de 99 pb.
La interpretación alélica se realizó de la siguiente manera: cuando hay presencia de sitio de restricción en el producto amplificado, entonces se observan dos fragmentos de
63 y 36 bp, que corresponde al alelo PON1192R; y si no presenta sitio de corte, se obtiene un fragmento de 99 pb que corresponde al alelo PON1192Q. La combinación de esos dos alelos dará tres posibles genotipos (Figura 1).
Análisis Estadístico
Los resultados fueron expresados en índices estadísticos y la desviación estándar. Se efectuó la comparación múltiple de medias mediante análisis de varianza (ANOVA) y la subsiguiente prueba de Tukey, si las varianzas grupales eran homogéneas, o según la prueba de Tamhane, en caso contrario. Las frecuencias genotípicas fueron determinadas por conteo directo. La prueba de chi-cuadrado se utilizó para determinar el grado de asociación entre variables cualitativas y para evaluar la magnitud de desviación de las frecuencias genotípicas respecto del equilibrio de Hardy-Weinberg. Se consideró significativo todo resultado cuyo valor de p era < 0,05. Se utilizó el paquete estadístico SPSS versión 20.
RESULTADOS
En la Figura 1 se muestra la imagen de electroforesis en gel de agarosa de los resultados típicos de la digestión con AlwI para el polimorfismo PON1192QR. Los productos de PCR que no fueron digeridos corresponden al genotipo QQ (carriles 1 y 2). Los productos de PCR que presentan los fragmentos de 99, 63 y 36 pb corresponden al genotipo QR (carriles 4, 7 y 8) y los fragmentos de 63 y 36 bp corresponden al genotipo RR (carriles 3 y 6).
Las frecuencias tanto genotípicas como alélicas de las variantes de la PON1 Q192R fueron obtenidas por conteo directo. El resultado del análisis se presenta en la Tabla 1, y como puede observarse, las frecuencias genotípicas se encuentran en equilibrio de Hardy-Weinberg (p = 0,864).
Las frecuencias genotípicas observadas según género se presentan en la tabla 2: la frecuencia del genotipo QQ es mayor en hombres (23,08%) que en mujeres (9,43%), mientras que los genotipos QR y RR son menores en hombres que en mujeres (38,46 %, 38,5% vs 49,1% y 41,5%, respectivamente); sin embargo las diferencias no son significativas por género en cuanto a la distribución de los genotipos (p = 0,246).
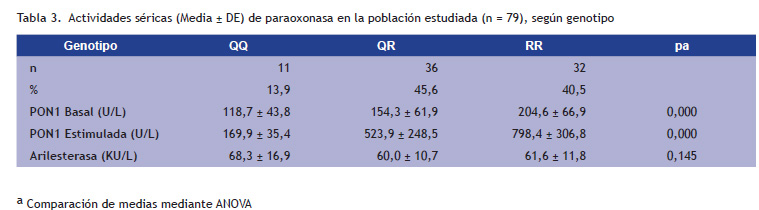
Los resultados del análisis de varianza, según el genotipo y su correspondiente actividad enzimática, se presentan en la Tabla 3. Se observan diferencias significativas en las comparaciones (p = 0,000), tanto las basales como las estimuladas con NaCl 1M. Sin embargo, en el caso de la actividad arilesterasa de la enzima, no se observan diferencias (p = 0,145).
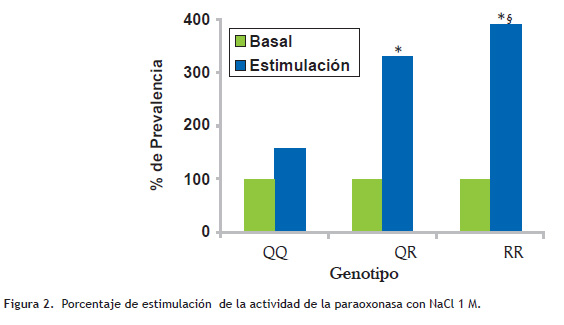
La respuesta de la enzima paraoxonasa a la estimulación por acción del NaCl muestra amplia diferencia respecto del genotipo; así el QQ presenta un % de estimulación de 56,6 ± 48,2; el QR de 229,7 ± 93,1 y el RR de 287,2 ± 79,0. El análisis de varianza de los porcentajes de estimulación pone en evidencia alta significación estadística en las diferencias entre las medias de los tres grupos (p = 0,000) Figura 2.
Los resultados son expresados como medias. La media del grupo Basal corresponde al 100%. * p = 0,000 respecto a QQ, § p = 0,015 respecto a QR. Comparación de medias mediante ANOVA, prueba de Tukey.
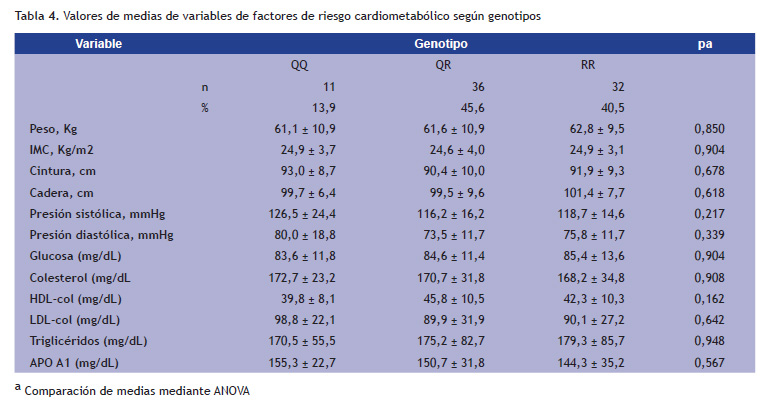
La Tabla 4 muestra los valores de las medias de las diferentes variables en relación al genotipo; como se observa, no hay diferencia significativa en peso, IMC, cintura, cadera y presión entre los tres genotipos. No se detectaron diferencias significativas entre los genotipos con respecto a los valores medios de glucosa, colesterol, HDL-C, LDL-col y APO A1; en relación al nivel de triglicéridos, se observa que los valores son menores en orden según el fenotipo QQ<QR< RR , pero sin significación estadística entre genotipos.
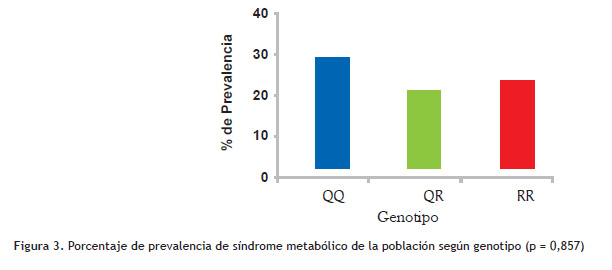
Se encontraron 17 sujetos que califican para SM dentro del grupo (n = 79) y que representan el 21,5 % de prevalencia. La figura 3 grafica la prevalencia de SM según el genotipo. Los resultados parecen indicar una mayor prevalencia de SM en el alelo Q que en el R; sin embargo no se observó ninguna asociación significativa entre genotipo y SM (p = 0,857).
DISCUSIÓN
Numerosos estudios se han realizado sobre la relación entre los polimorfismos de PON1- Q192R y enfermedades cardiovasculares (16). Según un meta-análisis de 35 estudios, Wheeler et al. (17) reportaron que la variante PON1 192R estaría asociado con mayor riesgo de enfermedad cardiovascular, por lo que esa variante genética ha sido propuesta como un factor de riesgo; y debido también que esta isoforma es más susceptible a ser inactivada por estrés oxidativo, a diferencia del alelo Q, que estaría involucrado en la protección contra la aterosclerosis (10).
En nuestro estudio hemos encontrado una frecuencia de 13,9 % para el genotipo QQ; 45,6% para el genotipo QR y 40,5% para el genotipo RR, y con una frecuencia alélica de 0,633 para el alelo R. Estos resultados difieren a los reportados por Cataño et al (18), quienes indicaron que la frecuencia del alelo Q PON1 192 fue mayor que la del R PON1 192 (0,54 y 0,46, respectivamente) en una población de Lima.
La frecuencia del alelo Q PON1 192 hallada en el presente estudio (0,367) fue inferior a la encontrada en otras poblaciones: norte americanos (USA) (0,728), europeos (0,7129, promedio de 8 países incluyendo España), hindúes, (0,820), etíopes (0,592), mestizos mejicanos (0,525), chilenos (0,663), brasileños de origen europeo (0,693), costarricenses (0,757), etc. (18).
La población estudiada en este trabajo es mestiza y la distribución de los genotipos va a variar según el grado de mestizaje, es decir de la mezcla de la población indígena peruana con poblaciones euro-asiáticas, principalmente españolas.
Nuestros resultados de la frecuencia del alelo R (0,633) son similares a los encontrados en los nativos Oji-Cree (0,766) y entre la población Inuit (0,700), ambas en Canadá (19); teenek (0,520) y huichol (0,590) (grupos indígenas ubicados en las regiones Huasteca y Nayarit, respectivamente, de México) (20); la tribu Cayapa ecuatoriana (0,789) (21) y entre algunos pueblos amazónicos de Brasil (0,730) (22). Del mismo modo, con algunos grupos del este de Asia, como China (0,632) (23), Japón (0,660) (24) y Corea (0,620) (25). Según los datos observados, la distribución del alelo R podría reflejar la cercanía genética que tienen entre sí esas poblaciones, hipótesis ya planteada por otros estudios previos (18). Hasta donde sabemos, este es el primer estudio sobre polimorfismos de la enzima paraoxonasa en poblaciones que viven en las grandes alturas en nuestro país. Es necesario estudiar la distribución del genotipo de PON 1 Q192R en poblaciones nativas de las grandes alturas, de ascendencia quechua, aymara y de otros grupos étnicos.
En el presente estudio se ha evaluado las actividades de la enzima PON 1 (paraoxonasa basal y estimulada, y esterasa) en relación con sus correspondientes genotipos. Las actividades paraoxonasa, tanto basal como estimulada, fueron mayores en el genotipo RR, seguido por el heterocigoto QR y la más baja en los genotipos QQ, y la diferencia es estadísticamente significativa (p = 0,000). La alta respuesta a la estimulación de la actividad entre los genotipos con alelo 192 R de esta población podría deberse a una característica adaptativa a las grandes alturas. Asimismo, sería interesante estudiar otros grupos poblacionales a diferentes niveles de altitud para ver si hay una posible presión selectiva de los genotipos. Por otro lado, la actividad esterasa de la enzima no fue afectada por el polimorfismo PON 1 Q192R (Tabla 3), lo que coincide con lo hallado por otros autores (26).
La asociación del polimorfismo PON1 Q192R con los niveles de lípidos séricos ha sido estudiada por diversos investigadores, con resultados contradictorios. Por ejemplo, algunos han encontrado una asociación significativa entre variantes genéticas de la paraoxonasa y cambios en los niveles de HDL-col y en las concentraciones de triglicéridos en poblaciones relativamente aisladas (26), sin embargo, otros no hallaron ese vínculo (27). En un estudio clásico, que se llevó a cabo con los Hermanos Hutterian o huteritas, una población de América del Norte, aislada por sus creencias religiosas, se encontró que el genotipo PON1 se asociaba significativamente con variaciones en la concentración de HDL-C, LDL-C, triglicéridos y apo-B (26). Entre ellos, los homocigotos QQ tenían significativamente menor nivel de TG, LDL-C y apo-B que los heterocigotos QR y homocigotos RR. Ruiz et al. (1995) (28) estudiaron a poblaciones caucásicas con diabetes tipo 2 y encontraron que los genotipos QR y RR tuvieron mayor concentración HDL-C y APO A1 en comparación con genotipos QQ. En contraste, Agachan et al (2004) (27) informaron que el genotipo PON1 192 no tuvo efecto alguno sobre los niveles de lípidos tanto en los controles como en los pacientes con diabetes tipo 2.
Con el fin de conocer si el polimorfismo PON1 tiene alguna influencia en el perfil lipídico de nuestra población de estudio, se determinaron los niveles séricos del perfil lipídico y APO A1. Así, luego de analizarse los datos mediante análisis de varianza, no se encontró ninguna asociación entre los genotipos PON1 y la concentración de lípidos séricos y APO A1. Por otra parte, el alelo PON1 192R ha sido considerado un factor de riesgo para desarrollar obesidad y trastornos cardiometabólicos (29). Sin embargo, en este estudio no se encontró diferencia significativa en la prevalencia de SM entre los tres grupos.
La alta prevalencia de este alelo encontrada en una poblacion sin antecedentes de enfermedades cardiovasculares ni diabetes es dificil de interpretar, debido a que existen otros factores como estilo de vida que modulan el metabolismo y por ende, la salud y enfermedad.
Estudios realizados en poblaciones andinas han encontrado que la prevalencia de los principales factores de riesgo cardiovascular es menor que en poblaciones del nivel del mar (30) y que la incidencia de enfermedad coronaria así como del infarto al miocardio también es menor en personas que viven a gran altitud (31). Sin embargo, es posible que los individuos portadores del alelo * R podrían estar en mayor riesgo de estas enfermedades cuando se exponen a condiciones particulares, como migrar hacia la costa, donde se produce un cambio en sus costumbres, patrones alimentarios y actividad física, etc.
En conclusión, nuestros resultados indican que en esta población andina aparentemente sana hay mayor prevalencia del alelo PON1 R, el cual se considera como un factor de riesgo de ECV. En tanto, el polimorfismo genético PON1192 no tiene influencia sobre los niveles del perfil lipídico y APO A1.
Sin embargo, debido a las limitaciones del presente trabajo, se sugiere estudiar la distribución del polimorfismo PON1 192 con muestras de mayor tamaño en otras poblaciones que viven en las grandes alturas. Subsiguientes estudios permitirán esclarecer si hay o no correlación entre el polimorfismo del gen PON1 y el riesgo de padecer enfermedades cardiometabólicas en las poblaciones peruanas.
REFERENCIAS BIBLIOGRÁFICAS
1. Draganov DI, Teiber JF, Speelman A, Osawa Y, Sunahara R, La Du B. N. Human paraoxonases (PON 1, PON 2, and PON 3) are lactonases with overlapping and distinct substrate specificities. J Lipid Res. 2005; 46(6):1239-47. [ Links ]
2. Mackness B, Durrington PN, Mackness MI. Human serum paraoxonase. Gen Pharmacol. 1998; 31(3):329–36 [ Links ]
3. Mackness MI, Arrol S, Durrington PN. Paraoxonase prevents accumulation of lipo-peroxides in low-density lipoprotein. FEBS Letts. 1991; 286(1):152–4. [ Links ]
4. Watson AD, Berliner JA, Hama SY, La Du BN, Fault KF, Fogelman AM, et al. Protec¬tive effect of high density lipoprotein associated paraoxonase. Inhibition of the bio¬logical activity of minimally oxidized low-density lipoprotein. J Clin Invest. 1995; 96(6):2882–91. [ Links ]
5. Aviram M, Rosenblat M, Bisgaier CL, Newton RS, Primo-Parmo SL, La Du BN. Paraoxonase inhibits high-density lipoprotein oxidation and preserves its functions. A possible peroxidative role for paraoxonase. J Clin Invest. 1998; 101(8):1581-90. [ Links ]
6. Deakin SP, Bioletto S, Bochaton-Piallat ML, James RW. HDL- associated paraoxonase-1 can redistribute to cell membranes and influence sensitivity to oxida¬tive stress. Free Radic Biol Med. 2011; 50(1):102–9. [ Links ]
7. Goswami B, Tayal D, Gupta N, and Mallika V. Paraoxonase: a multifaceted biomolecule. Clin Chim Acta. 2009; 410(1):1–12. [ Links ]
8. Adkins S, Gan KN, Mody M, La Du BN. Molecular basis for the polymorphic forms of human serum paraoxonase/arylesterase: glutamine or arginine at position 191, for the respective A or B allozymes. Am J Hum Genet 1993; 52(3): 598- 608. [ Links ]
9. Serrato M, Marian AJ. A variant of humanparaoxonase/ arylesterase (HUMPONA) gene is a risk factor for coronary artery disease. J Clin Invest 1995; 96(6): 3005–8 [ Links ]
10. Mackness B. Mackness M, Aviram M, Paragh G. The Paraoxonases: Their Role in Disease Development and Xenobiotic Metabolism. Vol 3. The Netherlands: Springer, 2008. p. 51–60 [ Links ]
11. Wang M, Lang X, Zou L, Huang S, Xu Z. Four genetic polymorphisms of the paraoxonase gene and risk of coronary heart disease: a meta-analysis based on 88 case–control studies. Atherosclerosis. 2010; 214(2):377-85. [ Links ]
12. Williams L. Third Report of the National Cholesterol Education Program (NCEP) Expert Panel on Detection, Evaluation, and Treatment of High Blood Cholesterol In Adults (Adult Treatment Panel III) final report. Circulation. 2002; 106(25):3143-421. [ Links ]
13. Hernández AF, Gonzalvo MC, Gil L, Rodrigo L, Villanueva E, Pla A. Distribution profiles of paraoxonase and cholinesterase phenotypes in a Spanish population. Chem Biol Interact. 1999;119–120: 201-209 [ Links ]
14. Eckerson HW, Whyte CM, La Du BN. The human serum paraoxonase/ arylesterase polymorphism. Am. J. hum. Genet. 1983; 35(6):1126−38.
15. Humbert R, Adler DA, Disteche CM, Hassett C, Omiecinski C, Furlong C. The molecular basis of the human serum paraoxonase activity polymorphism. Nat Genet. 1993;3(1):73- 6. [ Links ]
16. Hassan MA, Al-Attas OS, Hussain T, Al-Daghri NM, Alokail MS, Mohammed AK, et al. The Q192R polymorphism of the paraoxonase 1 gene is a risk factor for coronary artery disease in Saudi subjects. Mol Cell Biochem. 2013;380(1-2):121-8 [ Links ]
17. Wheeler JG, Keavney BD, Watkins H, Collins R, Danesh J. Four paraoxonase gene polymorphisms in 11212 cases of coronary heart disease and 12786 controls: meta-analysis of 43 studies. Lancet. 2004; 363(9410):689–95. [ Links ]
18. Cataño HC, Cueva JL, Cardenas AM, Izaguirre V, Zavaleta AI, Carranza E, et al. Distribution of paraoxonase-1 gene polymorphisms and enzyme activity in a Peruvian population. Environ Mol Mutagen. 2006;47(9): 699–706. [ Links ]
19. Hegele RA. Genetic prediction of atherosclerosis: Lessons from studies in native Canadian populations. Clin Chim Acta. 1999;286(1):47-61 [ Links ]
20. Gamboa R, Zamora J, Rodriguez–Perez JM, Fragoso JM, Cardoso G, Posadas–Romero C, et al: Distribution of paraoxonase PONÍ gene polymorphisms in Mexican populations. Its role in the lipid profile. Exp Mol Pathol. 2006; 80(1): 85–90. [ Links ]
21. Scacchi R, Corbo RM, Rickards O, De Stefano GF. New data on the world distribution of paraoxonase (PON1 Gln 192 -- Arg) gene frequencies. Hum Biol. 2003;75(3):365-73. [ Links ]
22. Santos N, Ribeiro-dos-santos A, Santos S. Frequency of the Q192R and L55M polymorphisms of the human serum paraoxonase gene (PON1) in ten Amazonian Amerindian tribes. Genetics and Molecular Biology. 2005; 28(1): 369. [ Links ]
23. Sanghera D, Aston C, Saha N, Kamboh M. DNA polymorphism in two paraoxonase genes (PON1 and PON2) are associated with the risk of coronary heart disease. Am J Human Genet. 1998; 62(1):36-44 [ Links ]
24. Sato H, Ito Y, Ueyama J, Kano Y, Arakawa T, Gotoh M, et al. Effects of Paraoxonase 1 gene polymorphisms on organophosphate insecticide metabolism in Japanese pest control workers. J Occup Health. 2016; 58(1): 56–65 [ Links ]
25. Pejin-Grubiša, I. HDL-Associated paraoxonase 1gene polymorphisms as genetic markers for wide spread diseases. In S. Frank and G. Kostner , Lipoproteins – Role in Health and Disease. 2012, p. 431 – 44.
26. Hegele RA, Brunt JH, Connelly PW. A polymorphism of the paraoxonase gene associated with variation in plasma lipoproteins in a genetic isolate. Arterioscler Thromb Vasc Biol. 1995;15(1): 89–95. [ Links ]
27. Agachan B, Yilmaz H, Karaali Z, Isbir T. Paraoxonase 55 and 192 polymorphism and its relationship to serum paraoxonase activity and serum lipids in Turkish patients with non-insulin dependent diabetes mellitus. Cell Biochem Funct. 2004; >22(3):163–8 [ Links ]
28. Ruiz J, Blanche H, James RW, Garin MC, Vaisse C, Charpentier G, et al. Gln-Arg 192 polymorphism of paraoxonase and coronary artery disease in type 2 diabetes. Lancet. 1995; 346:869–72. [ Links ]
29. Senti M, Tomas M, Fitó M, Weinbrenner T, Covas MI, Sala J, et al. Antioxidant paraoxonase 1 activity in the metabolic syndrome. J Clin Endocrinol Metab. 2003; 88(11):5422-6 [ Links ]
30. Segura L, Regulo C, Parodi J. Factores de Riesgo de las Enfermedades Cardiovasculares en el Perú. Estudio TORNASOL. Rev Peru Cardiol. 2006; 32(2):82-128. [ Links ]
31. West J, Schoene R, Luks A, Milledge J. High Altitude Medicine and Physiology. 5nd ed. CRC Press, Taylor & Francis Group Boca Raton, FL, 2013. p. 275-277 [ Links ]
Fuentes de financiamiento
Este artículo ha sido financiado por los autores.
Conflictos de interés:
Los autores declaran no tener ningún conflicto de interés.
Correspondencia:
Elizabeth Carranza Alva
Dirección: Instituto Nacional de Biología Andina Facultad de Medicina – UNMSM. Av. Alfonso Ugarte 880. Lima, Perú.
Teléfono: 431-3355
Correo electrónico: ecarranzalva@gmail.com
Recibido: 15 de febrero de 2017
Aprobado: 01 de abril de 2017