Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Horizonte Médico (Lima)
Print version ISSN 1727-558X
Horiz. Med. vol.18 no.2 Lima Apr./Jun. 2018
http://dx.doi.org/10.24265/horizmed.2018.v18n2.03
ARTÍCULO ORIGINAL
Patrón de clonalidad mediante ERIC-PCR y REP-PCR de Escherichia coli y Klebsiella pneumoniae productores de betalactamasas de espectro extendido, aisladas de pacientes con infección urinaria intrahospitalaria. Hospital Regional Lambayeque, Perú
Clonality pattern assessed by ERIC-PCR and REP-PCR in extended-spectrum beta- lactamase-producing Escherichia coli and Klebsiella pneumoniae isolates from patients with nosocomial urinary tract infections. Hospital Regional Lambayeque, Peru
Kelly Lelia López-Ramírez 1b ORCID iD: https://orcid.org/0000-0002-9640-2439, Kevin Colbert Díaz-Maldonado 1b ORCID iD: https://orcid.org/0000-0002-3216-0338, Martha Arminda Vergara Espinoza 1b ORCID iD: https://orcid.org/0000-0003-4395-1212, Olivia Santamaría- Veliz 1a ORCID iD: https://orcid.org/0000-0003-4098-3139, Luis Miguel Serquén-López 2a ORCID iD: https://orcid.org/0000-0002-4141-9719, Bustamante Canelo Olinda 3b ORCID iD: https://orcid.org/0000-0003-3244-5939, Franco Ernesto León-Jimenez 4c ORCID iD: https://orcid.org/0000-0002-9418-3236, Franklin-Rómulo Aguilar-Gamboa 5b ORCID iD: https://orcid.org/0000-0003-1943-5613
1. Universidad Nacional Pedro Ruiz Gallo, Laboratorio de Microbiología. Lambayeque, Perú.
2. Hospital Regional Lambayeque, Dirección de Investigación, Laboratorio de Biología Molecular. Lambayeque, Perú.
3. Hospital Regional Lambayeque, Laboratorio de Bacteriología. Lambayeque, Perú.
4. Universidad Católica Santo Toribio de Mogrovejo, Escuela de Medicina Humana. Chiclayo, Perú.
5. Hospital Regional Lambayeque, Dirección de Investigación, Laboratorio de Inmuno-microbiología experimental. Chiclayo, Perú. a. Biólogo.
b. Microbiólogo.
c. Médico Internista, Epidemiólogo Clínico.
RESUMEN
Objetivo: Determinar el patrón de clonalidad mediante ERIC-PCR y REP-PCR en aislamientos de Escherichia coli y Klebsiella pneumoniae, productores de betalactamasas de espectro extendido (BLEE), aisladas de pacientes con infección urinaria del Hospital Regional Lambayeque durante julio a noviembre de 2015.
Materiales y métodos: Se analizaron 30 aislados clínicos conformados por E. coli y K. pneumoniae procedentes de los servicios de emergencia, medicina, cirugía y pediatría del HRL, La relación clonal, se evaluó mediante ERIC-PCR y REP- PCR. Para las agrupaciones se empleó el algoritmo UPGMA utilizando el software Quantity One-BIORAD, generando los dendrogramas con la unión de los perfiles electroforéticos obtenidos por ambas herramientas de tipificación. Resultados: Del análisis molecular se discriminaron tres patrones clonales predominantes en E. coli y dos en K. pneumoniae. Conclusiones: El estudio revela la diseminación clonal de microorganismos potencialmente patógenos en el servicio de emergencia, donde urge implementar medidas para su prevención y control.
Palabras claves: Escherichia coli; Klebsiella pneumoniae; betalactamasas; infecciones urinarias; infección hospitalaria (Fuente: DeCS BIREME).
ABSTRACT
Objective: To determine the clonality pattern assessed by ERIC-PCR and REP-PCR in extended-spectrum beta-lactamase (ESBL)-producing Escherichia coli and Klebsiella pneumoniae isolates from patients with urinary tract infections at the Hospital Regional Lambayeque (HRL) from July to November 2015.
Materials and methods: A total of 30 ESBL-producing E. coli and K. pneumoniae clinical isolates collected from the HRL’s emergency, medicine, surgery and pediatrics services were assessed. The clonal relationship was determined using the ERIC-PCR and REP-PCR markers. For clusters, UPGMA algorithm with Bio Rad Quantity One 1-D analysis software was used, thus generating dendrograms through the union of the electrophoretic profiles obtained by both molecular markers. Results: From the molecular analysis, three predominant clonal patterns were found in E. coli and two in K. pneumoniae. Conclusions: The study reveals the clonal dissemination of potentially pathogenic microorganisms in the emergency service, where it is urgent to implement measures for their prevention and control.
Keywords: Escherichia coli; Klebsiella pneumoniae; beta-lactamases; urinary tract infections; cross infection (Source: MeSH NLM).
INTRODUCCIÓN
Las infecciones intrahospitalarias (IIH) son aquellas que se suscitan luego de 48 a 72 horas del ingreso de un paciente a un servicio; ello produce aumento en la morbimortalidad, particularmente en imnunodeprimidos, pacientes operados y pacientes con dispositivos médicos invasivos. La infección del tracto urinario es la IIH más frecuente, y es principalmente originada por cepas de Escherichia coli y Klebsiella pneumoniae productoras de betalactamasas de espectro extendido (1,2). Debido a su capacidad de diseminación, existe limitación en las opciones terapéuticas disponibles (3).
Patrones fenotípicos de resistencia antimicrobiana similares pueden sugerir que varios aislamientos provienen de una misma cepa, sin embargo esto no es concluyente. Para ello, es necesario realizar la caracterización molecular o genotipificación de los mismos, y aunque la electroforesis de campo pulsado (PFGE) es la técnica más empleada para este cometido, existen en la actualidad otras técnicas moleculares que permiten analizar con precisión y reproducibilidad la relación clonal existente entre aislamientos bacterianos (4). A este respecto, el genoma de las enterobacterias contiene dos tipos de secuencias cortas, repetidas y esparcidas, llamadas ERIC (Enterobacterial Repetitive Intergenic Consensus) y secuencias REP (Repetitive Extragenic Palindromic), las cuales han sido usadas ampliamente en tipificación, análisis genético y en la identificación de brotes infecciosos bacterianos (5).
Las pruebas ERIC y REP PCR (reacción en cadena de la polimerasa), están basadas en la amplificación de las secuencias de ADN a través de la PCR, usando cebadores específicos para cada tipo de secuencia. El polimorfismo detectado resulta de la variabilidad en la repetición de dichas secuencias y de la distancia entre copias contiguas, causadas por inserciones o deleciones del ADN. Usando estas secuencias ha sido posible, discriminar serotipos estrechamente relacionados de la misma especie, y grupos de cepas no relacionadas clonalmente, ya que poseen un gran poder discriminatorio (6,7).
En el Hospital Regional Lambayeque (HRL) durante el 2015 se tuvo una incidencia acumulada de infección del tracto urinario (ITU) de 11.2 por cada 1000 pacientes, superior al promedio nacional (3.31 para hospitales nivel III.1) y de estos más del 60 % fueron causadas por enterobacterias productoras de betalactamasas de espectro extendido (BLEE) tanto en los servicios de hospitalización como en áreas críticas (datos sin publicar). Sin embargo, se desconoce si estos aislamientos corresponden a casos aleatorios o relacionados, por lo que se necesita establecer similitudes filogenéticas que permitan determinar la presencia de clones. Ante ello, el objetivo del estudio fue caracterizar, mediante ERIC-PCR y REP-PCR, cepas de Escherichia coli y Klebsiella pneumoniae productoras de BLEE, aisladas de pacientes con infección urinaria intrahospitalaria del Hospital Regional Lambayeque durante los meses de junio a diciembre de 2015.
MATERIALES Y MÉTODOS
Estudio descriptivo trasversal. Durante el periodo de julio a noviembre de 2015 por medio de un muestreo no probabilístico consecutivo. Se seleccionaron un total de 20 aislamientos de E. coli y 10 de K. pneumoniae productores de BLEE, obtenidos de pacientes con infección urinaria, procedentes de los servicios de Emergencia, Medicina, Cirugía y Pediatría del HRL. Se tomaron en cuenta los aislamientos de pacientes que desarrollaron ITU después de 48 horas de estancia hospitalaria. Las muestras de orina fueron sembradas en el medio CLED (Cysteine lactose electrolyte deficient). Se tomaron como criterios de positividad la presencia de reacción inflamatoria y un crecimiento mayor a 105 UFC/ml en placa, luego las colonias fueron identificadas mediante métodos convencionales. No se consideraron los aislamientos que se obtuvieron de un mismo paciente.
Se realizó el antibiotipado por el método de Kirby Bauer en placa Muëller Hinton y la interpretación se realizó de acuerdo a los parámetros establecidos por la guía Clinical and Laboratory Standars Institute (CLSI) (8). Mientras que la producción de BLEE fue confirmada por el método de Jarlier según el Comité de la Sociedad Francesa de Microbiología (9).
Para la validación de los resultados se utilizó la cepa control K. pneumoniae 700603, según lo recomendado por el CLSI (8).
Se extrajo elADN de ambas cepas los cuales fueron suspendidos en 50 µL de H O PCR y conservados en refrigeración de 4 °C o -20 °C hasta su posterior análisis (10,11).
Los patrones de clonalidad fueron obtenidos mediante las técnicas ERIC-PCR y REP-PCR, empleando para el primero primers forward ERIC 1 (5'-ATGTAAGCTCCTGGGGATTCA-3'), y reverse ERIC 2 (5'-AAGTAAGTGACTGGGGTGAGAGCG-3'), mientras que para el segundo, primers forward REP 1R-I (5'-IIIICGICGICATCIGGC-3') y reverse REP2-I (5'-ICG ICTTATCIGGCCTAC-3') (7).
La preparación del mix de reacción se realizó con un volumen final de 12,5 µL (5,12). Se utilizó Master Mix 2X Promega (1X) que contuvo Taq (0.625 U), dNTPs (200 µM) y MgCl2 (1,5 mM), además se agregó Primer Forward y Reverse (1 µM/µL) y ADN, adicionalmente se optimizó con MgCl2 (1 mM) y Taq Polimerasa Gentaq (0,375 U). Las condiciones termodinámicas para ambas herramientas de tipificación constaron de un paso inicial de desnaturalización de 94 °C por 7 minutos, seguido por 45 ciclos de: desnaturalización a 94 °C por 1 minuto, hibridación de 41.2 °C para ERIC y 40.3 °C para REP por 1 minuto y extensión a 72 °C por 5 minutos, además de un paso de extensión final a 72 °C por 7 minutos.
Los productos amplificados se separaron electroforé- ticamente en un gel de agarosa al 2 % usando un marcador de peso molecular de 100 pb. Las condiciones fueron: Pre- corrida 70V por 10 minutos, corrida a 140V por 70 minutos y post-corrida 70V por 10 minutos. Luego los geles fueron teñidos por 15 minutos en una solución que contenía bromuro de etidio (0.5 mg/mL).
Los perfiles electroforéticos generados por ERIC-PCR y REP-PCR fueron fotodocumentados con el escáner molecular Pharos Fx®, la posición de las bandas se normalizó empleando un marcador de peso molecular de 100 pb. Las imágenes se analizaron mediante el software Quantity One® BIORAD.
El cálculo de similitud entre cada par de aislamientos de la misma especie se dio utilizando el coeficiente de Dice generando una matriz de similitud. Con estos datos se llevó a cabo un agrupamiento UPGMA (Unweighted Pair Group Method using Arithmetic Averages). Este procedimiento se evidenció gráficamente en un dendrograma. Para la formación de agrupaciones se utilizaron los criterios de Tenover et al. (13).
Consideraciones éticas
El protocolo del estudio fue evaluado y aprobado por el comité de ética de la dirección de investigación del Hospital Regional Lambayeque con código 212-030-15. Se siguieron los criterios y normas éticas establecidas en la Declaración de Helsinki. Todos los procedimientos del presente se enfocaron en preservar la privacidad de los pacientes de los cuales se aislaron las bacterias objeto de estudio.
RESULTADOS
Durante el periodo de estudio se obtuvieron 30 aislamientos de origen hospitalario conformados por 20 aislamientos E. coli y 10 de K. pneumoniae productores de BLEE causantes de infecciones urinarias. Mediante la prueba de susceptibilidad antimicrobiana de los 20 aislamientos de E. coli, se obtuvo una resistencia del 100 % a los antibióticos cefotaxima, ceftriaxona, ciprofloxacino y trimetoprim/ sulfametoxazol y 60 % a ceftazidima, susceptibilidad intermedia del 50 % en aztreonam y amoxicilina/ ácido clavulánico, y se observó una sensibilidad del 100 % con meropenem y cefoxitin.
En los 10 aislamientos de K. pneumoniae se registró un 100 % de resistencia a los antibióticos cefotaxima, ceftriaxona, cefepime, ciprofloxacino, gentamicina y trimetoprim/ sulfametoxazol, seguido de ceftazidima con 90 % y aztreonam con 80 % de resistencia, el 40 % presentaron susceptibilidad intermedia a amoxicilina/ ácido clavulánico, y se evidenció mayor sensibilidad para meropenem y cefoxitin en 100 % y 90 % respectivamente.
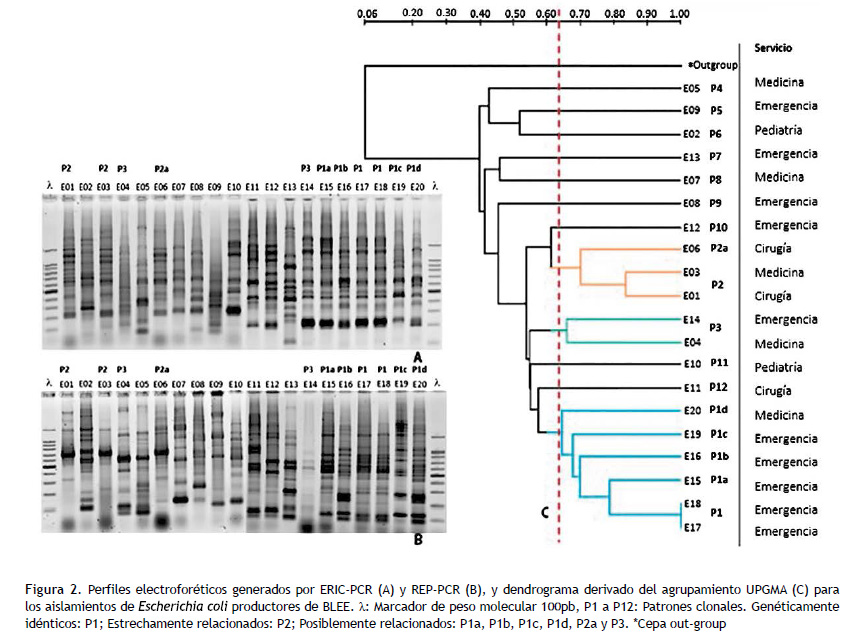
Dentro de los patrones clonales de E. coli, el patrón P1 agrupó seis aislamientos, dos de ellos (E17 y E18) fueron discriminados como genéticamente idénticos, con un coeficiente de similitud del 1.00, los cuatro aislamientos restantes (E15, E16, E19 Y E20) correspondieron a subpatrones clonales (P1a, P1b, P1c y P1d) discriminados como posiblemente relacionados. El patrón P2, agrupó tres aislamientos, dos de ellos (E01 y E03) fueron discriminados como estrechamente relacionados, y el aislamiento restante (E06) fue considerado como un subpatrón clonal (P2a) discriminado como posiblemente relacionado; el patrón P3, agrupó dos aislados (E04 y E14) que fueron discriminados como posiblemente relacionados; los patrones no relacionados P4, P5, P6, P7, P8, P9, P10, P11 y P12 agruparon un solo aislamiento cada uno (Figura 1).
Se confirmó la producción de BLEE en todos los aislados utilizando el Test de Jarlier y se detectó que el 65 % de aislamientos de E. coli evidenciaron sinergismo con tres cefalosporinas, el 35 % con dos (Cefotaxima y ceftriaxona), y no hubo sinergismo con solo una cefalosporina, mientras el 80 % de aislamientos de K. pneumoniae presentaron sinergismo con tres cefalosporinas, un 10 % con dos (Ceftazidima y ceftriaxona) y 10 % con una (Cefotaxima).
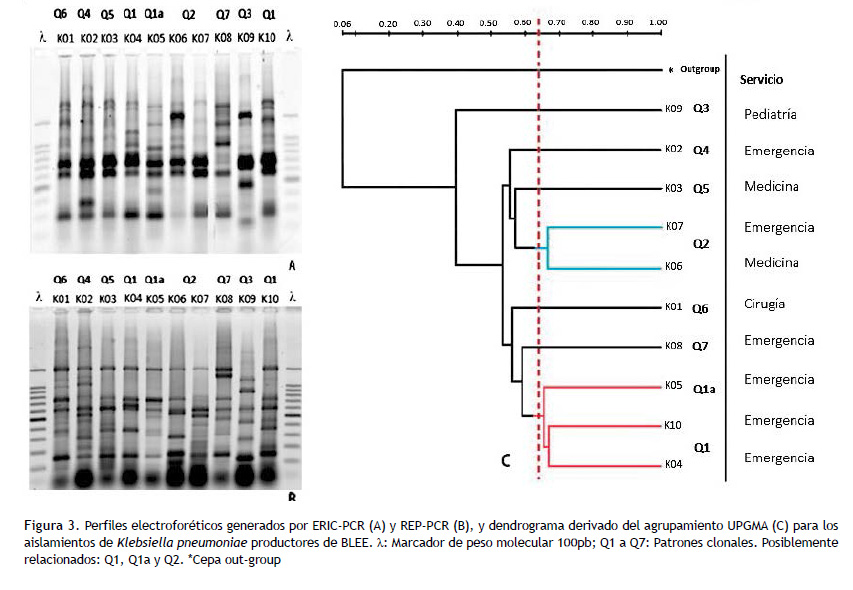
Los perfiles electroforéticos generados por ERIC-PCR y REP-PCR discriminaron doce patrones clonales (P1-P12) de los 20 aislamientos de E. coli (12/20) y siete patrones clonales (Q1-Q7) de 10 aislamientos para K. pneumoniae (7/10), con un coeficiente de similitud del 0.64 tomando en cuenta los polimorfismos generados. Además, se obtuvo un poder de discriminación de 0.91 y 0.93 para las agrupaciones de E. coli y K. pneumoniae y un coeficiente de correlación cofenético de 0.95 y 0.97, respectivamente. Las agrupaciones para cada especie se ilustran en Figuras 2 y 3.
Para K. pneumoniae productores de BLEE, el patrón Q1 agrupó tres aislamientos, dos de ellos (K04 y K10) fueron discriminados como posiblemente relacionados y el aislamiento restante (K05) considerado como un subgrupo clonal Q1a, se discriminó como posiblemente relacionado.
El patrón Q2 agrupó dos aislamientos (K06 y K07) discriminados como posiblemente relacionados; los patrones no relacionados Q3, Q4, Q5, Q6 y Q7 agruparon una aislamiento cada uno (Figura 3).
DISCUSIÓN
Los ambientes hospitalarios suelen prestar las condiciones adecuadas para albergar patógenos multirresistentes, los cuales emplean diversas vías de diseminación que son facilitadas por las características propias de cada servicio. En la actualidad, las bacterias productoras BLEE se han convertido en uno de los principales problemas en pacientes de áreas no críticas, debido al incremento de morbimortalidad con la que estos se relacionan (3). Ante ello, la vigilancia de infecciones nosocomiales (IN) es una práctica que puede reducir de forma efectiva las tasas de infección. Así lo demostraron Li et al., quienes evaluaron el impacto de dicha vigilancia en las IN, resaltando la importancia de que ésta se realice tomando en cuenta las peculiaridades de cada nosocomio (14).
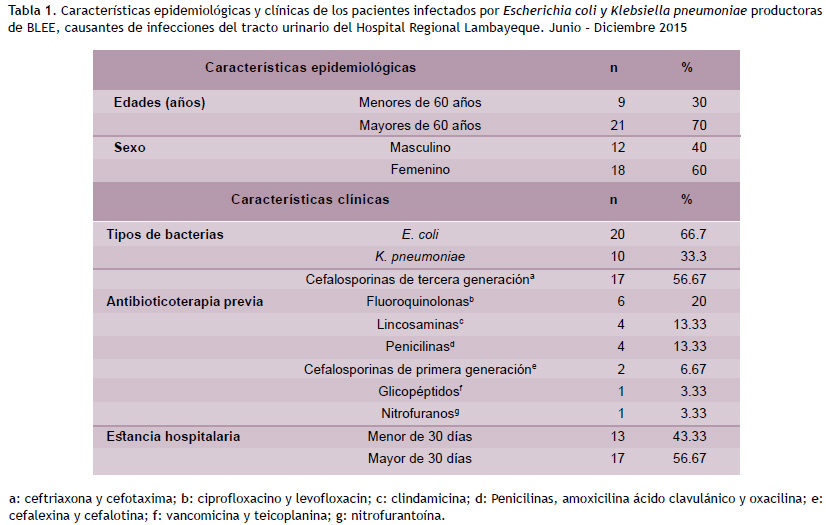
Los pacientes internados por periodos superiores a siete días en el servicio de emergencia presentaron más del 50 % de infecciones urinarias causadas por E. coli y K. pneumoniae productoras de BLEE. De estas, 17/30 presentaron estancia mayor a 30 días (Tabla 1).
Esto es similar a lo encontrado por Rubio et al. en un hospital de Madrid, quienes tras 2 años de investigación reportaron que los pacientes infectados por bacterias productoras de BLEE presentaban una estancia media de 47 días (1), lo que reafirma el hecho conocido de que cuanto más tiempo permanece un paciente hospitalizado, mayor es el riesgo de adquirir este tipo de microorganismos. El mismo autor destaca que el sexo femenino y una edad promedio de 71 años fueron características resaltantes en su población de estudio (1), lo que coincide con los datos obtenidos en el presente estudio, donde las bacterias productoras de BLEE causantes de ITU de los distintos servicios, se hallaron en mujeres con edades superiores a los 60 años.
El uso previo de cefalosporinas de tercera generación y fluoroquinolonas fueron las características más frecuentes halladas en los pacientes infectados por estas bacterias, lo cual coincide con lo reportado anteriormente por Cassier et al. (15). El uso de estos antimicrobianos debería considerarse de alto riesgo para la selección de este tipo de resistencia, haciendo urgente la búsqueda de nuevas alternativas terapéuticas. A este respecto en la actualidad se buscan nuevos principios activos, sin embargo el estudio de los mismos puede requerir décadas para su aplicación. Por ello lo más práctico es buscar drogas ya disponibles o combinaciones de estas, poco ensayadas, que puedan disminuir el impacto de este problema de manera más rápida (16,17).
El estudio de la relación clonal de microorganismos nos permite entre otras cosas, conocer el origen de los mismos, discriminar si su diseminación es vertical u horizontal, e identificar poblaciones. ERIC y REP – PCR son en la actualidad herramientas de tipificación empleadas en este tipo de estudios (18). De esta manera, tras emplear estas técnicas en los aislamientos de E. coli productores de BLEE, se identificaron 3 patrones predominantes P1, P2 y P3, mientras que en K. pneumoniae productores de BLEE, predominaron Q1 y Q2. Si bien los patrones tuvieron valores de similitud menores en relación a otros estudios (5), esto hallaría explicación en los eventos genéticos como mutaciones puntuales, inserciones o deleciones que ocurren de manera espontánea y al azar, y que pueden alterar el patrón de bandas. Así mismo, se demuestra que 5 clones tuvieron éxito en su diseminación y circularon en los servicios del HRL durante el periodo de estudio, siendo P1 y Q1 hallados exclusivamente en el servicio de emergencia, lo cual evidencia que este servicio presenta las condiciones apropiadas para albergar estos microorganismos.
En cuanto a la relación filogenética de los aislamientos, se pudo discriminar como genéticamente idénticos a dos ellos pertenecientes a E. coli productores de BLEE (E17 y E18), ubicados dentro del patrón P1. Al obtener aislados clasificados como genéticamente idénticos, se puede asumir que estos, sin duda, son clones (19). Ante esta evidencia, se pudo descubrir que un linaje de E. coli productor de BLEE se transmitía entre los pacientes internados en este servicio, y aunque se desconocen las vías, es muy probable que se dé por transmisión indirecta de un paciente a otro o por manos del personal sanitario. Sin embargo, son necesarios mayores estudios para determinar las vías de transmisión y el verdadero impacto de la diseminación clonal intrahospitalaria.
La confiabilidad de los datos y la reproducibilidad de las observaciones son cruciales para un sólido estudio de patrón de clonalidad. La información que cada herramienta de tipificación imparte al estudio, es considerada una medida útil para distinguir un aislamiento de otro. En los ensayos se obtuvo un poder de discriminación de 0.91 y 0.93 para las agrupaciones de E. coli y K. pneumoniae productores de BLEE respectivamente. Considerando que el número máximo de discriminación es 1.0, y comparados con el poder de discriminación de 0.98 de la PFGE presentado por un estudio previo (6), se demostró la aceptabilidad de los datos generados mediante ERIC-PCR y REP-PCR. Esto revela que las herramientas de tipificación fueron muy discriminativas, permitiendo reconocer a los aislamientos no relacionados como distintos.
Con el fin de demostrar la seguridad de las agrupaciones generadas en el dendrograma, se utilizó la prueba conocida como índice de correlación cofenético o Test de Mantel (20). Con ello, los coeficientes de correlación cofenético resultaron ser 0.95 y 0.97 para E. coli y K. pneumoniae productores de BLEE respectivamente. Esto se evidenció al compararse la matriz de similitud con la matriz cofenética, definiendo que hubo una adecuada representación de las similitudes genéticas por parte de los dendrogramas, es decir, no se generaron distorsiones en la gráfica, indicando que el algoritmo de agrupamiento UPGMA fue el correcto. Cabe resaltar que las herramientas de tipificación fueron altamente resolutivas, generando gran cantidad de bandas, esto a diferencia de los coeficientes de similitud obtenidos mediante el agrupamiento UPGMA los cuales fueron en su mayoría relativamente bajos.
En conclusión, ERIC-PCR y REP-PCR, revelaron la presencia de tres patrones clonales predominantes en E. coli, evidenciando mayor diseminación clonal en el servicio de emergencia durante el mes de octubre del 2015, y dos patrones clonales predominantes en K. pneumoniae, procedentes de los servicios de emergencia y medicina. Estas herramientas de tipificación son métodos alternativos al PFGE los cuales permiten realizar análisis de patógenos en ambientes hospitalarios.
Los resultados sugieren que el menor tránsito de pacientes, el número de camas y el tipo de terapia antimicrobiana empleada principalmente en el área de emergencia podrían estar relacionadas con el albergue y diseminación de este tipo de microorganimos. Sin embargo, se necesitan mayores estudios que indaguen las causas y vías de diseminación de estas bacterias, a fin de establecer medidas de contención que permitan, si bien no erradicar estos microorganismos, reducir su frecuencia.
REFERENCIAS BIBLIOGRÁFICAS
1. Rubio I, Martin E, Domingo D, López M, Larrañaga E. Extended spectrum betalactamase producing bacteria in a tertiary care hospital in Madrid: caracterlogy, risk factors and antimicrobial susceptibility patterns. Emerg Health Threats J. 2012;5(1):1–6. [ Links ] Disponible en: http://dx.doi.org/10.3402/ehtj.v5i0.11589
2. Yamile A, Celis B. Escherichia coli uropatogénica resistente a múltiples antibióticos: un problema de salud pública. Rev Fac Nac Salud Pública. 2012;30(1):74-77. [ Links ] Disponible en: http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S0120-386X2012000400020
3. Escalante J, Síme A, Díaz C. Características clínicas y epidemiológicas en pacientes con infección intrahospitalaria por bacterias productoras de betalactamasas de espectro extendido. Rev Peru Epidemiol. 2013;17(1):1–6. [ Links ] Disponible en: http://www.redalyc.org/articulo.oa?id=203128542008
4. Ramazanzadeh R, Zamani S, Zamani S. Genetic diversity in clinical isolates of Escherichia coli by enterobacterial repetitive intergenic consensus (ERIC)-PCR technique in Sanandaj hospitals. Iran J Microbiol. 2013;5(2):126–131. [ Links ] Disponible en: http://ijm.tums.ac.ir/index.php/ijm/article/view/622
5. Bailón H, Sacsaquispe R. Caracterización molecular de cepas de Klebsiella pneumoniae productoras de BLEE causantes de infección intrahospitalaria en el servicio de neonatología de un hospital de Lima, Perú. Rev Med Hered. 2013;24:101–108. [ Links ] Disponible en: http://www.scielo.org.pe/scielo.php?pid=S1018-130X2013000200002 &script=sci_arttext
6. Castro N, Alonso A, Silva J, Armenta A. Validación de dos variantes de la técnica rep-PCR para la tipificación molecular de aislados de Enterobacter cloacae productores de β– lactamasas de espectro extendido. Bioquimia. 2009;33(4):165-174. [ Links ] Disponible en: http://www.redalyc.org/articulo.oa?id=57612691002
7. Versalovic J, Koeuth T, Lupski J. Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes. Nucleic Acids Res. 1991;19(24):6823–6831. [ Links ] Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/ PMC329316/
8. Clinical and Laboratory Standards InstitutePerformance Standards for Antimicrobial Susceptibility Testing; Twenty - Fifth Informational Supplement. M100-S25. 2015; 35(3). [ Links ]
9. Comité de L’Antibiogramme de la Societe Francaise de Microbiologie. Les recommandations. CA-SFM. Soc Franç Microbiol. 2010.
10. Current Protocols in Molecular Biology. 2003. Disponible en: http://www.aun.edu.eg/molecular_biology/Proceeding_Dec2011/Current%20Protocols%20in%20Mol.%20Biol..pdf [ Links ]
11. Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratories. 2001;3. [ Links ]
12. Wan L, Wang Z, Yan Q, Wang X, Lei Y, Zuo L, et al. Genetic diversity of Escherichia coli isolated from commercial swine farms revealed by enterobacterial repetitive intergenic consensus PCR (ERIC-PCR) and repetitive extragenic palindrome PCR (REP-PCR). Afr J Biotechnol. 2011;10(51):10543–10550. [ Links ] Disponible en: https://www.ajol.info/index.php/ajb/article/view/96008
13. Tenover FC, Arbeit RD, Goering RV, Mickelsen PA, Murray BE, Persing DH, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis: Criteria for bacterial strain typing. J Clin Microbiol. 1995;33(9):2233-2239. [ Links ] Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC228385/
14. Li Y, Gong Z, Lu Y, Hu G, Cai R, Chen Z. Impact of nosocomial infections surveillance on nosocomial infection rates: A systematic review. Int J Surg. 2017;42:164-169. [ Links ] Disponible en: http://dx.doi.org/10.1016/j.ijsu.2017.04.065
15. Cassier P, Lallechére S, Aho S, Astruc K, Neuwirth C, Piroth L, et al. Cephalosporin and fluoroquinolone combinations are highly associated with CTX-M β-lactamase producing Escherichia coli: a case control study in a French teaching hospital. Clin Microbiol Infect. 2011; 17:1746-1751. [ Links ] Disponible en: http://onlinelibrary.wiley.com/doi/10.1111/j.1469-0691.2010.03349.x/full
16. Afridi FI, Farooqi BJ. Activity of beta-lactam beta-lactamase inhibitor combinations against extended spectrum Beta-lactamase producing Enterobacteriaceae in urinary isolates. J Coll Physicians Surg Pak. 2012; 22(6):358-362. [ Links ] Disponible en: https://www.ncbi.nlm.nih.gov/pubmed/22630093
17. Matthews PC, Barrett LK, Warren S, Stoesser N, Snelling M, Scarborough M, et al. Oral fosfomycin for treatment of urinary tract infection: a retrospective cohort study. BMC Infect Dis. 2016;16:566. [ Links ] Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5057270/
18. Mantilla JR, García I, Espinal PA, Valenzuela EM. Estandarización y evaluación de tres sistemas de rep-PCR para la tipificación de Klebsiella pneumoniae. Rev Col Cienc Quím Farm. 2004;33(1):48-58. [ Links ] Disponible en: http://revistas.unal.edu.co/index.php/rccquifa/article/view/1662
19. Rivas J, Redondo C, Alonso G. Genotyping of enterobacterias strains from 4 healthcare centers in Caracas. Act Cient de la Soc Venz de Bioanal Espec. 2006;9(2):3–7. [ Links ]
20. García S, Puigbo P. DendroUPGMA: A dendrogram construction utility. Biochemistry and Biotechnology Departament. 2010. Disponible en: http://genomes.urv.cat/UPGMA/index.php?entrada=Example3 [ Links ]
Fuentes de financiamiento: Este artículo ha sido financiado por los autores.
Conflictos de interés: Los autores declaran no tener ningún conflicto de interés.
Correspondencia:
Franklin Rómulo Aguilar Gamboa
Dirección: Av. Arequipa 940- José Olaya- Chiclayo
Teléfono: 971339765
Correo electrónico: faguilar@hrlamb.gob.pe
Recibido: 17 de enero de 2018
Evaluado: 19 de enero de 2018
Aprobado: 06 de marzo de 2018