Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Horizonte Médico (Lima)
Print version ISSN 1727-558X
Horiz. Med. vol.19 no.4 Lima Oct./Dic. 2019
http://dx.doi.org/10.24265/horizmed.2019.v19n4.03
ARTÍCULO ORIGINAL
Análisis de asociación entre polimorfismos (rs941798 y rs914458) del gen PTPN1 y diabetes tipo 2 en familias de Lima-Perú
Analysis of the association between PTPN1 gene polymorphisms (rs941798 and rs914458) and type 2 diabetes in families from Lima – Peru
Enrique Eduardo Sanchez-Castro 1 ORCID iD: http://orcid.org/0000-0002-2319-5192; Renato LaTorre-Ramírez 1 ORCID iD: http://orcid.org/0000-0001-8900-2085; Carlos Patricio Padilla Rojas 2 ORCID iD: http://orcid.org/0000-0002-0562-0431; Frank Lizaraso-Soto 3 ORCID iD: http://orcid.org/0000-0001-9993-9998; Jorge Calderón Ticona 4; José Solís Villanueva 4; Wilser Andrés García-Quispes 1ORCID iD: http://orcid.org/0000-0001-6628-2985; Mónica Yolanda Paredes Anaya*1 http://orcid.org/0000-0002-9038-2270
1. Universidad Nacional Mayor de San Marcos, Facultad de Ciencias Biológicas, Laboratorio de Genética de Enfermedades Metabólicas. Lima, Perú.
2. Instituto Nacional de Salud, Laboratorio de Biotecnología y Biología Molecular. Lima, Perú.
3. Universidad de San Martín de Porres, Facultad de Medicina. Lima, Perú.
4. Hospital Nacional Arzobispo Loayza, Servicio de Endocrinología. Lima, Perú.
* Autor corresponsal
RESUMEN
Objetivo: Analizar la asociación de los polimorfismos de un solo nucleótido (SNP) rs941798 y rs914458 del gen PTPN1 con la diabetes tipo 2 en familias peruanas de Lima.
Materiales y métodos: Veintitrés tríos familiares fueron captados en el Hospital Nacional Arzobispo Loayza. Se extrajeron muestras de sangre periférica para obtener el ADN y luego las frecuencias alélicas y genotípicas de los SNP. La genotipificación de los SNP se realizó mediante secuenciación. El análisis de asociación basado en familias entre los SNP y la diabetes tipo 2 se realizó con el programa FBAT.
Resultados: Se observaron los 3 genotipos posibles para cada SNP, rs941798 (A>G) y rs914458 (G>C). Las pruebas de asociación basada en familias a nivel alélico mostraron al alelo A del SNP rs941798 asociado a la diabetes tipo 2 (p = 0.023) bajo uno de los modelos evaluados; no obstante, tras la corrección de Bonferroni para comparaciones múltiples, esta asociación se perdió. No se evidenció asociación entre los SNP y la enfermedad en ningún nivel (alélico, genotípico o haplotípico).
Conclusiones: No se encontraron evidencias de asociación significativa entre los SNP rs941798 y rs914458 del gen PTPN1 con la DT2 en familias peruanas de Lima, en ninguno de los niveles estudiados (alélico, genotípico y haplotípico).
Palabras clave: Diabetes mellitus tipo 2; Proteína tirosina fosfatasa no receptora tipo 1; Marcadores genéticos; Polimorfismo de nucleótido simple (Fuente: DeCS BIREME).
ABSTRACT
Objective: To analyze the association that PTPN1 gene single nucleotide polymorphisms (SNPs) rs941798 and rs914458 have with type 2 diabetes in Peruvian families from Lima.
Materials and methods: Twenty-three (23) families consisting of three members each were recruited at the Hospital Nacional Arzobispo Loayza. Peripheral blood samples were collected to obtain the DNA, and then the allele and genotype frequencies of the SNPs. SNP genotyping was performed using the sequencing method. The family-based analysis of the association between SNPs and type 2 diabetes was conducted using the family-based association test (FBAT) program.
Results: Three (3) possible genotypes were observed for each SNP, i.e. rs941798 (A>G) and rs914458 (G>C). In one of the assessed models, the family-based association tests showed at the allele level that allele A of SNP rs941798 is associated with type 2 diabetes (p = 0.023). However, after using the Bonferroni correction for multiple comparisons, this association was lost. No association was demonstrated between the SNPs and the disease at any level (allele, genotype or haplotype).
Conclusions: No evidence of significant association was found between PTPN1 gene SNPs rs941798 and rs914458 and type 2 diabetes at the studied levels (allele, genotype or haplotype) in Peruvian families from Lima.
Keywords: Diabetes mellitus, type 2; Protein tyrosine phosphatase, non-receptor type 1; Genetic markers; Polymorphism, single nucleotide (Source: MeSH NLM).
INTRODUCCIÓN
La diabetes, con la diabetes de tipo 2 (DT2) como manifestación más frecuente (90 % de los casos), se ha convertido en un importante problema de salud a nivel mundial, y es considerada una pandemia (1).
En Perú, la diabetes es la tercera principal causa específica de mortalidad (2). Asimismo, resulta ser la sexta causa de ceguera y la primera causa, tanto de enfermedad renal crónica como de amputaciones no traumáticas de miembros inferiores (3). Si bien la investigación global sobre la genética de la diabetes ha tenido un progreso significativo en lo que va del siglo XXI, y se han podido identificar varios loci asociados a la enfermedad (4), los trabajos sobre este tema en el Perú son todavía escasos. Si consideramos que, aproximadamente, 50 % de las personas que padecen de DT2 no han sido diagnosticadas (5) y que este hecho incrementa el riesgo de sufrir de comorbilidades (6,7), se entiende la imperativa necesidad de encontrar métodos diagnósticos más efectivos y específicos para cada población.
Varios miembros de la familia de proteínas tirosina fosfatasas (PTP) clase I están involucrados en la regulación del metabolismo (8). En particular, la proteína tirosina fosfatasa 1B (PTP1B), codificada por el gen PTPN1, se ha considerado como un objetivo terapéutico ideal para la DT2, ya que regula, directamente, los receptores de insulina (9,10). En cuanto a la asociación genética de PTPN1 con la DT2, polimorfismos ubicados en este gen (cromosoma 20q13) han sido asociados con la enfermedad, aunque no para todas las poblaciones evaluadas (11). La varianza entre poblaciones es una constante en cuanto a la asociación de marcadores moleculares, debido a que la genética que caracteriza a las poblaciones de una determinada región afecta en gran medida las asociaciones genéticas, por lo que la validación de cualquier marcador genético de una enfermedad para cada población en particular es imperativa para su aprovechamiento práctico. Cabe resaltar, de todos modos, que se ha evidenciado incluso una variante (1484insG o rs16989673) en PTPN1 con implicancia funcional (12), así como 20 polimorfismos de un solo nucleótido (SNP) asociados a la sensibilidad a la insulina y al valor de la glucosa en ayunas en individuos hispano-estadounidenses (13).
Los SNP estudiados en esta investigación fueron el rs941798 (A>G, intrón 1) y el rs914458 (G>C, a 10 kb en el extremo 3’ de PTPN1), que han mostrado estar asociados con la DT2 y con otros fenotipos metabólicos relacionados a diabetes en otras poblaciones (14,15).
El objetivo de nuestro estudio es analizar la asociación de variantes genéticas (SNP rs941798 y rs914458) del gen PTPN1 con la DT2 en familias peruanas de Lima.
MATERIALES Y MÉTODOS
Diseño y población de estudio
Este estudio incluyó 23 familias nucleares (tríos familiares padres-hijo) procedentes de Lima, Perú. La captación de familias se llevó a cabo en el Servicio de Endocrinología del Hospital Nacional Arzobispo Loayza, donde todos los probandos fueron previamente diagnosticados con DT2. A partir de cada probando (hijo con DT2) se captaron las familias nucleares (que incluyen a los progenitores de cada probando, quienes podían tener o no el diagnóstico de DT2).
Variables y mediciones
Las variables de este estudio son las frecuencias alélicas y genotípicas de los SNP rs941798 y rs914458. Para obtenerlas se extrajo ADN desde sangre total y se genotipificó los SNP mediante secuenciación.
Extracción de ADN
Se colectaron muestras de sangre en tubos vacutainer con EDTA como anticoagulante. El ADN genómico fue extraído utilizando el kit de extracción PureLink™ Genomic DNA Mini (Invitrogen™), según las recomendaciones del fabricante. Las muestras de ADN genómico obtenidas fueron almacenadas a -20 °C.
Genotipificación
Se diseñaron los cebadores 5’TTCCTCCCATTCACACTCC- AAA3’ (forward), 5’GTTGACATGGTGACAGGAGGCC3’ (reverse) y 5’CAGCTGGAAATAGGTGGGTTCC3’ (forward), 5’GGAAGA-CTCGTCAGACACCTCG3’ (reverse), para la amplificación de fragmentos de 242 pb y 234 pb de PTPN1, que contenían los SNP rs941798 y rs914458, respectivamente. Para ambos marcadores la amplificación se realizó en un volumen de reacción de 20 µL que contenía buffer PCR 1X, 200 µmol de cada dNTP, 1,5 mM de MgCl2, 0,25 U de Applied Biosystems™ AmpliTaq Gold™, 0,5 µL de cada cebador y 40 ng de ADN genómico. Las condiciones de amplificación para ambos SNP fueron 95 °C por 2 min de denaturación inicial, seguido de 40 ciclos de 95 °C por 30 s, 63,2 ºC (para el rs941798) y 64 ºC (para el rs914458) por 30 s de hibridización y 72 °C por 10 s; y, finalmente, con 72 °C por 10 minutos de extensión. Los fragmentos amplificados se visualizaron en geles de agarosa al 1,5 % teñidos con bromuro de etidio. Luego, estos fueron purificados con el kit High Pure PCR Cleanup Micro. La genotipificación se realizó con el kit ABI PRISM® SNaPshot™ Multiplex, según las recomendaciones del fabricante, y con los cebadores 5’-CCATTCACACTCCAAATTGTGCACCAAAGT-3’ (forward),5’-TGGAGAACCATCCTCACAAAACTAGGATGA-3’(reverse)y5’-GACTG ACTCTGTGTCTTCATGCCATCGGACGGAGGAG-3’(forward), 5’-AGGGCGGTGACTCGCACTGAAGCTGAGAAA-3’ (reverse) para SNP rs941798 y rs914458, respectivamente.
Análisis estadístico
Para cada SNP se calcularon las frecuencias genotípicas. Mediante la prueba exacta del equilibrio Hardy-Weinberg (EHW) se evaluó si los SNP estaban en equilibrio para las muestras parentales. La frecuencia de los haplotipos se estimó mediante el programa HAPLO (16), que implementa el método esperanza-maximización (EM).
La asociación entre los marcadores y la enfermedad se analizó con la prueba de asociación basada en familias (FBAT) (17,18). El análisis FBAT, bajo los modelos aditivo y dominante en el programa FBAT (19), se aplicó bajo dos hipótesis principales (H : sin asociación, sin ligamiento y H0: sin asociación, con ligamiento), a niveles de alelos, genotipos y haplotipos. Siempre se asumió una prevalencia del 6,3 % por la enfermedad en la población. Inicialmente, se consideró significativo a p < 0,05; no obstante, se aplicó la corrección de Bonferroni para comparaciones múltiples mediante la fórmula alfa = 0,05/N, donde N es el número hipótesis totales, y se tomó el nivel de significancia corregido como p < 0,0125.
Consideraciones éticas
Todos los participantes firmaron el consentimiento informado aprobado para esta investigación por el Comité de Ética del Hospital Nacional Arzobispo Loayza (N°134-CEI-HNAL-2008).
RESULTADOS
La muestra consistió en 23 tríos familiares. La proporción de sexos hombre/mujer es de los probandos 0,70/0,30. La edad (años), descrita con la media y la desviación estándar (DE), fue de 43,3 (DE 8,6) en los probandos, y de 69,8 (DE 10,0) en los progenitores.
Se observaron los 3 genotipos posibles para cada SNP, rs941798 (A>G) y rs914458 (G>C), las frecuencias del alelo menos común para el total de individuos estudiados fue 0,493 y 0,225, respectivamente.
Con respecto al EHW, las frecuencias genotípicas del SNP rs941798 mostraron una desviación significativa (P=0,01). Por el contrario, para el SNP rs914458 las frecuencias genotípicas se encontraron en EHW (P=0,186). El patrón de herencia mendeliano se cumplió en el 100 % de la muestra.
Prueba de asociación basada en familias
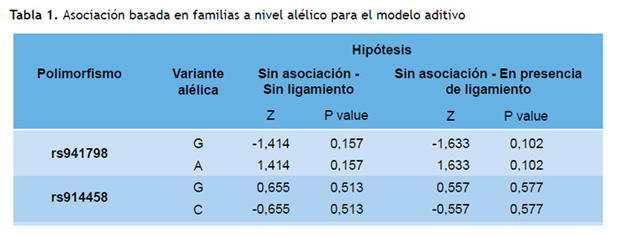
Con respecto a los alelos, se realizaron las pruebas de asociación a nivel alélico mediante la FBAT para ambos SNP, bajo las 2 hipótesis nulas (sin asociación – sin ligamiento y sin asociación – en presencia de ligamiento) para el modelo aditivo (Tabla 1). No se observó ninguna asociación significativa entre ninguno de los SNP y la DT2 en esta prueba.
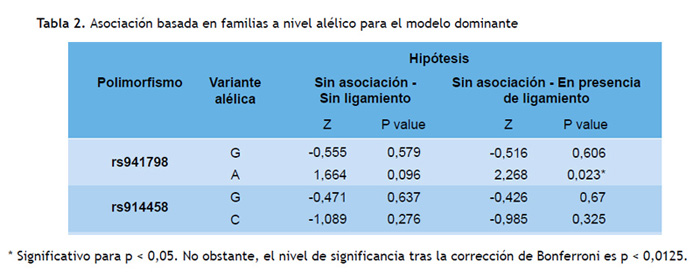
De igual manera, se realizaron las pruebas de asociación a nivel alélico mediante la FBAT para ambos SNP, bajo las 2 hipótesis nulas (sin asociación – sin ligamiento y sin asociación – en presencia de ligamiento) para el modelo dominante (Tabla 2). Se observó una asociación significativa entre el SNP rs941798 y la DT2 para el alelo A (se considera la hipótesis nula "no asociación" – "en presencia de ligamiento") en el modelo dominante (p = 0,023), (Tabla 2); no obstante, esta se perdió tras aplicar la corrección de Bonferroni. No se observó ninguna asociación significativa entre el SNP rs914458 y la DT2.
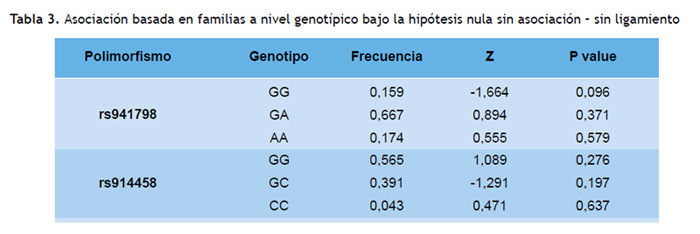
Con respecto a los genotipos, se realizaron las pruebas de asociación a nivel genotípico mediante la FBAT para ambos SNP, bajo la hipótesis nula "sin asociación – sin ligamiento", sin observarse ninguna asociación significativa entre ninguno de los SNP y la DT2 (Tabla 3).
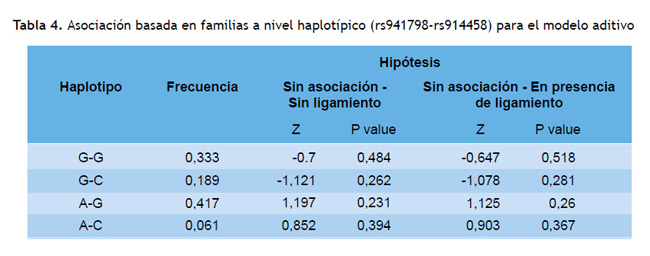
El análisis de haplotipos se realizó bajo las 2 hipótesis nulas (sin asociación – sin ligamiento y sin asociación – en presencia de ligamiento) para el modelo aditivo (Tabla 4). No se observó ninguna asociación significativa entre ninguno de los haplotipos y la DT2.
DISCUSIÓN
Estudios alrededor del mundo, y las réplicas de estos, evidencian la asociación que existe entre diferentes variantes genéticas y DT2; sin embargo, solo una pequeña parte de la heredabilidad total de la DT2 se ha podido explicar mediante estas investigaciones (20).
Si bien los estudios de asociación genética pueden incluir cualquier variante genética, se recomienda que se utilicen las que tienen potencial connotación funcional (21). Según esta recomendación, se decidió estudiar variantes en PTPN1.
Las frecuencias alélicas y la estructura genética de cada población pueden influir en las asociaciones genéticas, por lo que es necesario corroborar las encontradas en una determinada población, con el estudio del marcador en otras poblaciones.
Este es el caso de varios SNP en PTPN1, para los que se han observado polimorfismos asociados con la enfermedad, aunque no para todas las poblaciones evaluadas (11). Precisamente, una de las ventajas de realizar un estudio de asociación basado en familias es la reducción de los efectos dados por factores de confusión, así como por la estratificación poblacional (16,17).
Con respecto al SNP rs941798, el alelo A fue el único que evidenció una asociación significativa (P = 0,23) para el modelo dominante bajo la hipótesis "sin asociación - en presencia de ligamiento" (tabla 2). Si bien, luego de la corrección del nivel de significación estadística, no podemos afirmar la existencia de asociación entre este SNP y la DT2 (P > 0,0125), existen estudios anteriores realizados en hispano- estadounidenses (13) y caucásicos estadounidenses (15) que lo encuentran asociado con esta enfermedad. Por otro lado, para la población francesa (14) no se evidenció asociación entre el SNP rs941798 y la DT2, pero sí con rasgos asociados a la DT2. Además, en una investigación en familias escandinavas (22), tampoco se observó ninguna asociación significativa entre el SNP rs941798 a nivel genotípico (Tabla 3) y la DT2, lo que coincide con nuestros resultados.
Con respecto al SNP rs914458, no se observó ninguna asociación significativa con la DT2 ni a nivel alélico (Tablas 1 y 2) ni a nivel genotípico (Tabla 3), lo que ya había sido reportado para la población peruana, aunque en un estudio poblacional (23). Esto refuerza el hallazgo de que el SNP rs914458 y la DT2 en la población peruana de Lima no evidenciarían asociación. De manera similar, con respecto al análisis de haplotipos, no se observó ninguna asociación significativa entre ninguno de los haplotipos con DT2 (Tabla 4).
Si bien el cromosoma 20 ha sido asociado con la DT2 hace ya 20 años (24) y PTPN1 (20q13) ha mostrado una repercusión funcional en el desarrollo de DT2 (12), existen reportes en los que la gran mayoría de SNP en este gen no muestra evidencias de asociación con DT2 (25,26). Los resultados de este trabajo han coincidido con dichos reportes.
Finalmente, el presente estudio no encontró evidencias de asociación significativa entre los SNP rs941798 y rs914458 del gen PTPN1 con la DT2 en familias peruanas de Lima, en ninguno de los niveles estudiados (alélico, genotípico y haplotípico).
Fuentes de financiamiento: Este artículo ha sido financiado por INNOVATE-PERÚ [Contract #401-PNICP-PIAP-2014] y la Universidad Nacional Mayor de San Marcos.
Conflicto de interés: Los autores declaran no tener ningún conflicto de interés.
REFERENCIAS BIBLIOGRÁFICAS
1. International Diabetes Federation. IDF Diabetes Atlas. 8th edition. Brussels: International Diabetes Federation; 2017. [ Links ]
2. Ministerio de Salud del Perú. Análisis de las causas de mortalidad en el Perú, 1986-2015. 1ra edición. Lima: Ministerio de Salud del Perú; 2018. 226 p. [ Links ]
3. Villena JE. Epidemiología de la diabetes mellitus en el Perú. Diagnóstico. 2016; 55(4): 173-81. [ Links ]
4. Kwak SH, Park KS. Recent progress in genetic and epigenetic research on type 2 diabetes. Exp Mol Med. 2016; 48(3): e220. [ Links ]
5. Beagley J, Guariguata L, Weil C, Motala AA. Global estimates of undiagnosed diabetes in adults. Diabetes Res Clin Pract. 2014; 103(2): 150-60. [ Links ]
6. Kalyani RR, Saudek CD, Brancati FL, Selvin E. Association of diabetes, comorbidities, and A1C with functional disability in older adults: results from the National Health and Nutrition Examination Survey (NHANES), 1999–2006. Diabetes care. 2010; 33(5): 1055-60. [ Links ]
7. American Diabetes Association. Standards of medical care in diabetes-2016 abridged for primary care providers. Clin Diabetes. 2016; 34(1): 3-21. [ Links ]
8. Gurzov EN, Stanley WJ, Brodnicki TC, Thomas HE. Protein tyrosine phosphatases: molecular switches in metabolism and diabetes. Trends Endocrinol Metab. 2015; 26(1): 30-9. [ Links ]
9. Feldhammer M, Uetani N, Miranda-Saavedra D, Tremblay ML. PTP1B: a simple enzyme for a complex world. Crit Rev Biochem Mol Biol. 2013; 48(5): 430-45. [ Links ]
10. Sun J, Qu C, Wang Y, Huang H, Zhang M, Li H, et al. PTP1B, a potential target of type 2 Diabetes Mellitus. Mol Biol. 2016; 5(4): 174. [ Links ]
11. Tsou RC, Bence KK. The Genetics of PTPN1 and Obesity: insights from mouse models of tissue-specific PTP1B deficiency. J Obes. 2012; 2012: 1-8. [ Links ]
12. Di Paola R, Frittitta L, Miscio G, Bozzali M, Baratta R, Centra M, et al. A variation in 3′ UTR of hPTP1B increases specific gene expression and associates with insulin resistance. Am J Hum Genet. 2002; 70(3): 806-12.
13. Palmer ND, Bento JL, Mychaleckyj JC, Langefeld CD, Campbell JK, Norris JM, et al. Association of protein tyrosine phosphatase 1B gene polymorphisms with measures of glucose homeostasis in Hispanic Americans: the insulin resistance atherosclerosis study (IRAS) family study. Diabetes. 2004; 53(11): 3013-9. [ Links ]
14. Cheyssac C, Lecoeur C, Dechaume A, Bibi A, Charpentier G, Balkau B, et al. Analysis of common PTPN1 gene variants in type 2 diabetes, obesity and associated phenotypes in the French population. BMC Med Genet. 2006; 7(1): 44. [ Links ]
15. Bento JL, Palmer ND, Mychaleckyj JC, Lange LA, Langefeld CD, Rich SS, et al. Association of protein tyrosine phosphatase 1B gene polymorphisms with type 2 diabetes. Diabetes. 2004; 53(11): 3007-12. [ Links ]
16. Hawley M, Kidd KK. HAPLO: a program using the EM algorithm to estimate the frequencies of multi-site haplotypes. J Hered. 1995; 86(5): 409-11. [ Links ]
17. Rabinowitz D, Laird NM. A unified approach to adjusting association tests for population admixture with arbitrary pedigree structure and arbitrary missing marker information. Hum Hered. 2000; 50(4): 211-23. [ Links ]
18. Laird NM, Horvath S, Xu X. Implementing a unified approach to family-based tests of association. Genet Epidemiol. 2000; 19(Suppl.1): 36-42. [ Links ]
19. Horvath S, Xu X, Laird NM. The family based association test method: strategies for studying general genotype-phenotype associations. Eur J Hum Genet. 2001; 9(4): 301-6. [ Links ]
20. Prasad RB, Groop L. Genetics of type 2 diabetes-pitfalls and possibilities. Genes (Basel). 2015; 6(1): 87-123. [ Links ]
21. Vassy JL, Hivert MF, Porneala B, Dauriz M, Florez JC, Dupuis J, et al. Polygenic type 2 diabetes prediction at the limit of common variant detection. Diabetes. 2014; 63(6): 2172-82. [ Links ]
22. Florez JC, Agapakis CM, Burtt NP, Sun M, Almgren P, Råstam L, et al. Association testing of the protein tyrosine phosphatase 1B gene (PTPN1) with type 2 diabetes in 7,883 people. Diabetes. 2005; 54(6): 1884-91. [ Links ]
23. Paredes Anaya M, García-Quispes WA, Lizaraso Soto F, Padilla Rojas C, Torres-Gonzales D, Calderón Ticona J, et al. Análisis de asociación genética entre el SNP RS914458 del gen proteína tirosina fosfatasa no receptor tipo 1 (PTPN1) y diabetes tipo 2 en población peruana. Horiz Med. 2014; 14(4): 31-6. [ Links ]
24. Ghosh S, Watanabe RM, Hauser ER, Valle T, Magnuson VL, Erdos MR, et al. Type 2 diabetes: evidence for linkage on chromosome 20 in 716 finnish affected sib pairs. Proc Natl Acad Sci USA. 1999; 96(5): 2198–203. [ Links ]
25. Traurig M, Hanson RL, Kobes S, Bogardus C, Baier LJ. Protein tyrosine phosphatase 1B is not a major susceptibility gene for type 2 diabetes mellitus or obesity among Pima Indians. Diabetología. 2007; 50(5): 985–9. [ Links ]
26. Bodhini D, Radha V, Ghosh S, Majumder PP, Mohan V. Lack of association of PTPN1 gene polymorphisms with type 2 diabetes in South Indians. J Genet. 2011; 90(2): 323–6. [ Links ]
Correspondencia: Mónica Yolanda Paredes Anaya
Dirección: Laboratorio de Genética de Enfermedades Metabólicas (115). Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos. Calle Germán Amézaga 375, Lima 15081, Perú.
Teléfono: +51-1-6197000 (ext. 1502)
Correo electrónico: mparedesa1@unmsm.edu.pe
Recibido: 03 de mayo de 2019.
Evaluado: 05 de julio de 2019.
Aprobado: 05 de agosto de 2019.