INTRODUCCIÓN
Los persistentes o células persistentes bacterianas son variantes transitorias de una población genéticamente homogénea, que pueden sobrevivir a la exposición de estrés, como el tratamiento antibiótico, y generan latencia celular 1. En contraste con la resistencia causada por mutaciones genéticas, la tolerancia a los antibióticos por los persistentes se produce por la varianza fenotípica del tipo salvaje.
La reinoculación (crecimiento) de células persistentes regenera la población sensible original (2,3, por lo que los persistentes son considerados variantes fenotípicas pero no mutantes 4 o productos de un fenómeno epigenético no heredado 5-8 (Figura 1). Este fenotipo fue descrito por primera vez en 1942 por Hobby et al. cuando encontraron que el 1 % de Staphylococcus aureus sobrevivió al tratamiento con penicilina 9. En 1944, Joseph Bigger observó los mismos resultados en infecciones estafilocócicas tratadas con penicilina y acuñó el término "persistentes" para denominar a estas células tolerantes 10-12. Chain y Duthie, en 1945, aseguraron que la penicilina no mataba por completo a Staphylococcus spp. hasta que se usaron tratamientos más largos, y que las células de fase estacionaria (es decir, células que no proliferaban) eran casi completamente insensibles a la penicilina 13. En los años siguientes, el tema de persistencia fue rápidamente olvidado y se tomó mayor interés a la relación entre resistencia bacteriana y el descubrimiento de nuevos antibióticos (era dorada, desde 1940 a 1960). Las observaciones de persistentes fueron ignoradas debido a la dificultad de aislarlos, su naturaleza transitoria, su baja cuantía y similitud con el tipo de célula viable pero no cultivable 14. Sin embargo, con el descubrimiento y selección de mutantes de alta persistencia a la ampicilina en Escherichia coli realizado por Harris Moyed en 1980, y con ayuda de estudios moleculares, se llegó a comprender mejor este fenómeno que, desde entonces, se ha observado en muchas especies bacterianas 1,15,16.
Una población bacteriana inicialmente está formada por células sensibles a los antibióticos (color rojo) las cuales pueden ser erradicadas después de la terapia antibiótica y solo las que adquirieron resistencia volverían a crecer, de modo que la nueva población estaría formada únicamente por células resistentes (color negro) como resultado de la selección de subpoblaciones que han adquirido genes de resistencia o que ya los poseían. La misma población bacteriana puede contener también células persistentes (color azul) originadas después del tratamiento antibiótico por un cambio de fenotipo sensible a fenotipo tolerante y que, al volver a crecer sin ninguna exposición al estrés, exhibirían el mismo patrón de sensibilidad que la población original (Figura 1).
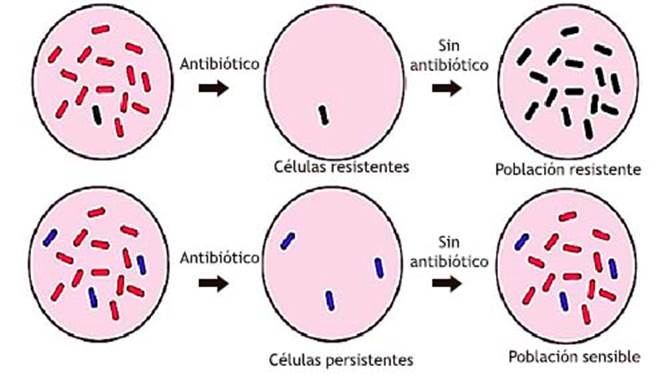
Figura 1 Comparación entre resistencia y persistencia. Modificado a partir del artículo Role of persister cells in chronic infections: Clinical relevance and perspectives on anti-persister therapies
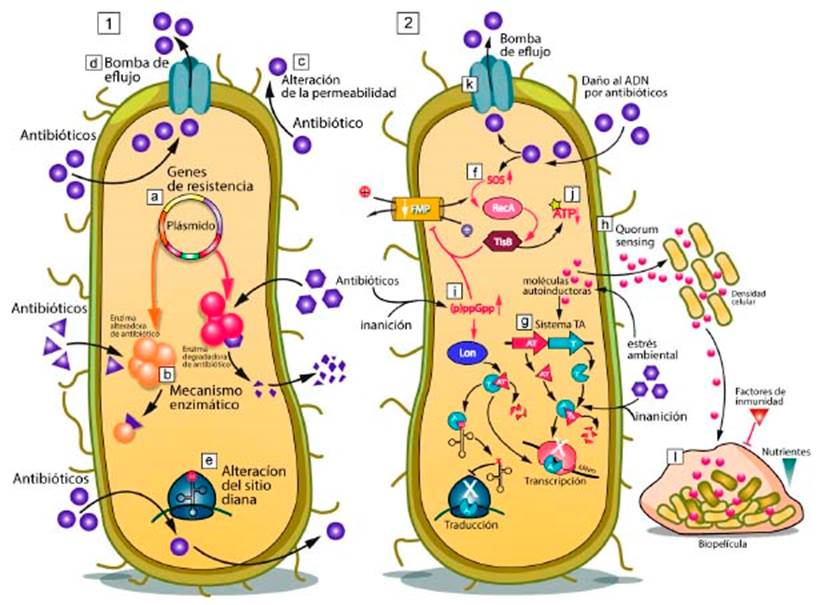
La presencia de persistentes puede ser causa importante de infecciones bacterianas crónicas 17, lo que da lugar a la recalcitrancia, que se ha relacionado con un aumento en el riesgo de aparición de resistencia a los antibióticos durante el tratamiento 3. Claramente, la persistencia debe ser una causa de preocupación global, ya que agrava la perspectiva de la actual crisis antibiótica, que se estima provocará hasta 10 millones de víctimas al año para el 2050 18,19. Los estudios de persistencia bacteriana tienen similitudes en términos de mecanismos, tratamiento, tanto en animales, parásitos, hongos, virus, células tumorales, por lo que los nuevos fármacos y vacunas son críticos para mejorar el tratamiento 20. A pesar de su descubrimiento hace más de 70 años, los mecanismos de persistencia bacteriana son complejos y poco conocidos. Estudios recientes han identificado una serie de genes y vías que intervienen en la formación de persistentes (Figura 2). Estas incluyen módulos toxina-antitoxina (TA), respuesta estricta por (p) ppGpp, reparación o protección del ADN (respuesta SOS), metabolismo reducido del fosfato, estrés celular, limitación de nutrientes, producción de energía alternativa, inhibición en la síntesis de proteínas y ácidos nucleicos, degradación de proteínas, transportadores/sistemas de eflujo, reguladores transcripcionales. Tensiones más generales, como pH extremo, choque térmico, radiación, defensa antioxidante, fagos, las biopelículas y macrófagos, también se han asociado a la formación de persistentes (3,4,11,20-26.
La resistencia bacteriana implica una serie de cambios genéticos heredados (genes de resistencia, plásmidos) mecanismos enzimáticos (enzima alteradoras, degradadoras), cambios estructurales y funcionales (impermeabilidad, bombas de eflujo), alteraciones de sitio diana, que hacen ineficiente la terapia antibiótica. En contraste, la persistencia bacteriana implica una variación fenotípica no heredada, que se presenta en genes y mecanismos (respuesta SOS, sistemas TA, detección de quorum, respuesta estricta (p) ppGpp, metabolismo reducido del ATP, bombas de eflujo, formación de biofilms) que se activan en respuesta a una gran variedad de tensiones ambientales de estrés y permiten su adaptación y supervivencia (Figura 2).
¿Qué es persistencia bacteriana?
La persistencia bacteriana es un conjunto grande y heterogéneo de fenómenos fisiológicos que ocurre en una subpoblación pequeña de células bacterianas salvajes llamadas células persistentes o persistentes bacterianos 27-30. Estas células se forman en respuesta a una gran variedad de tensiones ambientales de estrés (como la limitación de nutrientes o exposición al daño por antibióticos), se adaptan con éxito al cambio ambiental 3, se vuelven tolerantes y expresan un rango epigenético no heredado (inmutable) a su progenie 6, rasgo que las diferencia de la resistencia antimicrobiana, en la que sí existe una verdadera mutación (Figura 2). La aparición de estas células es espontánea y ocurre en alguna parte del ciclo celular como variantes fenotípicas raras de tipo salvaje 31, fenotipo de resistencia, subpoblación tolerante 2,7,29,32 o denominadas por algunos autores como células multirresistentes a los medicamentos (33-34, que pueden tener una morfología indistinguible a las células susceptibles a los antibióticos (fenotipo salvaje) 27. Estas células se caracterizan por tener un estado metabólico latente o quiescente (metabolismo reducido), con funciones deprimidas o carecer de ellas (transcripción, traducción, bajo niveles de producción de energía ATP y fuerza motriz protónica (Figura 2) 28,33,35-37.
La latencia es una característica primordial en los persistentes, y se define como el estado que implica el cierre metabólico reversible (metabolismo inactivo) que permite a las células escapar de la acción letal de los antibióticos 37, donde muchos sitios diana están inactivos, lo que explica la ineficacia de estos últimos. Sin embargo, el concepto de que los persistentes son bacterias latentes no replicantes suele generar conflictos y es tema aún de debate, y ha sido cuestionado
en los últimos años por varios estudios (23 que definen persistencia como un fenómeno mucho más complejo en términos de latencia 31, donde en lugar de una aparente falta de crecimiento (metabolismo inactivo) en persistentes, estos sí presentarían actividad metabólica activa, que se manifiesta en la sobreexpresión y aumento en la actividad de las bombas de eflujo para las drogas (Figura 2) 20,38,39, capacidad de replicación y una aparente estabilidad de su población como resultado de un equilibrio dinámico entre muerte y replicación bacteriana, lo que hace mucho más compleja la definición de persistencia que se asocia a múltiples fenómenos metabólicos adicionales, lo que la aleja de ser un simple sinónimo de latencia 23,25.
Modelos de clasificación
Los persistentes bacterianos pueden clasificarse en dos grupos, tipo I y tipo II. Los persistentes de tipo I se forman después de la fase estacionaria, en respuesta a desencadenantes externos como la falta de nutrientes, y no pueden volver al fenotipo normal sensible; tienen una frecuencia de 1 % en biopelículas bacterianas y cultivos de fase estacionaria 28. Los persistentes de tipo II son de crecimiento lento, se forman durante la fase exponencial (con una frecuencia de 0,0001 a 0,001 %) 28 y se cree que derivan de fluctuaciones aleatorias en el ruido transcripcional y traslacional por un cambio de fenotipo sin intervención de desencadenantes externos y pueden retomar el fenotipo normal 11,21. Aunque los tipos I y II resultan útiles para la clasificación, vale la pena señalar que este fenotipo es mucho más heterogéneo. Los persistentes pueden adoptar diferentes tamaños y formas, desde una morfología regular o alterada (cocoide, granular, filamentosa) hasta morfologías atípicas, pleomórficas o deficientes de pared celular como las formas L 40,41 generadas como parte del ciclo de vida de las bacterias estresadas y que han sido implicadas en infecciones persistentes por biofilms20.
Mecanismos implicados en la formación de persistentes
Persistencia bacteriana mediante módulos toxina- antitoxina (TA)
Los módulos toxina-antitoxina (TA) son los principales modelos de persistencia bacteriana estudiados, sobre todo en bacterias gramnegativas 17, se conoce muy poco sobre la formación de persistentes mediante este modelo en grampositivos. Su función en las bacterias no se conoce con precisión, se ha sugerido que el sistema activa la muerte celular programada en respuesta al estrés, inanición, daño al ADN y exposición a los antibióticos. Por otro lado, se plantea que actúa como inhibidor de la síntesis de macromoléculas esenciales lo que genera latencia celular reversible al crecimiento 9,30. Los módulos TA están muy sobreexpresados en las células persistentes en comparación con el resto de la población 8. Por ejemplo, Escherichia coli K-12 contiene al menos 36 módulos TA 42 y el genoma de Mycobacterium tuberculosis contiene 88 módulos 43, más que cualquier otro patógeno humano 44.
Típicamente, están constituidos por dos genes expresados como un operón, codificados por genomas y plásmidos bacterianos 7,16; uno de los genes codifica una toxina estable (siempre una proteína) que interrumpe un proceso celular esencial, como la replicación, transcripción, traducción; y el otro gen codifica una antitoxina lábil (ya sea ARN o una proteína inestable), que neutraliza la toxina y también actúa como un autorregulador de su expresión (Figura 2) 12.
Estos sistemas se han encontrado en bacterias como E. coli que posee el gen HipA (un factor de persistencia crítico) que detiene la síntesis de ADN, ARN y proteínas, lo que inhibe el crecimiento en un 90 % de las células 28 y que es normalmente neutralizado por el gen HipB, un represor de la transcripción del gen HipA 15,45,46. Recientemente, se descubrió que bacterias como Bartonella schoenbuchensis, Yersinia enterocolitica y Pseudomonas aeruginosa producen toxinas cíclicas de AMP(Fic) que bloquean la actividad de la topoisomerasa IV y la ADN- girasa (que regulan el anudamiento, filamentación y concatenación del cromosoma bacteriano), y logran detener el crecimiento 47. También se encontró que los módulos toxina-antitoxina tipo II estaban regulados positivamente durante la infección de macrófagos por Salmonella typhimurium, y que la eliminación de estos conducía a una disminución significativa en los niveles de formación de persistencia inducida por el estrés oxidativo dentro de los macrófagos 48.
Respuesta SOS y formación de persistentes
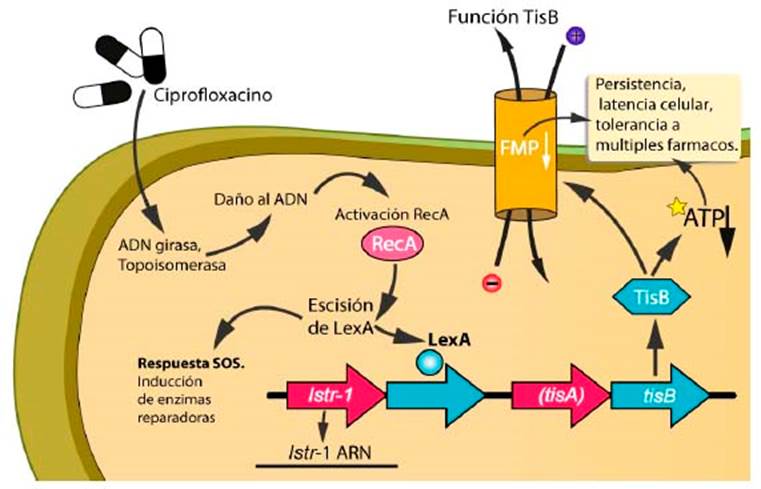
La respuesta SOS se desencadena por daño en el ADN y permite su reparación. Esta respuesta se puede considerar como un mecanismo importante de supervivencia bacteriana bajo condiciones de estrés. La respuesta SOS involucra genes que afectan los procesos celulares, como la recombinación y reparación del ADN, y también la patogénesis, la resistencia a los antimicrobianos y la producción de biopelículas 32. Las proteínas que componen el sistema SOS incluyen un represor transcripcional llamado LexA y una proteína activadora de unión al ADN, RecA (Figura 3). Sin embargo, otras proteínas pueden estar involucradas. El sistema SOS contribuye a la reparación del ADN pero también induce el desarrollo de módulos TA 49. Una de las principales toxinas responsable de la formación de persistentes en respuesta SOS, es la toxina TisB, un péptido hidrofóbico de 29 aminoácidos que se une a la membrana celular, interrumpe la fuerza motriz protónica, conduce a la disminución de los niveles de ATP y, como consecuencia, la inactivación celular (Figuras 2 y 3). Esta toxina está presente en el módulo TA de tipo I en E. coli, y se expresa en respuesta al daño causado en el ADN por antibióticos como el ciprofloxacino, que bloquea la actividad ligasa de la ADN girasa y topoisomerasa, para convertirlas en endonucleasas 12,34 que dañan al ADN, lo que induce la respuesta SOS que regula su reparación, y también causa la expresión de la toxina TisB a un nivel alto, lo que provoca una disminución en la fuerza motriz protónica y los niveles de ATP, y lleva a la célula a un estado persistente y tolerante a los antibióticos (Figura 3). Diversos estudios muestran que la administración por etapas de ciprofloxacino induce la formación de persistentes en una forma dependiente de TisAB, y que las células que producían la toxina son tolerantes a múltiples antibióticos. El descubrimiento de TisB, y su papel en la tolerancia antibiótica, abre una posibilidad intrigante de un vínculo más amplio entre otras respuestas de estrés y la formación de persistentes.
Detección de quorum y formación de persistentes
La detección de quorum, quorum sensing (QS) o red de comunicación bacteriana permite a las células modificar su comportamiento colectivo a través de pequeñas moléculas de señalización llamadas autoinductores (similares a las hormonas de los mamíferos) que favorecen la formación de células persistentes de acuerdo con los cambios en el entorno. Estas moléculas controlan procesos específicos como la formación de biopelículas (Figura 2), la expresión de genes que regulan virulencia, la producción de toxinas, la motilidad, la quimiotaxis, la competencia bacteriana (sistemas de secreción T3SS y T6SS), la bioluminiscencia, la producción de metabolitos secundarios, la transferencia de plásmidos y la esporulación, entre otros (32,50,51. La detección de quorum es importante debido al aumento de persistentes al entrar a la fase estacionaria, y porque permite controlar la densidad celular 52,53. El primer proceso fisiológico regulado por quorum sensing que se estudió fue la bioluminiscencia producida por dos bacterias marinas Vibrio fischeri y Vibrio harveyi , relacionada con el control de su densidad celular, que demuestra que la bioluminiscencia no es proporcional al aumento poblacional y que aumenta a medida que las bacterias entran a fase estacionaria 54. La relación de quorum sensing como inductor de persistencia en grupos bacterianos en un principio era rechazada; sin embargo, estudios posteriores encontraron que las moléculas con capacidad de detectar el quorum (como la piocianina y la N-(3-oxododecanoil)-homoserina lactona (3-OC12-HSL) en P. aeruginosa), aumentan el número de persistentes en una población polimicrobiana sin afectar persistencia en otras especies como E. coli o S. aureus5.
En S. aureus, el sistema Agr, un conjunto de cuatro genes (Agr A, B, C, D), produce oligopéptidos modificados que funcionan como autoinductores de detección de quorum con funciones muy específicas. Estas moléculas permiten que S. aureus exprese factores proteicos de adherencia y colonización de superficies cuando la densidad celular baja, asociado con la formación del fenotipo de persistencia mediante este sistema. Sin embargo, a elevadas concentraciones celulares (metabolismo activo) la síntesis de estos factores se reprime y la bacteria comienza a secretar sus factores de virulencia 24,32,50,55.
Otra molécula que detecta quorum en P. aeruginosa es la 2-amino acetofenona que también induce la formación de persistencia, muy probablemente, al cerrar la actividad de traducción 56. La piocianina producida por P. aeruginosa muestra inducción de persistencia en otras especies, como Acinetobacter baumannnii, en entornos polimicrobianos naturales. El efecto del indol apoya esta hipótesis. El indol es un metabolito producido en fase estacionaria e interviene en diferentes procesos como estabilización de plásmidos, síntesis de biopelículas y formación de esporas, actúa como una molécula que detecta e induce persistencia en especies como E. coli57. Dado que muchas bacterias producen indol, este mecanismo podría estar muy extendido. Esto se corrobora por el hecho de que Salmonella typhimurium, incapaz de producir indol por sí misma, puede interceptar la señal de E. coli y, posteriormente, utilizarla para aumentar los niveles de persistencia in vivo. Aunque hay diversos estudios que muestran que la persistencia inducida por la percepción del quorum es posible, todavía falta mucho por investigar en este contexto debido a la amplia complejidad del metabolismo bacteriano para apoyar esta hipótesis 11.
Papel de (p)ppGpp como regulador global y formador de persistentes
Las bacterias sintetizan (p) ppGpp (guanosina tetrafosfato y pentafosfato), llamadas moléculas reguladoras globales de alarma bacteriana "alarmone" 26, en respuesta estricta a una variedad de tensiones ambientales de estrés, como la limitación de nutrientes o el ataque antibiótico. Esto facilita la supervivencia, adaptación y formación de subpoblaciones bacterianas persistentes. Los (p)ppGpp son nucleótidos con gran influencia en la fisiología y metabolismo bacteriano, al regular directa o indirectamente muchos procesos celulares críticos, como la replicación, transcripción, traducción, división celular, formación de biofilms, motilidad, competencia, virulencia 58-61. Su función de alarma frente a tensiones ambientales, como la limitación de nutrientes, hace que aumente sus niveles intracelulares y se encuentre muy relacionado con módulos TA en especies bacterianas patógenas como E. coli. Los (p)ppGpp activan la proteasa Lon en respuesta a la inanición, la que degrada a las proteínas antitoxinas, y las toxinas liberadas pueden escindir ARNm, lo que frena la transcripción y traducción e induce la detención del crecimiento (Figura 2) 23,39,62-64.
Pero también tiene función de regulador en la estructura y formación de biopelículas en Pseudomonas aeruginosa y E. coli cuando estas bacterias se encuentran frente a estrés antibiótico e inanición de nutrientes 26,65,66. Los (p)ppGpp actúan, además, como reguladores estrictos en la detección de quorum (QS). Por ejemplo, en Pseudomonas aeruginosa, activan la transcripción del autoinductor detector de quorum N-(3-oxododecanoil)-homoserina lactona (3-OC12-HSL), y mejoran la adaptabilidad de las bacterias con la formación de biopelículas 67,68. En E. coli, los (p) ppGpp aumentan la producción de indol en respuesta al estrés oxidativo y la exposición antibióticos, pero también actúan como inhibidores de la síntesis de peptidoglucano y fosfolípidos (30, que son algunas de las funciones principales que contribuyen en la persistencia bacteriana. No obstante, su acción como reguladores globales de diversos procesos en el metabolismo bacteriano, sigue siendo tema de investigaciones actuales.
Persistencia bacteriana e infecciones persistentes
Las formas persistentes dificultan el tratamiento de muchas enfermedades. Varios factores, del agente patógeno y del huésped, contribuyen al establecimiento y mantenimiento de estas infecciones, incluida la resistencia a los antibióticos e inmunosupresión 3. Estas formas de persistencia también se pueden manifestar en pacientes inmunocompetentes, donde el patógeno se encuentra en sitios poco accesibles para los componentes del sistema inmune 69.
Una gran variedad de bacterias origina infecciones persistentes, como Mycobacterium tuberculosis, Salmonella, Chlamydia, Brucella, Borrelia, Pseudomonas, Escherichia coli, Staphylococcus, Streptococcus solo por indicar algunos grupos 23. Las infecciones bacterianas asintomáticas persistentes (subclínicas) o sintomáticas, pueden tener lugar en diversos tejidos dentro del huésped, como el sistema inmunitario, el sistema nervioso central (Treponema pallidum), donde causan meningitis (Neisseria meningitidis), en macrófagos o granulomas (Mycobacterium tuberculosis), en el estómago (Helicobacter pylori), la vesícula biliar (S. enterica subsp. enterica serovar Typhi) 70,71. Las infecciones asintomáticas persistentes también pueden ser causadas por bacterias latentes de crecimiento detenido y se asocian con recaídas de infecciones sintomáticas agudas. Por ejemplo, el 10 % de las personas que están infectadas con M. tuberculosis latente luego de una infección aguda inicial, recaerán durante su vida. La fiebre tifoidea se repite en el 15 % de los pacientes después del tratamiento con antibióticos, y luego de una infección del tracto urinario (ITU) causada por E. coli uropatógena (UPEC), el 27 % de las mujeres sufrirá una recaída. Otras infecciones bacterianas, incluida la amigdalitis estreptocócica, tienden a reaparecer después del tratamiento con antibióticos y es probable que tengan un período de latencia en el huésped 3.
Biopelícula y estado persistente
Una biopelícula es una población de células que crece en una superficie encerrada por una matriz extracelular de sustancias mucoides (exopolisacáridos, mucoproteínas, lípidos e, incluso, ADN extracelular, etc.) difíciles de erradicar y fuente de infecciones recalcitrantes 1,3,72-74 que causan una mortalidad aproximada de 100 mil personas al año con un costo de 28 a 54 mil millones de dólares en los Estados Unidos 75. Las biopelículas se forman en dispositivos médicos permanentes o dentro de los tejidos y son responsables de infecciones en catéteres, prótesis ortopédicas, válvulas cardíacas, oído medio (otitis), heridas que no cicatrizan y en pulmones de pacientes con fibrosis quística 69. Las células persistentes crecen exponencialmente en la biopelícula y se convierten en una fracción significativa de la población en la fase estacionaria (hasta 1 %) 17. Un biofilm está formado por células regulares y células persistentes que se liberan en los tejidos y entran en la circulación. Aunque los antimicrobianos matarían las células normales que crecen en la superficie de la biopelícula, los persistentes sobrevivirían (protegidas por la matriz del biofilm), reanudarían el crecimiento y repoblarían la biopelícula después de retirar el antimicrobiano (con una concentración antibiótica baja y sin señal de estrés) para mantener la infección a pesar de la terapia prolongada 12,13) (Figura 4). Se cree que varios factores contribuyen a la tolerancia antibiótica en las biopelículas, incluida la penetración limitada de los antibióticos a través de la matriz extracelular, el crecimiento lento de bacterias debido a la restricción de nutrientes y oxígeno, desechos metabólicos acumulados y cambios fisiológicos desencadenados por señalización de quorum entre células 7. Las biopelículas pueden bloquear la muerte mediada por el complemento y células del sistema inmune en muchas infecciones persistentes, como las asociadas a dispositivos permanentes 3.
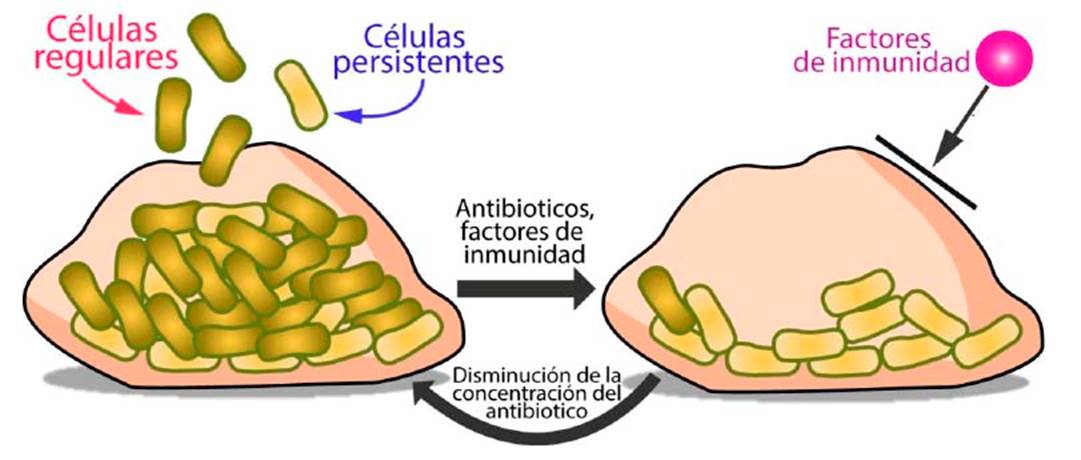
Figura 4 Modelo de persistencia bacteriana en biofilm causante de infecciones recurrentes. Modificado a partir del artículo Persister Cells
Varios microorganismos relevantes para la salud pública forman biopelículas. Entre ellos destacan S. aureus (también las cepas resistentes a la meticilina), S. epidermidis, E. coli, P. aeruginosa, Burkholderia cepacia, Salmonella spp. Vibrio cholera, Candida parapsilosis y C. albicans. Estos han sido descritos en vejiga, pulmón de pacientes con neumonía por fibrosis quística, y en vesícula biliar. En este sentido, la formación de persistentes es, probablemente, un factor importante en la recaída y recalcitrancia de los procesos infecciosos 3,76.
Eliminación ineficaz y tolerancia antibiótica en persistentes bacterianos
El fracaso en el tratamiento puede deberse a la falta de cumplimiento en la terapia por parte del paciente, la llegada deficiente del fármaco hacia el sitio diana, el estado de latencia celular, entre otros. Aunque se postula que la tolerancia a múltiples fármacos en persistentes es un fenómeno fenotípico, es posible que bajo ciertas condiciones los persistentes adquieran mutaciones y desarrollen resistencia genotípica 20.Las células resistentes crecen en presencia de niveles elevados de concentración antibiótica, que se manifiesta como un aumento en la concentración mínima inhibitoria (MIC), en cambio, los persistentes no tienen ningún efecto sobre la MIC, ya que estas células están en latencia y para matarlas, los antibióticos requieren objetivos activos (células en crecimiento exponencial), lo que explica la tolerancia. Esto muestra a este fenotipo como una importante primera línea de defensa contra los antibióticos antes de que se adquiera la resistencia antimicrobiana 11,12,23,69.
Explicar la causalidad entre la persistencia y el fracaso antibiótico es todavía un desafío. Un primer informe sobre la relación causa-efecto revela que P. aeruginosa aumenta los niveles de persistencia en pacientes con fibrosis quística debido al prolongado tratamiento antibiótico 77. Del mismo modo, los aislamientos de C. albicans en infecciones recurrentes de pacientes inmunocomprometidos, muestran un nivel de tolerancia más alto que los aislamientos de infecciones agudas, por selección de persistentes 78. Otro estudio muestra que los aislamientos de E. coli uropatógena, que a menudo albergan mutaciones de alta persistencia tipo hipA (el primer gen de persistencia descrito in vitro), causa la naturaleza crónica de las infecciones recurrentes o se selecciona durante el tratamiento antibiótico a largo plazo 21. Otra publicación, también relacionada a E. coli uropatógena, muestra que elevadas concentraciones de enrofloxacino y ciprofloxacino in vitro no tienen una acción eficaz en persistentes bacterianos, que permanecen viables, sin desarrollar colonias y adoptan una morfología filamentosa como una estrategia de adaptación y supervivencia (25.
La persistencia parece ser un fenómeno generalizado, sin embargo, diferentes especies bacterianas parecen tener diferentes capacidades de persistencia in vitro e in vivo, con distintos grados de dificultad para el tratamiento 20. Si bien se ha dado mayor atención a la resistencia antimicrobiana, la persistencia o la tolerancia a estos es igualmente importante e incluso mucho más compleja, lo que plantea retos importantes para el tratamiento 37.
Estrategias de tratamiento en infecciones bacterianas persistentes
Mientras que las diversas infecciones bacterianas parecen tener distintas capacidades de persistencia y diferentes grados de dificultad para el tratamiento, su curación debería tener distintos enfoques estratégicos indicados, como el tipo de fármaco usado, la terapia combinada, la alteración "despertar" del metabolismo persistente, la potenciación de la actividad antibiótica, la mejora del sistema inmune del huésped y el estado de las células diana. Todos estos factores mediarían un mejor resultado al tratamiento.
Una primera estrategia de tratamiento sería el tipo de fármaco usado y su acción en infecciones bacterianas persistentes. Estudios muestran que la colistina 34, así como los péptidos antimicrobianos catiónicos de arginina o triptófano, tienen un efecto notable en reducir el desarrollo de persistencia en E. coli por alteración de la membrana externa y en la erradicación de biopelículas 79. Recientemente, se divulgó que una pequeña molécula, la TCA1 inhibe la formación de biofilms en M. tuberculosis y mata persistentes, probablemente al centrarse en la inhibición de la síntesis de la pared celular 80. Otro fármaco poco convencional en el tratamiento de la tuberculosis es la pirazinamida, que actúa únicamente en persistentes no muertos por medicamentos que solo tienen efecto en micobacterias de crecimiento activo, y juegan un papel clave en la reducción de la terapia de 9 a 12 meses, a una de 6 meses. La pirazinamida inhibe la producción de energía, los procesos de transcripción y traducción, recicla ribosomas, degrada la acumulación de proteínas tóxicas bajo estrés y, tal vez, la síntesis de coenzima A, necesaria para la supervivencia de micobacterias persistentes 81,82. Es importante señalar que la pirazinamida inhibe también al parásito de la malaria en reposo (hipnozoito) en modelos murinos 83 y, a pesar de que existe un creciente interés por el desarrollo de fármacos para persistentes bacterianos, la pirazinamida es el único prototipo de fármaco demostrado, hasta ahora, que mejora el tratamiento de este tipo de infecciones 20.
Actualmente, los medicamentos usados contra el cáncer como la mitomicina C, el cisplatino, así como las antraciclinas, han demostrado ser efectivos como terapia para matar persistentes (al reticular y entrecruzar ADN mientras las células duermen) en diversos patógenos como E. coli, S. aureus y P. aeruginosa, tanto en condiciones de vida libre como en biofilms. Aún no se contrasta su acción en condiciones in vivo para curar infecciones bacterianas persistentes y, por ello, su aplicación de uso clínico solo está limitada para tratamientos contra el cáncer 11,35.
Un segundo método de tratamiento sería la terapia combinada. En teoría, podría ser efectiva en persistentes, sin embargo, la combinación de antibióticos a menudo falla en la erradicación completa. Existen informes en Borrelia burgdorferi que indican que la combinación de antibióticos contra cultivos en crecimiento (cefoperazona o cefuroxima y doxiciclina) con los antibióticos más potentes en las células que no crecen (daptomicina o daunomicina) tendrían un mejor efecto en persistentes (84. El uso de medicamentos que no requieren el metabolismo activo para operar, como el caso de la nitrofurantoína, también ha sobresalido como un fuerte candidato al matar persistentes bacaterianos en Listeria monocytogenes que quedan vivas por otros antibióticos 85, lo que demuestra que la terapia combinada puede ser un tratamiento eficaz contra la formación de persistentes y, a la vez, retardaría el desarrollo de resistencia antimicrobiana 86, pero las investigaciones aún son limitadas en este contexto.
Un tercer mecanismo para tratar las infecciones persistentes consistiría en "despertar" o alterar el estado metabólico de los persistentes para que puedan responder mejor al tratamiento tradicional. Algunas investigaciones muestran que la adición de azúcares o productos intermedios de la glucólisis como manitol, glucosa, fructosa o piruvato activan rápidamente el metabolismo en persistentes de E. coli y responden mejor al tratamiento por aminoglucósidos 23,87 (que tiene una mayor absorción al encontrarse activa la fuerza motriz protónica) pero la desventaja de este tratamiento, es que solo se ha demostrado con este tipo de antibiótico, pero no con otros. Estudios relacionados muestran que el glicerol, fuentes de carbono relativamente menos eficaces y nucleótidos tales como la timidina, también potenciarían la actividad de los aminoglucósidos en persistentes en ensayos in vitro, y tal enfoque debe ser validado en modelos animales a futuro 88. De forma similar, las células persistentes de P. aeruginosa también pueden despertarse con la adición de ácido cis-2-decenoico (cis-DA), lo que provoca un estallido en la síntesis de proteínas, para luego ser tratadas con ciprofloxacino 73,89, pero al igual que los estudios anteriores, debería ser validado en modelos animales.
Una cuarta estrategia de tratamiento buscaría mejorar la actividad de los antibióticos por ciertos agentes. Por ejemplo, el uso de aspirina, ibuprofeno o hierro que mejoran la actividad de la pirazinamida en infecciones por M. tuberculosis90,91. Ciertos estudios expresan que la plata, al producir especies reactivas de oxígeno, mejora la actividad de la vancomicina in vitro, como también en la eliminación de biopelículas 92. Queda por determinar cómo funcionan los compuestos y cómo pueden ser utilizados para el tratamiento mejorado de infecciones persistentes.
Finalmente, un quinto método sería aprovechar el sistema inmune del huésped para controlar los persistentes a través del mejoramiento de la inmunidad innata y adaptativa, modulación de citoquinas y vacunas inmunoterapéuticas que incluyan antígenos de ambas células, en crecimiento y células no crecientes (células persistentes) 20.
El redescubrimiento de persistentes y la intrigante posibilidad de que estos sean los principales culpables de infecciones crónicas y recurrentes, renovó el interés por comprender la naturaleza de estas células inusuales. Los principales conocimientos generados en persistencia bacteriana, sobre su fisiología, mecanismos de formación e importancia clínica, así como las estrategias de tratamiento, hasta el momento, son temas de investigación que requieren de equipos multidisciplinarios para lograr comprender mejor este fenómeno. Su naturaleza transitoria, las distintas hipótesis planteadas sobre su formación, fisiología, vínculo con infecciones recurrentes y el nexo con la resistencia antibiótica son todavía, en muchos casos, causa de confusión.
Los distintos experimentos que plantean los mecanismos de formación, como los que hemos expuesto (módulos toxina-antitoxina, señales SOS, (p)ppGpp, señales de estrés ambiental, los procesos deterministas, como la edad celular y la detección de quorum) tienen un enfoque orientador que trata de explicar su formación y su evolución en las enfermedades persistentes, sin embargo, los estudios aún son limitados, debido que la formación de persistentes entre uno y otro grupo bacteriano es de naturaleza heterogénea y solo ha sido demostrado in vitro, o, en algunos casos, in vivo (en modelos experimentales animales). Faltan todavía muchas más investigaciones que muestren y expliquen qué otros factores o mecanismos ayudan a su formación, la posibilidad de revertir el cambio de fenotipo (de latente a normal) y que sirvan como evidencia sustentable para aumentar su relevancia clínica y comprender mejor la actual crisis antibiótica.
La persistencia impone una carga significativa a la atención de salud pública, no solo en infecciones relacionadas con biopelículas, sino también en otras infecciones donde los patógenos están protegidos del sistema inmune, lo que ocasiona la naturaleza crónica, e incluso incurable, de muchas infecciones. Hasta el momento, la investigación que relaciona la persistencia bacteriana con enfermedades crónicas, recurrentes y la formación de biofilms, está limitada a algunos grupos bacterianos de importancia clínica. En este contexto, la posibilidad de que las bacterias persistentes sean la etiología de las enfermedades crónicas, que estas constituyan reservorios biológicos de las recidivas y que a partir de ellas podrían emerger mutantes resistentes, destaca la importancia de revolucionar las prácticas clínicas, los métodos de diagnóstico y la necesidad de desarrollar en un futuro nuevos antibióticos para erradicar esta subpoblación bacteriana tolerante