Introducción
En enero de 2020, durante la Conferencia Internacional de la Salud Mundial en Ginebra, la Organización Mundial de la Salud (OMS) anuncia el brote de un nuevo coronavirus (Coronavirus 2 del síndrome respiratorio agudo severo-SARS) como una emergencia de salud pública de importancia mundial 1,2,3. Este nuevo coronavirus se reportó en la ciudad china de Wuhan en diciembre de 2019 cuando aparecieron varios casos de neumonía de etiología desconocida que ocasionaron el síndrome respiratorio agudo severo 4,5,6,7,8, cuadro que la OMS declaró como una pandemia el 11 de marzo de 2020 9.
Por su rápido mecanismo de transmisión, la falta de una vacuna y un tratamiento con medicamentos probados, la COVID-19 ha llegado a cambiar un estilo de vida que la humanidad consideraba como "normal".
A mayo del 2020, la COVID-19 ha infectado a más de medio millón de personas en el mundo y más de 25 000 han perdido la vida, lo que ha obligado a más de tres mil millones de seres humanos a estar en aislamiento en sus casas 1-3,10.
Aunque en China la enfermedad se ha controlado, en otros países, incluido el Perú, el número de casos se incrementa. En nuestra nación el primer caso de COVID-19 se reportó el 6 de marzo de 2020 y para el 31 de mayo de 2020 se cuentan 155 671 casos y 4371 fallecidos 11.
Los modelos matemáticos se utilizan para entender las transiciones epidemiológicas críticas y predecir los parámetros. Con frecuencia, el ajuste de la curva epidemiológica, los datos de vigilancia durante la transmisión temprana y otros modelos epidemiológicos se han empleado para realizar pronósticos de la pandemia de la COVID-19 en el mundo 3,12-14.
De las técnicas estadísticas para predicción, el análisis de los datos de las series de tiempo, que se expresa simplemente como un conjunto de valores numéricos ordenados en el tiempo 15,16, tiene como objetivo revelar estadísticas confiables y significativas, y utilizar este conocimiento para predecir los valores futuros 17-19. De todos los modelos conocidos, el modelo pronóstico suavizado de Brown es apropiado para las series con una tendencia lineal y sin estacionalidad. Sus parámetros de suavizado son el nivel y la tendencia, que se admiten iguales. Por ello, este modelo es un caso específico del modelo de Holt y es muy análogo al modelo autorregresivo integrado de media móvil o ARIMA (por sus siglas en inglés, autoregressive integrated moving average) 20,21.
Realizar el cálculo de un pronóstico va a presentar errores, y en la práctica se procura minimizar los tipos de errores optando por el mejor método de pronóstico. Por ello, existen las medidas de precisión del pronóstico 21,22, las cuáles se utilizan para determinar qué tan eficaz es un pronóstico a través del cálculo de su precisión con respecto a los valores reales, es decir, la precisión o medida de error de un modelo se puede probar comparando los valores observados con los valores pronosticados 16. La determinación de la idoneidad del modelo seleccionado para el pronóstico se realiza analizando los errores de predicción. Existen muchas medidas de precisión del pronóstico, pero la mayoría de los autores recomiendan el empleo del error cuadrático medio y del porcentaje de error medio absoluto (que tiene la ventaja adicional de no ser afectado por los cambios de escala en la variable de estudio) 23.
Lixiang Li et al. 24) encontraron en Corea, Italia e Irán, que el error de pronóstico entre el modelo y los datos oficiales era bastante pequeño. En Colombia, Díaz-Pinzón 21) reportó que el error de pronóstico fue muy bajo y correspondió al MAPE (error porcentual medio absoluto) con un 0,03 %, lo cual ajusta el pronóstico al contexto de contagio real de pacientes con COVID-19 en ese país.
Algunas de las principales preocupaciones de los gobiernos y los responsables de las políticas de salud del mundo son ¿cuándo finalizará la propagación de la COVID-19?, ¿cuál es la tendencia de este brote?, ¿cuántas personas se infectarán y cuantos perderán la vida con COVID-19 día con día? Si bien estas preguntas se podrían responder con modelos predictivos 7,25, el presente estudio tuvo como objetivo realizar el análisis de la precisión del pronóstico de un modelo predictivo, el modelo suavizado de Brown, para predecir la propagación de la COVID-19 entre el 6 de marzo al 30 de mayo del 2020 en Perú.
Materiales y métodos
Diseño y población
El presente estudio fue descriptivo y estuvo basado en un análisis de series de tiempo correspondiente al período entre el 6 de marzo al 30 de mayo del 2020 en Perú. Se utilizó la información del número de casos positivos de COVID-19, que fue de 155 671 personas afectadas.
Variables y mediciones
Los datos provienen de la Sala situacional de COVID-19 del Perú, Instituto Nacional de Salud y Centro Nacional de Epidemiologia, Prevención y Control de Enfermedades del Ministerio de Salud 11 los cuales sirven para obtener la precisión del pronóstico de la propagación de la COVID-19.
Análisis estadístico
El programa estadístico SPSS versión 22 se utilizó para el procesamiento. Como método de predicción se analizó el modelo pronóstico suavizado de Brown que consiste en realizar dos suavizaciones exponenciales a partir de las cuales se calcula el pronóstico: la primera emplea los valores observados en la serie de tiempo, y la segunda emplea la serie que ha sido obtenida mediante la primera atenuación. Debido a que los valores calculados al realizar las dos primeras atenuaciones no son los datos considerados a obtener, es decir, que formarán las inferencias de los valores que se espera que tome la serie de tiempo en el futuro cercano, se emplea una notación distinta a la de la expresión final con la cual se calculan los valores que componen en realidad el pronóstico 21,23,26.
Las medidas de precisión que se calcularon fueron las siguientes: el error medio del pronóstico (EMP), el error medio al cuadrado (EMC), la desviación absoluta de la media (DAM) y el porcentaje de error medio absoluto (PEMA).
Consideraciones éticas
Los datos han sido obtenidos de la Sala Situacional COVID 19-Perú, del Instituto Nacional de Salud y Centro Nacional de Epidemiologia, Prevención y Control de Enfermedades del Ministerio de Salud. La identidad de los participantes no fue registrada. Esta información es de acceso público y abierto por lo que no se requiere de una aprobación ética.
Resultados
En la Tabla 1 se muestran el promedio, la desviación estándar, el valor mínimo y valor máximo de los diferentes errores de los 86 días de observación, entre el 6 de marzo del 2020 al 30 de mayo del 2020 en Perú. El porcentaje de error medio absoluto (PEMA) fue del 9,03 %.
Tabla 1 Precisión del pronóstico para la COVID-19, entre el 18 de abril al 30 de mayo del 2020. Perú
| Error medio del pronóstico (EMP) | Error medio al cuadrado (EMC) | Desviación absoluta de la media (DAM) | Porcentaje de error medio absoluto (PEMA) % | |
| Promedio | 156,7 | 506461,3 | 450,6 | 9,03 |
| Desviación estándar | 698,3 | 963528,2 | 554,0 | 33,14 |
| Mínimo | -1560 | 0 | 0 | 0 |
| Máximo | 2488 | 6190144 | 2488 | 300,00 |
Fuente: Sala Situacional COVID 19-Perú, del Instituto Nacional de Salud y Centro Nacional de Epidemiologia, Prevención y Control de Enfermedades del Ministerio de Salud
Se observa que la línea continua de color celeste (de los casos observados o contagio real) y la línea discontinua de color verde (de los casos pronosticados o pronóstico) son muy similares entre sí y permanecen, en la mayoría del recorrido, una sobre otra, entre el 6 de marzo al 30 de mayo del 2020. De igual manera, el coeficiente de determinación (R2) es igual a 0,8078, lo que indica que los datos se ajustan en un 80,78 % al modelo de predicción (Figura 1). Asimismo, se observa el pronóstico (de los próximos 30 días) del número de casos confirmados con intervalos de confianza del 95 %, el cual puede estar entre 287 284 y 432 977(Figura 1 y Tabla 2).
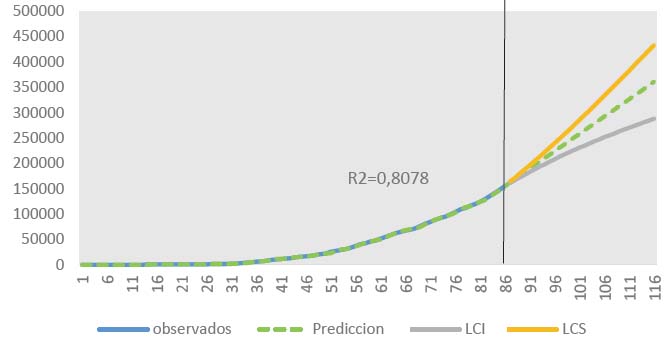
Figura 1 Casos observados (contagio real) y casos pronosticados (pronóstico de COVID-19) entre el 6 de marzo del 2020 al 30 de mayo del 2020, y pronósticos con intervalos de confianza del 95 % de nuevos casos con COVID-19 en Perú (en los próximos 30 días).
Tabla 2 Número pronosticado de casos nuevos diarios con COVID-19, con intervalos de confianza del 95 %
| Intervalo de confianza: 95 % | Intervalo de confianza: 95 % | ||||||
| Día | Pronóstico | LCI | LCS | Día | Pronóstico | LCI | LCS |
| 31/05/2020 | 162 384 | 160 961 | 163 807 | 15/06/2020 | 264 667 | 234 728 | 294 605 |
| 1/06/2020 | 169 203 | 166 728 | 171 678 | 16/06/2020 | 271 486 | 238 915 | 304 056 |
| 2/06/2020 | 176 022 | 172 325 | 179 718 | 17/06/2020 | 278 304 | 243 028 | 313 581 |
| 3/06/2020 | 182 840 | 177 774 | 187 907 | 18/06/2020 | 285 123 | 247 070 | 323 176 |
| 4/06/2020 | 189 659 | 183 087 | 196 231 | 19/06/2020 | 291 942 | 251 044 | 332 841 |
| 5/06/2020 | 196 478 | 188 277 | 204 679 | 20/06/2020 | 298 761 | 254 949 | 342 573 |
| 6/06/2020 | 203 297 | 193 352 | 213 242 | 21/06/2020 | 305 580 | 258 789 | 352 371 |
| 7/06/2020 | 210 116 | 198 318 | 221 914 | 22/06/2020 | 312 399 | 262 564 | 362 233 |
| 8/06/2020 | 216 935 | 203 183 | 230 687 | 23/06/2020 | 319 218 | 266 276 | 372 159 |
| 9/06/2020 | 223 754 | 207 950 | 239 557 | 24/06/2020 | 326 036 | 269 926 | 382 147 |
| 10/06/2020 | 230 572 | 212 625 | 248 520 | 25/06/2020 | 332 855 | 273 515 | 392 196 |
| 11/06/2020 | 237 391 | 217 211 | 257 572 | 26/06/2020 | 339 674 | 277 044 | 402 304 |
| 12/06/2020 | 244 210 | 221 711 | 266 709 | 27/06/2020 | 346 493 | 280 515 | 412 471 |
| 13/06/2020 | 251 029 | 226 129 | 275 929 | 28/06/2020 | 353 312 | 283 928 | 422 696 |
| 14/06/2020 | 257 848 | 230 467 | 285 228 | 29/06/2020 | 360 131 | 287 284 | 432 977 |
Fuente: Obtenido con el modelo suavizado de Brown
Discusión
Los modelos de series temporales cobran importancia para predecir la dinámica de propagación y pueden contribuir a que se tomen las medidas necesarias que ayuden a superar la crisis que atraviesan el Perú y otros países del mundo. Además, resulta beneficioso crear un modelo de pronóstico confiable y adecuado que sirva como referencia para decidir sobre las estrategias que se tomen en la emergencia sanitaria y sobre la asignación de recursos médicos. En este estudio, se analiza la precisión del pronóstico del modelo suavizado de Brown para predecir la propagación de la COVID-19 en el Perú; información muy relevante ya que, según la Universidad Johns Hopkins, nuestra nación está entre los nueve países del mundo con más casos de coronavirus, y en América del Sur es el segundo, después de Brasil 27.
En este estudio, se analiza la precisión del pronóstico del modelo suavizado de Brown para predecir la propagación de la COVID-19 en el Perú, información muy relevante ya que, según la Universidad Johns Hopkins, nuestro país está entre los nueve países del mundo con más casos de coronavirus, y en América del Sur es el segundo, después de Brasil 27.
Respecto a los resultados de nuestro estudio, el porcentaje de error medio absoluto (PEMA) fue del 9,03 %, lo que indica que el pronóstico del modelo está errado en dicho porcentaje; sin embargo, es posible observar en la figura 1 que, a pesar que el valor del PEMA no es muy bajo, el modelo parece ajustarse adecuadamente a los datos. El coeficiente de determinación (R2) nos indica en qué medida los datos se ajustan al modelo seleccionado, en este caso el R2 = 0,8078 significa que los datos se ajustan en un 80,78 %. El valor del error obtenido es alto comparado con el estudio de Díaz-Pinzón 21 que encontró un PEMA indican un mejor ajuste de los datos. El estudio realizado por Ceylan Z 16 menciona que el PEMA obtenido fue del 4,75 % para Italia; del 5,85 % para España, y del 5,63 % para Francia, pero a diferencia de este estudio, el modelo utilizado para determinar el error corresponde a los modelos ARIMA 20,21 que se utilizan ampliamente para la predicción de series temporales de enfermedades epidémicas como la fiebre hemorrágica con síndrome renal, la fiebre del dengue y la tuberculosis 16.
El PEMA es el promedio del error absoluto o diferencia entre el riesgo real y el pronóstico. Es indicado como un porcentaje de los valores reales 21 y puede aumentar significativamente si los valores que divide el error absoluto entre los datos reales se aproximan a 0. La razón por la cual se está tomando ese valor de medición del error del modelo de pronóstico es porque si un modelo individual no tiene los valores más bajos para las otras medidas de error, como se observa en la tabla 1, el PEMA es, generalmente, la medición más recomendable. El error de un modelo de pronóstico, nos ayuda a identificar que tan exacto es nuestro pronóstico o si nuestro pronóstico se encuentra subestimado o sobreestimado.
Sobre la precisión del pronóstico de la propagación de la COVID-19 en Colombia, Díaz-Pinzón 21 encontró que durante 40 días de observación, el error de pronóstico fue muy bajo lo que indica que la predicción tenía un alto grado de confiabilidad, y ajusta el pronóstico al contexto de contagio real de pacientes con COVID-19. En otro estudio realizado por Lixiang et al. 24 sobre el análisis de propagación y predicción de la COVID-19 en Corea, Italia e Irán encontraron que el error de pronóstico entre el modelo y los datos oficiales era bastante pequeño, por lo que concluyeron que el estudio realizado podía ser una ayuda para estos países al momento de tomar decisiones sobre la pandemia.
En nuestra investigación, el valor del error de pronóstico obtenido puede deberse a que el número de casos observados por día se obtiene de los resultados positivos; es decir, de las pruebas de reacción en cadena de polimerasa (PCR) como de las pruebas serológicas (IgM e IgG), las que tienen una sensibilidad demasiado baja en la primera semana desde la aparición de los síntomas como para desempeñar un papel principal en el diagnóstico de la COVID-19 28.
Existe una gran preocupación en cuanto a la capacidad resolutiva del sistema de salud peruano que ha colapsado, lo que indica que no se va a poder responder eficazmente a las necesidades de los pacientes infectados que requieren atención en cuidados intensivos. En el país, el número de personas infectadas (desde el 6 de marzo) sigue una tendencia ascendente, y aunque el número total de casos confirmados sigue aumentando, el gobierno ha abierto las puertas a la nación a una nueva vida normal o "nueva normalidad". Por otro lado, no se visualiza una tendencia a la baja y, al parecer, se necesita más tiempo para alcanzar la meseta esperada, de lo contrario, los problemas sanitarios, económicos y sociales serán inmanejables y podrían resultar en un desastre nacional.
Los modelos de series de tiempo pueden jugar un papel importante en el análisis de brotes y predicción de enfermedades. Las características de la información disponible permiten evaluar la calidad de las series y el aspecto necesario para poder comenzar el proceso de predicción 21 y, por supuesto, la exactitud de esta predicción depende de la precisión de los datos y de la adecuación del modelo estadístico aplicado. Si se quiere identificar un modelo de pronóstico que muestre un menor error, los tipos de error se tienen que medir varias veces con diferentes modelos de pronóstico, y así poder identificar el modelo con el menor error posible. Para una comparación más precisa y perspectivas futuras, los datos deben actualizarse en tiempo real.
El pequeño número de observaciones para este tipo de algoritmos de series de tiempo es una limitación del estudio. Por otro lado, no hay evidencia adicional que pueda confirmar si el número de personas infectadas es exacto ya que solo se trabajó con el número reportado de casos de COVID-19 que han sido notificados oficialmente. Si bien estos datos son parciales, son la única fuente de información disponible para comprender la dinámica de propagación de la enfermedad.
El modelo de pronóstico utilizado en este estudio solo toma en cuenta el número acumulado de casos de COVID 19. En otras epidemias se han aplicado modelos más complejos y de mayor precisión como el modelo SIR, que estima el número teórico de personas susceptibles de enfermar (susceptibles), el número de enfermos (infectados) y el número de personas que ya no pueden transmitir la enfermedad (recuperados o fallecidos) en una población a lo largo del tiempo 29-33.
A pesar de las limitaciones (como no contar con la precisión de los datos y no realizar varias veces la medición de los tipos de error con diferentes modelos de pronóstico), los hallazgos de este estudio aportan datos que deben servir de inicio para analizar otros modelos matemáticos de predicción más precisos y con los supuestos verificados y validados de manera adecuada por los especialistas, para que se pueda tener una visión realista de la dinámica de propagación de la COVID-19 en el Perú, de tal manera que, a medida que los pronósticos diarios de los infectados comiencen a concurrir, puede considerarse que el brote está bajo control. Sin embargo, cualquier desviación sistemática de la curva de pronóstico puede indicar que la epidemia está escapando al control 33,34.
En conclusión, el error de precisión o porcentaje de error medio absoluto (PEMA) fue del 9,03 %, con un coeficiente de determinación (R2) de 0,8078 que indica que los datos se ajustan en un 80,78 % al modelo evaluado.















