Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. v.15 supl.1 Lima nov. 2008
TRABAJOS ORIGINALES
Estatus taxonómico de Oenocarpus bataua (Euterpeae, Arecaceae) inferido por secuencias del ADN cloroplástico
Taxonomic status of Oenocarpus bataua (Euterpeae, Arecaceae) inferred from plastid sequences
Rommel Montúfar1 y Jean-Christophe Pintaud2
1 Lab. (113) de Genética Molecular, Escuela de Biología, Pontificia Universidad Católica del Ecuador. Av. 12 de Octubre 1076, Apartado 17-01-2184, Quito, Ecuador. Email: rjmontufar@puce.edu.ec
2 IRD, UR 188, UMR DIA-PC, 911 Avenue Agropolis, BP 64501, 34394 Montpellier Cedex 5, France Email: pintaud@ird.fr
Trabajo presentado al Simposio Internacional Las palmeras en el marco de la investigación para el desarrollo en América del Sur, del 07 al 09 de Noviembre 2007, Museo de Historia Natural, Universidad Nacional Mayor de San Marcos, Lima, Perú.
Resumen
Las relaciones evolutivas entre la especie Oenocarpus bataua (anteriormente denominada Jessenia bataua) y otras especies del género Oenocarpus fueron estudiadas mediante un análisis filogenético de la tribu Euterpeae (Arecaceae) en base a 3,6 kb de secuencias de espaciadores intergénicos del cloroplasto. Los cladogramas recuperados sugieren: (1) la monofilia de la tribu Euterpeae, (2) la inclusión de los individuos de O. bataua dentro del clado de Oenocarpus, y (3) la divergencia molecular entre O. bataua var. bataua y var. oligocarpus, sugiriendo que estos taxa podrían representar especies distintas, así como fueron descritos originalmente. Sin embargo, los patrones filogenéticos mencionados tuvieron un bajo soporte estadístico, por lo cual las conclusiones derivadas de estos permanecen tentativas.
Palabras claves: Arecaceae, Euterpeae, Oenocarpus bataua, Jessenia bataua, filogenia Molecular, Amazonía.
Abstract
With the aim of testing the evolutionary relationships between Oenocarpus bataua (formerly Jessenia bataua) and other Oenocarpus species (Arecaceae), a phylogenetic study of tribe Euterpeae (Arecaceae) was conducted using 3,6 kb of non-coding chloroplast DNA sequence data. The results of maximum parsimony analysis support the monophyly of Euterpeae and the inclusion of O. bataua within the Oenocarpus clade. Our results show substantial molecular divergence between O. bataua var. bataua and var. oligocarpus suggesting that, according to their original description, these two taxa may indeed represent different species. However, our phylogenies should be considered as tentative due to low statistical support.
Keywords: Arecaceae, Euterpeae, Oenocarpus bataua, Jessenia bataua, Molecular phylogeny, Amazonia.
Introducción
La tribu de palmeras Euterpeae esta constituida por cinco géneros (Euterpe, Oenocarpus, Neonicholsonia, Hyospathe y Prestoea), y aproximadamente 30 especies ampliamente distribuidas en la región neotropical (Govaerts y Dransfield 2005). El género Oenocarpus es el segundo más diverso dentro de Euterpeae, con aproximadamente 9 especies (O. bacaba Martius, O. balickii Kahn, O. bataua Martius, O. circumtextus Martius, O. distichus Martius, O. makeru Bernal, Galeano & Henderson, O. mapora Karsten, O. minor Martius, O. simplex Bernal, Galeano & Henderson). En particular, O. bataua (localmente conocida como ungurahua, ungurahui, seje, mil pesos, chapil) es una palmera ampliamente distribuida y abundante en los bosques tropicales del norte de América del Sur (Balick 1986, Henderson 1995).
Oenocarpus bataua es una palmera arborescente (hasta los 30 metros de alto), monoica, alógama, y altamente apreciada por las tribus Amerindias y la población local por sus frutos nutritivos, ricos en compuestos oleaginosos y proteicos, de los cuales se elaboran bebidas nutritivas. En la época de fructificación los frutos de O. bataua son comercializados en los mercados regionales de la cuenca Amazónica (Balick, 1986). A pesar de su potencial como una nueva fuente de aceites comestibles de alto valor nutritivo, el conocimiento sobre su biología y relaciones filogenéticas permanece pobremente estudiado.
La historia taxonómica del género Oenocarpus y de la especie O. bataua es confusa. El género Oenocarpus y la especie O. bataua fueron descritas en 1823 por Martius en su clásico tratamiento taxonómico Historia Naturalis Palmarum. Karsten en 1857 describe el género monoespecífico Jessenia, con la especie tipo J. polycarpa Karsten, sin hacer mención de su afinidad taxonómica con Oenocarpus (J. polycarpa es actualmente un sinónimo de O. bataua var. bataua). Posteriormente, Burret (1928) reevalúa y mantiene estos dos géneros, y hace una nueva combinación taxonómica: Jessenia bataua (Martius) Burret. Burret se basó en el tipo del endospermo (ruminado en Jessenia y homogéneo en Oenocarpus) y la forma de los tricomas en el envez de las pinnas (curvados en Jessenia) para discriminar Jessenia de Oenocarpus (Balick, 1986). A partir del trabajo de Burret (1928), el binomio Jessenia bataua ha sido ampliamente utilizado en la literatura botánica. Sin embargo, Wessels Boer (1965), incluye Jessenia en sinonimia de Oenocarpus, enfatizando la importancia de la peculiar inflorescencia hipuriforme que caracteriza todas las especies del complejo Oenocarpus / Jessenia.
Una moderna revisión taxonómica del complejo Jessenia/Oenocarpus, incluyendo datos morfológicos y marcadores bioquímicos (composición de flavonoides) fue realizada por Balick (1986). Este trabajo dio soporte a la separación taxonómica de estos géneros. A nivel morfológico, Balick (1986) separa Jessenia de Oenocarpus en base al número de estambres, a la morfología de los filamentos y anteras, al tipo de endospermo y fibras, y al indumento de las pinnas. El genero Jessenia fue mantenido separado de Oenocarpus también en Genera Palmarum (Uhl y Dransfield, 1987).
En las últimas décadas, nuevas expediciones y colecciones de palmeras en la región amazónica de Colombia (Bernal et al., 1991; Henderson, 1995) documentaron la existencia de una nueva especie de palmera con características morfológicas intermedias entre Jessenia y Oenocarpus. Oenocarpus makeru posee características morfológicas del género Oenocarpus; pero también un endospermo ruminado, carácter asociado al género Jessenia. Este descubrimiento debilitó la hipótesis de separación taxonómica entre Jessenia y Oenocarpus propuesta por Balick (1986); y en particular el tipo de endospermo como carácter clave para diferenciar estos dos géneros. Como consecuencia de estos descubrimientos, Henderson (1995) reduce de nuevo Jessenia en sinonimia de Oenocarpus. Posteriormente, Henderson (1999) desarrolló el primer análisis filogenético de la tribu Euterpeae en base a caracteres morfológicos y anatómicos. Este estudio confirmó el tratamiento de Jessenia y Oenocarpus como un solo género, y el consecuente restablecimiento del nombre Oenocarpus bataua.
Adicional a la controversial delimitación genérica entre Jessenia y Oenocarpus, la taxonomia intraespecífica de O. bataua no ha sido estudiada en detalle. Las dos variedades actuales (bataua y oligocarpus), establecidas por Henderson (1995), fueron originalmente descritas como dos especie diferentes (O. bataua / J. bataua y O. oligocarpus / J. oligocarpus). Grisebach y Wendland (1864 en Balick, 1986) describieron a la especie Jessenia oligocarpus a partir de una población proveniente de la isla de Trinidad. Wessels Boer (1965) transfirió J. oligocarpus en Oenocarpus manteniendo su rango de especie. Balick (1986) considera insuficientes los caracteres morfológicos usados para separar O. oligocarpus de O. bataua (tratado como Jessenia) a nivel de especies (forma y disposición de las pinnas, número de flores pistiladas en cada rachilla floral, número de estambres); y trata estas dos taxa como subespecies. Las poblaciones de Oenocarpus bataua del nororiente de las Guayanas son habitualmente asignadas a la variedad oligocarpus siguiendo la revisión de Wessels Boer (1965), aunque su identidad con la población insular tipo merecería un estudio detallado.
El objetivo de nuestro estudio fue explorar las relaciones filogenéticas dentro de la tribu Euterpeae; en particular si las poblaciones de la especie Oenocarpus bataua -anteriormente consideradas como Jessenia bataua- difieren molecularmente de otras especies del género Oenocarpus. Si las secuencias de los individuos de O. bataua y de otras especies de Oenocarpus forman un grupo monofilético; entonces, interpretaríamos este patrón como una evidencia molecular que corrobora la inclusión de Oenocarpus bataua (ex Jessenia bataua) dentro del género Oenocarpus como ha sido sugerido por Henderson (1995, 1999). En el caso contrario, si las secuencias de O. bataua y de otras especies de Oenocarpus forman dos grupos monofiléticos distintos; entonces, este patrón constituiría una evidencia molecular que apoya la diferenciación biológica entre O. bataua y otras especies del género Oenocarpus; y su consecuente transferencia al género Jessenia, como ha sido sugerido por Burret (1928) y Balick (1986).
Para probar esta hipótesis desarrollamos un análisis filogenético basado en secuencias regiones intergénicas del cloroplasto. El ADN cloroplástico ha sido utilizado para la inferencia filogenética dentro de la familia de palmeras (Baker et al., 1999; Asmussen y Chase, 2001; Hahn, 2002; Asmussen et al., 2006); en particular, las secuencias no codantes del cloroplasto han sido utilizadas exitosamente para resolver relaciones evolutivas entre géneros y especies en varios grupos de plantas. A nivel de especies su aplicación ha sido más limitada (Couvreur et al., 2006).
Material y métodos
Diez y ocho especies de la tribu Euterpeae fueron incluidas en el análisis filogenético. El género Oenocarpus fue representado por individuos de cuatro especies; mientras que la especie Oenocarpus bataua fue representada por tres especímenes correspondientes a O. bataua var. bataua y cuatro especímenes de O. bataua var. oligocarpus. Dos especies por cada uno de los géneros Euterpe, Hyospathe y Prestoea fueron incluidas en el análisis. El género monoespecífico Neonicholsonia fue representado por un solo espécimen. Adicionalmente, fueron incluidas ocho especies provenientes de otras tribus de la subfamilia Arecoideae, y una especie de la subfamilia Ceroxyloideae como grupo externo (Tabla 1).
Los tejidos vegetales utilizados para la extracción de ADN fueron colectados directamente en el campo y posteriormente secados con silica gel. El ADN total fue extraído con el DNeasy Plant Mini Kit (Qiagen). Los espaciadores intergénicos del cloroplasto utilizados para la filogenia (trnQ-rps16, trnD-trnT, and psbC-trnfM) fueron amplificados y secuenciados utilizando los cebadores (primers) presentados en la Tabla 2. El espaciador psbC-trnfM fue amplificado con dos pares de cebadores (Tabla 2). Las amplificaciones fueron realizadas con la técnica PCR utilizando los reactivos Failsafe Amplification Premix (Epicentre Technologies, Madison, WI) en 25 µl conteniendo: 0,2 µM de cada primer, 2,5 unidades de la Enzima Mix, 1X PreMix E, y 5 µl de ADN. El ciclo termal del PCR fue: 3 minutos de denaturación inicial a 95 °C, seguido de 35 ciclos de 95 °C por 30 segundos, 5062 °C de temperatura de annealing por 45 minutos, 72 °C como período de elongación por 2 minutos, y 5 minutos a 72 °C como extensión final. Los productos PCR fueron secuenciados en un laboratorio comercial (Genome Express, Grenoble, Francia). El alineamiento de secuencias individuales fue realizada con el programa Sequence Assembly (Lasergene, DNASTAR Inc., Madison, EE.UU). Procesamiento de secuencias y alineamiento de las mismas siguen las recomendaciones de Kelchner (2000): cuatro inserciones/deleciones (INDELs) y tres inversiones detectadas en las secuencias estudiadas fueron adicionalmente codificadas en una matriz binaria. Los análisis de maximum parsimonia (MP) fueron calculados sobre una matriz de bases (adenina, guanina, citosina, timina) y con la matriz binaria adicional para inserciones/deleciones e inversiones. El análisis aplicó un método heurístico con un stepwise addition y tree bisection-reconection algorithms (TBR) según la metodología implementada por el programa PAUP Versión 4.0b10 (Swofford, 2003). El soporte estadístico de cada clado fue evaluado a través de análisis de bootstrap con 1000 repeticiones. Con el objetivo de observar la similitud total entre taxa se realizó un análisis de Neighbor-joining utilizando la distancia de Jukes-Cantor. Valores bootstrap fueron calculados sobre 1000 repeticiones. El análisis Neighbor-joining fue realizado con el programa PAUP Versión 4.0b10 (Swofford, 2003).
Resultados
El espaciador trnD-trnT incluyó 927 pares de bases, con apenas 17 (1,8%) caracteres filogenéticamente informativos. Dos inserciones/deleciones de 2 y 30 pares de bases fueron detectados en esta secuencia: la primera inserción/deleción fue observada para dos especies de Oenocarpus (O. distichus, O. mapora); y la segunda estuvo restringida al clado Oenocarpus-O. bataua-Euterpe. Adicionalmente, una pequeña inversión de 6 pares de bases fue detectada en varias especies incluidas en el estudio. El espaciador trnQ-rps16 comprendió 1232 pares de bases, con 19 caracteres (1,5%) filogenéticamente informativos. Una pequeña inversión de 2 pares de bases fue detectada en este espaciador, la cual fue polimórfica para la tribu Euterpeae. Del espaciador psbC-trnfM fueron secuenciados 1464 pares de bases, con 13 caracteres (0,88%) filogenéticamente informativos. Dos pequeñas inversiones de 7 y 3 pares de bases fueron identificadas en este espaciador. Adicionalmente, una inserción/deleción de 6 pares de bases fue detectado para una especie de Euterpeae (Euterpe precatoria) y en Geonoma interrupta.
Una matriz de 3630 pares de bases (3623 pares de bases + 4 inserciones/deleciones + 3 inversiones), provenientes de tres espaciadores intergénicos secuenciados, con un 1,5% de caracteres filogenéticamente informativos fue utilizada en el análisis filogenético. El Análisis de Parsimonia de las secuencias intergénicas generó 26 árboles parsimoniosos, los cuales fueron construidos a través de 252 pasos, con un índice de consistencia de 0,60 y un índice de retención de 0,75. Un cladograma representativo de estos 26 árboles y el árbol de consenso (stricto consensus) es representado en la figura 1 con sus respectivos valores bootstrap.
La monofilia de la tribu Euterpeae fue resuelta con un bajo soporte estadístico (valor bootstrap = 52%). Los individuos de la especie O. bataua fueron incluidos dentro de un mismo clado con otros especímenes de Oenocarpus, sin embargo el clado Oenocarpus + O. bataua obtuvo un valor bootstrap menor al 50%. Euterpe fue resuelto como un grupo hermano de Oenocarpus (valor bootstrap = 65%). Hyospathe y Prestoea aparecen como un grupo monofilético (valor bootstrap < 50%). Las dos especies de Prestoea incluidas en el estudio formaron un grupo monofilético pero con un bajo soporte estadístico (valor bootstrap = 64%). Neonicholsonia es resuelta como hermana de los otros géneros de la tribu (valor bootstrap = 52%).
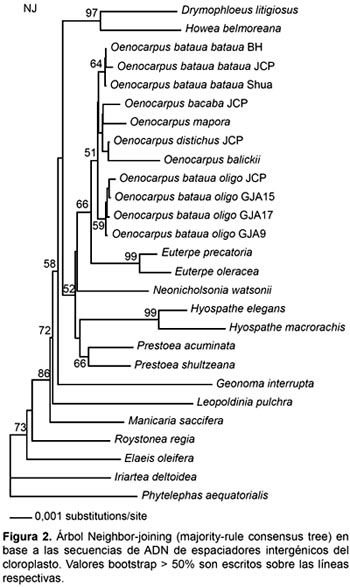
Dentro del clado Oenocarpus + O. bataua (Fig. 1B), los individuos del género Oenocarpus y de la especie O. bataua formaron una politomia. Un patrón de diferenciación molecular entre los individuos de O. bataua fue detectado. Los individuos de O. bataua var. bataua y aquellos de la var. oligocarpus formaron dos grupos monofiléticos separados sugiriendo la presencia de sinapomorfías para cada una de estas variedades; sin embargo, el soporte estadístico de este patrón permanece bajo (clado oligocarpus = 57% valor bootstrap, clado bataua = 64% valor bootstrap; Figura 1). Estos dos clados se diferenciaron por cuatro substituciones en el espaciador TrnD-TrnT and TrnQ-rps16 y por un polimorfismo de inversión de 7 pares de bases en el espaciador psbC-trnfM. El análisis Neighbor-joining muestra similares patrones de asociación entre taxa a los observados por el análisis filogenético (Fig. 2).
Discusión
Las secuencias intergénicas del cloroplasto utilizadas en nuestro estudio revelaron bajas tasas de divergencia molecular para las especies de la tribu Euterpeae (1,5% de caracteres filogenéticamente informativos). Los bajos niveles de diferenciación molecular en palmeras han sido previamente reportados por Wilson et al. (1990) y Baker et al. (1999). Sin embargo, en la tribu Euterpeae el nivel de diferenciación de las secuencias cloroplásticas estudiadas es aún más bajo comparado con el número de caracteres filogenéticamente informativos proporcionado por estas mismas secuencias para otros grupos de palmeras (Couvreur, comentario personal).
La filogenia molecular reporta de manera preliminar el origen monofilético de la tribu Euterpeae. Este resultado debe ser considerado como una evidencia molecular que soporta los resultados de Henderson (1999), quién propuso la monofilia de este grupo de palmeras basado en caracteres morfológicos. Sin embargo, la evidencia de este estudio debe ser considerada como preliminar debido a la ausencia de soporte estadístico en los patrones observados.
A nivel de géneros, cuatro grupos monofiléticos correspondientes a cada género de la tribu Euterpeae (Hyospathe, Euterpe, Prestoea, Oenocarpus + O. bataua) y una ramificación para Neonocholsonia fueron obtenidos; sin embargo, las relaciones filogenéticas entre géneros fueron parcialmente resueltas. Comparado con el trabajo de Henderson (1999), el cual que constituye el único estudio filogenético de referencia para la tribu Euterpeae se obtienen los siguientes comentarios. (1) Los clados de Hyospathe y Prestoea formaron un grupo monofilético en este estudio. Este resultado es controversial, ya que a nivel morfológico se ha sugerido que Prestoea es más próxima a Euterpe que a Hyospathe (Burret, 1929 en Henderson, 1999). (2) Euterpe constituye el género hermano del clado Oenocarpus + O. bataua; mientras que, Henderson (1999) reporta a Prestoea como el género hermano del clado Oenocarpus + Jessenia. (3) Neonicholsonia es el grupo hermano del clado Hyospathe + Euterpe + Prestoea + Oenocarpus + O. bataua. Los datos morfológicos de Henderson (1999) sugieren a Euterpe como el género hermano para todos los géneros de Euterpeae. (4) El resultado más relevante de esta filogenia molecular fue la inclusión de los especímenes de O. bataua y Oenocarpus en un mismo clado pero con ausencia de soporte estadístico. En base a las secuencias cloroplásticas estudiadas, no se detectaron sinapormorfias que diferencien O. bataua de otras especies del género Oenocarpus como dos entidades genéricas diferentes. Por consiguiente, no existe un soporte molecular en nuestros resultados que justifique la transferencia de O. bataua al género Jessenia con la combinación J. bataua, como fue postulado por Balick (1986) a partir de datos morfológicos y bioquímicos. Este resultado coincide con el trabajo de Henderson (1999).
A nivel intraespecífico, los individuos de Oenocarpus bataua var. bataua y O. bataua var. oligocarpus fueron agrupados en dos clados distintos (bataua y oligocarpus). Un patrón similar fue observado cuando se compara la similitud total de las secuencias entre los taxones (Fig. 2). Estos resultados sugieren una divergencia biológica entre las variedades bataua y oligocarpus; divergencia que ha sido previamente reportada por Grisebach y Wendland (1864 en Balick, 1986) en base a caracteres morfológicos. Grisebach y Wendland (1864 en Balick, 1986) reconocieron a las actuales variedades bataua y oligocarpus como dos especies distintas (J. bataua /O. bataua, y J. oligocarpus /O. oligocarpus). Si bien, la separación taxonómica entre estas especies fue basada en caracteres morfológicos ambiguos, los datos moleculares proporcionan una evidencia molecular preliminar a favor de una mayor divergencia biológica entre bataua y oligocarpus. Los resultados de este estudio sugieren la revisión de la hipótesis de Grisebach y Wendland (1864) en base a detallados estudios morfológicos y moleculares que permitan detectar el nivel de diferenciación biológica entre estas categorías intraespecíficas.
Conclusión
Los resultados de nuestro estudio permanecen preliminares debido a la falta de soporte estadístico. Es probable que con la inclusión de nuevas regiones cloroplásticas o nucleares se obtengan mayores niveles de resolución filogenética y soporte estadístico. Los resultados expuestos en este trabajo rescatan hipótesis antiguas sobre las relaciones evolutivas dentro de esta tribu de palmeras neotropicales. La evidencia molecular proporcionada en este trabajo, y la filogenia con caracteres morfológicos desarrollada por Henderson (1999) justifican el tratamiento taxonómico de Jessenia y Oenocarpus como una sola entidad genérica. Es necesario normalizar el uso correcto de los nombres taxonómicos para las especies de Oenocarpus y en particular para O. bataua y sus variedades; aún hoy en día, publicaciones científicas usan de forma indistinta nombres taxonómicos que causan confusión sobre la especie objeto de estudio. En relación a las categorías intraespecíficas de Oenocarpus bataua, se abre una nueva discusión en relación al estado taxonómico de las mismas. Un mayor esfuerzo dirigido a la caracterización morfológica, genética y la biología floral de las poblaciones de O. bataua permitirá definir el nivel taxonómico de las variedades bataua y oligocarpus.
Agradecimientos
Agradecemos a Betty Millán de la Universidad Nacional Mayor San Marcos (Perú) por su ayuda durante este proyecto. A Jean-Jacques de Granville y el personal del Herbario de Cayena por su apoyo durante la fase de campo en Guyana Francesa. A Andrew Henderson, Bill Hahn y Conny Assmusen quienes compartieron algunas secuencias con nosotros.
Literatura Citada
Asmussen C.B., J. Dransfield, V. Deickmann, A. Barfod, J.-C. Pintaud & W. Baker. 2006. A new subfamily classification of the palm family (Arecaceae): evidence from plastid DNA phylogeny. Botanical Journal of the Linnean Society 151: 15-38.
Asmussen C.B. & M.W. Chase. 2001. Coding and noncoding plastid DNA in palm systematics. American Journal of Botany 88: 1103–1117.
Baker W., C. Asmussen, S. Barrow, J. Dransfield & T. Anderson. 1999. A phylogenetic study of the palm family (Palmae) based on chloroplast DNA sequences from the trnL-trnF region. Plant Systematics and Evolution 219: 111–126.
Balick, M.J. 1986. Systematics and Economic Botany of the Oenocarpus-Jessenia (Palmae) complex. Advances in Economic Botany 3: 1–140.
Bernal R., G. Galeano & A. Henderson. 1991. Notes on Oenocarpus (Palmae) in the Colombian Amazon. Brittonia 43: 154–164.
Burret M. 1928. Die palmengattungen Oenocarpus Mart. und Jessenia Karst., nebst bemerkungen zu Archontophoenix H.Wendl. et Drude (einsichliesslich Loroma O.F.Cook). Notizbl. Bot. Gart. Berlin-Dahlem 10: 291–312.
Couvreur T., N. Billote, A.-M. Risterucci, C. Lara, Y. Vigouroux & et al. 2006. Close genetic proximity between cultivated and wild Bactris gasipaes Kunth revealed by microsatellite markers in Western Ecuador. Genetic Resources and Crop Evolution 53: 1361-1373.
Demesure B., N. Sodzi & J. Petit. 1995. A set of universal primers for amplification of polymorphic non-coding regions of mitochondrial and chloroplast DNA in plants. Molecular Ecology 4: 129–131.
Govaerts R. & J. Dransfield. 2005. World Checklist of Palms. Royal Botanic Gardens Kew.
Grivet D., B. Heinze, G.G. Vendramin & J. Petit. 2001. Genome walking with consensus primers: application to the large single copy region of chloroplast DNA. Molecular Ecology Notes 1: 345-349.
Hahn W. 2002. A phylogenetique analysis of the Arecoid Line of palms based on plastid DNA sequence data. Molecular Phylogenetics and Evolution 23: 189–204.
Henderson A. 1999. A phylogenetic analysis of Euterpeinae (Palmae) based on morphology and anatomy. Brittonia 51: 106–113.
Henderson A. 1995. The palms of the Amazon. Oxford: Oxford University Press.
Karsten H. 1857. Plantae Columbianae. Linnaea 28: 387–462.
Kelchner S.A. 2000. The evolution of non-coding chloroplast DNA and its application in plant Systematics. Annals of Missouri Botanical Garden 87: 482–498.
Martius K. P. F. von. 1823. Historia naturalis palmarum. Vol. 2. T. O. Weigel, Leipzig.
Swofford D.L. 2003. PAUP*: phylogenetic analysis using parsimony (* and other methods), version 4b10. Sinauer, Sunderland, Massachusetts, USA.
Uhl N.W. & J. Dransfield. 1987. Genera Palmarum. Allen Press, Lawrance, Kansas, USA.
Wessels Boer J.G. 1965. The indigenous palms of Suriname. E. J. Brill, Leiden.
Wilson M.A., B. Gaut & M.T. Clegg. 1990. Chloroplast DNA evolves slowly in the palm family (Arecaceae). Molecular Biology and Evolution 7: 303–314.

















