Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. v.15 n.2 Lima 2008
TRABAJOS ORIGINALES
Identificación molecular de Pichia guillermondii aislada de aguas ácidas de minas en el Perú y su resistencia a metales pesados
Molecular identification of Pichia guillermondii isolated from mine water acidic of Peru and its resistance to heavy metals
Jeanette Orbegozo, Michel Abanto, Ruth García y Pablo Ramírez
Laboratorio de Microbiología Molecular, Facultad de Ciencias Biológicas Universidad Nacional Mayor de San Marcos. Apartado 11-0058, Lima 11, Perú. E-mail Pablo Ramírez pramirezr@unmsm.edu.pe
Resumen
Las aguas ácidas de minas tienen una considerable diversidad de microorganismos eucariontes, entre ellos hongos y protistas; en particular, poco se conoce sobre la diversidad de levaduras en drenajes ácidos de minas peruanas. En el presente estudio se aisló y caracterizó la levadura 1MA9, mediante la amplificación y secuenciación del LSU D1/D2 del gen rRNA 26S. La cepa 1MA9 se identificó molecularmente como Pichia guillermondi, ésta presentó una resistencia alta a Mn2+ (>400 mM), intermedia a iones Zn2+ y Co2+ (400600 mM), pero fue sensible a iones Cu2+. Este trabajo representa el primer reporte de levaduras en ambientes acuáticos ácidos proveniente de minas peruanas.
Palabras claves: Pichia guilliermondii, LSU D1/D2, agua ácida de mina (AMD), resistencia a metales, levaduras.
Abstract
Microbe eukaryotes like fungi and protista are common in the drainage of mines. Few information is known on microbial diversity of acid drainages of Peruvian mines. In this work we isolated and characterized the 1M9 yeast. We utilized LSU D1/D2 of the 26S rRNA gene sequence phylogenetic analyses to characterize the diversity the yeast 1MA9 isolated. The strain 1MA9 was closely related to the Pichia guilliermondii. The yeast showed high resistance to Mn2+ (>400 mM), intermediate to ions Zn2+ and Co2+ (400600 mM), but sensible to ions Cu2+. This work provides the first data on yeasts from an aquatic acid environment of Peruvian mines.
Keywords: Pichia guilliermondii, LSU D1/D2, acid mine drainage, Heavy metals resistance, yeast.
Introducción
En las zonas mineras podemos encontrar las denominadas aguas ácidas (pH<3), las que contienen comunidades microbianas quimiolitotróficas que obtienen su energía de la oxidación de la pirita (FeS2) y producen H2SO4, convirtiendo al medio en extremadamente ácido. Además, estas aguas están asociadas a una alta concentración de metales, donde están presentes diversas comunidades de acidófilos extremos, entre ellas las levaduras. Pichia guilliermondii es una levadura con un estado vegetativo conocido como teleomorfo y habitan en ambientes terrestres y acuáticos (Russo et al., 2006), y poco se conoce sobre su diversidad en aguas ácidas de minas (AMD).
El empleo de técnicas convencionales como las pruebas microbiológicas de asimilación de carbono, de compuestos nitrogenados y requerimientos vitamínicos, son comunmente usadas para la identificación de levaduras, pero éstas no siempre son precisas ni reproducibles, y suelen llevar a datos inespecíficos debido a que las características morfológicas y fisiológicas están fuertemente influenciadas por las condiciones de cultivo. Es por ello, que en la actualidad el empleo de técnicas moleculares tales como análisis de microsatélites, el polimorfismo de longitud del DNA mitocondrial, el polimorfismo de longitud de los fragmentos de restricción del RNA ribosomal y el análisis de RNA de bajo peso molecular entre otros (Orberán, 2004), son la combinación de exactitud, rapidez y factibilidad que hacen de ellas herramientas ideales en la identificación de microorganismos.
En este reporte se emplea en la identificación y análisis filogenético de un aislado levaduriforme 1MA9, de 600 nucleótidos de la región variable (D1/D2) del extremo 5´ del gen de la subunidad mayor del rDNA (LSU). El LSU es parte del complejo de genes del rDNA y esta constituido por secuencias repetidas en tandem, organizándose en grupos los genes ribosomales en el genoma nuclear. Los genes ribosomales son altamente conservados, actualmente se sabe que están compuestos de una mezcla de regiones conservadas y divergentes, éstas últimas son llamadas regiones divergentes -D y tienen una orientación que va de 5 a 3 del rRNA maduro (Sonnenberg, 2007). Esta región ha sido secuenciada en la mayoría de ascomicetos y es común a todas las especies de levaduras, permitiendo un reconocimiento intraespecífico entre ellas, por ser una región altamente variable ayuda a una mejor identificación de las especies. En el caso de especies fuertemente relacionadas además es recomendable el secuenciamiento de la región del espacio intergénico (ITS) (Hall et al., 2003).
El uso de levaduras en el tratamiento de ambientes contaminados con metales pesados, resulta en la biosorción de los agentes tóxicos por la biomasa celular y/o el transporte dentro de la célula (Kaszycki et al., 2004). Esta capacidad se ha estudiado en Saccharomyces cerevisiae, que acumula cromo, cadmio y otros metales. También se ha reportado una cepa de Candida sp. resistente a Cr (VI), una de Rhodosporidium sp. aislada de desechos industriales y cepas silvestres de Pichia guilliermondii que son utilizadas en procesos de bioremediación de Cr (III) y Cr(VI) ya que presentan una alta tolerancia a éstos (Ksheminska et al., 2003).
El objetivo de este trabajo fue identificar y determinar los niveles de resistencia a metales pesados de una cepa de P. guilliermondii 1MA9 aislada de aguas ácidas de la minera Yanacocha con la finalidad de utilizarla en procesos de biorremediación y bioadsorción.
Materiales y métodos
Muestreo y aislamiento de levaduras
Se colectaron en botellas estériles, seis muestras de aguas ácidas de 300 mL de diferentes puntos de la Minera Yanacocha. Para el aislamiento de las levaduras, estas se sembraron por estriado en agar Saboraud 2% modificado, este medio tuvo la siguiente composición (g/L): 10 de peptona, 20 de glucosa y 20 de agar-agar. La modificación consistió en la adición de 4% de medio 9K para obtener un pH final de 4. Los cultivos se incubaron a 28 oC y por 5 días.
Extracción de DNA genómico
Para la extracción de DNA genómico se cultivó la levadura en medio líquido Saboraud 2% modificado y se utilizó el protocolo para levaduras del Wizard Genomic DNA Purification kit (Promega©). Este protocolo se basa en una lisis celular mediante enzimas, precipitación de DNA genómico con isopropanol, seguido de digestión enzimática de RNA y solubilización. Brevemente, se centrifugó un cultivo crecido por 20 h en medio líquido GPY a 13000×g por 2 min y el pellet se resuspendió en 293 μL de 50 mM EDTA. Seguidamente para digerir la pared celular se añadió 7,5 μL de liticasa (Sigma©, 20 mg/mL) y se incubó a 37 °C por 60 min. Se centrifugó a 13000×g por 2 min y se eliminó el sobrenadante. Al pellet resultante se añadió 300 μL de solución de lisis, seguidamente se añadió 100 μL solución de precipitación de proteínas y se incubó en hielo por 5 min para posteriormente centrifugar a 13000×g por 3 min. Posteriormente, al sobrenadante conteniendo el DNA se añadió 300 μL de isopropanol y se centrifugó a 13000×g por 2 min. Al pellet se añadió 300 μL de etanol al 70% y se centrifugó a 13000×g por 2 min y se recuperó el DNA secando el tubo por 15 min. Posteriormente se eliminó el RNA incubando las muestras con 1,5 μL de RNAsa a 37 °C por 15 min. Finalmente se almacenó el DNA a 4 °C.
Iniciadores y condiciones de PCR
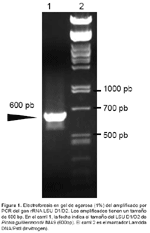
Las secuencias de los iniciadores para amplificar la región D1/D2 LSU ribosomal de Pichia guilliermondii fueron: F63 (5`-GCATATCAATAAGCGGAGGAAAAG-3`) y LR3 (5`-GGTCCGTGTTTCAAGACGG-3`). Para amplificar la secuencia D1/D2 LSU usamos el siguiente protocolo de PCR: 3 min a 95°C; 30 s a 95 °C; 30 s a 57 °C; 2 min a 72 °C, seguido de 30 ciclos, 5 min a 72 °C y finalmente tiempo indefinido a 4 °C. El tamaño del amplificado esperado fue de 600 bp.
Electroforesis de DNA en geles de agarosa
Para visualizar el DNA cromosomal o productos de PCR utilizamos geles de agarosa ultrapura (Gibco BRL©) al 1% en amortiguador TAE 0,5× (20 mM Tris-acetato pH 8,0; 0,5 mM EDTA). Las muestras de DNA se mezclaron con solución de carga (0,25% azul de bromofenol, 40% sacarosa). Como marcadores de estándares de peso molecular se utilizó Lamdda DNA/PstI (Invitrogen). La tinción se realizó durante 10 min en una solución de TAE 0,5× con bromuro de etidio a una concentración 0,5 μg/mL. Las bandas se visualizaron por la fluorescencia emitida al irradiar con luz ultravioleta de 320 nm.
Secuenciación e identificación molecular
La secuenciación se llevó a cabo por la empresa MacroGen USA y se depositó en la base de datos (GeneBank/EMBL/DDJB) con el número de acceso AM887520. La secuencia obtenida se comparó con las existentes en las bases de datos mediante el programa BlastN y se observó un 98% de identidad con P. guilliermondii YQ2, (Número de acceso EF644481) que nos permite afirmar que pertenece a esta especie.
Análisis filogenético
El análisis filogenético y evolutivo molecular fue llevado a cabo usando el programa MEGA v3.1 (Kumar et al., 2004). Previamente se hizo una búsqueda de las secuencias depositadas en Ribosomal Data Project y GenBank usando el programa BLASTN v2.0 (Altschul et al., 1997). Luego se realizó un alineamiento múltiple usando el programa CLUSTAL X (Thompson et al., 1997). La distancia evolutiva fue calculada mediante el modelo Kimura´s dos-parámetros, los árboles filogenéticos fueron reconstruidos por el método neighbour-joining y la topología y consistencia de los árboles fue evaluada por bootstrapping con 1000 réplicas utilizando el programa MEGA v3.1. Las posiciones de alineamiento con gaps y bases no identificadas fueron excluidas de los cálculos.
Evaluación de la resistencia a metales pesados
Se empleó el medio GPY modificado a pH 3, con la siguiente composición (g/L): 30 de glucosa, 10 de peptona de caseína y 5 de extracto de levadura, el pH del medio fue ajustado con 4,5 M de H2SO4 (1,1 mL de ácido para 100 mL de GPY; Nguyen et al., 2001). Este medio fue suplementado con zinc, manganeso, cobalto y cobre a una concentración final de 50, 100, 300, 400 y 500 mM. Los cultivos se incubaron a temperatura ambiente en agitación rotatoria a 170 rpm por 48 horas. El recuento de unidades formadoras de colonia en presencia de las distintas concentraciones de iones metálicos se realizó en placas de agar GPY a pH 3,0 (Nguyen et al., 2001).
Resultados
Aislamiento e identificación molecular de P. guilliermondii
El aislado 1MA9 se cultivó en medio Sabouraud glucosado al 2% y estos se sometieron a la identificación molecular mediante PCR y secuenciamiento del amplificado de 600 pb de la región variable (D1/D2) del gen LSU (Fig. 1), el cual ha sido usado en la identificación de la mayoría de levaduras ascomicetas. Al realizar la comparación de esta secuencia con otras existentes en la bases de datos se observó un 98% de identidad con la secuencia correspondiente a P. guilliermondii YQ2 (número de acceso EF644481) aislado de China. Por lo tanto, el análisis bioinformático de la secuencia del gen LSU D1/D2 permitió determinar la especie de la cepa aislada.
Filogenia molecular de P. guilliermondii 1MA9
En la mayoría de los estudios de sistemática molecular se utiliza solamente las primeras 600900 bases del gen LSU, que incluye los tres dominios divergentes (D1, D2, D3) que están entre las regiones más variables del gen completo.
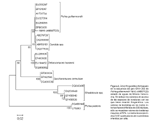
Según el análisis filogenético del gen LSU D1/D2 rRNA las levaduras se encuentran distribuidas en ramas o clados discretos según la clase a la cual pertenecen. La relación filogenética entre la cepa 1MA9 aislada en el presente estudio con otros aislados de otras partes del mundo se muestra en la figura 2, en ella se puede apreciar que la cepa en estudio está estrechamente relacionada con el clado de la especie P. guilliermondii formando un clado robusto (bootstrap de 1000), separado de la otra especie P. pastoris. A su vez podemos observar que todas las cepas de Candida sp., incluidas en nuestro análisis, conservan una relación con la cepa 1MA9.
Anteriores reportes dan cuenta de la presencia del género Cándida sp., en este tipo de ambientes y comentan que este es el mas abundante grupo de levaduras en un amplio rango de medios ambientes ácidos como en el Río Tinto, España (López et al., 2004), y otras especies que no han sido fáciles de identificar fueron incluidas en este género, tal vez por ello levaduras como la identificada en este trabajo no han sido reportados con anterioridad.
Por otro lado se observó que la secuencia del gen LSU de la cepa P. guilliermondii 1MA9, corresponde a un agrupamiento o cluster que exhiben una ramificación relativa con las cepas EU056297, EF191047, EF063135 y otros pertenecientes al género Pichia, formando un clado robusto (bootstrap de 1000), a su vez este clado se relaciona estrechamente con Candida, pero ambas ramificaciones se encuentran más relacionadas entre si que con otros géneros. Esta fuerte relación se puede deber a que P. guilliermondii es un estado asexual llamado teleomorfo del género Candida.
Este análisis filogenético es de interés para la tipificación de aquellos organismos cuya identidad es cuestionable o pueden determinarse por métodos fenotípicos, además nos permite tener un registro confiable de secuencias de nuevos aislados o especies raras que se podrán reconocer sólo por sus secuencias de ácidos nucleicos existentes en el GeneBank.
Tolerancia a metales pesados
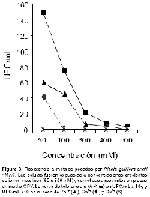
La cepa 1MA9 presentó elevados niveles de tolerancia a Mn2+ con un MIC >400mM y una gran sensibilidad a Cu2+ (<50mM), esto se ve reflejado en el número de colonias (UFC/ml) que se contabilizó en cada caso (Fig. 3). Interesantemente, se obtuvo mayores recuentos en agar GPY (pH 3) en el caso del Mn2+, en el cual a 50 mM se obtuvieron más de 14000 UFC/ml mientras que en el caso de Zn2+ y Co2+ se obtuvieron recuentos de entre 400 y 600 UFC/ml que representan menos de 20 veces en comparación al Mn2+. En cambio, la cepa en estudio resulto muy sensible a concentraciones de Cu2+ (≥ 50 mM), (Fig. 3).
Discusión
Las levaduras son de amplia distribución y sobreviven en ambientes tan hostiles por su tolerancia al stress, al calor, presión osmótica, oxidación, congelamiento, elevada concentración de metales. Esto se debe a múltiples factores entre ellos su genoma, los genes conocidos como heat-shock (choque térmico) juegan un importante rol en el incremento de la tolerancia a estas formas de stress; la composición y morfología de las hifas se cree que les sirvan para desarrollarse en zonas mineras, permitiéndoles anclarse en biofilms sobre pirita, además de producir iones carbonatos disueltos como alimento para los procariontes acidofilos quimiolitoautotroficos (Baker et al., 2004).
Cabe resaltar que la mayoría de investigaciones se orientan a estudiar a microorganismos procariontes y sólo unos pocos trabajos se han centrado en el estudio de la biodiversidad de eucariontes en estos ambientes. Por ello nuestro trabajo se enfocó en rescatar la importancia de estos organismos y hallamos una cepa con una elevada tolerancia al Mn2+ muy por encima de los demás metales de transición empleados en nuestra investigación (500 mM). Esto se debería a la importancia del manganeso en la actividad de diversas enzimas vitales para la célula, como las que se ubican en el aparato de Golgi, o en vesículas post-Golgi que son importantes para el correcto funcionamiento de la glicolisación de la membrana plasmídica y secreción de proteínas. Recientemente se ha demostrado que el complejo de glicosilación es vital para las levaduras y que la actividad de este complejo es dependiente de manganeso. Por lo tanto la deficiencia de este metal en estos compartimientos resulta en la falta de desarrollo de la levadura (Cohen et al., 2000).
La activación de vías de detoxificación de manganeso, controlada por el gen pmr1p, ayuda a eliminar los niveles tóxicos de éste, por un bombeo en el aparato de Golgi, logrando que el manganeso sea secretado. Trabajos con cepas mutantes para este gen, no son sólo sensibles al manganeso sino a metales como zinc, cobalto y cobre. En investigaciones similares en S. cerevisiae, se encuentra una respuesta ante la toxicidad del manganeso, con el secuestro del metal en las vacuolas. Siendo la participación de las vacuolas un importante mecanismo en el almacenamiento y detoxificación del manganeso en levaduras (Yang et al., 2005).
Por otro lado, la respuesta de aislado 1MA9 frente al ion zinc también fue satisfactoria esto se debería a la participación del zinc en el ensamblaje de cientos de proteínas como las polimerasas. Las polimerasas de las levaduras y algunos procariontes tienen zinc en su estructura formando los dedos de zinc. Otra enzima que contiene zinc es la fosfatasa alcalina, también esta presente en cofactores catalíticos. Pero la toxicidad del zinc puede involucrar la competición con otros iones por la actividad de sitios activos, enzimas o de proteínas de transporte intracelular, investigaciones recientes han reconocido que la resistencia al zinc es regulada por el gen ZRC1, que codifica una proteína de la membrana vacuolar, y que esta presente en bacterias, plantas y mamíferos. La proteína Zrc1 es requerida para la resistencia a más de 4 mM de zinc, sin embargo bajo condiciones de escasez de este metal la proteína Zrc1 se requiere para tolerar no más de 1 μM de zinc (Colin et al., 2003).
El comportamiento del aislado 1MA9 frente al cobalto es similar a otros reportes que explican la interacción de este metal con el hierro, en el caso de S. cerevisiae esto es regulado por el factor transcripcional Aft1p, el cual juega un rol importante en la tolerancia al cobalto, la permanente inducción de esta proteína conlleva a la resistencia del cobalto, se sabe que esta resistencia se correlaciona con el incremento intracelular del hierro y la sensibilidad de las células frente al cobalto se debe a la falta de acumulación del mismo. Además, el elevado nivel del hierro en el medio de cultivo de células mutantes para el gen AFT1 suprime la sensibilidad frente al cobalto, esto permite el incremento de la asimilación de cobalto a nivel intracelular (Jochen y Schweyen, 2002).Por lo expuesto, se puede asumir que la presencia del hierro en la resistencia del cobalto es vital, y en nuestro caso la falta de hierro en la composición del medio GPY usado en la evaluación de la resistencia del cobalto haya atribuido una baja respuesta a este metal esto se evidencia en el bajo número de unidades formadoras de colonias (UFC) (Fig. 3).
El comportamiento del aislado 1MA9 frente al ion cobre es similar a las observaciones realizadas en S. cerevisiae, resultando que el cobre es nocivo a altas concentraciones y la resistencia se hace presente en un intervalo de 210 mM, siendo un desencadenante apoptótico a concentraciones altas (Liang y Zhou, 2007). Cabe resaltar que la afinidad a este metal, al menos en levaduras, es mediado por dos proteínas de transporte de membrana codificado por los genes CTR1 y CTR3, y en recientes investigaciones en Schizosaccharomyces pombe se ha descubierto la participación de dos nuevos genes que codifican las proteínas Ctr4 y Ctr5 de la membrana plasmática y tendrían participación en el complejo de transporte de cobre (Zhou y Thiele, 2001).
Una herramienta importante en la identificación de nuestro aislado fue el gen LSU D1/D2 del rRNA. Existen reportes que respaldan el uso de esta secuencia en este tipo de estudio. Esta región en un buen marcador taxonómico por su alta divergencia (01%) en el domino D2 y que de acuerdo a nuestros resultados nos ha permitido la asignación de una taxa a la cepa 1MA9. Asimismo, la secuencia utilizada en la identificación de esta levadura ha permitido a otros autores diferenciar cepas coespecíficas y especies emparentadas como es el caso de Issatchenkia, Pichia y Saccharomyces (Daniel y Meyer, 2003).
Literatura citada
Altschul S., T. L. Madden, A. Schäffer, J. Zhang, Z. Zhang, W. Miller & D. Lipman. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucl Acids Res, 25:3389–3402.
Baker B., M.A. Lutz, S.C. Dawson, P.L. Bond & J.F. Banfield1. 2004. Metabolically active eukaryotic communities in extremely acidic mine drainage. Appl. Environ. Microbiol. 70:6264–6271.
Cohen A., H. Nelson & N. Nelson. 2000. The family of SMF metal ion transporters in yeast cells. J. Biol. Chem. 275: 33388-33394.
Colin W. M., M.A. Milanick & D.J. Eide. 2003. Induction of the ZRC1 Metal Tolerance Gene in Zinc-limited Yeast Confers Resistance to Zinc Shock. J. Biol. Chem. 278:15065-15072.
Daniel H.-M. & W. Meyer. 2003. Evaluation of ribosomal RNA and actin gene sequences for the identification of ascomycetous yeasts. Int. J. Food Microbiol. 86:61-78.
Hall L., S. Wohlfiel & G.D. Roberts. 2003. Experience with the MicroSeq D2 large-subunit ribosomal DNA sequencing kit for identification of commonly encountered, clinically important yeast species. J. Clin. Microbiol. 41: 5099-5102.
Jochen A.S. & R.J. Schweyen. 2002. The Yeast Iron Regulon Is Induced upon Cobalt Stress and Crucial for Cobalt Tolerante. J. Biol. Chem. 277:39649-39654.
Kaszycki P., D. Fedorovych, H. Ksheminska, L. Babyak, D. Wójcik, H. Koloczek & P.Kaszycki. 2004. Chromium accumulation by living yeast at various environmental conditions. Microbiol. Res. 159:11-17.
Ksheminska H., A. Jaglarz, D. Fedorovych, L. Babyak, D. Yanovych, P. Kaszycki & H. Koloczek. 2003. Bioremediation of chromium by the yeast Pichia guilliermondii: toxicity and accumulation of Cr (III) and Cr (VI) and the influence of riboflavin on Cr tolerance. Microbiol Res. 158: 59-67.
Kumar, S., K. Tamura & M. Nei. 2004. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Briefings in Bionformatics 5: 150-163.
Liang Q & B. Zhou. 2007. Copper and Manganese Induce Yeast Apoptosis via Different Pathways. Mol. Biol. Cell.18:4741-4749.
López A.I., A.E. González, M.C. Terrón & R. Amils. 2004. Ecological study of the fungal populations of the acidic Tinto River in southwestern Spain. Can. J. Microbiol. 50:923-34.
Nguyen V.A.T., K. Senoo, T. Mishima & M. Hisamatsu. 2001. Multiple tolerances of Rhodotorula glutinis R-1 to acid, aluminum ion and manganese ion, and its unusual ability of neutralizing acidic medium. Journal of Bioscience and Bioengineering 92:366-371.
Orberán T. 2004. Métodos moleculares de identificación de levaduras de interés biotecnológico. Rev. Iberoam. Micol. 21:15-19.
Russo G., D. Libkind, J. Sampaio & M. Van Broock. 2006. Levaduras del Río Agrio y El Lago Caviahue, un ambiente acuático ácido de origen volcánico (Neuquén, Argentina). Bol. Soc. Argent. Bot. 41: 167 - 175.
Sonnenberg R., A.W. Nolte & D. Tautz. 2007. An evaluation of LSU rDNA D1-D2 sequences for their use species identification. Front. Zool. 4:6.
Thompson J., T. Gibson, F. Pleioniak, F. Jeanmougin & D. Higgins. 1997. The Clustal_X interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucl. Acids Res. 25:4876–4882.
Yang M., L.T. Jensen, A.J. Gardner & V.C. Culotta. 2005. Manganese toxicity and Saccharomyces cerevisiae Mam3p, a member of the ACDP (ancient conserved domain protein) family. Biochem. J. 386: 479–487.
Zhou H.& D.J. Thiele. 2001. Identification of a Novel High Affinity Copper Transport Complex in the Fission Yeast Schizosaccharomyces pombe. J. Biol. Chem. 276:20529-35
___________________
Presentado: 04/03/2008
Aceptado: 09/05/2008