Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. v.17 n.3 Lima dic. 2010
Identificación molecular y actividad sobre sustratos cromogénicos de la venombina A del veneno de la serpiente peruana Bothrops atrox
Molecular identification and activity upon chromogenic substrates of a venombin A from Bothrops atrox Peruvian snake venom
Gustavo A. Sandoval1 ; Fanny Lazo1 ; Edith Rodriguez1 ; Armando Yarlequé1* y Russolina B. Zingali2
1 Laboratorio de Biología Molecular, Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos. Lima 11, Perú.
2 Laboratorio de Hemostasia y Venenos. Departamento de Bioquímica Médica. Universidad Federal de Rio de Janeiro. Rio de Janeiro - Brazil.
Resumen
En el presente trabajo se ha realizado la identificación molecular de la enzima similar a trombina (EST) del veneno de Bothrops atrox y se ha evaluado su actividad enzimática sobre diversos sustratos sintéticos. La enzima fue purificada utilizando tres pasos cromatrográficos, sobre Sephadex G-75, CM-Sephadex C-50 y Agarosa-PAB, determinándose su peso molecular por PAGE-SDS. La identificación molecular de la enzima aislada se realizó por la técnica de peptide mass fingerprinting basada en espectrometría de masas MALDI-TOF y posterior análisis in silico. Las actividades fibrinocoagulante y amidolítica fueron ensayadas sobre fibrinógeno bovino y BApNA, respectivamente, así como la hidrólisis sobre los sustratos cromogénicos específicos S-2238, S-2251 y S-2266. Como resultado de los ensayos bioquímicos y estructurales, la EST del veneno de B. atrox, presentó un peso molecular de 29,6 kDa. El análisis mediante espectrometría de masas de los péptidos obtenidos, permitió identificar a esta enzima como una venombina A, presentando una identidad del 75%. Del análisis de actividad enzimática, se obtuvo que la EST de B. atrox produjo coagulación del fibrinógeno bovino y presentó actividad sobre BApNA, S-2238 y S-2266, siendo incapaz de hidrolizar el sustrato S-2251. El empleo de estas aproximaciones estructurales y funcionales ha permitido lograr la identificación molecular del principal componente del veneno de B. atrox relacionado con su acción coagulante, así como evaluar en detalle la naturaleza de su actividad enzimática sobre diversos sustratos.
Palabras claves: Bothrops atrox, enzima similar a trombina, MALDI-TOF, sustratos cromogénicos.
Abstract
In this work, the thrombin-like enzyme (TLE) from Bothrops atrox has been identified by mass spectrometry and its enzymatic activity evaluated upon several synthetic substrates. The enzyme was purified to homogeneity using three chromatography steps on Sephadex G-75, CM-Sephadex C-50 and Agarose-PAB. Also, molecular weight by PAGE-SDS was determined. For molecular identification of this enzyme, mass spectrometry-based peptide mass fingerprinting was used and later in silico analysis. Enzymatic activities were determined using bovine fibrinogen, BApNA and also upon specific chromogenic substrates such as S-2238, S-2251 y S-2266. As a result of these biochemical and structural procedures, we obtained a TLE from B. atrox venom with a molecular weight of 29,6 kDa. Mass spectrometry analysis of obtained peptides, allow us to identify this enzyme as a venombin A, showing a 75% sequence homology. After recording enzymatic activity, this TLE showed coagulant activity on bovine fibrinogen and upon BApNA, S-2238 y S-2266, being unable to hydrolyze S-2251 substrate. Using this combination of structural and functional approaches, we have identified the main component of B. atrox venom related to its coagulant activity, as well as a detailed evaluation of its enzymatic activity upon several substrates.
Keywords: Bothrops atrox, Thrombin-like enzyme MALDI-TOF, chromogenic substrates.
Introducción
Las serpientes del género Bothrops habitan una extensa región de América y en el Perú se presentan hasta 17 especies, siendo el Jergón, Bothrops atrox (Linnaeus, 1758), la más abundante, peligrosa y causante del 90% de los accidentes ofídicos en el Perú (Loja et al. 2000, Yarlequé 2000). Como consecuencia de la mordedura se presenta dolor intenso en la zona afectada, edema, hemorragia y un severo descenso de la presión arterial que ocasiona la muerte; en otros casos una masiva necrosis que lleva a la amputación del miembro afectado (Lévano & Fernández 2004).
Estudios previos del veneno de B. atrox han determinado actividades esterásica, fibrinolítica y kininogenásica, las cuales se encuentran relacionadas con la marcada acción del veneno sobre la coagulación sanguínea (Loayza et al. 1985). Entre las enzimas relacionadas con esta acción se encuentran las similares a trombina (ESTs), las cuales de igual forma que la trombina, producen coágulos que bloquean la circulación sanguínea aunque su mecanismo de acción sea diferente, ya que preferentemente liberan sólo fibrinopéptidos A o B, mientras que la trombina produce la liberación de ambos fibrinopéptidos de la molécula del fibrinógeno, es decir, los fibrinopéptidos A y B (Braud et al. 2000; Lu et al. 2005). Esta enzima ha sido detectada en varios venenos botrópicos incluído B. atrox utilizando como sustratos plasma bovino como también fibrinógeno bovino y canino (Orejuela et al. 1991). En un estudio previo, Sandoval et al. (2010), lograron purificar la EST de B. atrox e identificar algunas propiedades bioquímicas como el peso molecular y el contenido de azúcares, así como su acción sobre ésteres de arginina como el BAEE.
Para el estudio de las propiedades estructurales y funcionales de las enzimas presentes en los venenos de serpiente se han usado diferentes aproximaciones siendo la más común la obtención de la secuencia de aminoácidos, así como la acción enzimática sobre sustratos naturales, las cuales tienen como desventaja el empleo de una gran cantidad de muestra para los respectivos análisis (Smolka et al. 1998). En este aspecto, el empleo alternativo de técnicas microanalíticas como la espectrometría de masas MALDI-TOF (matriz-assisted laser desortion/ionization - time-of-flight mass spectrometry) permiten la identificación de proteínas a través de la interpretación de los espectros de fragmentación de los péptidos generados a partir de su proteólisis (habitualmente con tripsina). Estos espectros son comparados con una base de datos de proteínas digeridas in silico y analizados con softwares específicos (ProFound, Mascot, etc.) (Ashcroft 2003). A su vez, el empleo de sustratos cromogénicos que imiten los sitios de corte de las enzimas sobre los sustratos originales permite lograr una mayor profundidad en el estudio del reconocimiento enzima-sustrato, importante en el diseño posterior de inhibidores sintéticos para estas proteínas (Castro et al. 2001).
En este contexto, en el presente trabajo se describe la identificación molecular de la enzima similar a trombina del veneno de B. atrox mediante espectrometría de masas MALDI-TOF, así como su actividad diferencial sobre diversos sustratos cromogénicos.
Materiales y métodos
Material Biológico.- Se utilizó el veneno de ejemplares adultos de Bothrops atrox procedentes de Pucallpa (Ucayali, Perú) y mantenidos en cautiverio en el serpentario Oswaldo Meneses del Museo de Historia Natural de la Universidad Nacional Mayor de San Marcos. El veneno fue extraído por presión manual de las glándulas venenosas, siendo luego liofilizado y conservado a 4 °C hasta su utilización en las pruebas experimentales correspondientes.
Cuantificación de proteínas.- La cantidad de proteína fue calculada midiendo la absorbancia de luz UV a 280 nm (Warburg & Christian 1941) en un espectrofotómetro Shimadzu UV 120-02. Además se empleó el método de Lowry et al. (1951) modificado (Loayza et al. 1985) utilizando un fotocolorímetro y albúmina sérica bovina como proteína estándar.
Purificación de la enzima similar a trombina.- Para la purificación de la EST del veneno de B. atrox se empleó la metodología según Sandoval et al. (2010). Se utilizó 200 mg de veneno crudo disuelto en buffer acetato de amonio 0,05 M, pH 6,0; el cual fue separado mediante filtración molecular en Sephadex G-75 (45,2 x 1,2 cm), luego por intercambio catiónico en CM Sephadex C-50 (35 x 1,2 cm) y finalmente usando cromatografía de afinidad en Agarosa-PAB (6 x 0,8 cm). Para el monitoreo de la purificación se empleó la actividad enzimática sobre BApNA, y las fracciones con mayor actividad fueron concentradas y mantenidas en congelación hasta su uso.
Evaluación de la pureza y determinación del peso molecular.- Se empleó la electroforesis en geles de poliacrilamida en condiciones denaturantes con SDS (PAGE-SDS), de acuerdo al método de Laemmli (1970), utilizando un equipo de electroforesis vertical en placa Mini-PROTEAN 3. La corrida electroforética se realizó aplicando 100 V constantes durante 1 h. Luego de transcurrido el tiempo, el gel fue transferido a una solución colorante de azul brillante de Coomassie al 0,1% por 10 min, para luego ser desteñido hasta evidenciar las bandas proteicas. El peso molecular de la enzima purificada fue estimado por comparación con una mezcla de proteínas para electroforesis en gel (Amersham Biosciences), conteniendo: fosforilasa b (97 kDa), albúmina sérica bovina (66 kDa), ovalbumina (45 kDa), anhidrasa carbónica (30 kDa) e inhibidor de tripsina (20,1 kDa).
Digestión in gel e identificación molecular mediante espectrometría de masas MALDI-TOF.- Luego de realizada la electroforesis en geles de poliacrilamida, la banda correspondiente a la enzima similar a trombina fue escindida y rehidratada con acetonitrilo 50% en bicarbonato de amonio 25 mM. La proteína fue reducida con ditiotreitiol (DTT) 10 mM en bicarbonato de amonio 25 mM a 57 °C durante 1 h, y S-carbamidometilada con iodoacetamida 55 mM en bicarbonato de amonio 25 mM a temperatura ambiente durante 1 h.
La digestión in gel fue llevada a cabo con tripsina 12,5 ng/µL a 37 °C durante 15 h. Posteriormente, se mezclaron 0,5 µL de los fragmentos peptídicos con 0,5 µL de ácido ?-ciano-4-hidroxicinámico en acetonitrilo 50% / TFA 1% (10 mg/mL) y luego fueron aplicados a un espectrómetro de masas Applied Biosystems 4700 Proteomics Analyzer, ABI, para su análisis mediante espectrometría de masas MALDI-TOF utilizando un rango de detección de 500 a 4500 Da. Mediante esta técnica se realiza la ionización de las moléculas y su obtención en fase gaseosa (desorción/ionización asistida por matriz, MALDI), para luego ser acelerados hacia un analizador y separados en función de su relación masa/carga (m/z) y determinando el tiempo de llegada a un detector (tiempo de vuelo, TOF).
Los pmfs (peptide mass fingerprints) recuperados del procedimiento anterior fueron graficados utilizando el programa Data Explorer Versión 4,4 (Applied Biosystems) y analizados empleando el programa on line MASCOT (http://www.matrixscience.com) (Perkins et al. 1999) siendo contrastados contra la base de datos de secuencias de proteínas para espectrometría de masas (MSDB). Mediante este análisis, los picos correspondientes a los pmfs son combinados in silico con los de proteínas conocidas permitiendo la identificación de la proteína digerida inicialmente.
Actividad Fibrinocoagulante.- Esta actividad se evaluó por la medida del tiempo de formación del coágulo originado por la acción de la enzima sobre el fibrinógeno bovino (Sigma-Aldrich Chemical Co) (Devi et al. 1972). Una unidad de actividad (U) se define como la inversa del tiempo de coagulación en segundos.
Actividad sobre Sustratos Cromogénicos.- En primer lugar se midió la actividad amidolítica empleando BApNA (Sigma-Aldrich Chemical Co) como sustrato (Fig. 1), midiéndose la liberación de p-nitroanilida a 405 nm (Erlanger et al. 1961). Una unidad de actividad (U) es expresada como µmoles de p-nitroanilida liberados por minuto.
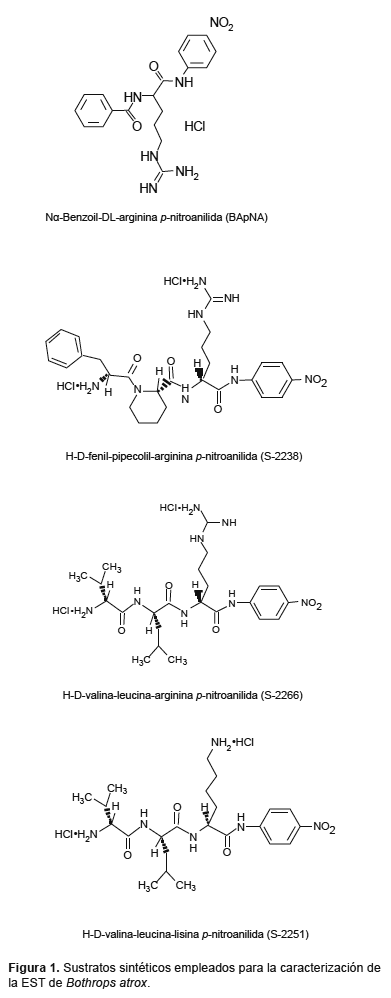
Posteriormente se emplearon los siguientes sustratos: S-2238 (H-D-fenilalanil-L-pipecolil-L-arginina-p-nitroanilida, sustrato cromogénico para trombina), S-2251 (H-D-valil-leucil-lisina-p-nitroanilida, sustrato cromogénico para plasmina) y S-2266 (H-D-valil-leucil-arginina-p-nitroanilida, sustrato cromogénico para kalikreína glandular) provenientes de Chromogenix (Mölndal, Suecia), disueltos en buffer Tris-HCl 0,05 M; pH 7,5 conteniendo NaCl 0,15 M, a una concentración final de 0,1 mM (Fig. 1). La hidrólisis de los sustratos sintéticos por el veneno crudo y por la enzima purificada fue medida en una lectora de microplacas Thermomax (Molecular Devices, Menlo Park, CA, USA), siguiendo la absorbancia a 405 nm a 37 °C durante 20 min a intervalos de 10 segundos.
Resultados
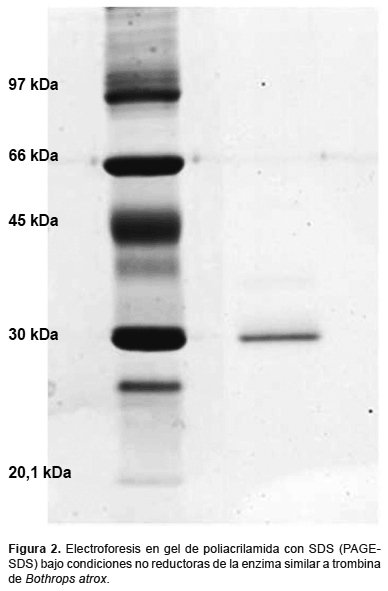
Purificación de la enzima y estimación del peso molecular.- Siguiendo los pasos descritos por Sandoval et al. (2010) se pudo obtener la enzima similar a trombina de B. atrox al estado homogéneo, la cual fue monitoreada por su actividad sobre el sustrato sintético BApNA. El ensayo electroforético en PAGE-SDS mostró que la enzima purificada corresponde a una única banda homogénea con un peso molecular aproximado de 29,6 kDa (Fig. 2).
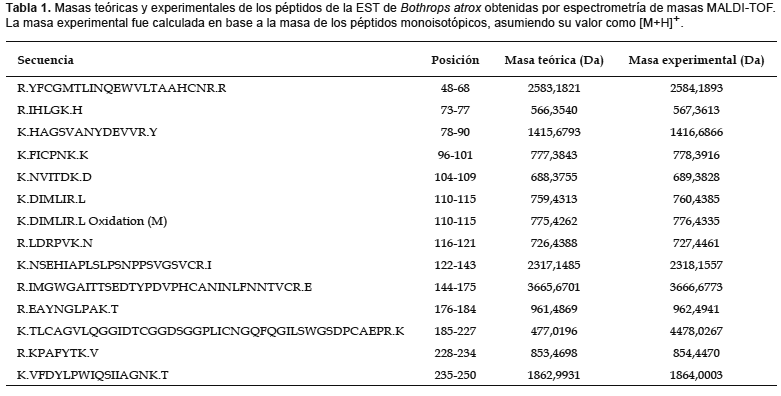
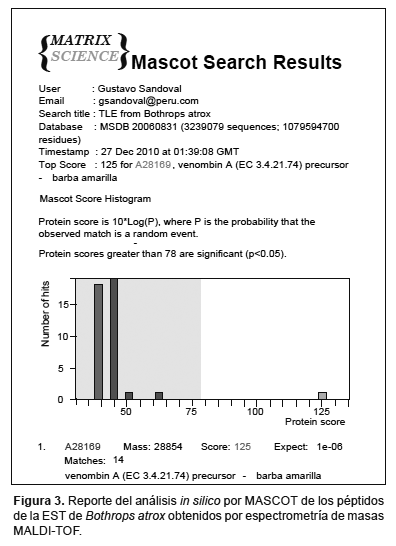
Identificación de la enzima purificada mediante espectrometría de masas MALDI-TOF.- Del análisis por espectrometría de masas MALDI-TOF, se pudieron recuperar 16 fragmentos peptídicos producto de la digestión de la enzima purificada, con valores de masas comprendidos entre los 500 y 4500 Da (Tabla 1). Estos valores obtenidos fueron utilizados para la identificación de la proteína mediante el programa MASCOT (opción: peptide mass fingerprinting). Como resultado del análisis in silico se obtuvo un score de alineamiento de 125 (expect: 10-6) con la proteína denominada venombina A (EC 3.4.21.74) (Fig. 3). Los péptidos recuperados comprenden un 75% del total de su secuencia.
Actividad sobre diferentes sustratos.- La EST purificada fue capaz de coagular el fibrinógeno bovino; comparando los tiempos de coagulación obtenidos con el veneno crudo y la enzima purificada se encontraron actividades específicas de 1,26 y 5,50 U/mg respectivamente, siendo la actividad de la enzima purificada 4,37 veces más que la actividad del veneno crudo (Tabla 2).
La enzima purificada también mostró una gran capacidad para hidrolizar sustratos sintéticos como el BApNA (0,874 U/mg), siendo la actividad purificada 25,5 veces respecto a la actividad mostrada por el veneno crudo (Tabla 2).
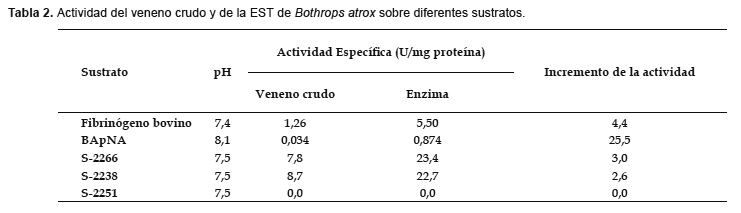
En el caso de las actividades sobre otros sustratos sintéticos específicos éstas fueron de 8,7 y 22,7 U/mg sobre S-2238 para el veneno crudo y la enzima purificada respectivamente, y de 7,8 y 23,4 U/mg sobre S-2266 para el veneno crudo y la enzima purificada respectivamente. No se encontró actividad sobre S-2251 bajo las mismas condiciones de trabajo tanto para el veneno crudo como para la enzima purificada (Tabla 2).
Discusión
Los venenos de serpientes de la familia Viperidae, presentan cantidades significativas de enzimas similares a trombina, las cuales son serinoproteasas y son similares a la trombina tanto en sus propiedades físicoquímicas como en los aminoácidos de su sitio activo, ya que contienen residuos reactivos de serina, ácido aspártico e histidina, haciéndolos susceptibles a agentes específicos que modifican su acción enzimática (Braud et al. 2000).
En el presente trabajo la purificación de la enzima similar a trombina (EST) del veneno de B. atrox consistió de una combinación de técnicas cromatográficas de filtración molecular, las que permiten separar las moléculas según su peso molecular; intercambio iónico, donde las moléculas son discriminadas de acuerdo a su carga eléctrica; y finalmente mediante afinidad enzima-inhibidor, utilizando p-aminobenzamidina (PAB). Esta molécula es un inhibidor competitivo específico de serinoproteasas, mediante la cual se pueden retener a las fracciones con afinidad permitiendo la eliminación de contaminantes. Del análisis electroforético de las fracciones obtenidas se observó una sola banda proteica de 29,6 kDa, indicando la homogeneidad del preparado (Fig. 2). Esta enzima tiene un mediano peso molecular, pero se encuentra dentro del rango de pesos moleculares reportados para varias enzimas similares a trombina aisladas de venenos de serpiente (Sandoval et al. 2010).
Para la identificación molecular de la enzima purificada se empleó el análisis por espectrometría de masas MALDI-TOF; que permite obtener información de la masa molecular e información estructural del compuesto analizado.
Del análisis por espectrometría de masas de las bandas de gel correspondientes a la EST de B. atrox y la posterior búsqueda en bases de datos, se obtuvo un score de alineamiento de 125 (expect: 10-6) con la venombina A (EC 3.4.21.74), EST del veneno de B. moojeni (Fig. 3). Esta proteína, a diferencia de la trombina, convierte el fibrinógeno en fibrina liberando sólo el fibrinopéptido A (Itoh et al. 1988). Esta característica le confiere un efecto defibrinogenante permitiendo que la fibrina sea rápidamente degradada a través del proceso fibrinolítico y eliminado mediante la orina (Lu et al. 2005). Actualmente es usado en el campo clínico para el tratamiento de enfermedades trombóticas .
Este análisis nos permitió además, ubicar y alinear los fragmentos producto de la digestión de la enzima en estudio. Los fragmentos recuperados cubrieron un 75% de la secuencia de la venombina A indicando una alta homología entre las secuencias de ambas proteínas. La venombina A es la enzima similar a trombina del veneno de B. moojeni y su secuencia de aminoácidos exhibe una homología significativa con enzimas proteolíticas como la tripsina, la kalikreina pancreática y la trombina, lo cual indica que se trata de un miembro de la familia de las serinoproteasas (Itoh et al. 1998).
A fin de complementar los resultados estructurales obtenidos por espectrometría de masas MALDI-TOF, se evaluó la actividad enzimática de la EST purificada sobre un sustrato natural como el fibrinógeno, así como sobre sustratos cromogénicos, los cuales contienen residuos de aminoácidos que imitan los sitios de corte de la enzima sobre el sustrato original. A fin de facilitar la medición de la actividad, dichos sustratos presentan un grupo cromogénico en el extremo carboxilo terminal, el cual es liberado como parte de la hidrólisis y posteriormente detectado por espectrofotometría.
En el presente trabajo se evaluó la actividad enzimática de la EST de B. atrox sobre diversos sustratos sintéticos que contienen como grupo cromogénico a la p-nitroanilida (Fig. 1). Durante los diversos pasos de purificación se empleó el sustrato BApNA el cual permite la caracterización de enzimas del grupo de las serinoproteasas como la tripsina y quimiotripsina y al cual pertenecen las enzimas similares a trombina de venenos de serpiente (Castro et al. 2001, 2004). Las fracciones que mostraron más actividad sobre este sustrato fueron ensayadas en su capacidad para coagular el fibrinógeno bovino obteniéndose resultados positivos indicando que ambas actividades están presentes en la enzima en estudio. En el caso de los sustratos sintéticos S-2266, S-2238 y S-2251, su empleo nos permitió determinar la especificidad en reconocimiento de los aminoácidos unidos al grupo cromogénico p-nitroanilida por parte de la enzima similar a trombina de B. atrox. Como se muestra en la Tabla 2, la enzima fue capaz de hidrolizar los sustratos S-2266 (sustrato para kalikreína glandular) y S-2238 (sustrato para trombina), mientras que fue incapaz de hidrolizar el sustrato S-2251 (sustrato para plasmina). En base a las características estructurales de estos sustratos podemos afirmar que la enzima presenta una especificidad para hidrolizar sustratos que contengan preferentemente arginina como el aminoácido que se une al grupo cromogénico a diferencia de la lisina la cual se encuentra en la estructura del S-2251, lo cual constituye una evidencia adicional de que esta enzima pertenece al grupo de las serinoproteinasas. Este análisis también fue realizado utilizando la enzima similar a trombina de Lachesis muta muta empleando sustratos cromogénicos con aminoácidos diversos en su estructura (Magalhaes et al. 2003). El hecho de que la enzima purificada hidrolice sustratos específicos para dos enzimas diferentes como la kalikreina y la trombina indican por un lado que nuestra enzima presenta características de la trombina como la de coagular el fibrinógeno, mientras que por otro está relacionada con las enzimas del grupo tripsina/kalikreína. Esto fue demostrado por Itoh et al. (1988) para el caso de batroxobina, la enzima similar a trombina de la serpiente B. moojeni mediante el análisis de la secuencia nucleotídica del gen.
De esta forma, mediante una combinación de aproximaciones estructurales y funcionales se ha podido caracterizar la naturaleza química y enzimática de la EST de B. atrox, identificándola como una venombina A, serinoproteasa con un rol importante en el envenenamiento por viperídos del género Bothrops y cuya detección y neutralización resultan de interés para el diagnóstico y tratamiento de accidentes ofídicos.
Agradecimientos
El presente trabajo fue desarrollado gracias al financiamiento otorgado a Gustavo Sandoval por la Red de Macrouniversidades de América Latina y el Caribe a través de una beca de investigación en la Universidad Federal de Rio de Janeiro como parte del desarrollo de su tesis de maestría en Biología Molecular.
Literatura citada
Ashcroft A.E. 2003. Protein and peptide identification: the role of mass spectrometry in proteomics. Nat. Prod. Rep. 20(2):202-215.
Braud S., C. Bon C & A. Wisner. 2000. Snake venom proteins acting on hemostasis. Biochimie. 82(9-10):851-859.
Castro H.C., D.M. Silva, C. Craik C, et al. 2001. Structural features of a snake venom thrombin-like enzyme: thrombin and trypsin on a single catalytic platform? Biochim Biophys Acta. 1547(2):183-195.
Castro H.C., R.B. Zingali, M.G. Albuquerque, et al. 2004. Snake venom thrombin-like enzymes: from reptilase to now. Cell Mol Life Sci. 61(7-8):843-856.
Devi A., S. Banerjee & A.L. Copley. 1972. Coagulant and esterase activities of thrombin and Bothrops atrox venom. Toxicon. 10(6):563-573.
Erlanger B.F., N. Kokowsky & W. Cohen. 1961. The preparation and properties of two new chromogenic substrates of trypsin. Arch Biochem Biophys. 95:271-278.
Itoh N., N. Tanaka, I. Funakoshi, et al. 1988. Organization of the gene for batroxobin, a thrombin-like snake venom enzyme. Homology with the trypsin/kallikrein gene family. J Biol Chem. 263(16):7628-7631.
Laemmli UK. 1970. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227(5259):680-685.
Lévano J. & R. Fernández. 2004. Diagnóstico y tratamiento de los accidentes por animales ponzoñosos. Instituto Nacional de Salud, Ministerio de Salud, Lima, Perú. 1era edición. 74 Pp.
Loayza S., Y. Morante, S. Campos, et al. 1985. Enzimas proteolíticas en el veneno de las serpientes peruanas Lachesis muta y Bothrops atrox. Bol Soc Quim Perú. 52(3):151-163.
Loja D., R. Avilés, Y. Necochea, et al. 2000. Ofidismo por Bothrops atrox: Estudio clínico-epidemiológico. Diagnóstico. 38(5):261-265.
Lowry O.H., N.J. Rosebrough, A.L. Farr, et al. 1951. Protein measurement with the Folin phenol reagent. J Biol Chem. 193(1):265-275.
Lu Q., J.M. Clemetson & K.J. Clemetson. 2005. Snake venoms and hemostasis. J Thromb Haemost. 3 (8):1791-1799.
Magalhaes A., R.N. Ferreira, M. Richardson, et al. 2003. Coagulant thrombin-like enzymes from the venoms of Brazilian and Peruvian bushmaster (Lachesis muta muta) snakes. Comp Biochem Physiol B Biochem Mol Biol. 136(2):255-266.
Orejuela P., A. Zavaleta, M. Salas, et al. 1991. Thrombin-like activity in snake venoms from Peruvian Bothrops and Lachesis genera. Toxicon. 29(2):1151-1154.
Perkins D.N., D.J. Pappin, D.M. Creasy, et al. 1999. Probability-based protein identification by searching sequence databases using mass spectrometry data. Electrophoresis. 20(18):3551-3567.
Sandoval G., L. Lerma, E. Rodríguez, et al. 2006. Purification of polyclonal antibodies against Bothrops atrox Peruvian snake venom ("Jergon") by affinity chromatography. Rev Soc Quim Perú. 72(3):140-149.
Sandoval G.A., N. Ruiz, F. Lazo, et al. 2010. Isolation and partial characterization of a thrombin-like enzyme from Bothrops atrox Peruvian snake venom "Jergon". Rev Soc Quim Perú. 76(2):156-164.
Smolka M.B., S. Marangoni, B. Oliveira, et al. 1988. Purification and partial characterization of a thrombin-like enzyme, balterobin, from the venom of Bothrops alternatus. Toxicon. 36(7):1059-1063.
Warburg O. & W. Christian. 1941. Isolierung and Kristallisation der Garungs ferments enolase. Biochem Z. 310: 384-421.
Yarlequé A. 2000. Las serpientes peruanas y sus venenos. Fondo Editorial, Universidad Nacional Mayor de San Marcos. Lima-Perú. 78 Pp.
Correspondencia:
1 Laboratorio de Biología Molecular, Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos.
Ciudad Universitaria, Av. Venezuela Cdra. 34 s/n. Apartado 110058, Lima 11, Perú.
Email Armando Yarlequé: ayarleque48@gmail.com
Presentado: 01/11/2009
Aceptado: 15/12/2010
Publicado online: 21/01/2011













