Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. v.18 n.2 Lima ago. 2011
TRABAJOS ORIGINALES
Divergencia intraespecífica y código de barras de ADN en Systrophia helicycloides (Gastropoda, Scolodontidae)
Intraspecific divergence and DNA barcodes in Systrophia helicycloides (Gastropoda, Scolodontidae)
Pedro Romero y Rina Ramírez
Departamento de Malacología y Carcinología, Museo de Historia Natural y Laboratorio de Sistemática Molecular y Filogeografía, Facultad de Ciencias Biológicas. Universidad Nacional Mayor de San Marcos. Av. Arenales 1256, Apartado 14-0434, Lima 14, Perú.
Email Pedro Romero: quipu.romero@gmail.com
Resumen
El DNA barcoding es un análisis que se basa en la comparación de distancias genéticas para identificar especies utilizando principalmente un segmento del gen Citocromo C Oxidasa I (COI). Los retos para la identificación surgen al estudiar grupos que presentan gran diversidad genética como los moluscos. Por ello, los objetivos de nuestra investigación fueron estimar la divergencia intraespecífica en el molusco terrestre amazónico Systrophia helicycloides (Gastropoda, Scolodontidae) y evaluar la utilización de los códigos de barras de ADN en la identificación molecular de esta especie. Las secuencias de nucleótidos fueron comparadas con las bases de datos Genbank y BOLD (Barcode of Life Data Systems). Se realizó un análisis de distancia genética mediante Neighbour Joining. Systrophia helicycloides presentó dos grupos de haplotipos con distancias genéticas intraespecíficas mayores a 4%. También se observó una superposición entre las distancias intraespecíficas y las interespecíficas. La gran divergencia intraespecífica puede estar relacionada a la rápida variación del genoma mitocondrial, la distribución poblacional de los moluscos la cual permite el aislamiento y diferenciación genética, y la presencia de polimorfismos ancestrales. Los perfiles COI enviados a la base de datos BOLD son los primeros registros para esta especie y permitieron diferenciar a Systrophia helicycloides de otras especies. Estos perfiles corroboran la gran variación que ocurre en el genoma mitocondrial de moluscos terrestres por lo que la asignación de especies en este grupo precisa de la combinación entre los valores de divergencia genética, la evaluación de sitios informativos y los estudios de taxonomía convencional.
Palabras clave: Systrophia, Scolodontidae, COI, códigos de barra de ADN, Neighbour Joining.
Abstract
DNA barcoding analysis is based on the comparison of genetic distances to identify species using a segment of Cytochrome C Oxidase I (COI) gene. Species identification through DNA barcoding challenges problems in groups with high genetic diversity as molluscs. Thus, our aim was to estimate intraspecific divergence in the Amazonian land snail Systrophia helicycloides (Gastropoda, Scolodontidae) and evaluate the use of DNA barcoding in molecular identification of this land snail. Nucleotide sequences were compared with Genbank and BOLD (Barcode of Life Data Systems) databases. We conducted distance analyses using the Neighbour Joining method. Systrophia helicycloides showed two groups of haplotypes and intraspecific genetic distances higher than 4%. We observed an overlap between intraspecific and interspecific distances. The high divergence may be related to rapid mutation rate in the snail mitochondrial genome, to population distribution that influences genetic isolation and differentiation, and to ancestral DNA polymorphisms. COI profiles uploaded in BOLD are the first records of this species and can identify Systrophia helcycloides from other species. These profiles corroborated the high variation in the land snail genome. Therefore, species identification in this group needs a combined analysis of genetic distances, informative sites, and conventional taxonomy.
Keywords: Systrophia, Scolodontidae, COI, DNA barcodes, Neighbour Joining.
Introducción
Existe una urgente necesidad de investigar los trópicos para conocer y entender la diversidad de los moluscos terrestres y demás grupos de invertebrados (Solem & van Bruggen 1984). Los invertebrados representan más del 99% de la diversidad animal, siendo los moluscos el segundo filo más diverso (Lydeard et al. 2004). Sin embargo, la mayoría de estudios de conservación en el bosque húmedo tropical se han enfocado principalmente en grupos de vertebrados. Poco se conoce de la historia natural de los moluscos terrestres de Sudamérica; por ejemplo, en la familia Scolodontidae, una familia de moluscos terrestres carnívoros endémicos de Sudamérica, sólo existe un trabajo que ha revisado en conjunto su taxonomía, sistemática y biogeografía (Ramírez 1993).
Systrophia (Pfeiffer 1855) es un género de moluscos terrestres perteneciente a la familia Scolodontidae (Baker 1925). Los escolodóntidos se distribuyen principalmente en Sudamérica hasta los 4400 m de altitud, algunas especies se encuentran en América Central y las Indias Orientales (Ramírez 1993). Estos moluscos son miembros conspicuos de la biota del bosque lluvioso tropical.
La utilización de una metodología basada en secuencias de ADN podría facilitar y economizar el proceso de identificación de las especies (Tautz et al. 2003). Así se desarrolló el concepto de códigos de barras de ADN (DNA barcodes), utilizando una secuencia de ADN corta que permitiera identificar las especies de manera eficiente. Se utiliza una porción del gen mitocondrial COI (citocromo C oxidasa subunidad I) de aproximadamente 700 pb debido a la existencia de primers universales (Folmer et al. 1994) y una alta tasa de mutación en la tercera posición de los codones. Además, esta sección casi no presenta inserciones o deleciones (indels), por tanto, los alineamientos son mucho más sencillos de realizar al no presentar gaps (Hebert et al. 2003).
La divergencia genética entre especies ha sido evaluada por Johns et al. (1998) encontrando una divergencia de 2% para especies de vertebrados utilizando el marcador citocromo b (cyt b). Hebert et al. (2004) propusieron un límite de 2,7% en el caso de especies de aves, indicando también que la divergencia interespecífica sería 10 veces mayor a la intraespecífica. En el caso de invertebrados, Hebert et al. (2003) propusieron un límite interespecífico de 3% para insectos. Davison et al. (2009) reportaron una gran variación en la secuencia COI entre moluscos terrestres; la divergencia intraespecífica promedio fue de 2,6% (0,0 – 8,1%) y la interespecífica de 10% (2,4 – 17,6%). Estos mismos autores también propusieron que el límite de divergencia entre las especies de moluscos sería de 4% con una probabilidad de error de identificación de entre 34 y 44%.
BOLD (Barcode of Life Data Systems) es una base de datos desarrollada por el grupo de Paul Hebert en la Universidad de Ontario, Canadá (Ratnasingham & Hebert 2007), destinada a la adquisición, mantenimiento y análisis de secuencias de códigos de barra de ADN. Para obtener una identificación eficiente basada en códigos de barra de ADN se debe tener en cuenta que los perfiles COI (1) deben ser únicos para cada especie, (2) los haplotipos de la especie deben pertenecer a un mismo grupo y (3) deben tener una distancia genética intraespecífica menor a 4% (Hebert et al. 2004; Davison et al. 2009). El objetivo de nuestra investigación fue evaluar la divergencia intraespecífica en el molusco terrestre amazónico Systrophia helicycloides (dOrbigny 1835) (Fig. 1), comparado con otros perfiles COI de las bases de datos Genbank y BOLD.

Material y métodos
En marzo de 2009, fueron colectados ejemplares de Systrophia helicycloides en dos localidades del departamento de Madre de Dios: Centro de Investigación y Capacitación del Río Los Amigos (CICRA) (12°3409"S, 70°0601"W) y Reserva Amazónica Inkaterra (ITA) (12°3234"S, 69°0312"W). Adicionalmente, fueron utilizados dos ejemplares de Scolodonta sp. colectados en el 2008 en la localidad de Shatuyacu, Juan Guerra (06°3501"S, 76°1955"W) del departamento de San Martín. Los especímenes fueron fijados en etanol de 96° y depositados en el Departamento de Malacología y Carcinología del Museo de Historia Natural – UNMSM (Lima, Perú).
La extracción y precipitación de ADN siguió el protocolo descrito por Romero (2010), la amplificación de ADN se realizó mediante PCR utilizando los primers de Folmer et al. (1994):
-
LCO 1490 (5'-GGTCAACAAATCATAAAGATATTGG-3')
-
HCO 2198 (5'-TAAACTTCAGGGTGACCAAAAAATCA-3')
Los amplicones fueron purificados y secuenciados por Macrogen Inc. (MD, USA).
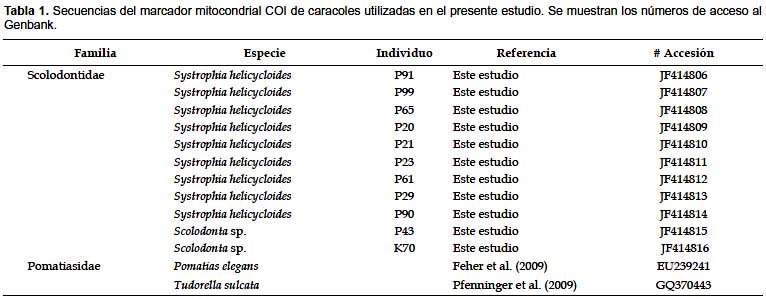
Las secuencias obtenidas fueron editadas con el programa Chromas (McCarthy 1996). El ensamblaje de ambas hebras de ADN se realizó mediante el software CAP3 (Huang & Madan 1999). Se obtuvieron 9 secuencias de Systrophia helicycloides: 3 de CICRA y 6 de Inkaterra, además de 2 secuencias de Scolodonta sp., las cuales están depositadas en la base de datos Genbank con los códigos de accesión JF414806 – JF414816. Los códigos de acceso de las secuencias se muestran en la Tabla 1.
Cada secuencia obtenida fue comparada contra la base de datos Genbank (http://www.ncbi.nlm.nih.gov/Genbank/)
por medio de la herramienta BLASTn. Esta búsqueda permitió comparar la similitud entre las secuencias obtenidas con las almacenadas en la base de datos, además de descartar errores por contaminación.Para el análisis de distancias genéticas se utilizaron los haplotipos encontrados en el conjunto de secuencias. Las secuencias de Pomatias elegans (EU239241) y Tudorella sulcata (GQ370443) (Pomatiasidae) fueron utilizadas como grupos externos. El alineamiento múltiple de secuencias, paso necesario para obtener una hipótesis de homología posicional que permita realizar comparaciones entre las secuencias, se hizo en el programa ClustalX 2.0 (Larkin et al. 2007) y la construcción del árbol fue realizada con el método de distancia Neighbour Joining (NJ) en el programa MEGA (Tamura et al. 2007), utilizando el modelo de sustitución nucleotídica Kimura 2-parámetros (K2P) (Kimura 1980). La evaluación de la topología encontrada se realizó mediante el análisis de bootstrap con 5000 réplicas (Felsestein 1985). El análisis de distancia intraespecífica e interespecífica se realizó en MEGA.
Las secuencias fueron ingresadas al Barcode of Life Data Systems (BOLD) (http://www.barcodinglife.org/),
servidor que permite la identificación de especies mediante el análisis de la divergencia genética entre las secuencias de ADN. Se debe introducir al menos tres secuencias por especie para realizar los análisis. Las secuencias fueron comparadas con toda la información disponible en BOLD (All Barcodes Record Database) la cual incluye todas las secuencias COI disponibles en la base de datos con una longitud mínima de 500 pb.Resultados
La herramienta BLASTn confirmó la amplificación del gen mitocondrial COI en todos los individuos. Las secuencias de S. helicycloides resultaron similares a los géneros de moluscos terrestres Natalina y Gulella de las familias Rhytididae y Streptaxidae, respectivamente. Las secuencias de Scolodonta sp. resultaron similares al género Carychium (Ellobiidae) Los porcentajes de identidad son en promedio menores a 90%. Dentro de las 9 secuencias de S. helicycloides se encontraron 6 haplotipos (Tabla 2), los cuales fueron diferentes de los dos haplotipos encontrados en Scolodonta sp., a su vez estos once haplotipos fueron diferentes de las secuencias de los grupos externos.
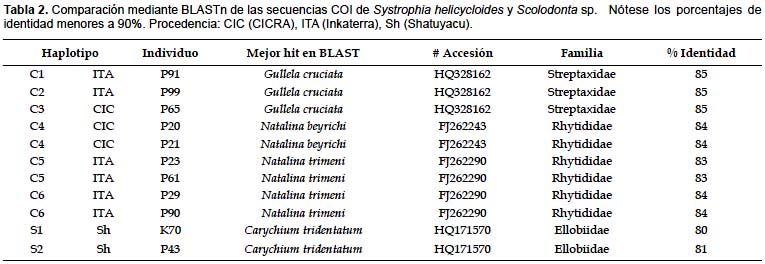
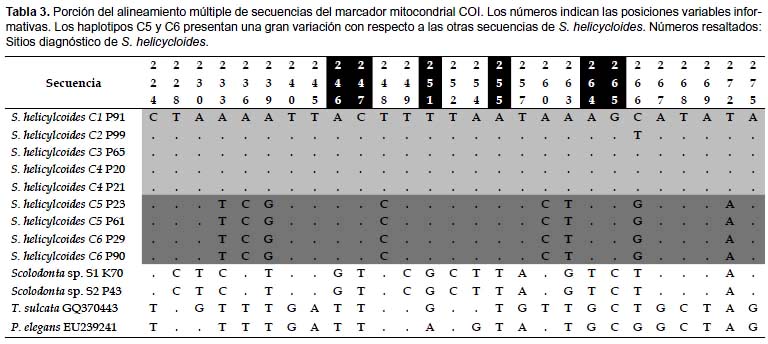
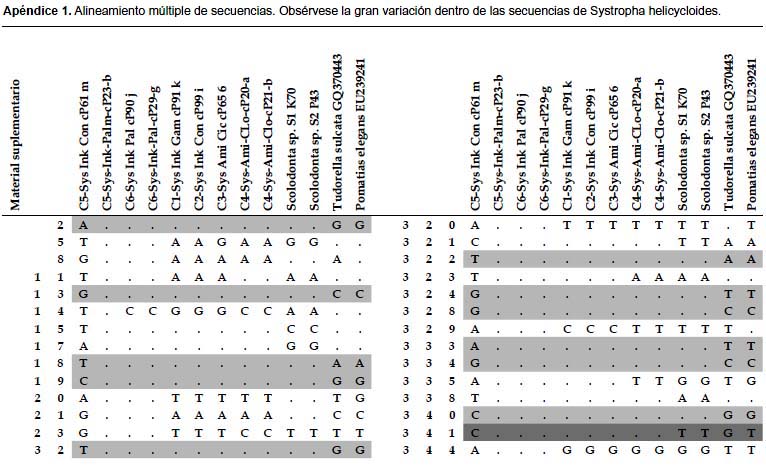
El alineamiento múltiple resultó en 623 sitios presentando 357 posiciones conservadas, 266 sitios variables y 232 sitios informativos. La Tabla 3 presenta una porción del alineamiento en la cual se pueden observar la similitud entre los haplotipos C1, C2, C3 y C4 de S. helicycloides. Los haplotipos C5 y C6 presentan una gran variación con respecto a los demás haplotipos de esta especie. El alineamiento además permite observar sitios informativos tales como las posiciones 246, 247, 251, 255, 264 y 265 que sirven como posiciones de diagnosis de S. helicycloides (Tabla 3). Los miembros de Scolodontidae (Systrophia y Scolodonta) también presentan sitios diagnósticos: posiciones 224, 240, 245, 267, 268, 269, 275 (Tabla 3, Apéndice 1).
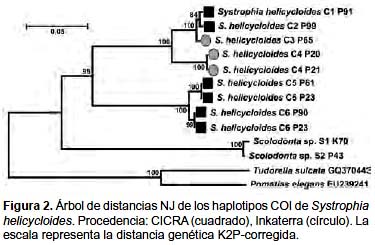
El análisis intraespecífico de los haplotipos COI se presenta en la Figura 2. Los haplotipos de S. helicycloides formaron un grupo con un alto valor de bootstrap (95%). Dentro de este grupo se forman dos sub-grupos, uno con los haplotipos C1-C4 (bootstrap: 100%) y otro con los haplotipos C5 y C6 de Inkaterra (bootstrap: 100%). Dentro del grupo C1-C4 se pueden diferenciar los haplotipos provenientes de Inkaterra C1 y C2 (bootstrap: 84%) de los de CICRA. Las secuencias de Scolodonta sp. forman un grupo alejado de las demás secuencias de S. helicycloides (bootstrap: 100%).
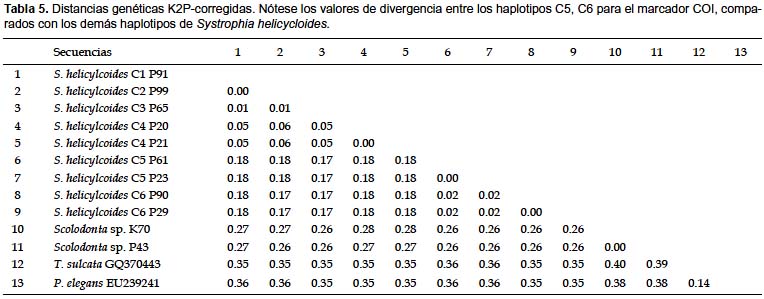
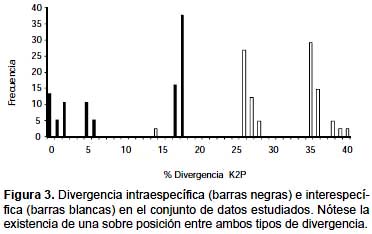
No se encontraron secuencias altamente similares en la base de datos BOLD (Tabla 4). Las secuencias de S. helicycloides obtenidas en este estudio son los primeros perfiles COI enviados a la base de datos del BOLD de esta especie. Esta plataforma necesita un mínimo de 3 secuencias para realizar la identificación, nosotros realizamos los análisis de distancia con las 9 secuencias obtenidas. La divergencia promedio entre todas las secuencias de S. helicycloides osciló entre 0,0 – 18,0% (Tabla 5). Sin considerar a los haplotipos divergentes C5 y C6, la distancia fluctuó entre 0,0 – 5,0% superando el valor de 4% considerado como límite específico (Tabla 5). La divergencia entre especies resultó entre 14,0 – 40,0%. Existe una ligera superposición entre los valores de divergencia intraespecífica e interespecífica (Fig. 3).
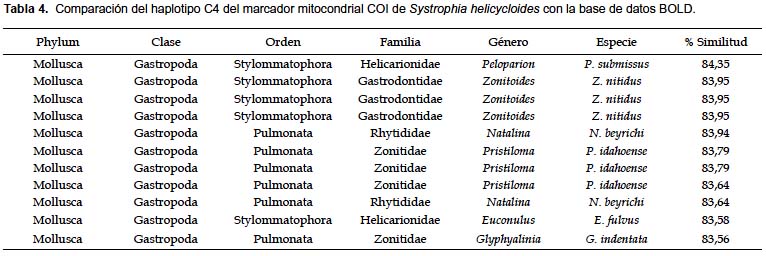
Los porcentajes de similitud, al comparar las secuencias con la base de datos BOLD, resultaron menores a 85%. Como ejemplo, la tabla 4 presenta los resultados de la comparación del haplotipo C4 con las secuencias disponibles en la base de datos. Según BOLD, las secuencias presentaron una mayor similitud con las familias de moluscos terrestres Helicarionidae, Gastrodontidae, Rhytididae, y Zonitidae.
Discusión
El análisis de distancias mediante Neighbour Joining diferenció a los haplotipos C5 y C6 (COI), correspondientes a individuos de Inkaterra, de los demás haplotipos de S. helicycloides, lo cual es reflejo de la gran cantidad de mutaciones con respecto a las demás secuencias (divergencia intraespecífica promedio mayor a 4%). Tal divergencia puede ser explicada por la presencia de polimorfismos ancestrales, los cuales pueden ser conservados a lo largo del tiempo debido a la distribución poblacional agregada característica de los moluscos terrestres (Thomaz et al. 1996), o por una elevada tasa de mutación (Chiba 1999). La extrema divergencia también puede deberse a una mayor acumulación de mutaciones dado que los moluscos terrestres presentan los tamaños de genoma más pequeños reportados para los metazoos celomados (Lydeard et al. 2000). Dentro de los moluscos, son los Heterobranchia (Hazprunar 1985), el grupo al cual pertenecen los gasterópodos pulmonados como Systrophia, los que poseen los genomas mitocondriales más pequeños (Kurabayashi & Ueshima 2000).
Se ha criticado mucho que un simple valor de distancia genética pueda discriminar especies diferentes (Rubinoff et al. 2006) por lo que se prefiere un análisis no sólo basado en distancias, sino en la observación de caracteres compartidos entre las secuencias (sinapomorfías) (De Salle et al. 2005). Las secuencias pueden proveer sitios informativos que potencian el análisis filogenético. En nuestro caso, se encontró que las secuencias de S. helicycloides poseen sitios informativos y diagnósticos de la especie (Tabla 3), los cuales sólo se presentan en los haplotipos encontrados (C1-C6) y que pueden ser utilizados para discriminarlas de otros taxones. La agrupación entre Systrophia y Scolodonta también es sustentada por mutaciones comunes (Tabla 3) y, a su vez, es soportada por la morfología de la rádula (Ramírez 1993), y por el uso de marcadores nucleares (Ramirez et al. 2011).
Las secuencias de COI de S. helicycloides presentaron una divergencia promedio mayor al 4%, aún sin considerar los haplotipos divergentes. Sin embargo, todas las secuencias conformaron un grupo separado de los demás taxones (Fig. 2). A pesar de la gran variación entre las secuencias COI de S. helicycloides, éstas no tienen un alto porcentaje de similitud (>95%) con ninguna otra secuencia dentro de la base de datos Genbank o BOLD (Tablas 1 y 4), demostrando el vacío de información para Scolodontidae y familias evolutivamente cercanas.
Resultados similares fueron encontrados por Romero (2010) para el marcador mitocondrial 16S rRNA en S. helicycloides, para los mismos individuos aquí estudiados con el marcador COI, donde se reporta que a pesar de las diferencias a nivel de secuencia, no existen diferencias en la morfología externa entre de los individuos. Por otro lado, si analizamos los niveles de divergencia de los haplotipos C5 y C6, éstos alcanzan valores similares a los presentes en las comparaciones interespecíficas (Tabla 5, Fig. 3).
Debido a la gran variación entre las secuencias del ADN mitocondrial en los moluscos, Davidson et al. (2009) propusieron que el COI podría ser de utilidad en la discriminación de los grupos dentro de Stylommatophora. Estos autores también propusieron cambiar el valor de 2% asignado por Hebert et al. (2003) por 4%. Aún utilizando este valor, las diferencias entre las secuencias de S. helicycloides resultan mayores a lo esperado para la comparación intraespecífica.
Siendo Mollusca uno de los filos más diversos, se debe tener mayor precaución en la asignación de valores de divergencia para la discriminación de especies. Ocurre lo contrario en el caso de vertebrados (Hebert et al. 2004), pues éstos poseen una gran cantidad de registros en Genbank o BOLD y presentan una mayor homogeneidad dentro de sus secuencias COI, lo que permite una asignación más confiable del límite de divergencia intraespecífica.
El perfil COI obtenido para S. helicycloides corrobora la gran variación que ocurre en el genoma mitocondrial de moluscos terrestres. La asignación de especies en este grupo precisa de una combinación entre los valores de divergencia genética, la evaluación de sitios informativos y los estudios de taxonomía convencional.
La rápida variación del genoma mitocondrial combinada con la distribución poblacional que permite el aislamiento y diferenciación, los procesos de selección natural que permiten la preservación de esta variación, además de la presencia de polimorfismos ancestrales (Thomaz 1996), deben ser tomados en cuenta para explicar la extrema variación intraespecífica presente en moluscos terrestres.
Agradecimientos
A la Amazon Conservation Association (ACA) y al Concejo Nacional de Ciencia, Tecnología e Innovación tecnológica (CONCYTEC) por las becas otorgadas a P.R. Al CSI-UNMSM (VRI N° 081001071, 091001041), y a la ONG Inkaterra Asociación (ITA) por el apoyo a R.R. Agradecemos a V. Borda y C. Calderón por la ayuda en las colectas. A J. Purisaca y H. Méndez por el apoyo logístico en Inkaterra. Los permisos de colecta en áreas no protegidas fueron proveídos por SERNANP.
Literatura citada
Baker H. 1925. Agnathomorphous Aulacopoda. Nautilus 38: 86–89.
Chiba S. 1999. Accelerated evolution of land snails Mandarina in the oceanic Bonin Islands. Evolution 53: 460-471.
Davison A., R. Blackie & G. Scothern. 2009. DNA barcoding of stylommatophoran land snails: a test of existing sequences. Molecular Ecology Resources 9: 1092–1101.
DeSalle R., M. Egan & M. Siddall. 2005. The unholy trinity: taxonomy, species delimitation and DNA barcoding. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 2360 (1462): 1905-1916.
Feher Z., K. Szabo, M. Bozso & Z. Penzes. 2009. Recent range expansion of Pomatias rivulare (Eichwald, 1829) (Mollusca: Pomatiidae) in Central-Eastern Europe. Acta Zool. Academ. Sci. Hung. 55 (1): 67-75.
Felsestein J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39: 783-791.
Folmer O., M. Black, W. Hoeh, R. Lutz & R. Vrijenhoek. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Mol. Mar. Biol. Biotechnol. 3(5): 294-299.
Haszprunar G. 1985. The Heterobranchia ― a new concept of the phylogeny of the higher Gastropoda.
Zeitschrift für zoologische Systematik und Evolutionsforschung 23 (1): 15–37.Hebert P., A. Cywinska, S. Ball & J. deWaard. 2003. Biological identifications through DNA barcodes.
Proc. R. Soc. Lond. B Biol. Sci. 270: 313–322.Hebert P., M. Stoeckle, T. Zemlak & C. Francis. 2004. Identification of Birds through DNA Barcodes. Plos Biology, 2 (10): e312.
Huang X. & A. Madan. 1999. CAP3: A DNA sequence assembly program. Genome Res. 9: 868-877.
Johns G & J. Avise. 1998. A comparative summary of genetic distances in the vertebrates from the mitochondrial cytochrome b gene. Mol. Biol. Evol. 15: 1481–1490.
Kimura M. 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences.
J. Mol. Evol. 16: 111–120.Kurabayashi A. & R. Ueshima. 2000. Complete sequence of the mitochondrial DNA of the primitive opisthobranch gastropod Pupa strigosa: systematics implication of the genome organization. Mol. Biol. Evol. 17:266-277.
Larkin M., G. Blackshields, N. Brown, R. Chenna, P. McGettigan, H. McWilliam, F. Valentin, I. Wallace, A. Wilm, R. Lopez, J. Thompson, T. Gibson & D. Higgins. 2007. Clustal W and Clustal X version 2.0. Bioinformatics 23 (21): 2947–2948.
Lydeard C., W. Holznagel, M. Schnare & R. Gutell. 2000. Phylogenetic analysis of molluscan mitochondrial LSU rDNA sequences and secondary structures. Mol. Phyl. Evo. 15(1): 83-102.
Lydeard C., R. Cowie, W. Ponder, A. Bogan, P. Bouchet, S. Clark, K. Cummings, T. Frest, O. Gargominy, D. Herbert, R. Hershler, K. Perez, B. Roth, E. Strong & F. Thompson. 2004. The Global Decline of Nonmarine Mollusks. BioScience 54 (4): 321-330.
McCarthy C. 1996. Chromas: version 2.0. Technelysium PTY, Australia.
dOrbigny A. 1835. Synopsis terrestrium et fluviatilium molluscorum, in suo per American meridionalem itinere. Magazine de Zoolgie 5: 1-44.
Pfeiffer L. 1855. Versuch einer Anordnung der Heliceen nach natürlichen Gruppen. Malakozoologische Blätter. 1: 221 – 223.
Pfenninger M., E. Vela, R. Jesse, M. Elejalde, F. Liberto, F. Magnin & A. Martinez-Orti. 2009. Temporal speciation pattern in the western Mediterranean genus Tudorella P. Fischer, 1885 (Gastropoda, Pomatiidae) supports the Tyrrhenian vicariance hypothesis. Mol. Phylogenet. Evol. 54 (2): 427 – 436.
Ramírez R. 1993. A generic analysis of the family Systrophiidae (Mollusca, Gastropoda). Taxonomy, Phylogeny and Biogeography. Master Thesis on Ecology and Systematics. University of Kansas.
Ramirez R., J. Ramirez & P. Ramírez. 2011. The enigmatic phylogenetic position of Scolodontidae: a thrid stylommatophoran clade. In: Sociedade Brasileira de Malacologia. (Org.). Tópicos em Malacologia - Ecos do XIX EBRAM. Technical Books Ltda, Rio de Janeiro. Pp. 124-131.
Ratnasingham S. & P. Hebert. 2007. BOLD: The Barcode of Life Data System (www.barcodinglife.org). Molecular Ecology Notes 7(3): 355-364.
Romero P. 2010. Filogeografía de Systrophia helicycloides: El reflejo de la dinámica del bosque lluvioso tropical en los genes 16S rRNA y COI de los moluscos terrestres. Tesis, Magíster en Biología Molecular. Facultad de Ciencias Biológicas. Universidad Nacional Mayor de San Marcos.
Rubinoff D., S. Cameron & K. Will. 2006. A genomic perspective on the shortcomings of mitochondrial DNA for "barcoding" identification. J. Hered. 97: 581-594.
Solem A. & A. van Bruggen, eds. 1984. World-wide snails: Biogeographical studies on non-marine Mollusca. E. Brill, W. Backhyus, Leiden. Pp. 6 – 22.
Tamura K., J. Dudley, M. Nei & S. Kumar. 2007. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Molecular Biology and Evolution 24: 1596-1599.
Tautz D., P. Arctander, A. Minelli, R. Thomas & A. Vogler. 2003. A plea for DNA taxonomy. Trends Ecol. Evol. 18: 70-74.
Thomaz D., A. Guiller, & B. Clarke. 1996. Extreme Divergence of Mitochondrial DNA within Species of Pulmonate Land Snails.
Proc. R. Soc. Lond. B. 263 (1368): 363-368.
Apéndice 1


Presentado: 22/02/2011
Aceptado: 20/07/2011
Publicado online: 25/08/2011













