Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.19 no.3 Lima dic. 2012
NOTA CIENTÍFICA
Implementación de dos métodos de recuento en placa para la detección de colifagos somáticos, aportes a las metodologías estándar
Implementation of two plate count methods for detection of somatic coliphages and contributions to the standard methodologies
Melissa Solano Barquero, Luz María Chacón Jiménez, Kenia Barrantes Jiménez y Rosario Achí Araya
Universidad de Costa Rica, Instituto de Investigaciones en Salud (INISA), Sección de Infección-Nutrición. Apartado 11501-2060, San Pedro Montes de Oca, San José, Costa Rica.
Resumen
Los métodos de recuento en placa capa doble y capa simple de agar, para la cuantificación de colifagos somáticos en aguas, fueron implementados utilizando como base metodologías estándar. Diferentes variables fueron ensayadas, lo cual permitió la precisión en algunos pasos no incluidos en metodologías estándares. De los hallazgos de mayor importancia, se exponen las consecuencias de utilizar un cultivo de Escherichia coli excesivamente concentrado y se describe la obtención de un cultivo en fase logarítmica en solo 4 horas de incubación, ajustando la concentración a una densidad óptica de 0,3 a 600nm (3,1 x 108UFC/ mL), o a un McFarland 1 (3,0 x108UFC/ mL). Se determinó que los controles de colifagos deben ser almacenados a -70 °C para reducir su degradación y que se deben evitar cantidades superiores a 20 mL de mezcla de reacción por plato de Petri, para reducir las burbujas que pueden interferir con la lectura de unidades formadoras de placas (UFP). Se demostró que los colifagos de las muestras de agua pueden almacenarse 48 horas a 4 °C sin que sufran degradación y que en las muestras con altas concentraciones de colifagos no se observa UFP porque se da una lisis confluente de la capa bacteriana. No se encontraron diferencias significativas en la recuperación de colifagos al utilizar un método u otro, pero dichos métodos deben ser evaluados por medio de controles, antes de aplicarlos directamente en el análisis de muestras de agua.
Palabras clave: colifagos somáticos; capa doble de agar; capa simple de agar; aguas; indicadores virales.
Abstract
Two plate count methods, double layer and single layer of agar for quantification of somatic coliphages in water, were implemented using standard methodologies. Several variables were tested and provided valuable information that was not included in standard methodologies. The most important findings are described, such as the effect of using an excessively concentrated culture of E. coli and production of a log phase culture in only 4 hours of incubation, adjusting the concentration to an optical density of 0.3 at 600 nm (3.1 x 108 CFU / mL), or to McFarland 1 (3.0 x 108 CFU / mL). It was determined that coliphages controls must be stored at -70 °C to reduce its degradation. Quantities of reaction mixture exceeding 20 mL per Petri dish must be avoided to prevent interfere with bubbles during the counting of plate forming units (PFU). It was demonstrated that coliphages isolated from water samples can be stored for 48 hours at 4 °C without any degradation, and PFU are not observed in samples with high concentrations of coliphages, because a confluent lysis of the bacterial layer. There was no significant difference in the recovery of coliphages using doble layer or single layer methods, but such methods should be evaluated by means of controls, before applying them directly in the analysis of water samples.
Keywords: somatic coliphages; double agar layer; simple agar layer; water; viral indicator.
Introducción
La contaminación de aguas superficiales con aguas negras, representa un alto riesgo de transmisión de enfermedades, ya que las deposiciones fecales humanas y de animales contienen diversos tipos de microorganismos enteropatógenos, entre estos podemos encontrar bacterias, protozoarios y varios tipos de virus entéricos (Borrego et al. 1990). De estos últimos se calcula que más de 100 tipos pueden ser transmitidos a través del agua (Fong & Lipp 2005).
La falta de tratamiento de las aguas residuales domésticas procedentes de ciudades, genera una gran contaminación en mares y ríos, lo cual merece especial atención, ya que estos últimos son fuente de abastecimiento de agua para consumo humano. En general, se estima que en las aguas sometidas a tratamiento para potabilización solamente se logra eliminar entre el 50 y el 90% de los virus presentes, ya que por lo general éstos son muy resistentes a dichos procesos (Bofill et al. 2005, Okoh et al. 2010).
En décadas pasadas, la mayoría de los estudios sugerían que las infecciones de origen hídrico se encontraban principalmente asociados a contaminación con bacterias o protozoarios; sin embargo, posteriormente se observó un incremento gradual en el número de casos registrados cuyo agente etiológico eran los virus, y en la actualidad se consideran a los virus como los responsables del mayoria de casos de origen hídrico (Bofill et al. 2005, Sinclair et al. 2009). Posiblemente este incremento fue consecuencia de una mejora en los métodos diagnósticos, como la aplicación de métodos de biología molecular (v.g. Reacción de Cadena de la Polimerasa -PCR) para la detección de patógenos ambientales; pero, también podría deberse a un incremento real en la incidencia, debido a cambios en la epidemiología de los virus, el aumento de la población y la contaminación de las aguas (Bofill et al. 2005, Sinclair et al. 2009).
Aunque es claro que los virus trasmitidos por vía hídrica, representan una amenaza para la salud pública, en la mayoría de las normativas su detección no ha sido considerada como hídrico. Por lo general, se analiza únicamente la presencia de indicadores bacterianos, cuya desventaja es que no reflejan el comportamiento de patógenos virales, ni de otros parásitos como protozoarios (Borrego et al. 1987, Vivier 2004).
Parte de las razones que han llevado a excluir a los virus en el análisis de aguas, es que estos presentan una baja concentración aún en grandes volúmenes de agua, lo cual los hace difíciles de detectar en muestras ambientales (Reynolds 2001). Ademas, aunque los métodos para la detección de virus en aguas han mejorado, ellos suelen ser muy laboriosos, como es el caso del cultivo celular, o se encuentran aún en la fase de estandarización como sucede con la Reacción en Cadena de la Polimerasa PCR (Bofill et al. 2005, Mooijmana et al. 2005) además, la implementación de los mismos representa una fuerte inversión económica (Campos-Pinilla et al. 2008).
Sin embargo, debido al riesgo para la salud que implica la presencia de virus entéricos en las aguas, así como por el incremento en los brotes de origen hídrico asociados a virus, es importante contar con metodologías de detección viral, rápidas y económicas, principalmente en países en desarrollo. Dentro de las opciones, existen metodologías basadas en el uso de indicadores virales, como los colifagos, cuya presencia correlaciona con la existencia de virus entéricos patógenos de humanos.
Los colifagos son virus que infectan y se replican en Escherichia coli (Armon & Kott 1996) del tracto gastrointestinal de organismos de sangre caliente (Paz 2003). Los colifagos somáticos incluyen una gran variedad de fagos que pertenecen a las familias Myoviridae, Siphoviridae, Podoviridae, Microviridae (Okafor 2011). Estos fagos han sido utilizados como indicadores de virus entéricos, ya que comparten muchas características con estos virus, como lo es su composición, morfología y modo de replicación, lo cual los hace comportarse de manera similar en cuanto a su persistencia ambiental, su comportamiento ante las tensiones ambientales, variación estacional y grado de resistencia a la cloración. De esta forma, una muestra positiva por colifagos reúne las condiciones para la presencia de virus entéricos y por lo tanto, la probabilidad de que también sea positiva para estos virus es alta (Cole 2003).
El análisis de colifagos somáticos también ha sido utilizado como indicador de contaminación fecal y marcador en el proceso de tratamiento de aguas, ya que provienen del tracto gastrointestinal de organismos de sangre caliente y se ha observado que tras el proceso de tratamiento de las aguas, se eliminan a un ritmo comparable a los virus entéricos (Skraber et al. 2004, Campos-Pinilla et al. 2008). Los colifagos son además, más resistentes a los procesos de tratamiento de agua, por lo cual algunos autores sugieren que su detección es de mayor utilidad que las bacterias fecales (Okoh et al. 2010, Brezina & Baldini 2008).
Para la detección de colifagos se han desarrollado métodos como la inducción de liberación de β–galactosidasa (Stanek & Falkinham 2001), bioluminiscencia (Guzmán et al. 2009) y fluorescencia (Salter et al. 2010), entre otros. Sin embargo, los protocolos de referencia siguen siendo los métodos de capa doble de agar y capa simple de agar, que son más económicos.
Los métodos de conteo en placa, como capa doble y capa simple de agar, se basan en la determinación de la concentración viral, mediante el conteo de unidades formadoras que placas (UFP), las cuales son zonas de aclaramiento circular que se producen en la capa bacteriana, debido a la lisis o destrucción de la E. coli mediada por los colifagos. Inicialmente una partícula de colifagos infecta a su bacteria hospedera, se replica y posteriormente se disemina, generando una zona circular sin crecimiento de la bacteria.
Existen protocolos estandarizados para el análisis de colifagos somáticos, por capa doble y capa simple de agar, como el 9224 C y 9224 E (APHA 2005), el 1602 (EPA 2001), y el método ISO 10705–2 (ISO 1999), los cuales han sido aplicados en muchos laboratorios con algunas variaciones. Sin embargo, estas metodologías no explican algunas de las variables a las que se enfrenta un laboratorio al implementarlas.
El objetivo de este trabajo es describir el proceso con la finalidad de informar sobre algunos aspectos y recomendaciones que no se incluyen en las metodologías estándar y que son importantes para la implementación de los ensayos
Material y métodos
Microorganismos utilizados.– Se utilizó la bacteria Escherichia coli 13706 ATCC y el colifago Ф174 ATCC. Los controles de colifagos se ajustaron a una concentración entre 20 – 80 UFP/ mL y se almacenaron a –70 °C, sin la adición de crioprotector.
Metodologías de referencia.– A continuación se describen brevemente las metodologías de referencia utilizadas para la implementación de los ensayos de capa doble de agar y capa simple de agar
Capa doble de agar:
-Protocolo 9224 B, Standard Methods for the Examination of Water and Wastewater (SMEWW) (APHA 2005). Se preparó un cultivo bacteriano en fase logarítmica, inoculando una asada del cultivo congelado de E. coli (ATCC 13706) en caldo tripticasa soya (CTS) que se incubó a 36,5±2 °C, 16 horas y se transfirió un inóculo a CTS fresco incubándolo 6 horas más. Para replicar los fagos el cultivo de la bacteria se inoculó con 1 mL del colifago rehidratado y fueron incubados juntos durante 4 horas. Posterior a esto, la suspensión colifagos-bacteria o la muestra de agua a analizar, fueron filtrados con filtros de 0,22 μm pretratados con extracto de carne. Se realizó diluciones seriadas del filtrado y se mezcló 1 mL de las mismas, 0,1 mL del cultivo de la bacteria y 3 mL de Agar Tripticasa Soya (ATS) al 0,7%, se homogenizó, se vertió en platos de Petri que contenían una capa de ATS al 1,5 % y fueron incubados toda la noche. El procedimiento se realizó por quintuplicado para cada dilución.
Capa simple de agar:
-Protocolo 1602, Agencia de Protección Ambiental de los Estados Unidos (EPA 2001). Este método de referencia difiere del anterior en cuanto a la preparación del cultivo bacteriano ya que utiliza ácido nalidíxico, que al inhibir el crecimiento de otras bacterias hace que se pueda evitar la filtración de la muestra. Sin embargo, los pasos de esta metodología que se siguieron fueron los siguientes: se preparó un cultivo bacteriano en CTS a partir de una asada de cultivo congelado y se incubó 18 – 20 horas a 36±1 °C. A partir de este cultivo se transfirió una alícuota a CTS fresco y se incubó durante 4 horas o hasta que alcanzó una densidad óptica (DO) de 0,1 – 0,5. Se preparó 100 mL de ATS 1,2 % el cual fue inoculado con 0,5 mL de cloruro de magnesio, 10 mL del cultivo de E.coli y 100 mL de la muestra de agua.
La mezcla fue homogenizada y dispensada en 8 a 10 en platos de Petri. Los platos se incubaron 36±1 °C por 16 – 24 horas.
-Protocolo 9224 E, SMEWW (APHA, 2005). La bacteria se preparó y la muestra se filtró según lo descrito en el Protocolo 9224 B SMEWW para capa doble de agar. Los 100 mL de la muestra fueron inoculados con 5 mL de cloruro de calcio (CaCl2), 10 mL de la bacteria en fase logarítmica y 100 mL de ATS 1,2 %, la mezcla se dispensó en platos de Petri. Los platos fueron incubados a 36,5 ±1 °C toda la noche.
Metodologías implementadas.– En esta sección se detallan las metodologías implementadas en nuestro laboratorio. La implementación de los métodos se realizó inicialmente con muestras de agua destilada inoculada y posteriormente utilizando agua de río, en este caso se utilizó el agua del río Purires, ubicado en la provincia de Cartago, Costa Rica. Todas las muestras fueron analizadas en dos momentos distintos: el día en que fueron colectadas y posterior a un almacenamiento de 48 horas a 4 °C.
Capa doble de agar
Para preparar el cultivo de la bacteria en fase logarítmica, se colocó 3 asadas del cultivo congelado de la bacteria en CTS y se incubó a 35 °C (incubadora Lab–line Imperial III modelo 310) en agitación hasta obtener una densidad óptica DO= 0,3. Para replicar los colifagos, uno de los cultivo de la bacteria fue inoculado con 1 mL del colifago Ф174 ATCC rehidratado y fueron incubados a 35 °C durante 4 horas, posteriormente se filtró en un sistema de filtración al vacío (Sartorius Stedim®) con filtros de acetato de celulosa (Sartorius) de 0,22 μm, pretratados con extracto de carne (OXOID) al 1,5%. El filtrado se distribuyó en alícuotas de 1 mL, en tubos cónicos tipo Eppendorff, y almacenadas a –70 °C. Posteriormente para el recuento del título de colifagos se realizó diluciones decimales seriadas las cuales fueron mezcladas con la E. coli y con el agar y distribuidas en platos Petri como se especifica en el Protocolo 9224 B del SMEWW para capa doble de agar. Para el análisis de muestras de agua se siguió el mismo procedimiento pero sin replicación de los colifagos, por lo que el procedimiento inició con la filtración de la muestra.
Capa simple de agar
El cultivo de la bacteria se preparó y la muestra se filtró como se especificó para la implementación de los ensayos de capa doble de agar. Para determinar el título de colifagos, 100 mL de la muestra de agua filtrada fueron inoculados con CaCl2, el cultivo de la bacteria hospedera y el agar, como se describió en el Protocolo 9224 E del SMEWW.
Variables ensayadas.–
(1) Optimización del cultivo bacteriano en fase logarítmica, para la replicación y recuento de los colifagos. Inicialmente el cultivo en fase logarítmica se preparó según el protocolo de APHA, posteriormente se ajustó para que el cultivo primario tuviera una concentración semejante a McFarland 1, lo cual equivale a aproximadamente a 3x108UFC/ mL. Por último, se preparó un cultivo bacteriano en fase logarítmica con una DO=0,3 a 600 nm, utilizando un biofotómetro.
(2) Edad del cultivo bacteriano. Se prepararon cultivos bacterianos con 4 horas de incubación y otros con 24 horas de incubación. Para preparar el cultivo de 24 horas, se inoculó una asada del cultivo congelado de E.coli en CTS y se incubó durante 16 horas a 36,5 ±1 °C, posteriormente se transfirió una alícuota a CTS fresco y se incubó 4 horas a la misma temperatura, el cultivo se almacenó en refrigeración y al día siguiente se transfirió otra alícuota a CTS fresco, se incubó durante 4 horas y se le ajustó la DO a 0,3.
(3) Aplicabilidad del rango de densidad óptica del cultivo bacteriano, para la amplificación y el recuento de colifagos. Se realizó el recuento de colifagos por capa doble de agar utilizando cultivos de bacteria hospedera con DO= 0,1, DO= 0,5 y DO= 0,3.
(4) Importancia de la concentración de bacterias en fase logarítmica, en el proceso de replicación y filtración de los colifagos. Posterior a la replicación de los colifagos se determinó el título de la suspensión, por el método capa doble de agar. La suspensión de colifagos sobrante, se hizo pasar nuevamente por el sistema de filtración cuyos filtros se encontraban tapizados con la bacteria hospedera E. coli. Lo anterior con el fin de establecer si existía disminución en el conteo de UFP debido a la captura de los fagos entre los restos bacterianos.
(5) Temperatura de almacenamiento de los colifagos. Concentrados de colifagos con título conocido fueron almacenados a tres temperaturas distintas: temperatura ambiente, 4 °C y –70 °C. Esta última temperatura se ensayó con la adición de glicerol (Sigma®) al 20% y sin añadir este crioprotector. Después de 30 días se realizó nuevamente el recuento de colifagos. A los tubos a –70 °C también se les realizó el recuento tras 7 meses de almacenamiento.
(6) Concentración del colifago. Se realizó el recuento de colifagos en filtrados altamente concentrados del virus, así como, en diluciones de los mismos, para determinar posibles diferencias en la distribución y forma de las UFP.
(7) Volumen de mezcla de reacción. En cada plato de Petri de 10 cm de diámetro, se vertió volúmenes de mezcla de reacción de 15, 20, 25, 30, 35, 40 y 45 mL, con el fin de evaluar si el grosor de la capa de agar afecta la observación de UFP.
Análisis estadístico.- Para los análisis estadísticos se aplicó ANOVA o t-Student, con el programa SPSS Statistics versión 17.0. En todos los casos se trabajó con un nivel de confianza de 95%.
Resultados
(1) Optimización del cultivo bacteriano en fase logarítmica, para la replicación y recuento de los colifagos.- Al aplicar la metodología original de APHA la densidad de la sobrecapa bacteriana obtenida impidió la observación de UFP. El cultivo de la bacteria hospedera en fase logarítmica se logró optimizar, regulando su concentración al comparar con un McFarland 1 (3,0x108 UFC/ mL) o midiendo una densidad óptica de 0,3 (3,1x108 UFC/ mL), con la ayuda de un biofotómetro. El inóculo bacteriano obtenido con las modificaciones aplicadas, generó una capa bacteriana menos densa que permitió la observación de las UFP.
(2) Optimización de la edad del cultivo bacteriano.- Se realizó un ensayo en el que se comparó los resultados del recuento al utilizar cultivos bacterianos con 4 horas y con 24 horas de incubación, obteniéndose 4,5 x 1010 UFP/ mL y 4,6 x 1010 UFP/ mL respectivamente, para lo cual se determinó que tampoco existe diferencia estadísticamente significativa (p= 0,5).
(3) Aplicabilidad del rango de densidad óptica (0,1 – 0,5), para la generación del cultivo en fase logarítmica.- Al utilizar la DO más baja sugerida por el método de la EPA (DO= 0,1), se obtuvo una sobrecapa bacteriana menos confluente en el plato de Petri, que no delimita bien las UFP, por lo cual estas tienden a traslaparse unas con otras. Cuando se utilizó DO= 0,5 se obtuvo UFP bien definidas, pero de menor tamaño que las observadas cuando la DO fue cercana a 0,3.
(4) Importancia de la concentración de bacterias en fase logarítmica en el proceso de replicación y filtración de los colifagos.- El título de la suspensión de colifagos replicados fue inicialmente de 3,8 x 103 UFP/ mL, dicho conteo disminuyó a 3,4 x 101 UFP/ mL, cuando la suspensión se hizo pasar nuevamente por el sistema de filtración cuyos filtros se encontraban tapizados con restos de la bacteria hospedera.
(5) Temperatura de almacenamiento de los colifagos.- Un concentrado de colifagos de título 2,2 x 104UFP/ mL, fue evaluado para determinar su estabilidad después de un mes de almacenamiento a diferentes temperaturas (4 °C, temperatura ambiente y –70 °C). En el caso de –70 °C se evaluó bajo dos condiciones, una utilizando 20% de glicerol y otra sin glicerol. Se obtuvo como resultados 6,1 x 101UFP/ mL a temperatura ambiente; 2,8 x 103 UFP/ mL a 4° C; 2,1 x 104 UFP/ mL a –70° C sin adicionar glicerol y 2,1 x 104 UFP/ mL a –70 °C adicionando glicerol. El almacenamiento durante un mes a temperatura ambiente y a 4 °C fueron significativamente menores (p<0,0001), en tanto que a –70 °C utilizando glicerol y sin la adición del mismo, la pérdida de colifagos no fue significativa (p= 0,8 y p= 0,9 respectivamente) independientemente de si se aplica o no crioprotector (p= 0,9) (Fig. 1).
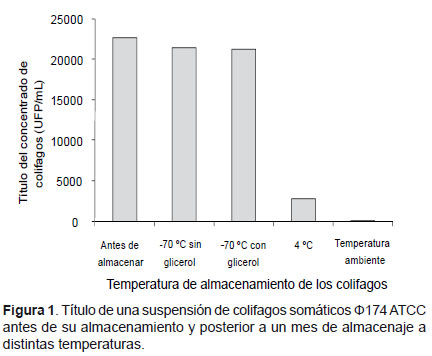
Después de 7 meses de almacenamiento a –70 °C y sin añadir crioprotector, el título disminuyó de 2,2 x 104 UFP/ mL a 7,2 x 102UFP/ mL.
(6) Observación de resultados según la concentración de colifagos. - Cuando la cantidad de colifagos fue 103UFP por plato de Petri o superior, la sobrecapa bacteriana desaparece por completo (lisis confluente), obteniéndose un plato de Petri con una superficie de agar brillante y traslúcido. Debido a esta lisis de la capa bacteriana, el recuento máximo obtenido por el método capa doble de agar fue de 200 UFP en cada plato de Petri, mientras que por el método capa simple de agar fue de 350 UFP por plato de Petri.
(7) Estandarización del volumen de mezcla de reacción, vertido en cada plato de Petri.- En el método de capa simple de agar se observó que volúmenes de 30, 35, 40 y 45 mL por plato generaron gran cantidad de burbujas que interfirieron en la lectura de las UFP, mientras que con volúmenes de 15, 20 y 25 mL, la cantidad de burbujas generadas es mínima, de manera que no limita la lectura de las UFP
(8) Repetibilidad de los métodos.- No hubo diferencias estadísticamente significativas para los 3 ensayos realizados por el método capa doble de agar (p= 0,7) (Tabla 1). Todos los recuentos realizados por el métodos capa simple de agar mostraron el mismo orden de magnitud 1,7x 107UFP/mL, 1,6 x 107; 2,4 x 107y 3,8x 7UFP/mL.
Comparación de métodos.- Para un concentrado de colifagos se realizó 3 ensayos por el método capa doble de agar y en paralelo se realizó 4 repeticiones por el método capa simple de agar, obteniendo como promedios 3,8 x 107UFP/ mL y 2,4 x 107UFP/ mL, respectivamente. No hubo diferencias estadísticamente significativas en la recuperación de colifagos, por ambos métodos (p= 0,1).
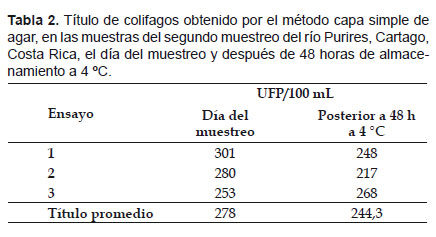
Análisis y conservación de las muestras de agua de río.- En el primer muestreo del río Purires se obtuvo un título de colifagos de 2,5 x 102 UFP/100 mL el día del muestreo y posterior a un almacenamiento de 48 h a 4 °C se obtuvo el mismo resultado. Para el segundo muestreo el recuento de colifagos se realizó por triplicado, tanto el día del ensayo como posterior al almacenamiento de 48 h (Tabla 2). No se encontró diferencia significativa entre los resultados obtenidos antes y después de almacenar la muestra (p= 0,8).
En un tercer muestreo realizado en el Río Purires se determinó tanto la concentración de coliformes fecales, como la de colifagos, obteniéndose los siguientes resultados: 280 NMP/100 mL y 233 UFP/100 mL respectivamente.
Discusión
Los métodos de capa simple y capa doble de agar descritos en SMEWW (APHA 2005) y EPA (EPA 2001) han sido ampliamente utilizados con algunas modificaciones en diferentes laboratorios (Paz et al. 2003, Muniesa & Jofre 2004). En el caso de esta investigación, se determinó que los métodos se ven afectados por muchas variables como la densidad óptica del cultivo bacteriano, temperatura de almacenamiento del fago, condición fisiológica de la bacteria hospedera, relación en la concentración colifagos-E.coli, entre otros aspectos; pero una vez que se tienen controladas estas variables, los resultados se comportan de manera repetible.
La bacteria hospedera E.coli debe encontrarse en fase logarítmica para que el virus se replique adecuadamente, ya que la generación de nuevas partículas virales depende de la replicación del material genético bacteriano (Breitbart et al. 2005). Para la reproducción de colifagos por el método capa doble de agar descrito por APHA, el cultivo secundario de E. coli se incuba 6 horas antes de inocularlo con los colifagos; sin embargo, si se compara este tiempo de incubación con las curvas de crecimiento descritas para E. coli (Ramírez et al. 2005), estas sugieren que dicha bacteria no se encuentra en fase de crecimiento exponencial, sino en fase estacionaria, en la cual hay equilibrio entre la muerte y división de la bacteria o ella simplemente deja de dividirse (Prescott 2002), lo que podría afectar la replicación del fago. Además, períodos de incubación prolongados generan cultivos bacterianos muy densos, como sucedió al aplicar el método de APHA, esto hace que las filtraciones sean más lentas y que aumenten las pérdidas en la recuperación de los colifagos, porque éstos pueden quedar atrapados entre los restos bacterianos dispuestos sobre los filtros de 0,22 μm; y como se observa en nuestros resultados, al hacer pasar una solución de colifagos por filtros tapizados con los restos de la bacteria hospedera, la concentración de los fagos disminuye 2 logaritmos en el título de los colifagos, de aquí la importancia de que la incubación de la E. coli-colifago se realice entre 4 y 6 horas para evitar un cultivo excesivamente denso.
La alta densidad bacteriana también afecta el recuento de colifagos, ya que cuando el cultivo es muy denso, la sobrecapa bacteriana es confluente, por lo cual aunque el virus logre la lisis de las de E. coli, el exceso de bacterias impide la observación de las zonas de aclaramiento (UFP). El ajuste de la concentración bacteriana mediante la aplicación de McFarland 1 (aproximadamente 3x10<8 UFC/ mL), mejoró la lectura de la prueba, ya que se logró obtener un cultivo bacteriano menos concentrado. Por esta razón se propone como un método alternativo, principalmente para laboratorios que no cuenten con biofotómetro. Otros métodos de referencia como el ISO 10705-2, propone la reducción de los tiempos de incubación de la bacteria (ISO 1999).
El uso del biofotómetro fue de utilidad para ajustar de manera exacta la concentración de bacterias en el medio de cultivo. Los resultados de recuento de colifagos en las dos densidades ópticas extremas recomendadas por el método de la EPA (0,1 y 0,5), no produjeron resultados adecuados para la ejecución del ensayo, mientras que al utilizar un cultivo de bacteria hospedera con valores de DO cercanos a 0,3 produjo una sobrecapa bacteriana óptima que permitió una adecuada delimitación de las UFP; aún cuando el recuento de las UFP era alto.
En cuanto a la optimización de la edad del cultivo bacteriano, se demostró que se puede lograr un cultivo de bacteria hospedera en fase logarítmica en 4 horas, obteniendo los mismos resultados que al utilizar un cultivo con más días de incubación que también se encuentre en fase logarítmica. Esto es de gran importancia ya que de esta manera el tiempo de preparación del cultivo bacteriano se reduce de 3 días (24 horas en total, según protocolo APHA) a 4 horas. La preparación de la bacteria hospedera el mismo día del ensayo, ha sido descrito para la determinación de colifagos somáticos según el manual de ISO 10705–2 (ISO, 1999) y para la evaluación los bacteriófagos de Bacteriodes fragilis (Araujo et al. 2001).
La temperatura óptima para el almacenamiento de polífagos es de –70 °C, lo cual es un hallazgo de importancia ya que esta información no se especifica en los métodos de referencia (APHA 2005, EPA 2001, ISO 1999), los datos que se encuentran en la literatura son escasos y basados en ensayos con otros tipos de bacteriófagos (Feng et al. 2003, Méndez et al. 2002). A pesar de que –70 °C fue la temperatura más adecuada para almacenar los colifagos, si se observó una pérdida de los mismos tras 7 meses de almacenamiento.
Para una adecuada lectura de los resultados es importante revisar minuciosamente los platos de Petri para notar las diferencias entre un plato negativo y uno con una alta concentración de colifagos, ya que ninguno presenta UFP; además es importante que cada plato contenga entre 15 y 25 mL de agar para evitar la formación de burbujas producto del metabolismo de E. coli (McFaddin 2003) que interfieren en la lectura de las UFP.
Una vez que variables como tiempo de incubación de la bacteria, y temperatura de almacenamiento de fagos se han optimizado, los métodos de capa doble y capa simple de agar se comportan de manera repetible. Capa simple de agar es una técnica más rápida y más económica, sin embargo, su utilidad es limitada cuando la contaminación de la fuente de agua es alta y no se ha diluido la muestra previamente.
En los ensayos realizados para esta investigación no se encontró diferencias significativas al comparar los recuentos de colifagos obtenidos por ambos métodos.
El protocolo de la EPA (EPA 2001) menciona que la muestra de agua debe ser analizada como máximo 48 horas posterior a su recolección, otros autores han mencionado que la muestra de agua puede mantenerse a 5 – 8 °C incluso por 3 días sin presentar una pérdida significativa, ni aumento de colifagos (Méndez et al. 2002, Muniesa & Jofre 2004, Jofre 2009). Para el caso de esta investigación se demostró que las muestras de agua de río, pueden ser almacenadas durante 48 horas, a 4 °C posterior a su filtración, sin que se reduzca el número de colifagos.
Por otro lado la similitud encontrada entre el número de colifagos y el número de coliformes fecales concuerda con los resultados encontrados para aguas crudas, en otras investigaciones (Paz-y-Miño et al. 2003, Campos et al. 2008, Skraber et al. 2004). Sin embargo, no se cuenta con una interpretación de los resultados obtenidos para las aguas de río, debido a que no se ha establecido un valor límite para colifagos en aguas crudas. Las Guías para la Calidad del Agua de Bebida, de la Organización Mundial de la Salud, indican que se han encontrado concentraciones de colifagos somáticos de 109 UFP/100 mL en aguas de desecho y de 104 UFP/100 mL en aguas de ríos y lagos (OMS 2008). Por otro lado, estudios realizados en ríos de diferentes regiones han encontrado concentraciones de colifagos en un orden de magnitud de 101UFP/100 mL, 103UFP/100 mL (Paz-y-Miño et al. 2003, Skraber et al. 2004).
En este trabajo, las concentraciones más altas de colifagos (102/100 mL) fueron encontradas en los puntos más lejanos a la naciente del río Purires, donde se observó captación directa del agua para el riego de lechugas. Esto representa un riesgo para la salud debido a elevada probabilidad de que virus entéricos y otros agentes patógenos estén presentes en esta agua, lo cual es de especial importancia siendo la lechuga un alimento de consumo crudo. A pesar de esta situación, en Costa Rica no hay estudios epidemiológicos de las aguas, ni controles sobre las captaciones directas que se hacen en los ríos, aún cuando estas se realizan en zonas evidentemente contaminadas. Por lo tanto, la implementación y ensayo de estos métodos de conteo en placa viene a ser un paso inicial para realizar estudios de contaminación de estas fuentes de agua.
Como se determinó en esta investigación, es importante que antes de iniciar el análisis de la presencia de colifagos somáticos en las fuentes de agua, los laboratorios implementen y corroboren la funcionalidad de los ensayos basados en métodos estándar.
Literatura citada
Araujo R., M. Muniesa, J. Méndez, et al. 2001. Optimisation and standardisation of a method for detecting and enumerating bacteriophages infecting Bacteroides fragilis. J Virol Methods. 93:127–136. [ Links ]
Armon R., Kott Y. 1996. Bacteriophages as indicators of pollution. Crit Rev Env Sci Tec. 26:299–335. [ Links ]
APHA (Asociación Americana de Salud Pública). 2005. Standard Methods for the Examination of Water and Wastewater. 21ra ed. United Book Press, Washington. Pp 9–75 a 9–81. [ Links ]
Bofill S.M, P.C. Clemente, N.G. Albiñana, et al. 2005. Efectos sobre la salud de la contaminación de agua y alimentos por virus emergentes humanos. Rev. Esp. Salud Pública. 79:253–269. [ Links ]
Borrego J., M. Moriño, A. Vicente, R. Cónax & P. Romero. 1987. Coliphages an indicador of fecal pollution in water. It relationship with indicator an pathogenic microorganism. Water Res. 21:1473–1480. [ Links ]
Borrego J., R. Conax, E. Moriño, et al. 1990. Coliphages as indicador of fecal pollution in water. Their survival and productive infectivity in natural aquatic environments. Water Res. 24:111–116. [ Links ]
Breitbart M., F. Rohwer & S. Abedon. 2005. Phage ecology and bacterial pathogenesis. In: M. Waldor, D. Friedman, S. Adhya, eds. Phages: their role in bacterial pathogenesis and biotechnology. Washington: ASM Pres. Pp 67–70. [ Links ]
Brezina S.S., & M.D. Baldini. 2008. Detection of somatic coliphages as indicators of faecal contamination in estuarine waters. Rev. Argent. Microbiol. 40(1):72–4. [ Links ]
Campos–Pinilla C., M. Cárdenas, & A. Guerrero. 2008. Comportamiento de los indicadores de contaminación fecal en diferente tipo de aguas de la sabana Bogotá (Colombia). Universitas Sturrgart. 13:103–108. [ Links ]
Cole D., S. Long & M. Sobsey. 2003. Evaluation of F+ RNA and DNA coliphages as source–specific indicators of fecal contamination in surface waters. Appl. Environ. Microbiol. 69:6507–6514. [ Links ]
EPA (Agencia de Protección Ambiental de los Estados Unidos). 2001. (en línea). Method 1602: Male–specific (F+) and somatic coliphage in water by single agar layer (SAL) procedure. <http://www.epa.gov/nerlcwww/1601ap01.pdf>. Acceso 31/05/2012. [ Links ]
Feng Y., S. Ong & J. Hu. 2003. Effects of pH and temperature on the survival of coliphages MS2 and Qb. J Ind Microbiol Biotechnol. 30:549–552. [ Links ]
Fong T. & E. Lipp. 2005. Enteric viruses of humans and animals in aquatic environments: health risks, detection, and potential water quality assessment tools. Microbiol Mol Biol Rev. 69:357–371. [ Links ]
Guzmán C., A. Costán, F. Lucena & Jofre J. 2009. Detection of somatic coliphages through a bioluminescence assay measuring phage mediated release of adenylate kinase and adenosine 5–triphosphate. J Virol Methods. 161:107–113. [ Links ]
ISO/DIS 10705–2. 1999. Water Qualyty– Detection and enumeration of bacteriophages– Part 2: Enumeration of somatic coliphages. [ Links ]
Jofre J. 2009. Is the replication of somatic coliphages in water environments significant? J Appl Microbiol. 106:1059–1069. [ Links ]
McFaddin FJ. 2003. Pruebas bioquímicas para la identificación de bacterias de importancia clínica. 3ª ed. Buenos Aires: Editorial Médica Panamericana. Pp 302. [ Links ]
Mendez J., J. Jofre, F. Lucena, et al. 2002. Conservation of phage reference materials and water samples containing bacteriophages of enteric bacteria. J Virol Methods. 106:215–224. [ Links ]
Mooijmana K., Z. Ghameshlou, M. Bahar, et al. 2005. Enumeration of bacteriophages in water by different laboratories of the European Union in two interlaboratory comparison studies. J Virol Methods. 127:60–68. [ Links ]
Muniesa M. y J. Jofre. 2004. Factors influencing the replication of somatic coliphages in the water environment. Antonie Van Leeuwenhoek. 86:65–76. [ Links ]
Okafor N. 2011. Environmental Microbiology of Acuatic and Waste Sistems. Springer; 1st Edition. Pp.170. [ Links ]
Okoh A., I. Sibanda & S. Gusha. 2010. Inadequately Treated Wastewater as a Source of Human Enteric Viruses in the Environment. Int J Environ Res Public Health. 7(6):2620–2637. [ Links ]
OMS (Organización Mundial de la Salud). 2008. Guidelines for drinking water quality. < http://www.who.int/water_sanitation_health/dwq/fulltext.pdf>. Acceso 30/05/2012. [ Links ]
Paz-y-Miño M., C. Barzola, C. Lazcano, M. Ponce, J. León. 2003. Colifagos como indicadores de contaminación fecal y de remoción bacteriana en la potabilización del agua. Rev. Peru. Biol. 10:133–144. [ Links ]
Prescott L., J. Harley & D. Klein. 2002. Microbiología. 5ª ed. Madrid: McGraw–Hill. Pp 119–121 [ Links ]
Ramírez J.S., G.F. Contreras & C.E. Gómez. 2005. La fase estacionaria en la bacteria Escherichia coli. Rev. Latinoam. Microbiol. 47:92–101. [ Links ]
Reynolds K. 2001. Introducción a las enfermedades microbianas propagadas a través del agua. Revista Agua Latinoamérica. 1:38–39. [ Links ]
Salter R., G. Durbin, E. Conklin, J. Rosen, et al. 2010. Proposed modifications of Environmental Protection Agency method 1601 for detection of coliphages in drinking water, with same–day fluorescence–based detection and evaluation by the performance–based measurement system and alternative test protocol. Appl. Environ. Microbiol. 76:7803–7810. [ Links ]
Sinclair R. G., E. L. Jones & C.P. Gerba. 2009. Viruses in recreational water–borne disease outbreaks: a review. Journal of Applied Microbiology, 107:1769–1780. [ Links ]
Skraber S., B. Grassilloud & C. Gantzer. 2004. Comparison of coliforms and coliphages as tools for assessment of viral contamination in river water. Appl. Environ. Microbiol. 70:3644–3649. [ Links ]
Stanek J. & J. Falkinham. 2001. Rapid coliphage detection assay. J Virol Methods. 91:93–98. [ Links ]
Vivier J., M. Ehlers y W. Grabow. 2004. Detection of enteroviruses in treated drinking water. Water Res. 38:2699–2705. [ Links ]
Email:
Melissa Solano:melissa.solano_b@ucr.ac.cr
Luz María Chacón:luz.chacon@ucr.ac.cr
Kenia Barrantes: kenia.barrantes@ucr.ac.cr
Rosario Achí: rosario.achi@ucr.ac.cr
Presentado: 20/06/2012
Aceptado: 05/10/2012
Publicado online: 15/01/2013














