On-line version ISSN 1727-9933
Rev. peru biol. vol.20 no.2 Lima Aug. 2013
Aislamiento y caracterización de Escherichia coli O157:H7 a partir de carne molida de bovino en Lima-Perú
Isolation and characterization of Escherichia coli O157:H7 from ground beef cattle in Lima-Peru
Carmen R. Méndez1, Germán Vergaray1, Hilda Y. Morante2, Paulo R. Flores1 y Roger A. Gamboa1
1 Laboratorio de Control de Calidad de Alimentos, Aguas y Ambientes; Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos. Ciudad Universitaria de San Marcos,Av. Venezuela s/n, Lima 01. Correspondencia: Calle Laredo 372- Centro Comercial Monterrico- Surco. Lima 33. Carmen R. Méndez: cmendezf08@hotmail.com
2 Laboratorio de Química de los Alimentos, Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos.
Germán Vergaray: germanvergaray@gmail.com
Hilda Y. Morante: hildamoranteo@gmail.com
Paulo R. Flores: flopaulo@gmail.com
Roger A. Gamboa: roggeiro17@hotmail.com
Resumen
El objetivo del estudio fue aislar y caracterizar E. coli O157:H7 a partir de carne molida fresca de bovino obtenida en diferentes mercados de abastos. Se analizaron 195 muestras; para el aislamiento y enumeración de E. coli se utilizó la técnica del Numero Más probable mediante tubos múltiples; para el aislamiento y caracterización de E. coli O157:H7 el enriquecimiento selectivo y el análisis bioquímico, las colonias características se confirmaron mediante pruebas serológicas. Para determinar la presencia de shigatoxina (stx1, stx2) e intimina (eae A) se empleó la técnica de PCR multiplex en tiempo real y para enterohemolisina la prueba de hemólisis. El 87.18% de la muestras fue positivo para E. coli y el 77.95% presentó un recuento igual o superior a 50 NMP/g. Se obtuvieron 3 (1.54%) cepas de E. coli O157:H7, una stx1 +/ stx2 +/ eaeA – y enterohemolisina -, una stx 1 +/ stx2 -/ eaeA + y enterohemolisina – y la otra stx1 -/ stx2 -/ eaeA – y enterohemolisina +. También se obtuvieron 4 cepas (2.05%) de E. coli O157 no H7, ninguna presentó factores de virulencia. El estudio reveló el riesgo potencial de que E. coli O157:H7 afecte a la población de Lima.
Palabras clave: Escherichia coli O157:H7, bovino, carne molida, factores de virulencia.
Abstract
The aim of the study was to isolate and characterize E. coli O157: H7 from fresh ground beef obtained in different food markets. 195 samples were analyzed, for isolation and enumeration of E. coli was used most probable number technique using multiple tubes, for the isolation and characterization of E. coli O157: H7 selective enrichment and biochemical analysis, characteristic colonies were confirmed serologically. To determine the presence of shigatoxin (stx1, stx2) and intimin (eaeA) was done with multiplex real time PCR and for enterohemolysin the hemolysis test. The 87.18% of the samples were positive for E. coli and 77.95% had a count greater than or equal to 50 NMP/g. Were obtained 3 (1.54%) strains of E. coli O157: H7, one stx1 +/ stx2 +/eaeA - and enterohemolysin -, one stx1 +/stx2 -/eaeA + and enterohemolysin - and the other stx1 -/stx2 -/eaeA - and enterohemolysin +. Also obtained 4 (2.05%) strains of E. coli O157 non H7, no virulence factors presented. The study found that the risk potential E. coli O157: H7 affecting the population of Lima.
Keywords: Escherichia coli O157:H7, cattle, ground beef, virulence factors.
Introducción
Aunque Escherichia coli es una bacteria que habita el intestino humano y de animales de sangre caliente y usualmente se comporta como comensal, en las últimas décadas han aparecido grupos que causan patologías diarreicas, denominadas E. coli diarreogénicas o E. coli patógenas. Las cepas patógenas poseen factores de virulencia que sumados al tipo de enfermedad que producen han permitido agruparlas en patotipos (Nataro & Kaper 1998): E. coli enteropatógena (EPEC), E. coli enterotoxigénica (ETEC),E. coli enteroinvasiva (EIEC), E. coli de adherencia difusa (DAEC), E. coli enteroagregativa (EAEC) y E. coli shigatoxigénica (STEC).
Bajo el nombre de Escherichia coli shigatoxigénica (STEC) se conoce a un grupo de bacterias asociadas a enfermedades transmitidas por alimentos y reportadas tanto en países desarrollados como en vías de desarrollo (Frenzen et al. 2005, Rivas 2008, Clark et al. 2010). En el humano pueden causar colitis hemorrágica, síndrome urémico hemolítico (SUH) y púrpura trombocitopénica por las citotoxinas denominadas toxinas Shiga, cuya síntesis está codificada por genes stx de bacteriófagos lisogénicos que se insertan en su genoma en forma estable (Strockbine et al. 1986). Las toxinas Shiga (stx1, stx2 y variantes) están relacionadas estructural y funcionalmente con la toxina Shiga de Shigella dysenteriae, y dañan el endotelio vascular, principalmente de los pequeños vasos del colón, riñón y sistema nervioso central; constituyen los principales factores de virulencia responsables de la patogenicidad de la cepa (O´brien & Hornes 1987, Karmali 1989, Phillips et al. 2000, Pistone et al. 2006, Morato Bergamini et al. 2007).
El grupo STEC presenta otros 2 factores de virulencia que aumentan su patogenicidad. La intimina que es una proteína de membrana externa de 97 KDa codificada por el gen cromosomal (eae), y es responsable de la adherencia íntima a las células epiteliales y del barrido de las microvellosidades de la mucosa colónica (lesión por adherencia y esfacelación A/E). El otro factor es la enterohemolisina, que es una proteína de la familia RTX de las citolisinas formadoras de poros, es codificada por el gen ehxA del plásmido de 60 MDa-pO157 (Karch et al. 1987, Donnenberg et al. 1993, Schmidt et al. 1995).
El subgrupo EC enterohemorrágico (EHEC) es el serotipo más agresivo para el humano y según la WHO, una cepa STEC para ser considerada EHEC deberá producir intimina y enterohemolisina; existen más de 150 serotipos, el más frecuente a nivel mundial es E. coli 0157:H7.
Escherichia coli O157:H7 fue reconocido como patógeno humano después del brote de colitis hemorrágica ocurrido en 1982 en Estados Unidos de Norteamérica, y que tuvo como vehículo de transmisión a hamburguesas de carne de bovino, el serotipo fue aislado de los pacientes y del alimento (Riley et al. 1983). Desde entonces se han reportado numerosos brotes de colitis hemorrágica y sus complicaciones en Estados Unidos de Norteamérica, Canadá, Reino Unido, Dinamarca, Suecia, Alemania, España y Japón; países donde E. coli enterohemorrágica O157:H7 ha sido la principal causa (Dorn 1995, Siegler 1995, Mead & Griffin 1998, Michimo et al. 1999, Blanco et al. 2003).
Los rumiantes domésticos son portadores asintomáticos de E. coli O157:H7 y el ganado bovino es el principal reservorio para infecciones en humanos, en Europa y en Estados Unidos de Norteamérica alcanzan prevalencias de 12 y 28%. Caballos, cerdos y conejos también son considerados como portadores, pero no en el nivel de los rumiantes (Orskov et al. 1987, ICMSF 1996, Mora et al. 2003).
La carne de ganado bovino picada o molida e insuficientemente cocida, como la que se utiliza en hamburguesas, ha sido el vehículo más frecuente en los brotes epidémicos (Riley et al. 1983, Barret et al. 1994). En Argentina, se ha reportado E. coli O157:H7 en carne picada fresca (2.7%, Marzocca et al. 2006), y molida (1.2%, Roldán et al. 2007); en Uruguay en carne picada fresca (1.8%, Varela et al. 2008); en Colombia en carne molida (10%, Piedrahita et al. 2001); en Venezuela de carne molida (1.94% Bravo & Villalobos de Bastardo 2002) y en Paraguay en hamburguesas crudas (dos cepas [40%] Copes et al. 2009).
En el Perú, el primer hallazgo de E. coli O157:H7 se hizo en las heces de una lactante que provenía de Tacna, la cepa presentó los 3 factores de virulencia (Huapaya et al. 2001, Huguet et al. 2002). Mora et al. (2007) en un estudio de 102 muestras de carne molida de bovino en Lima metropolitana encontraron un 22.55% (23) positivo para E. coli O157; del total de positivos se analizaron 10 (43.48%) cepas, comprobándose que todas eran E. coli O157:H7 productoras de los 03 factores de virulencia.
El objetivo del presente estudio es determinar la presencia de cepas de E. coli O157:H7 en muestras de carne fresca molida de ganado bovino que se expende en diferentes distritos de Lima Metropolitana y determinar la presencia de factores de virulencia.
Material y métodos
Entre junio del 2009 y enero del 2011 se analizaron 195 muestras de carne fresca molida de ganado bovino colectadas en 65 puestos de venta elegidos al azar, ubicados en 33 mercados de abastos, localizados en 17 distritos de Lima metropolitana. En cada puesto se colectaron tres muestras, una cada seis meses. La carne era abastecida por tres frigoríficos y dos camales y estaba lista para su venta al público.
Para el aislamiento y enumeración de cepas de Escherichia coli se utilizó la técnica completa del número más probable mediante tubos múltiples FDA -BAM, (http://www.fda.gov/Food/FoodScienceResearch/LaboratoryMethods/ucm064948.htm). Para la caracterización y serotipificación de E. coli O157:H7 se empleó la metodología FDA - BAM, (http://www.fda.gov/Food/FoodScienceResearch/LaboratoryMethods/ucm070080.htm) realizándose las etapas de enriquecimiento y siembra, la primera con caldo EHEC suplementado con cefixime, cefsulodin y vancomicina y la segunda en Agar Mc Conkey sorbitol con cefixime y telurito. Se seleccionaron 10 colonias sospechosas de cada muestra, por ser sorbitol negativas; se transplantaron a agar TSYE y luego a agar EMB; se aislaron las colonias cuyas características eran de E. coli y se sometieron a las pruebas bioquímicas de indol (+), actividad de β-glucoronidasa (MUG) (-); TSI (A/A + Gas), citrato (-), LIA (A/A), Sorbitol (-). Las colonias presuntivas fueron tipificadas con antisueros 0157 y H7 – Probac. Se seleccionaron entre 5 a 7 colonias de cada muestra y se sembraron en Agar Mc Conkey.
Para determinar los factores de virulencia Shigatoxina (stx1, stx2) e intimina (eaeA) se empleó el método de PCR multiplex en tiempo real validado (Guion et al. 2008). Previamente se realizó la extracción del DNA a partir de un pool de 5 a 7 colonias típicas sembradas en agar Mc Conkey (Barletta et al. 2009); se le adicionó 100 µL de agua para PCR Finnzyme en un tubo Eppendorf y se sometió a 100 °C por 15 minutos, se dejó reposar a temperatura ambiente por 15 minutos y se centrifugó a 13000 rpm durante 15 minutos; luego se tomó 50 µL del sobrenadante y se almacenó a -20 °C para su conservación y uso posterior. Para la prueba de PCR se empleó una alícuota de 2 µL del lisado (utilizado como DNA molde), a la cual se le adicionó 23 µL de la mezcla maestra de PCR constituida por: 0.5 U de la enzima Phusion polymerase (Hot-Start DNA Polimerasa, Finnzyme), Phusion buffer con 200 µM de trifosfatos desoxinucleósidos, 4 mM MgCl2 y los primers previamente validados (Stx1: F: CTGGATTTAATGTCGCATAGTG, R: AGAACGCCCACTGAGATCATC, Amplicón 150 pb, Tm 87 °C; Stx2 F: GGCACTGTCTGAAACTGCTCC, R: TCGC-CAGTTATCTGACATTCTG, Amplición 255 pb, Tm 89.1 °C y eaeA F: ATGCTTAGTGCTGGTTTAGG, R: GCCTTCATCATTTCGCTTTC, Amplicón 248 pb y Tm 83.4 °C) y el agente SYBR green I como fluorocromo para el DNA. Los productos formados se diferencian por su temperatura de denaturación (Tm) y fueron graficados mediante el software Opticon Monitor como picos de fluorescencia después de 25 ciclos. La producción de enterohemolisina (EHEC ehxA) se demostró indirectamente mediante la prueba de hemólisis, para lo cual las colonias se sembraron en placas de agar sangre desfibrinada de carnero al 5% suplementado con 10 mM de cloruro de calcio. Las placas se incubaron a 37 °C durante 18 a 24 horas.
Se empleó como control positivo la cepa E. coli ATCC 918026 y como control negativo E. coli ATCC 25922.
Resultados
De las 195 muestras de carne fresca molida de bovino, el 87.18% (170) fue positivo para Escherichia coli y el 77.95% (152) presentó un recuento igual o superior a 50 NMP/g.
Del total de muestras, el 1.54% (03) presentó E. coli O157 :H7; se les denominó cepas CE 001, CE 002 y CE 003; los tres aislamientos se obtuvieron de muestras colectadas en mercados de los distritos de Comas, La Victoria y San Juan de Miraflores, respectivamente. Las tres cepas presentaron colonias sorbitol negativo, glucorinadasa negativo, movilidad positiva, indol positivo, hidrógeno sulfurado negativo, citrato negativo, Lisina descarboxilasa positiva y fermentación de glucosa y lactosa. El 2.05% (04) presentó E. coli O157 no H7.
De las E. coli O157:H7 la cepa CE 001 fue stx1 +/stx2 +/ eaeA -/enterohemolisina -; la cepa EC 002 fue stx1 +/stx2 -/eaeA +/enterohemolisina -, y la cepa CE 003 fue stx1 -/stx2 -/eaeA -/enterohemolisina +. De las cepas E. coli O157 no H7, todas fueron negativas para los factores de virulencia.
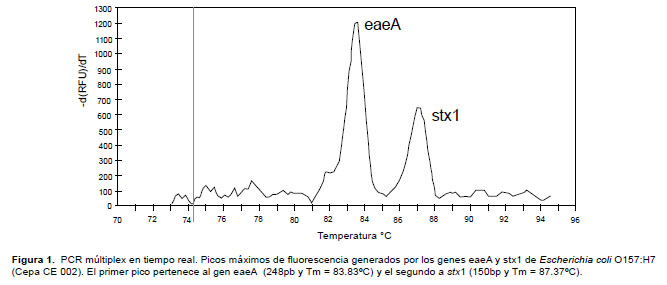
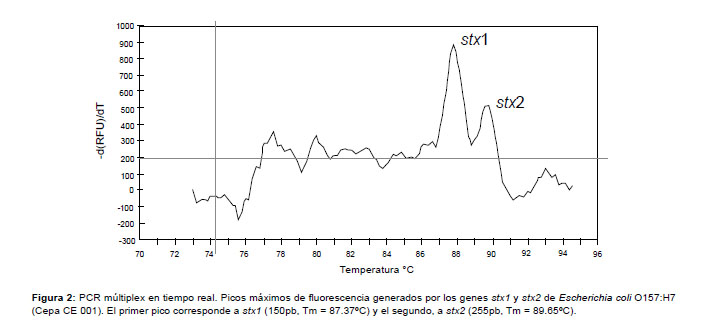
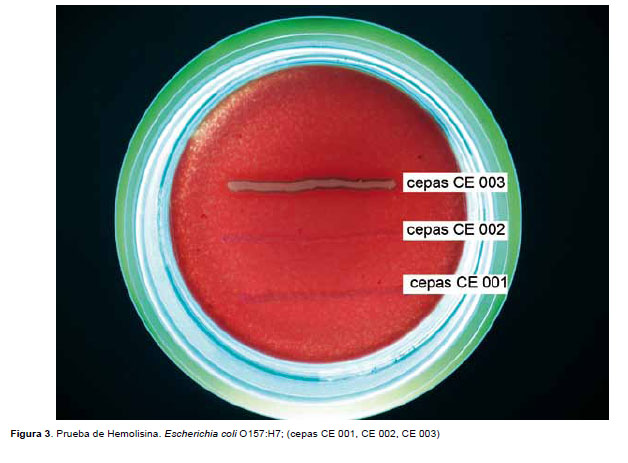
En el PCR multiplex en tiempo real, se evidenció la presencia de stx1 y eaeA en la cepa CE 002, mediante la formación de picos de fluorescencia generados por los genes stx1 y eaeA; el primer pico pertenece a eaeA y el segundo a stx1 (Fig. 1). En el caso de la cepa CE 001 se evidenció el pico generado por el gen stx1 y un segundo pico de fluorescencia generado por el gen stx2 (Fig. 2). En la cepa CE 003 se observó un halo de hemólisis alrededor del cultivo (Fig. 3).
Discusión
Se decidió analizar la carne molida fresca de bovino debido a que dicho ganado es considerado como el principal reservorio de E. coli O157:H7 a nivel mundial y a que la carne picada o molida insuficientemente cocida es considerada como el vehículo más frecuente de los brotes de colitis hemorrágica y de síndrome urémico hemolítico. La carne molida debido a la destrucción de sus fascies protectoras, aumento de sus superficies de contaminación y mayor manipulación es más susceptible a la contaminación microbiana que la carne entera.
En Lima metropolitana es elevado el consumo de carne de bovino principalmente picada o molida, posiblemente por la variedad de sus preparaciones y por su menor costo; dicha carne se considera estéril cuando el animal está vivo; sin embargo, nuestro estudio demostró que un elevado porcentaje (87.18%) de las muestras de carne molida que se expende en Lima está contaminada con E. coli y que un porcentaje algo menor (77,95%) tiene una contaminación superior a 50 NMP/g de E. coli, límite establecido por la NormaTécnica Sanitaria Peruana (R.M. N°. 591-2008-MINSA), por lo que es considerada inaceptable para el consumo humano y pone en evidencia las deficientes condiciones higiénicas en las que se ha procesado el alimento. Un porcentaje menor de contaminación con E. coli (55.8%) se reportó en Argentina (Cicuta et al. 2006), lo cual pone de manifiesto que con la aplicación de buenas prácticas de manipulación, disminuye la contaminación microbiana y mejora la calidad del alimento.
El 1.54% (03) de las muestras presentó el serotipo O157:H7; posiblemente una búsqueda más exhaustiva siguiendo otros procedimientos como la separación inmunomagnética o el VIP (Visual Immunoprecipitate Assay Eight Hours Method) (Blanco et al. 1996, Feldsine et al. 2002, Leotta et al. 2005, US/FIS 2005, Mora et al. 2007) podría haber aumentado el número de aislamientos. Este porcentaje es de interés epidemiológico para el Perú, aunque por ahora no es importante en salud pública (Huguet et al. 2002, Ochoa et al. 2011); sin embargo no se debe ignorar su elevada prevalencia a nivel mundial que incluye a países vecinos (Barret et al. 1994, Michimo et al. 1999, Blanco et al. 2004, Fernández & Padola 2012).
De las cepas aisladas ninguna presentó los 3 factores de virulencia (stx, eaeA y ehxA) considerados como característicos de E. coli O157:H7 y que se presentan con elevada frecuencia en las cepas aisladas en la mayoría de los países del mundo (Blanco et al. 2004, Lukásová et al. 2004, Varela et al. 2008). Por ello, las cepas aisladas por nosotros de acuerdo a la WHO (1997), no se incluirán en el grupoE. coli enterohemorrágico, porque para serlo deberían tener los 3 factores; además la cepa EC 003 no se incluye en el grupo E. coli shigatoxigénica por no presentar el gen stx. Se debe agregar la posibilidad de que mediante la PCR multiplex en tiempo real que empleamos no se detecte stx1 y stx2, tal como reportaron Varela et al. (2008), quienes obtuvieron positividad con ensayos de citotoxicidad y neutralización en cultivo de células Vero; nosotros no empleamos dichas técnicas.
En Lima, Mora et al. (2007) encontraron una elevada prevalencia (22.55%) en 102 muestras de carne molida de bovino, todas las cepas analizadas (10) produjeron los 03 factores de virulencia; Rivera et al. (2012) encontraron el serotipo E. coli O157 en el 1.75% (02) de 114 muestras de heces de bovino, una de las cepas producía stx1 y eaeA, y la otra sólo stx1. El muestreo de Mora et al. se efectuó entre Octubre del 2000 y Febrero del 2001, el de Rivera et al. entre Noviembre y Diciembre del 2010 y el de nuestro estudio entre Junio del 2009 y Enero del 2011. En los últimos 15 años no se han presentado brotes de la enfermedad en Lima, por lo que podría suponerse que se trató de una diseminación temporal del serotipo.
Los genes que codifican los factores de virulencia provienen de fagos y plásmidos; pueden no estar presentes (Wetzel & Le Jeune 2007), adquirirse (Muniesa & Jofre 2004) o perderse (Feng et al. 2001). Por lo tanto, es explicable que en países en los cuales no causan enfermedad o su prevalencia es baja se encuentren cepas de E. coli O157:H7 negativas a uno o más factores de virulencia como se demostró en Paraguay donde a partir de hamburguesas se aislaron 2 (40%) cepas: stx1 -/stx2 -/eae + y ehxA - (Copes et al. 2009); lo cual también se evidenció en nuestro estudio. Sin embargo, las cepas que carecen de factores de virulencia podrían adquirirlos por transferencia horizontal de genes, en el intestino del humano o de animales, especialmente rumiantes o en desagües domésticos (Muniesa & Jofre 2004). Echerichia coli debido a la plasticidad de su genoma es capaz de generar combinaciones de genes que conducen a la aparición de cepas muy agresivas (Blanco 2012). La presencia de cepas de E. coli O157 no H7, tambien fue reportado en el Perú por Huguet et al. (2002), quienes analizaron siete cepas que provenían de heces humanas y que carecían de factores de virulencia, como en nuestro estudio.
Se recomienda profundizar los estudios y aplicar un programa de vigilancia epidemiológica que permita detectar oportunamente la presencia de cepas de E. coli enterohemorrágica O157:H7 en carne de bovinos, tomando en consideración la que proviene de países en donde su prevalencia es elevada, con la finalidad de evitar la diseminación de dichas cepas cuya presencia es frecuente en el mundo e inclusive en países de América del Sur y que en un futuro cercano podrían constituir un problema importante de salud en el Perú.
Agradecimientos
Al Laboratorio de Referencia Nacional de Enteropatógenos/ CNSP/ Instituto Nacional de Salud y al Laboratorio de Enfermedades Entéricas y Nutrición – Instituto de Medicina Tropical A. von Humboldt de la UPCH, por su colaboración en la detección de los factores de virulencia. Al Vicerrectorado de Investigación y a Fundación San Marcos por haber contribuido al financiamiento del estudio y al manejo de los recursos económicos.
Literatura citada
Barletta F., T. Ochoa, L. Ecker L, et al. 2009. Validation of a five-colony pool analysis using Multiplex Real Time PCR for detection of diarrheagenic Escherichia coli. J Clin Microbiol. 47(6): 1915-1917. DOI: 10.1128/JCM.00608-09 [ Links ]
Barret J., H. Lior, J.H. Green et al. 1994. Laboratory Investigation of a multistate food-borne outbreak of Escherichia coli O157 :H7. J. Clin. Microbiol. 32 : 3013-3017. [ Links ]
Blanco J. 2012. Escherichia coli enteroagregativa O104:H4-ST678 productora de Stx2a. ¡Diagnóstico microbiológico ya, de este y otros serotipos de STEC/VTEC! Enferm. Infecc. Microbiol. Clin. 30(2): 84-89. DOI: 10.1016/j.eimc.2011.09.008 [ Links ]
Blanco J., M. Blanco, J.E. Blanco et al. 2003. Verotoxin producing Escherichia coli (VTEC) in Spain: Prevalence, serotypes and virulence genes of O157:H7 and non-O157 VTEC in ruminants, raw beef products, and human infections. Exp. Biol. Med. 228: 345-351. [ Links ]
Blanco J.E., M. Blanco, A. Mora et al. 1996. Detection of enterohaemhorragic Escherichia coli O157:H7 en minced beef using immunomagnetic separation. Microbio. SEM 12: 385-394. [ Links ]
Blanco M., N.L. Padola, A. Krüger et al. 2004. Virulence genes and intimin types of Shiga-toxin-producing Escherichia coli isolated from cattle and beef products in Argentina. Int. Microbiol. 7: 269-276. [ Links ]
Bravo V.J.B. & L.B. Villalobos de Bastardo. 2002. Escherichia coli enterohemorrágica en productos cárnicos comercializados en el mercado municipal de Cumaná, Venezuela. Rev. Soc. Ven. Microbiol. 22 (2): 119-121. [ Links ]
Cicuta M.E., N. Deza, W.R. Roibón et al. 2006. Detección de Escherichia coli productor de toxina Shiga en reses bovinas y carne molida de Corrientes, Argentina. Rev. vet. 17(1): 20-25. [ Links ]
Clark W.F., J.M. Sontrop, J.J. Macnab et al. 2010. Long term risk for hypertension, renal Impairment and Cardiovascular Disease after gastroenteritis from drinking water contaminated with Escherichia coli O157:H7: a Prospective cohort study. BMJ 341:c6020: 1-09. DOI: 10.1136/bmj.c6020 [ Links ]
Copes J., K. Pellicer, G. Del Hoyo et al. 2009. Primer aislamiento de E. coli O157:H7 a partir de hamburguesas en Paraguay. Analecta Veterinaria 29 (1): 11-14. [ Links ]
Donnenberg M., S. Tzipori, M. Mc Kee et al. 1993. The role of the eae gene of enterohaemorragic E. coli in intimate attachment in vitro and in a porcine model. J. Clin. Invest. 92: 1418-1424. [ Links ]
Dorn R.C. 1995. Escherichia coli O157:H7. JAVMA 206: 1583-1585. [ Links ]
Feldsine P.T., D. E. Kerr, S.C. Leung et al. 2002. Visual Immunoprecipitate Assay Eight hours Method for detection of Enterohemorrhagic Escherichia coli O157:H7 in Raw and Cooked beef (modification of AOAC official method 996.09): Collaborative study. Journal of AOAC International 85 (5): 1029-1036. [ Links ]
Feng P., M. Dey, A. Abe et al. 2001. Isogenic Strains of Escherichia coli O157:H7 that has lost both Shiga toxin 1 and 2 genes. Clin. Diagn. Lab. Inmunol. 8(4): 711-717. [ Links ]
Fernández D., Padola N. 2012. Escherichia coli verocitotoxigénico: varias cuestiones ... y los tambos tamboién. Rev. Argent. Microbiol. 44(4): 312- 323. [ Links ]
Frenzen P.D., A. Drake & F.J. Angulo. 2005. Economic cost of Illness due to Escherichia coli O157 Infections in the United States. J. Food Prot. 68: 2623-2630. [ Links ]
Guion C.E., T. Ochoa, C.M. Walker et al. 2008. Detection of Diarrheagenic Escherichia coli by Use of Melting-Curve Analysis and Real-Time Multiplex PCR. J. Clin. Microbiol. 46(5): 1752-1757. DOI: 10.1128/JCM.02341-07 [ Links ]
Huapaya B., J. Huguet, v. Suárez et al. 2001. Primer Aislamiento de Escherichia coli O157:H7 enterohemorrágica en el Perú. Rev. Med. Exp. 18 (1-2): 38-39. [ Links ]
Huguet J., B. Huapaya & E. Salazar. 2002. Determinación de factores de virulencia asociados a Escherichia coli enterohemorrágica en cepas peruanas aisladas entre 1999 – 2001. Rev. Peru Med. Exp. Salud Publica 19 (2): 63-67. [ Links ]
ICMSF (International Comission on Microbiological Specifications for Foods) 1996. Intestinally pathogenic Escherichia coli. In Microorganisms in foods 5. Characteristics of microbiol pathogens, Gaythersburg. M.D: Aspen Publishers, Inc. Pp: 126-140. [ Links ]
Karch H., J. Heesemann, R. Laufs et al. 1987. A plasmid of enterohemorrhagic Escherichia coli O157:H7 is requerid for expression of a new fimbrial antigen and for adhesion to epithelial cells. Infect. Immun. 55: 455-469. [ Links ]
Karmali M.A. 1989. Infection by verocytoxin-producing Escherichia coli. J. Clin. Microbiol. 2(1): 15-38. [ Links ]
Leotta G.A., I. Chinen, S. Epszteyn et al. 2005. Validación de una técnica de PCR múltiple para la detección de Escherichia coli productor de toxina Shiga. Rev. Arg. Microbiol. 37:1-10. [ Links ]
Lukásová J., B. Abraham & S. Cupáková. 2004. Occurrence of Escherichia coli O157 in Raw Material and Food in Czech Republic. J. Vet. Med. B 51: 77-81. DOI: 10.1111/j.1439-0450.2004.00727.x [ Links ]
Marzocca M.A., P.L. Marucci, M.G. Sica et al. 2006. Detección de Escherichia coli O157:H7 en carne picada fresca y hamburguesas congeladas. Rev. Argent. Microbiol. 38: 38-40. [ Links ]
Máttar S., J. Pulido, E. Vásquez et al. 1997. Enteropathogenic Escherichia coli strains isolated in Colombia from children with diarrhoea: Serotypes and drug-resistence. Medical science research 25(2): 615-617. [ Links ]
Mead P.S. & P.M. Griffin. 1998. Escherichia coli O157:H7. Lancet 352: 12071212. DOI:10.1016/S0140-6736(98)01267-7 [ Links ]
Michimo H., K. Arakaki, S. Minami et al. 1999. Massive Outbreak of Escherichia coli O157:H7 infection in schoolchildren in Sakai City, Japan, associated with consumption of White radish sprouts. Am. J. Epidemiol. 150: 787-796. [ Links ]
Mora A., M. Blanco, J.E. Blanco et al. 2003. Prevalencia, serotipos y genes de Virulencia de los Escherichia coli verotoxigénicos (O157 y No O157) en el ganado y carne de bovino. Rev. Epidem. Med. Prev. 1: 34-36. [ Links ]
Mora A., S.L. León, M. Blanco, J.E. Blanco, C. López, G. Dahbi, A. Echeita, E.A. González & J. Blanco. 2007. Phage types, virulence genes and PFGE profiles of Shiga toxin-producing Escherichia coli O157:H7 isolated from raw beef, soft cheese and vegetables in Lima (Peru). International Journal of Food Microbiology 114 (2): 204-210. doi:10.1016/j.ijfoodmicro.2006.09.009. [ Links ]
Morato Bergamini A.M., M. Simoes, K. Irino et al. 2007. Prevalence and characteristics of Shiga toxin-producing Escherichia coli (STEC) strains in ground beef in Sao Paulo, Brazil. Braz. J. Microbiol. 38 (3): 553-556. DOI: 10.1590/S1517-83822007000300032 [ Links ]
Muniesa M. & J. Jofre. 2004. Abundance in Sewage of Bacteriophages Infecting Escherichia coli O157:H7. Methods Mol. Biol. 268: 79-88. DOI: 10.1385/1-59259-766-1:079 [ Links ]
Nataro J.P. & Karper J.B. 1998. Diarrheagenic Escherichia coli. Clin. Microbiol. Rev. 11: 142-201. [ Links ]
Obrien A.D. & R.K. Hornes. 1987. Shiga and Shiga-like toxins. Microbiol. Rev. 51: 206-220. [ Links ]
Ochoa T.J., E.H. Mercado, D. Durand et al. 2011. Frecuencia y patotipos de Escherichia coli diarrogénica en niños peruanos con y sin diarrea. Rev. Peru Med. Exp. Salud Publica 28(1): 13-20. [ Links ]
Orskov F., I. Orskov & J.A. Villar. 1987. Cattle as reservoir of verotoxinproducing Escherichia coli O157:H7. Lancet: 276. [ Links ]
Phillips A.D., S. Navabpour, S. Hicks et al. 2000. Enterohaemorrhagic Escherichia coli O157:H7 target Peyer´s patches in humans and cause attaching/effacing lesions in both human and bovine intestine. Gut 47: 377-381. DOI: 10.1136/gut.47.3.377 [ Links ]
Piedrahita D., T. Márquez & S. Mattar. 2001. Detección de Escherichia coli O157:H7 en poblaciones porcinas, canal bovina y productos cárnicos en el Departamento de Córdoba. MVZ-CORDOBA 6 (2): 119-126. [ Links ]
Pistone V., P. Nuñez, J. Boccoli et al. 2006. Papel de la toxina Shiga en el síndrome urémico hemolítico. Medicina (Buenos Aires) 66 (Suplemento III): 11-15. [ Links ] [ Links ]
RM. N°. 591-2008-MINSA. Aprueban : «Norma sanitaria que establece los criterios microbiológicos de calidad sanitaria e inocuidad para los alimentos y bebidas de consumo humano » EL Peruano, Normas Legales, 29 de agosto de 2008 : 378827.
Riley L.W., R.S. Remis, S.D. Helgerson et al. 1983. Hemorrhagic colitis associated with a rare Escherichia coli serotype. N. Engl. J. Med. 308: 681-685. DOI: 10.1056/NEJM198303243081203 [ Links ]
Rivas M., S. Sosa-Estani, J. Rangel et al. 2008. Risk Factors for Sporadic Shiga Toxin-producing Escherichia coli Infections in Children, Argentina. Emerg. Infect. Dis. 14(5): 763-771. DOI: 10.3201/ eid1405.071050 [ Links ]
Rivera F.P., E. Sotelo, I. Morales et al. 2012. Short Communication: Detection of Shiga toxin producing Escherichia coli (STEC) in healthy cattle and pigs in Lima- Perú. J. Dairy Sci. 95: 1166-1169. doi:10.3168/ jds.2011-4662 [ Links ]
Roldán M.L., I. Chinen, J.L. Otero et al. 2007. Aislamiento, caracterización y subtipificación de cepas de Escherichia coli O157:H7 a partir de productos cárnicos y leche. Rev. Argent. Microbiol. 39:113-119. [ Links ]
Schmidt H., L. Beutin & H. Karch. 1995. Molecular analysis of the plasmid encoded hemolysin of Escherichia coli O157:H7 strain 933. Infect. Immun. 63: 1055-1063. [ Links ]
Siegler R.L. 1995. The Hemolytic Uremic Syndrome. Pediatr. Clin. North Am. 42: 1505-1529. [ Links ]
Strockbine N.A., L.R.M. Marques, J.W. Newland et al. 1986. Two toxin- converting phages from Escherichia coli O157:H7 strain 933 encoded antigenically distinct toxins with similar biological activities. Infect. Immun. 53: 135-140. [ Links ]
US/FIS. United States Food Safety. 2005. Office of Laboratory QA/QC Division. Laboratory Guidebook FSIS Procedure for the use of Escherichia coli O157:H7 and O157:NM (non motile) Screening tests. [ Links ]
Varela G., I. Chinen, P. Gadea et al. 2008. Detección y caracterización de E. coli productor de toxina Shiga a partir de casos clínicos y de alimentos en Uruguay. Rev. Argent. Microbiol. 40: 93-10. [ Links ]
Wetzel A.N. & J.T. Le Jeune. 2007. Isolation of Escherichia coli O157:H7 strains that do not produce Shiga toxin from bovine, avian, and environmental sources. Letters in Appl. Microbiol. 45: 504-507. DOI: 10.1111/j.1472-765X.2007.02228.x [ Links ]
WHO (World Health Organization). 1997. Consultation on prevention and control of enterohemorrahagic (EHEC) infections. In: World Health Organization. Proceedings of the Report of a WHO. Consultation, Geneva, Switzerland. [ Links ]
Presentado: 22/02/2013
Aceptado: 25/08/2013
Publicado online: 09/12/2013
 Cited by SciELO
Cited by SciELO  Similars in
SciELO
Similars in
SciELO  uBio
uBio 















