Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.20 no.3 Lima dic. 2013
TRABAJOS ORIGINALES
Agrotransformación y evaluación de la resistencia a Phytophthora infestans en Solanum tuberosum L. variedad Désirée
Agro-transformation and evaluation of resistance to Phytophthora infestans in Solanum tuberosum L. variety Désirée
Jeanette Orbegozo, María Lupe Román, Cristina Rivera, José Carlos Tovar, Willmer Pérez, Soledad Gamboa, Greg Forbes, Jan Kreuze y Marc Ghislain
Laboratorio de Biotecnología Aplicada, Centro Internacional de la Papa (CIP), Apartado 1558, Lima 12, Perú.
E-mail Jeanette Orbegozo: jorbegozoramirez@gmail.com
E-mail María Lupe Román: m.roman@cgiar.org
E-mail Cristina Rivera: c.rivera@cgiar.org
E-mail José Carlos Tovar: josectovar@gmail.com
E-mail Willmer Pérez: w.perez@cgiar.org
E-mail Soledad Gamboa: s.gamboa@cgiar.org
E-mail Greg Forbes: g.forbes@cgiar.org
E-mail Jan Kreuze: j.kreuze@cgiar.org
E-mail Marc Ghislain: m.ghislain@cgiar.org
Resumen
El Oomiceto Phytophthora infestans (Mont.) de Bary, agente causal de la enfermedad denominada tizón tardío, es el principal responsable del déficit en rendimiento y producción en el cultivo de papa a nivel mundial; una de las alternativas a considerar en la lucha contra este patógeno es la integración de secuencias completas de genes R en el genoma de la papa a través de Agrotransformación. El gen Rpi-blb2 (gen R) de la especie silvestre Solanum bulbocastanum Dunal, presenta una amplia resistencia a los aislamientos de P. infestans, haciéndolo un importante candidato en los estudios de mejoramiento genético en plantas. En el presente trabajo se describe la introducción del gen Rpi-blb2 por Agrobacterium tumefaciens en el genoma de la papa variedad Désirée, la caracterización molecular de 29 eventos transformados e infección de plantas completas con el aislamiento POX067 de P. infestans obtenido en el Perú. Los eventos Désirée [Rpi-blb2] 4 y Désirée [ Rpi-blb2] 30, presentaron una resistencia considerable frente a la infección de P. infestans, comprobando de esta manera la transferencia del gen Rpi-blb2 de una especie silvestre a una cultivada mediante transformación genética.
Palabras clave: papa; Phytophthora infestans; Agrobacterium tumefaciens; gen Rpi-blb2.
Abstract
The Oomycete Phytophthora infestans (Mont.) de Bary, the causal agent of the disease known as late blight, is primarily responsible for the decreased in production performance and potato crops worldwide. The integration of the complete R genes sequences in the potato genome using Agro-transformation appears an alternative to be considered in the fight against this pathogen. The Rpi-blb2 gene (R gene) from the wild species Solanum bulbocastanum Dunal shows a broad resistance to isolates of P. infestans, making it an important candidate for plant breeding studies. This paper reports the integration of the Rpi-blb2 gene into potato var. Désirée genome by Agrobacterium tumefaciens - mediated transformation system, the molecular characterization of 29 events transformed and whole plant infection with isolate POX67 of P. infestans from Peru. Désirée events [Rpi-blb2] 4 and Désirée [Rpi-blb2] 30, showed a substantial resistance to P. infestans infection confirming complete transfer of the Rpi-blb2 gene from a wild species to a cultivated species by genetic transformation.
Keywords : potato; Phytophthora infestans; Agrobacterium tumefaciens; Rpi-blb2 gene.
Introducción
Desde el inicio del uso de la bacteria Agrobacterium tumefaciens en los años 80, se revolucionó la obtención de plantas resistentes a diversos patógenos de una manera rápida y sencilla, siendo considerado hasta la fecha como un proceso importante en la biotecnología vegetal (Petti et al. 2009). Asimismo, la búsqueda de nuevas fuentes de resistencia ha impulsado el uso de genes de la misma especie o de una familia cercana a la planta que se desee modificar (Storck et al. 2012). Un ejemplo de ello son los genes R (genes de resistencia) de la familia de genes NB-LRR (Nucleotide binding and leucine-rich repeat), provenientes de la especie silvestre Solanum bulbocastanum (Collinge et al. 2008, Jacobs et al. 2010; Pel et al. 2009). Los genes R de S. bulbocastanum más representativos son Rpi-blb1, Rpi-blb2 y Rpi-blb3, y se encuentran localizados en los cromosomas 8, 6 y 4 respectivamente (Naess et al. 2000, Song et al. 2003). El interés que se tiene en estos genes radica en la resistencia que confieren ante el tizón tardío, enfermedad ocasionada por el Oomiceto Phytophthora infestans y descrita como la más devastadora en el cultivo de la papa (Garelik 2002, Fry 2008).
La lucha contra P. infestans es continua debido a su elevado potencial evolutivo y debido a que en su genoma posee regiones altamente repetitivas, las cuales presentan características muy dinámicas que les permite secretar proteínas efectoras (AVR) facilitando su colonización y patogénesis en la planta hospedera (Haas 2009). En consecuencia, la obtención de plantas con resistencia a P. infestans es apremiante. Es por este motivo, que el estudio de los genes R a tenido gran atención, como es el caso del gen Rpi-blb2, el cual presenta un amplio espectro de resistencia a P. infestans (van der Vossen et al. 2005). Se ha demostrado que este gen evolucionó más recientemente que el gen RB / Rpi-blb1 y codifica una proteína de 1,267 aminoácidos, cuya secuencia génica presenta 82% de similitud con el gen Mi-1 de tomate, el cual también confiere resistencia a nematodos, áfidos y a la mosca blanca (Nombela et al. 2003, Lokossou et al. 2010).
En el 2011, el grupo BASF (Alemania) obtuvo el cultivo de papa Fortuna, a partir de la introducción de genes Rpi-blb2, así como otros genes no reportados de S. bulbocastanum y otras especies silvestres. Sin embargo, de estas investigaciones sólo se conocen notificaciones de avances, y en la actualidad el grupo ha retirado su solicitud de liberación del cultivo en la Unión Europea por temas de mercado (Gillund et al. 2013). Debido a esta situación poco se conoce acerca de la resistencia que puede conferir este gen en cultivos de interés, es por ello que el presente trabajo provee de información actual en la generación de plantas transgénicas de S. tuberosum var. Désirée con el gen Rpi-blb2, así como la evaluación de la resistencia frente la infección con el aislado POX067 de P. infestans.
Materiales y métodos
Material vegetal.- Las plántulas de papa var. Désirée fueron obtenidas del banco de germoplasma del Centro Internacional de la Papa (CIP) (CIP800048). Se usaron explantes tipo hoja-peciolo y entrenudos con cuatro semanas de su propagación in-vitro. El aislamiento POX067 (Oxapampa, Pasco), se obtuvo de S. tuberosum var. Amarilis, perteneciente al tipo de apareamiento A1 y linaje clonal EC-1 (Pérez et al. 2001).
Diseño del plásmido pCIP95.- La secuencia completa del gen Rpi-blb2 fue obtenida del GenBank (número de accesión DQ122125.1), y sintetizado por la empresa Entelechon GmbH (Alemania). Posteriormente este gen fue clonado en el plásmido pCAMBIA1300 obteniéndose el plásmido pCIP95 (Figura 1). Por transformación de shock térmico se introdujo el plásmido en la cepa DH5α de E. coli y por electroporación en la cepa EHA105 de A. tumefaciens (Hood et al. 1993, Sambrok & Russell 2001).
Evaluación de la sensibilidad de los explantes de S. tuberosum var. Désirée al agente selector higromicina.- Por cada tipo de explante se usó 30 unidades sin transformar, las cuales fueron colocadas en un medio de regeneración semisólido (4.60 g/L de sales Murashigue & Skoog, 2% de glucosa, 0.02 mg/L de ácido giberélico, 0.02 mg/L de ácido naftalen acético, 2 mg/L de ribósido de zeatina, pH 5.6 y 0.23% de gelride) complementado con una gradiente de concentración del antibiótico higromicina: 1, 5, 10, 15, 20, 25 y 30 mg/L. El control negativo fue la var. Désirée (no transformada) colocada en el medio de propagación sin antibiótico. El cambio de placas se realizó cada 15 días.
Transformación de papa.- La transformación fue vía organo génesis directa, siguiendo el protocolo establecido por Medina et al. 2003, con modificaciones para la var. Désirée. Las plantas fueron propagadas in-vitro en magentas en un medio líquido de propagación (4.3 g/L de sales Murashige & Skoog, 0.4 mg/L de tiamina, 2 mg/L de glicina, 0.5 mg/L de ácido nicotínico, 0.5 mg/L de piridoxina, 0.1 mg/L de ácido giberélico, 2% de sacarosa y pH 5.6).
La bacteria A. tumefaciens conteniendo el plásmido pCIP95 fue sembrada en el medio semisólido Luria-Bertani (LB) (1% de bacto-triptona, 0.5% de extracto de levadura, 1% NaCl, pH 7.5 y 2.5 g/L de agar) con 100 mg/L de kanamicina, a 28 °C por 48 horas.
Los explantes fueron cortados de forma transversal con una cuchilla estéril que previamente fue puesta en contacto con una colonia de A. tumefaciens. Los explantes tratados fueron transferidos a un medio que contenía: 4.60 g/L de sales Murashige & Skoog, 2% de sacarosa, 30 mg/L de acetosiringona, pH 5.6 y 0.23% de gelride, sin antibiótico de selección, a 18 – 22 °C por 24 horas en oscuridad. Posteriormente, éstos fueron colocados en el medio de regeneración semisólido con el antibiótico higromicina y 200 mg/L de carbenicilina. Cada 15 días el material vegetal fue transferido a placas conteniendo un medio de regeneración nuevo. Una vez obtenidos los regenerantes fueron individualizados para su posterior evaluación. Los cálculos de las eficiencias de regeneración y transformación fueron obtenidos según García et al. (2008) y Luo et al. (2006), respectivamente.
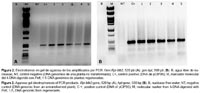
Caracterización molecular por PCR.- El proceso de extracción de DNA se realizó siguiendo lo descrito por Doyle y Doyle (1990). Para la reacción en cadena de la polimerasa (PCR) se usaron los siguientes sets de cebadores: gen Rpi-blb2: Blb2-F: 5'- AATACGCAAACCGCCTC-3' y Blb2-R: 5'-AGCTTCAGATCCTTGGCC-3' (Vector NT 9.1.0), para el gen hpt: hpt-F: 5'-TCCATCACAGTTTGCCAGTGATACA-3' y hpt-R: 5'-ATGAAAAAGCCTGAACTCACCGCGA-3' (Nishizawa et al. 1999). El volumen final de la reacción de PCR fue de 15μL, conteniendo 10ng de DNA, 1X PCR buffer (Promega©), 0.4 μM de cada cebador, 0.5 μM dNTPs y 1 U de la enzima Taq DNA polimerasa (Promega©). El programa de PCR usado fue: 95 °C por 5 min, la temperatura de hibridación para ambos cebadores fue de 56 °C con 34 ciclos de 95 °C por 1 min, 56 °C por 45 s y 72 °C por 1 min, y una elongación final de 72 °C por 5 min. Una vez terminada la amplificación los productos de PCR se mezclaron con solución de carga (0.25% azul de bromofenol, 40% sacarosa) y separados en geles de agarosa al 1% en amortiguador TBE 1X (20 mM Tris-borato y 0.5 mM EDTA, pH 8.0). Los tamaños del producto de PCR fueron comparados con el peso molecular del marcador Lamdda DNA previamente digerido con PstI. El gel fue teñido con bromuro de etidio para la visualización de los fragmentos de DNA a través de la irradiación del gel con luz UV (Sambrok & Russell 2001). Imágenes del gel donde se observan los fragmentos de DNA obtenidos por PCR fueron capturadas con el equipo EpiChemi3 Darkroom para su posterior análisis.
Ensayo de infección con P. infestans.- Se usaron 2 plantas por evento de transformación de la segunda propagación vegetativa, de un mes y medio de siembra. Los ensayos de infección se realizaron en los cubículos de bioseguridad del CIP bajo condiciones controladas de temperatura 16 – 18 °C, 12 h /12 h de exposición de luz/oscuridad y 60 – 75% de humedad relativa. La infección se realizó por aspersión de la planta completa con una suspensión de 3000 esporangios/mL del aislamiento POX067 de P. infestans. Una vez inoculadas las plantas, estas fueron colocadas en cámaras con humedad relativa alta (> 90%) para promover y aumentar el desarrollo del patógeno. Las plantas de S. tuberosum var. Désirée (susceptible) y el clon CIP 387164.4 (resistente) fueron usadas como control negativo y positivo, respectivamente. Las evaluaciones fueron realizadas por el Laboratorio de Patología del CIP al quinto día post infección (dpi). El daño foliar observado en cada uno de los clones se reportó en porcentaje y los valores promedios fueron analizados usando la siguiente escala: Altamente resistente= ≤10%, resistente= ≥11 – 20%, medianamente resistente = ≥21 – 50%, medianamente susceptible =50 – 65%, susceptible =>65 – 80%, altamente susceptible =>80%.
Resultados y Discusión
Determinación de la dosis letal de higromicina y transformación genética.- A través de los ensayos de sensibilidad al antibiótico higromicina se determinó que la dosis letal para los explantes hoja-peciolo y entrenudos no transformados fue de 5 mg/L y 10 mg/L, respectivamente. Las soluciones del antibiótico en las concentraciones descritas arriba fueron usadas de manera directa sobre los explantes, sin necesidad de adicionar un paso previo de precultivo, el cual incrementa la aparición de eventos considerados escapes (Chaudhry y Rashid 2010). Durante los ensayos de transformación se obtuvieron 29 regenerantes a partir de 898 explantes transformados (364 entrenudos y 534 hoja-peciolo), reportándose una eficiencia de regeneración de 1.5% para hoja-peciolo y 5.7% para entrenudos. Así también, se registro una eficiencia de transformación de 1.5% en hoja-peciolo y 5% para entrenudos.
La caracterización molecular de los 29 regenerantes obtenidos, identificó 26 eventos positivos conteniendo el gen de interés. De este análisis se concluye que los explantes tipo entrenudos presentaron una mejor respuesta de eficiencia de transformación (18 eventos positivos) que los explantes hoja-peciolo (8 eventos positivos). Esta observación confirma lo descrito por Beaujean et al. (1998), quienes al trabajar con las variedades Désirée, Bintje y Kaptah Vandel, identificaron que los explantes tipo entrenudos presentan una mejor respuesta a las condiciones in vitro debido a que son menos sensibles al daño durante los pasos de manipulación de transformación y tienen un alto poder regenerativo a diferencia de las hojas (Sarker et al. 2009, Soto et al. 2007). En relación al uso de higromicina durante el proceso de obtención de transformantes, se concluye que la eficiencia en ambos tipos de explantes fueron bajas en el presente trabajo esto se determinó al comparar los resultados obtenidos con ensayos reportados previamente donde se describe el uso del mismo antibiotico (M'Hamdi et al. 2003, Kashani et al. 2012). Estas diferencias pueden deberse a múltiples factores asociados a las condiciones de transformación o al tipo de explante usado (Lagunes 2009). No obstante la principal causa puede deberse a que los ensayos de regeneración y transformación son genotipo-dependiente del tipo de cultivo que se use en los experimentos a realizar (Cingel et al. 2010).
Análisis de eventos transformados por reacción en cadena de la polimerasa (PCR).- De los 29 eventos obtenidos en el primer experimento de transformación, 26 (90%) eventos fueron positivos por PCR para las amplificaciones de los genes Rpi-blb2 y hpt (Figuras 2A y 2B). Los eventos restantes fueron considerados como escapes, esto se puede deber a una proliferación de algunas células no transformadas presentando una resistencia o tolerancia natural ante el agente selector (López y Chaparro 2007). Así también es posible que la superficie de estos explantes no haya tenido un contacto perenne con el medio de cultivo haciendo favorable su desarrollo (Monserrat et al. 2001).
Respuesta de resistencia a la infección con P. infestans.-
Del total de los 26 eventos transformados, solo 13 presentaron aclimatación total y produjeron tubérculos en invernadero. Los eventos Désirée [Rpi-blb2]: 4 y Désirée [blb2] 30 registraron un 42.5% y 37.5% de severidad, respectivamente (Figura 3). La respuesta de hipersensibilidad (HR) inducida por la infección en estos dos eventos no estuvo correlacionada con una completa resistencia al patógeno, otorgando sólo una resistencia moderada. Esto pudo deberse a que el aislamiento POX067 presenta una gran variedad de genes Avr (genes de avirulencia de P.infestans) incluyendo a los Avr8 y Avr9 identificados previamente en infecciones en S.demissum ( Villamon et al., 2005). Es probable que estos genes Avr incrementen la agresión del aislado POX067durante la infección a la var. Désirée. Por lo general, la respuesta HR es asociada con otro tipo de respuestas que cooperan para restringir el desarrollo del patógeno (Chen & Halterman, 2011). Es por ello que la muerte celular y la resistencia pueden actuar de manera independiente, indicando que múltiples respuestas, así como la presencia de más de un gen R por parte de los hospederos, serían necesarias para lograr obtener resistencia o inmunidad. La detección de los eventos Désirée [Rpi-blb2] 4 y Désirée [Rpi-blb2] 30, muestra el éxito logrado en el presente trabajo debido a que las infecciones se realizaron en una población pequeña (13 plantas transgénicas) y con el aislamiento POX067 el cual es considerado una raza compleja y agresiva.
Agradecimientos
El presente trabajo fue realizado gracias al financiamiento de la Agencia de los Estados Unidos para el Desarrollo Internacional (USAID).
Literatura citada
Beaujean A., R.S Sangwan, A. Lecardonnel & B.S. Sangwan-Norreel. 1998. Agrobacterium-mediated transformation of three economically important potato cultivars using sliced internodal explants: an efficient protocol of transformation. Journal of Experimental Botany 49 (326): 1589–1595. doi: 10.1093/jxb/49.326.1589.
Chaudhry Z., & H. Rashid. 2010. An improved Agrobacterium mediated transformation in tomato using hygromycin as a selective agent. African Journal of Biotechnology 9 (13): 1882-1891. doi: http://dx.doi.org/10.4314%2Fajb.v9i13.
Chen Y. & D.A Halterman. 2011. Phenotypic characterization of potato late blight resistance mediated by the broad-spectrum resistance gene RB. Phytopathology 101(2):263-70. doi: 10.1094/PHYTO-04-10-0119.
Cingel A., B. Vinterhalter, D. Vinterhalter, et al. 2010. Agrobacterium-mediated transformation of two Serbian potato cultivars (Solanum tuberosum L. cv. Dragačevka and cv. Jelica). African Journal of Biotechnology 9(30):4644-4650. doi: 10.5897/AJB09.1241.
Collinge D.B., O.S Lund & H.T Christensen. 2008. What are the prospects for genetically engineered, disease resistant plants?. Eur J Plant Pathol 121:217–231. Doi: 10.1007/s10658-007-9229-2.
Doyle J.J & J.L. Doyle. 1990. Isolation of plant DNA from fresh tissue. Focus 12:13-15. DOI: 10.1313/1-92559-287-7:141.
Fry W., 2008. Phytophthora infestans: the plant (and R gene) destroyer. Mol Plant Pathol 9(3):385-402. doi: 10.1111/j.1364-3703.2007.00465.x. [ Links ]
García R., D. Somonte, Z. Zaldúa, et al. 2008. Efficient regeneration and Agrobacterium tumefaciens mediated transformation of recalcitrant sweet potato (Ipomoea batatas L.) cultivars. AsPac J. Mol. Biol. Biotechnol 16 (2): 25-33. doi: 10.1313/1-259-627-7:111.
Garelik G. 2002. Agriculture. Taking the bite out of potato blight. Science 298(5599):1702-1704. doi: 10.1126/science.298.5599.1702 [ Links ]
Gillund F., A. Hilbeck, O.G Wikmark, L. Nordgard & Bohn T. 2013. Genetically Modified Potato with Increased Resistance to P. infestans - Selecting Test Species for Environmental Impact Assessment on Non-Target Organisms. Biosafety Report 2013/01. Available online: http://genok.no/wp-content/uploads/2013/03/Biosafety_Report_2013_01.pdf. Accessed 23.01.2013
Haas B.J, S. Kamoun, M.C. Zody, R H. Y. Jiang, et al. 2009. Genome Sequence and Analysis of The Irish Potato Famine Pathogen Phytophthora Infestans. Nature 461(7262): 393-398. doi:10.1038/nature08358
Hood E. E., S. B. Gelvin, L. S. Melchers, & A. Hoekema. 1993. New Agrobacterium helper plasmids for gene transfer to plants. Transgenic Res 2:208-218. DOI:10.1007/BF01977351.
Jacobs M.M., B. Vosman, V.G Vleeshowers, R.G Viser, B, et al. 2010. A novel approach to locate Phytophthora infestans resistance genes on the potato genetic map. Theor Appl Genet. 120(4):785-96. doi: 10.1007/s00122-009-1199-7.
Kashani K., M.J Javaran, M. Mohebodini, A. Moieni; M. Sheikhi & D. Abad 2012. Regeneration and Agrobacterium-mediated transformation of three potato cultivars (Solanum tuberosum cv. Désirée Agria and Marfona) by human proinsulin gene. AJCS 6(7):1212-1220.ISSN 1835-2693.
Lagunes F.E. 2009. Transformación genética de ajo (Allium sativum L.) mediante Agrobacterium tumefaciens. Transformación genética de ajo (Allium sativum L.) mediante Agrobacterium tumefaciens. Tesis, Magister en Recursos Genéticos y Productividad, mención en Ciencias. Colegio de Postgraduados Campus Montecillo, Texcoco, Edo. de México. p.95. http://www.cm.colpos.mx/2010/images/tesis_p/fisiologiaV/resumen/resumen_transformaci%F3n.pdf [ Links ]
Lokossou A.A, H. Rietman, M. Wang, P. Krenek, et al. 2010. Diversity, distribution, and evolution of Solanum bulbocastanum late blight resistance genes. Mol Plant Microbe Interact. 23(9):1206-16. doi: 10.1094/MPMI-23-9-1206.
López A. & A. Chaparro. 2007. A system for transformation potato plants (Solanum tuberosum sp. andigena var. Pastusa suprema) mediated through Agrobacterium tumefaciens. Agronomía Colombiana 25(1):16-25. ISSN 0120-9965.
Luo H.R., M. Santa Maria, J. Benavides, D.P Zhang, Y.Z Zhang & M. Ghislain. 2006. Rapid genetic transformation of sweetpotato (Ipomoea batatas (L.) Lam) via organogenesis. African Journal of Biotechnology. 5(20):1851-1857. ISSN 1684–5315.
MHamdi M., C. Rouvière, J. Rojas-Beltran, P. Du Jardin. 2003. Optimisation de la transformation génétique de la ponme de terre par Agrobacterium tumefaciens. Utilisation de la résistance à lhygromycine comme marqueur sélectif. Biotechnol. Agron. Soc. Environ. 7 (3–4):183–188. ISSN 1370-6233.
Medina-Bolivar F., R. Wright, V. Funk, et al. 2003. A non-toxic lectin for antigen delivery of plant-based mucosal vaccines. Vaccine. 21(9-10):997-1005. doi. org/10.1016/S0264-410X(02)00551.
Monserrat E., V. Marfa, E. Melé & J. Messenguer. 2001. Study of different antibiotic combinations for use in the elimination of Agrobacterium with kanamycin selection in cRNAation. Plant Cell Tissue and Organ Culture 65: 211-220. DOI:10.1023/A:1010630726444.
Naess S., J. Bradeen, S. Wielgus, et al. 2000. Resistance to late blight in Solanum bulbocastanum is mapped to chromosome 8. Theor Appl Genet.10: 697–704. DOI: 10.1007/s001220051533.
Nishizawa Y., A. Kawakami, T. Hibi, D. He, N. Shibuya & E. Minami. 1999. Regulation of the chitinase gene expression in suspension-cultured rice cells by N-acetylchitooligosaccharides: differences in the signal transduction pathways leading to the activation of elicitor-responsive genes. Plant Mol Biol. 39(5):907-914. DOI: 10.1023/A:1006161802334.
Nombela G., V.M Williamson, & M. Muniz. 2003. The root-knot nematode resistance gene Mi-1.2 of tomato is responsible for resistance against the whitefly Bemisia tabaci. Mol. Plant Microbe.16(7):645-9. doi.org/10.1094/MPMI.2003.16.7.645.
Pel M.A., S.J Foster, T.H Park, H. Rietman, et al. 2009. Mapping and cloning of late blight resistance genes from Solanum venturii using an interspecific candidate gene approach. Mol Plant Microbe Interact. 22(5):601-15. doi: 10.1094/MPMI-22-5-0601.
Pérez W., J. Gamboa, Y. Falcon, M. Coca, et al. 2001. Genetic structure of Peruvian populations of Phytophthora infestans. Phytopathology.91:956-965. doi: 10.1094/PHYTO.2001.91.10.956.
Petti C., T. Wendt, C. Meade & E. Mullins. 2009. Evidence of genotype dependency within Agrobacterium tumefaciens in relation to the integration of vector backbone sequence in transgenic. Phytophthora infestans-tolerant potato. J Biosci Bioeng.107(3):301–306.doi.org/10.1016/j.jbiosc.2008.11.012
Sambrook J. & D. Russell. 2001. Molecular Cloning: a Laboratory Manual 3er ed. Cold Spring Harbor Laboratory Press, New York, USA. [ Links ]
Sarker S.R., M. Hossain & F. Shirin. 2009. Precise Incubation Period Increases the Agrobacterium Mediated Transformation Efficiency in Potato (Solanum tuberosum L.) cvs. Cardinal and Atlas. Plant Tissue Cult. & Biotech. 19(2): 227-235. DOI. 10.3329/ptcb.v19i2.5440.
Song J., J.M Bradeen, S.K Naess, et al. 2003. Gene RB cloned from Solanum bulbocastanum confers broad spectrum resistance to potato late blight. Proc Natl Acad Sci U S A. 100(16):9128-9133. doi: 10.1073/pnas.1533501100.
Soto N., G.A Enríquez, A. Ferreira, et al. 2007. Efficient transformation of potato stems segments from cultivar Désirée using phosphinothricin as selection marker. Biotecnología Aplicada. 24:139-144. ISSN: 0864-4551.
Storck T., T. Böhme & H. Schultheiss. 2012. Fortuna et al. Status and per-Status and perspectives of GM approaches to fight late blight. PPO-Special.15: 45 – 48. DOI: 09.11/j.6531-413.2012.02927.
Van Der Vossen E.A., J. Gros, A. Sikkema, M. Muskens, et al. 2005. The Rpi-blb2 gene from Solanum bulbocastanum is an Mi-1 gene homolog conferring broad-spectrum late blight resistance in potato. Plant J. 44(2):208-222. DOI: 10.1111/j.1365-313X.2005.02527.x
Villamon F.G., D.M. Spooner, M. Orrillo, E. Mihovilovich, W. Perez, & M. Bonierbale. 2005. Late blight resistance linkages in a novel cross of the wild potato species Solanum paucissectum (series Piurana). Theor. Appl. Genet. 111:1201-1214. DOI 10.1007/s00122-005-0053-9.
Presentado: 18/12/2013
Aceptado: 15/01/2014
Publicado online: 14/03/2014