Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.20 no.3 Lima dic. 2013
TRABAJOS ORIGINALES
Diversidad genética de papas nativas ( Solanum spp.) conservadas en cultivares nativos del Perú
Genetic diversity of native potatoes ( Solanum spp.) conserved in landraces from Peru
Julián Soto3, Tulio Medina2, Yeny Aquino2, Rolando Estrada1*
1 Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos, Ciudad Universitaria de San Marcos Av. Venezuela s/n. Apartado 110058, Lima 11 – Perú.
2 Instituto Nacional de Investigación Agraria-INIA, Av. La Molina # 1981. Apartado Postal 2791, La Molina, Lima - Perú
3 Centro Internacional de la Papa, CIP-Lima. Av. La Molina 1895 La Molina Apartado 1558, Lima 12, Perú
*Autor para correspondencia
Email: Rolando Estrada: restradaj@gmail.com
Email: Julián Soto: julian4881@yahoo.es
Resumen
El presente trabajo analiza el grado de diversidad genética utilizando 18 marcadores micro-satélites, de una muestra aleatoria de 79 variedades nominales de papa nativa (Solanum spp.) procedentes de cinco regiones políticas del Perú (Ayacucho, Cajamarca, Cusco, Huancavelica y Puno), cultivadas en "chacras" de agricultores que colaboraron con el proyecto "Conservación in situ de los cultivos nativos y sus parientes silvestres". De los 18 marcadores, 17 amplifica-ron un solo locus polimórfico, siendo el promedio de alelos por locus de 8.79. Se obtuvo una similitud media de 0.62 y rangos de agrupamiento que varíaron desde 0.41 a 0.98. Para los 19 loci registrados se obtuvo un total de 166 alelos. La región de Cuzco presentó el mayor número de alelos (130 alelos). De los 166 alelos caracterizados, 72 alelos (43.37%) fueron comunes o compartidos con las 5 regiones de colecta. La región de Puno presento el mayor numero de alelos exclusivos (8 alelos). Las 42 variedades nominales de S. tuberosum subsp. andigena tuvieron una diversidad promedio de 0.74 y las 18 variedades nominales de S. x chaucha una diversidad promedio de 0.70. Los valores de polimorfismo (PIC = 0.55 – 0.85) y los índices de diversidad genética obtenidos indicarían que los microsatélites evaluados logran identificar altos niveles de diversidad genética, pero a la vez no son suficientes para discriminar grupos diferenciados por procedencia o especies. Nuestros análisis indican que existe un alto grado de diversidad genética y corroboran los resultados obtenidos de los inventarios y caracterizaciones morfológicas realizadas in situ; también podemos concluir que existiría un pool de genes común que se encontrarían ampliamente distribuidos entre las regiones estudiadas.
Palabras claves: Conservación in situ; Diversidad genética; papa nativa; microsatélites; SSR.
Abstract
This paper analyzes the genetic diversity of 79 accessions of native potato varieties (Sola-num spp.) using 18 microsatellite markers. A random sample from Ayacucho, Cajamarca, Cusco, Huancavelica and Puno from "chacras" of farmers who collaborated with the "In situ conservation of native crops and wild relatives" were used. 17 markers amplified one single polymorphic locus, the mean number of alleles per locus was 8.79. The mean similarity was 0.62 and clustering indexes varied between 0.41 and 0.98. 19 loci showed a total of 166 alle-les. Cuzco had the highest number of alleles (130 alleles). Of the 166 characterized alleles, 72 alleles (43.37%) were common or shared with 5 sampling sites. Puno had the highest number of exclusive alleles (8 alleles). The 42 varieties of S. tuberosum subsp. andigena showed a mean diversity of 0.74 and 18 varieties of S. x chaucha an average diversity of 0.70. Polymorphism (PIC = 0.55 to 0.85) and genetic diversity indices show that microsatellites evaluated can identify high levels of genetic diversity, but also are not sufficient to discriminate differentiated by origin or species groups. Our analyzes indicate a high genetic diversity and are consistent with inventories and morphological characterizations performed in situ, we can also conclude that there would be a common pool of genes would be found widely distributed among the regions studied.
Keywords: In situ conservation; genetic diversity; native potato; microsatellites; SSR.
Introducción
El género Solanum tiene más de 300 especies que forman tubérculos, entre especies cultivadas y silvestres; sin embargo el género, considerado altamente polimórfico y muy complejo (Linnaeus 1753 en Ochoa 1990) incluye alrededor de 2400 especies alrededor del mundo (Ochoa 1999).
En los Andes, el género está representado por ocho especies cultivadas y alrededor de 200 silvestres. La rica diversidad de las especies cultivadas está incluida en una serie poliploide (2n = 24, 36, 48 y 60), que incluye unas 4000 variedades comestibles, con alto potencial genético para el rendimiento y amplia adaptabi-lidad a diferentes climas, lo que le ha permitido convertirlo en uno de los cultivos de mayor importancia para la alimentación mundial (Estrada 2000, Hawkes 1962). A pesar del gran poli-morfismo que existe entre las ocho especies cultivadas de papa (Fig. 1), estas tienen como características comunes el producir numerosos tubérculos, de gran tamaño y agradables al paladar (Matsubayashi 1991), lo que las distingue de las especies silves-tres, que poseen gran diversidad de caracteres y que pueden ser incorporarlos en las especies cultivadas mediante cruzamientos o manipulaciones genéticas.
La alta variabilidad de caracteres de las especies silvestres incluye tolerancias y resistencias a estrés biótico y abiótico, que han permitido mejorar las variedades comerciales desde el punto de vista nutricional, agronómico, industrial y farmacéutico. Es por ello que cada vez se incrementa más el interés por conocer las características morfológicas, bioquímicas y moleculares de las especies silvestres y cultivadas para prevenir la erosión genética de las mismas.
En el Perú existe una gran diversidad cultural ligada a la conservación de la biodiversidad, en este contexto el proyecto "Conservación in situ de los cultivos nativos y sus parientes silvestres" ("Proyeto in situ", llevado a cabo entre los años 2001- 2006 en el Instituto Nacional de Innovación Agraria, INIA), estuvo orientado a reforzar la conservación de los cultivos nati-vos en las chacras, identificar los factores que lo hacen posible y elevar el nivel de conciencia sobre su valor biológico, cultural y nutricional en el ámbito local y nacional, contando con la participación de las comunidades campesinas del país (INIA 2005 a-e). Los resultados del proyecto permitieron sustentar el alto grado de diversidad que presentan estos cultivos y fueron la base científica más importante para justificar la necesidad de protección que debe ejercer el Estado en los aspectos culturales y biológicos sobre los cultivares de papa nativa.
Dentro de este contexto, se consideró necesario afianzar los resultados obtenidos en la caracterización morfológica de las papas nativas con el uso de marcadores moleculares. Para el caso de la papa, los microsatélites vienen siendo usados hace más de 18 años para la genotipificación de este cultivo. Provan (1996), fue uno de los primeros en estudiar el potencial de los micro-satélites para el análisis de la diversidad genética de cultivares de papa. Por otro lado, Milbourne et al. (1997) compararon la eficiencia de tres marcadores basados en PCR para diferenciar variedades de papa tetraploide llegando a la conclusión que los microsatélites presentaban mayor polimorfismo y mayores venta-jas que los AFLP y RAPD. En el Perú, Ghislain et al. (2001) en el Centro Internacional de la Papa (CIP), lograron seleccionar 18 iniciadores para secuencias microsatélites de papa, con un alto grado informativo y son este grupo de iniciadores los que fueron utilizados en el presente trabajo.
Materiales y métodos
Material Vegetal y Extracción de ADN.- Se colectaron al azar tubérculos de 79 variedades nominales de papa nativa (Solanum spp.) perteneciente a agricultores colaboradores del " Proyecto in situ" y procedentes de cinco regiones politicas: ocho variedades del distrito de Luricocha y 17 del distrito de Vinchos en la Región Ayacucho, 14 variedades del distrito de Ocongate en la Región Cusco, 15 variedades del distrito de Pomata en la Región Puno, 13 variedades del distrito de Huasmín en la Región Cajamarca y 12 variedades del distrito de Yauli en la Región Huancavelica. De las 79 variedades, se identificaron 6 especies de papa cultivada: S. tuberosum subsp. andigena (42 variedades), S. stenotonum subsp. stenotonum (5 variedades) yS. stenotonum subsp. goniacalyx (4 variedades), S. phureja (2 variedades), S. ajanhuri (1 variedad), S. x chaucha (19 variedades) y S. x curtilobum (6 variedades). El ADN fue aislado a partir de hojas jóvenes y frescas mediante el método CTAB modificado de Doyle y Doyle (1990).
Amplificación de microsatélites.- Se utilizaron 18 iniciadores microsatélites identificados como altamente informativos por Ghislain et al. (2004) y desarrollados por Milbourne et al. (1998).Lascondicionesdeamplificaciónsellevaronacabosegún protocolos del CIP (CIP 1997, Ghislain et al. 2001) adaptados de Provan et al. (1996). La mezcla de reacción constó de: 20 ng de ADN, 0.5 μM de cada iniciador, 2.5 mM de MgCl2, 0.2 mM de dNTPs, 1X de Taq buffer y 0.5 U de Taq polimerasa en un volumen total de 20 L. El programa de amplificación se desarrolló de la siguiente manera: 3 min. a 94 °C, dos minutos a la temperatura de alineamiento, 1.5 minutos a 72 °C, 30 ciclos de 1 minuto a 94 °C, dos minutos a la temperatura de alineamiento, 1.5 minutos a 72 °C y una elongación final de 5 minutos a 72 °C, utilizando un termociclador modelo PTC100 (MJ Reasearch Inc.). La temperatura de alineamiento fue usada según los datos reportados por Ghislain et al. (2001, 2004).
Los productos de amplificación fueron separados en geles denaturantes de poliacrilamida (acrilamida 6%, bisacrilamida 0.3%, Urea 7M y TBE 1X) mediante electroforesis vertical en un sistema de secuenciamiento modelo S2-Gibco aplicándole un voltaje de 1600 V (40W). Se utilizó como marcador de peso la reacción de secuenciamiento del plásmido pUC-18 (Kit de secuenciamiento -Promega) y la detección de los fragmentos se realizó mediante tinción con nitrato de plata (Promega Corporation 1996).
Análisis de datos.- El patrón de bandas obtenido para cada iniciadorfueregistradoenunamatrizbinariadondealasbandas presentes se les asignó el valor 1 y las ausentes el valor 0. Se calculó el índice de similaridad DICE (Dice 1945, Nei y Lei 1979) y se realizó un análisis de agrupamiento UPGMA mediante el programa NTSYSpc ver 2.0 (Rohlf 1993), además se realizó un análisisbootstrapconayudadelprogramaWinBoot(Yap1992). Las variedades analizadas se agruparon según su procedencia, para determinar la cantidad de alelos que presentaban del total registrado, se analizó la presencia de los alelos compartidos entre las cinco regiones políticas de colecta y se determinó el rango de alelos comunes y exclusivos en cada región.
Por último, se determinó el índice de diversidad genética (Nei 1973) para las especie S. tuberosum subsp. andigena (4x) y S. x chaucha (3x), por ser las que poseen mayor número de variedades en la muestra analizada. Teniendo en cuenta que su valor varía entre 0 a 1, siendo los valores cercanos a 1 los de máxima diversidad.
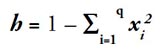
xi = frecuencia del alelo i
q = Nº de alelos observados en el locus
h = probabilidad que 2 alelos tomados al azar de la población sean diferentes (Nei 1973)
Resultados
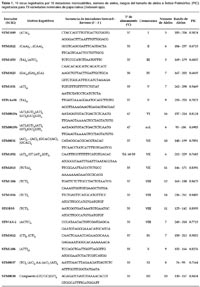
Número de alelos amplificados por locus e individuos no definidos.- De los 18 iniciadores microsatélites utilizados, 17 amplificaron un solo locus polimórfico mientras que el iniciador STM0019 amplificó 2 loci. El rango de alelos encontrados por locus, fue desde 5 (STM1049 y STM1053) a 16 alelos (STM0019a), siendo el promedio de alelos por locus de 8.79. Además los valores superiores a 0.5 encontrados (0.55 – 0.85) en el Contenido de Información Polimórfica (PIC) indican que los microsatélites evaluados son muy informativos detectando variaciones genéticas en el cultivo de papa (Tabla 1).
De las 79 variedades nominales analizadas, 16 no concordaron con el número de alelos esperados para un marcador según la especie y ploidia asignada, siendo el iniciador STM2013 el que presentó con mayor frecuencia este fenómeno(13variedades nominales). Además, se identificaron 5 variedades nominales (4 accesiones diploides S. stenotomum subsp stenotomum y 1 accesión triploide S. x chaucha) que no concordaron con la especie asignada, debido a que el número de alelos para la mayoría de iniciadores excedía a lo esperado para cada especie.
Análisis de agrupamiento.- Se obtuvo una similitud media de0.62yampliosrangosdeagrupamientoquevaríandesde0.41 a 0.98. El agrupamiento fue reforzado por medio del análisis de bootstrap para ver la consistencia de los grupos formados. Aunque se usaron 18 marcadores microsatélites repartidos por todo el genoma de la papa y se analizaron 19 locus muy polimórficos, las variedades nominales no lograron diferenciarse en grupos homogéneos. Sin embargo, en un rango de similitud de 0.51 a 0.72, se logró determinar 18 pequeños grupos en los cuales la mayoría guardan alguna relación de nombre, lugar y/o especie (Fig. 2).
De estos 18 grupos, 7 grupos (I, IV, VIII, IX, X, XIII y XVIII) presentaron una consistencia de media a alta (46.1 a 83.8%) según el análisis bootstrap. Los grupos restantes presentaron una consistencia media a baja (39.6 a 10.4%). Con respecto a la procedencia del material colectado y a su identidad taxonómica, se observaron 5 grupos formados con accesiones pertenecientes a la misma región política y con cierta relación de especie (VII, VIII, X, XII y XVII).
Se encontraron 4 variedades, posiblemente duplicadas: a) 2 accesiones de S. x curtilobum procedente de Cusco y b) 2 accesiones de S. x chaucha, una procedente de Ayacucho y la otra de Cusco.
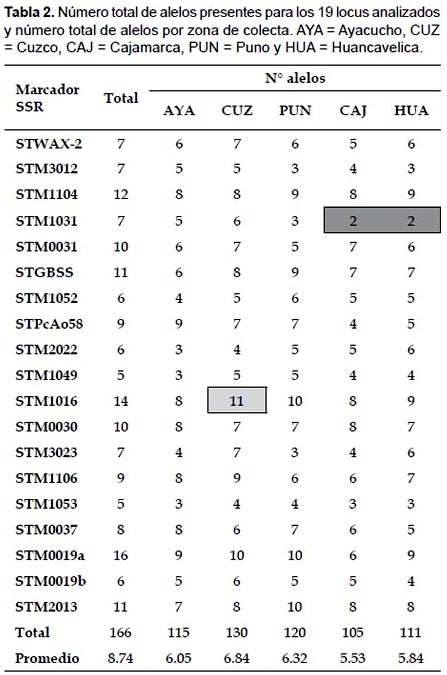
Riqueza alélica.- Para los 19 loci registrados se obtuvo un totalde166alelos.La región de Cusco presentó el mayor número de alelos (130 alelos), seguido de Puno con 120, Ayacucho con 115, Huancavelica con 111 y Cajamarca con 105 alelos. Cabe resaltar que se analizó un mayor número de muestras de Ayacucho, sin embargo no se encontró un mayor número de alelos en esa región; y más aún, solo 4 de los 19 locus analizados presentaron un mayor número de alelos en Ayacucho (Tabla 2)
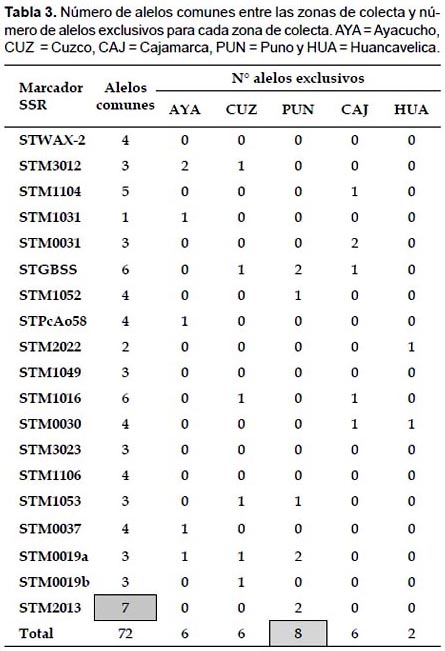
Alelos comunes y exclusivos.- De los 166 alelos caracterizados,72alelos(43.37%)fueroncomunesocompartidosconlas5 regiones de colecta. Se registraron alelos exclusivos (aquellos que solo se encontraron en una de las 5 regiones) en todas las zonas de colecta y para 15 de los 18 iniciadores, siendo los iniciadores STWAX-2, STM1049 y STM1106 los que no presentaron alelos exclusivos (Tabla 3). La región de Puno presento el mayor numero de alelos exclusivos (8 alelos).
Índice de Diversidad genética. - Para el caso de las 42 variedades nominales de S. tuberosum subsp. andigena, el valor promedio de diversidad fue de 0.74 y para las 18 variedades nominales de S. x chaucha, el valor promedio de diversidad fue de 0.70, siendo S. tuberosum subsp . andigena ligeramente más diversa que S. x chaucha.
Discusión
Iniciadores multilocus
En papas tetraploides, Milbourne el al. (1998) describieron la existencia de iniciadores que amplifican varios loci en simultaneo (multilocus); como es el caso del iniciador STM0019 que presentados regiones bien diferenciadas anteriormente reportado (Andrade2001,Ames2003,Ghislainetal.2001).Elincremento del número alelos esperados en el iniciador STM2013 para 13 accesiones, indicaría la posibilidad de ser un marcador multilocus, presentando un locus bien definido y otro con problemas de especificidad en la región amplificada, por lo que presentaría bandas sobrepuestas y una gran cantidad de alelos nulos, esto también fue sugerido por Ghislain (1999) y Andrade (2001).
Individuos no definidos
La probable explicación para que 5 variedades nominales no concuerden con el número de alelos esperado sería una mala clasificacióntaxonómica insitu yporlotantonoconcuerdanconla especie asignada. Aunque también deberíamos tomar en cuenta que los campesinos no siembran los cultivos de papa separándolos por especie y no tienen cuidado de la hibridación natural que puede sufrir este cultivo (Sevilla 2004, Brush et al. 1981, Jackson et al. 1978) lo que estaría ayudando a cometer errores involuntarios en la caracterización. Esta característica natural de la papa de formar híbridos espontáneos interespecíficos podría hacer que fenotípicamente un híbrido sea reconocido como alguna especie determinada debido a que expresa característica de uno de sus progenitores pero que genotípicamente comparte regiones genómicas de sus dos especies parentales.
Análisis de agrupamiento
La baja similitud media obtenida (0.62) y los amplios rangos de agrupamiento nos indican una alta variación genética entre las accesiones evaluadas. Si bien existen ciertas relaciones entre zonas de procedencia y/o especie, la consistencia de estos grupos no es fuerte, por lo que no se puede afirmar grandes relaciones genéticas entre las variedades nominales agrupadas.
Estos resultados sugieren que los 18 iniciadores microsatélites no fueron suficientes para lograr una completa diferenciación en grupos homogéneos por especies, zona de procedencia ni por ploidía. Este fenómeno ya fue observado por Raker y Spooner en el 2002, donde encontraron que muchas accesiones de S. tuberosum subsp. andigena (tetraploides) se mantenían agrupadas junto con sus ancestros diploides sin formar grupos definidos. Además debemos tener en cuenta el alto porcentaje de hibridación natural presente en las especies de papa nativa y que posiblemente los marcadores moleculares evaluados no se encuentren ligados a regiones genéticas que puedan diferenciar estas especies.
La presencia de variedad es duplicada sindicaría la existencia de un pequeño grado de sobreestimación de la diversidad genética, específicamente en el caso de las 2 variedades que provienen de la misma región. Por otro lado, las 2 variedades duplicadas que provienen de lugares diferentes podrían explicarse por los mecanismo de intercambio de semillas que se efectúan entre los agricultores (trueques,ferias regionales y nacionales) referenciado en los informes del Proyecto in situ (INIA 2005a-e).
Riqueza alélica
La respuesta más probable para que las cinco regiones presenten valores similares de polimorfismo, además de presentar cantidades similares de alelos totales, de alelos exclusivos y de no formar grupos homogéneos, probablemente se deba a la selección realizada por los agricultores (realizado desde los primeros pobladores andinos) que tienden a conservar diversos cultivares de papa; pero que entre todas ellas comparten las mismas características de utilidad comercial y de consumo (sabor, tamaño, productividad, etc.) que hace que prevalezca un pool de genes específico, además no todos los agricultores mantienen las mismas variedades, pero sí todas ellas se encuentran dentro de un solo pool de genes utilitarios.
En el caso de Ayacucho, el tener un mayor número de muestras no significó encontrar un mayor número de alelos. Por lo que se puede afirmar que conservar el mayor número de variedades en una región no necesariamente significará conservar la mayor cantidad de variación genética, ya que la diversidad genética dentro de una especie no se distribuye homogéneamente a través de los ambientes donde se desarrolla (Hodgkin 1995).
Por otro lado, casi la mitad de alelos caracterizados son comunes para las 5 regiones de colecta, lo que indicaría que estos alelos son fijos para las especies de papa nativa y se encuentran ampliamente distribuidas en la zona andina del Perú.
Diversidad genética de las especies evaluadas
Los valores de diversidad genética encontrados para las especies analizadas (S. tuberosum subsp. andigena y S. x chaucha) son altos y reafirman el alto grado de diversidad genética mantenido en la población muestreada. En promedio el valor de diversidad genética es más alto en S. tuberosum subsp. andigena (0.74)en comparación con el valor obtenido para S.xchaucha (0.70),probablemente esto se deba aun menor número de muestras analizadas por parte de la especie triploide, pero tampoco podemos descartar la naturaleza genética de la misma especie y sus diferencias de ploidía como responsables del grado de diversidad.
Los resultados nos indican que los microsatélites evaluados para papa nativa logran identificar altos niveles de diversidad genética dado por los valores de polimorfismo (PIC = 0.55 – 0.85) y los índices de diversidad genética obtenidos, pero a la vez no son suficientes para discriminar grupos diferenciados por procedencia y/o especies.
Asimismo, el análisis de agrupamiento, la riqueza alélica y los valores de diversidad genética obtenidos indican que existe un alto grado de diversidad genética y corrobora los resultados obtenidos de los inventarios y caracterizaciones morfológicas realizadas en las chacras de los agricultores; también podemos concluir que debido a los niveles similares de riqueza alélica, de alelos exclusivos y el gran porcentaje de alelos compartidos para las cinco regiones de colecta, existiría un pool de genes común y fijo para las especies de papa nativa y que se encontrarían ampliamente distribuidos entre las chacras de los campesinos.
Agradecimientos
El presente trabajo ha sido realizado en el Instituto Nacional de Investigación Agraria–INIA de la Dirección de Investigación Agraria – DIA en la Sub dirección de Recursos Genéticos y Biotecnología – SUDIRGEB, en el Centro Experimental La Molina - Lima, dentro del marco del Proyecto Conservación in situ de los cultivos nativos y sus parientes silvestres, con el apoyo financiero del Fondo Mundial para el Medio Ambiente – FMAM, la Cooperación Italiana y el Gobierno Peruano. Los autores agradecen la colaboración del Centro Internacional de la Papa por permitirnos usarlos programa sinformáticos necesarios para este trabajo.
Literatura citada
Ames M. 2003. Validación de la selección de la colección núcleo de Solanum tuberosum subsp. andigena mediante el uso de marcadores microsatélites. Tesis para optar el Titulo Profesional de Biólogo-Universidad Nacional Federico Villareal, Lima, Perú. pp. 48-63. [ Links ]
Andrade D. 2001. Selección de la colección núcleo para Solanum phureja mediante el uso de marcadores microsatélites. Tesis para optar el Titulo Profesional de Biólogo-Universidad Nacional Agraria La Molina, Lima, Perú. pp. 51-71 [ Links ]
Brush S. B., Carney, H. J. & Huamán, Z. 1981. Dynamics of Andean potato agriculture. Econ. Bot. 35:70-88. [ Links ] En: Estrada N. 2000. La biodiversidad en el mejoramiento genético de la papa. Pp:41-42
CIP (Centro Internacional de la Papa). 1997. Protocolos de laboratorio de biología molecular-Tipicación genética. En: Ghislain, M., Zhang, D. & Herrera, M. R (edit). Departamento de Recursos Genéticos. Manual de Capacitación CIP. Lima, Perú. pp. 30. [ Links ]
Dice L.R. 1945. Measures of the amount of ecologic association between species. Ecology, 26: 297-302. http://dx.doi.org/10.2307/1932409 [ Links ]
Doyle J. J. & J. L. Doyle. 1990. Isolation of DNA from small amounts of plant tissues. BRL Focus 12: 13-15, modified at the Forest Biotechnology Lab at NCSU.
Estrada R. 2000. La biodiversidad en el mejoramiento genético de la papa. PROINPA/CIA/CIP. Bolivia. pp. 21-88. [ Links ]
GhislainM., D. Zhang, D. Fajardo, Z. Huaman & R. Hijmans. 1999. Marker-assisted sampling of the cultivated Andean potato Solanum phureja collection using RAPD markers. Genetic Resources and Crop evolution, 46 (6): 547-555. DOI 10.1023/A:1008724007888
Ghislain M., D.M. Spooner, F. Rodríguez, F. Villamón, J. Nuñez, C. Vásquez, R. Waugh & M. Bonierbale. 2004. Selection of highly informative and user-friendly microsatellites (SSRs) for genotyping of cultivated potato. Theor Appl Genet (2004) 108:881–890. DOI 10.1007/s00122-003-1494-7
Ghislain M., F. Rodríguez, F. Villamón, J. Núñez J., R. Waugh & M. Bonierbale1. 2001. Establishment of microsatellite assays for potato genetic identification In: Scientist and farmer: partners in research for the 21th century. Technical Progress Report (1999-2000). International Potato Center, Lima, Perú. pp. 167-174.
Hawkes J.G. 1962. The origin f Solanum jusepczukii Buk and Solanum curtilobum Juz. et Buk. Z. Pflanzenzucht 47: 1-14. [ Links ]
Hawkes J.G. 1994. Origins of cultivated potatoes and species relationships. In: Potato Genetics. Centre for Agriculture and Biosciences-CAB Internacional University Press UK Cambridge. Pp. 3-42. [ Links ]
Hodgkin T. 1995. Some current issues in the conservation and use of plant Genetic Resources. In: Molecular Genetic techniques for plant genetic resources. Report of IPGRI workshop 9-11. Ayad W G, Hodgkin T, Jaradat A & Rao V R (Eds). October 1995. Rome, Italy. pp.3-22 [ Links ]
Instituto Nacional de Investigación Agraria-INIA. 2005a. Informe Final de cierre del Proyecto Conservación in situ de cultivos nativos y sus parientes silvestres 2005. SUDIRRGG-Estación Experimental Andenes. Cusco, Perú [ Links ]
Instituto Nacional de Investigación Agraria-INIA. 2005b. Informe Final de cierre del Proyecto Conservación in situ de cultivos nativos y sus parientes silvestres 2005. SUDIRRGG-Estación Experimental Canaan. Ayacucho, Perú [ Links ]
Instituto Nacional de Investigación Agraria-INIA. 2005c. Informe Final de cierre del Proyecto Conservación in situ de cultivos nativos y sus parientes silvestres 2005. SUDIRRGG-Estación Experimental Ilpa. Puno, Perú [ Links ]
Instituto Nacional de Investigación Agraria-INIA. 2005d. Informe Final de cierre del Proyecto Conservación in situ de cultivos nativos y sus parientes silvestres 2005. SUDIRRGG-Estación Experimental Baños del Inca. Cajamarca, Perú [ Links ]
Instituto Nacional de Investigación Agraria-INIA. 2005e. Informe Final de cierre del Proyecto Conservación in situ de cultivos nativos y sus parientes silvestres 2005. SUDIRRGG-Estación Experimental Santa Ana. Huancayo, Perú. [ Links ]
Jackson M. T., P. R. Rowe & J.G. Hawkes. 1978. Crossability relationships of Andean potato varieties of three ploidy levels. Euphytica 27:541-551. En: Estrada N. 2000. La biodiversidad en el mejoramiento genético de la papa. pp:41-42 Rev. peru. biol. 20(3): 215 -222 (March 2014) Soto et al.
Matsubayashi M. 1991. Phylogenetic relationships in the potato and its related species. In: Chromosome engineering in Plants: genetics, breeding andevolution.PartB:Developmentinplantgeneticsandbreeding. Edited by T. Tsuchiya and P. K. Gupta. Elsevier science Publisher B. V.; Amsterdan – Printed in the Netherlands. Pp. 93-118. [ Links ]
Milbourne D., R. Meyer, A.J. Cllins, L.D. Ramsay-Gebhardt & R. Waugh. 1998. Isolation, characterization and mapping of simple sequence repeat loci in potato. Mol. Gen. Genet (1998) 259: 233-245. DOI 10.1007/s004380050809
Milbourne D., R. Meyer, J.E. Bradshaw, E. Baird, N. Bonar, J. Provan, W. Powell & R. Waugh. 1997. Comparison of PCR-based marker system for the analysis of genetic relationships in cultivated potato. Molecular Breeding 3:127-136. DOI 10.1023/A:1009633005390
Nei M. & Lei W.H. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc. Natl. Acad. Sci. USA, 76: 5269-5273 [ Links ]
Nei M. 1973. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences of the USA: 3321-3323
Ochoa C M. 1999. Las Papas de Sudamérica: Perú. CIP. 1999. Kansas (USA). Allen Press. 1036 p. [ Links ]
Ochoa Carlos M. 1990. The potatoes of South America: Bolivia. Cambridge University Press.512 pp [ Links ]
Promega Corporation.1996.SilversequenceDNAsequencingsystem.Manual técnico, EEUU. pp. 19 [ Links ]
Provan J., A. Kumar, L. Shepherd, W. Powell & R. Waugh. 1996. Analysis of intra-specific somatic hybrids of potato (Solanum tuberosum) using simple sequence repeats. Plant Cell Reports 16:196-199. DOI 10.1007/BF01890866
Provan J., W. Powell & R. Waught. 1996. Microsatellite analysis of relationships within cultivated potato (Solanum tuberosum). Theorical and Applied Genetics. Springer-Verlag. 92:1078-1084. DOI10.1007/ BF00224052
Raker C M & Spooner D.2002.Chileantetraploidcultivatedpotato,Solanum tuberosum, is distinct from the Andean populations: Microsatellite data. Crop Sci. 42:1451-1458. doi:10.2135/cropsci2002.1451 [ Links ]
Rohlf F. 1993. NTSYS pc. Numerical taxonomy and multivariate analysis system. Exeter Softyware. New York. E.E.U.U. [ Links ]
Sevilla R & Holle M. 2004. Recursos Genéticos Vegetales. Ed. Torre Azul. Lima, Perú. [ Links ]
Yap I.V. 1992. Winboot UPGMA bootstrapping for binary data. PHYLIP Copyright 1986-1991 by the University of Washington and Joseph Felsentein. [ Links ]
Presentado: 23/07/2012
Aceptado: 14/11/2012
Publicado online: 14/03/2014