Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.21 no.1 Lima 2014
http://dx.doi.org/http://doi.org/10.15381/rpb.v21i1.8250
http://doi.org/10.15381/rpb.v21i1.8250
TRABAJOS ORIGINALES
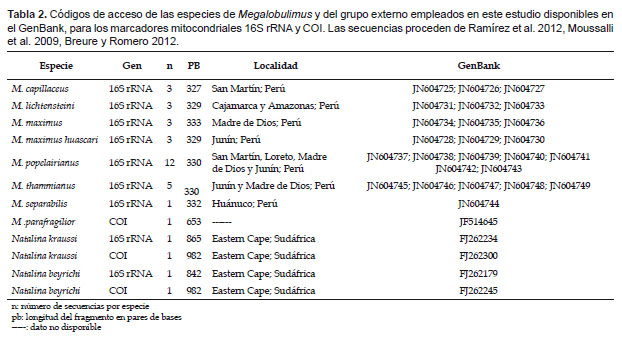
Análisis morfológico del sistema reproductor e identificación molecular a través de los marcadores mitocondriales COI y 16S rRNA de Megalobulimus oblongus (Mollusca, Strophocheilidae) de Colombia
Morphological analysis of the reproductive system and molecular identification by mitochondrial markers COI and 16S rRNA of Megalobulimus oblongus (Mollusca, Strophocheilidae) of Colombia
Erika Jaramillo Roldán1,3, Jessika López Martínez1,3, Rina Ramírez2, Luz Elena Velásquez Trujillo1,3
1 Grupo de Microbiología Ambiental, Escuela de Microbiología, Universidad de Antioquia, Medellín, Colombia.
2 Laboratorio de Sistemática Molecular y Filogeografía, Facultad de Ciencias Biológicas; y Museo de Historia Natural, Universidad Nacional Mayor de San Marcos, Av. Arenales 1256, Lima-11, Perú.
3 Programa de Estudio y Control de Enfermedades Tropicales _ PECET, Sede de Investigación Universitaria _ SIU, Medellín, Colombia.
Autora para correspondencia: Rina Ramírez, Email: rina_rm@yahoo.com
Email Jessika López: lopezmartinezjesica@gmail.com
Email Erika Jaramillo: tatajaramillo217@gmail.com
Email Luz Velásquez: luzelena333@yahoo.com
Resumen
En este trabajo se hizo el análisis morfológico y molecular a 28 caracoles terrestres de Megalobulimus oblongus, colectados en diferentes departamentos de Colombia, depositados en una colección de referencia. Para la caracterización morfológica, los animales se disecaron al estéreomicroscopio. Se describieron el sistema reproductor y la concha. Del sistema reproductor se tomaron medidas a las estructuras que lo componen. De la concha se describió la forma, el color, el número de espiras y la ornamentación e igualmente se tomaron medidas básicas usando un calibrador digital. Para el análisis molecular se usaron dos marcadores mitocondriales, el 16S rRNA y el gen Citocromo Oxidasa C subunidad I (COI). Ambos marcadores confirmaron la presencia de un único haplotipo en los ejemplares, aun para individuos situados en regiones biogeográficas diferentes y distantes. Este estudio sugiere que en Colombia M. oblongus se encuentra en peligro, por lo que urgen investigaciones sobre reproducción, genética poblacional y biogeografía que esclarezcan su situación en el país. Demuestra además que las colecciones de referencia y los bancos de tejidos son fuentes de información de gran valor, ya que permiten conocer aspectos relacionados con el riesgo en que se encuentran las especies y que sirven de insumo para el diseño de acciones de conservación.
Palabras claves: 16S rRNA; COI; caracterización morfológica; Megalobulimus oblongus; Colombia.
Abstract
In this work was done morphological and molecular analysis to 28 land snails of Megalobulimus oblongus, collected in different departments of Colombia, deposited in a reference collection. For morphological characterization, the animals were dissected in a stereomicroscope. The reproductive system and the shell were described. Measures were taken to structures of the reproductive system. Of the shell were described its shape, color, number of whorls and ornamentation and equally basic measures were taken using a digital caliper. For molecular analysis were used two mitochondrial markers, 16S rRNA and cytochrome C oxidase subunit I (COI). Only one haplotype was obtained for each marker, even for individuals of different and distant biogeographical regions. This study suggests that M. oblongus is in danger, therefore are urgent investigations about reproduction, population genetics and biogeography to clarify its situation in Colombia. It also demonstrates that the reference collections and tissue banks are sources of valuable information since they allow knowing aspects related with the species´ risk that serve as an input for the design of conservation actions.
Keywords: 16S rRNA; COI; morphology; Megalobulimus oblongus; Colombia.
Introducción
La familia Strophocheilidae (Hausdorf & Bouchet 2005) (Mollusca: Gastropoda: Stylommatophora) de origen gondwánico, incluye los moluscos terrestres del género Megalobulimus Miller 1878, endémicos de la región Andina y Amazónica Suramericana (Leme 1975), considerados los más grandes del continente.
Las clasificaciones taxonómicas de las especies de Megalobulimus, realizadas en los siglos XVIII y XIX, con base en los caracteres morfológicos de la concha, describieron 50 especies, siete de las cuales fueron registradas para Colombia: M. popelairianus (Nyst 1845), M. thammianus (Martens 1876), M. senezi (Jousseaume, 1887), M. oblongus (Müller 1774), M. auritus, M. valenciennesii (Pfeiffer 1842) y M. pudicus (Müller, 1774). La distribución geográfica en Colombia varía según la especie, por ejemplo M. thammianus se señala para la región andina, M. oblongus y M. popelairianus para las regiones Caribe, Andina, Amazonia y Orinoquia. En tanto que M. valenciennesii solo se reporta para la amazonia; se desconoce la procedencia de M. auritus, M. pudicus y M. senezi (Daniel 1942, Bequaert 1948, Vera 2008, Restrepo 2009, Linares & Vera 2012).
La validez de las determinaciones taxonómicas específicas de los Megalobulimus fue cuestionada por Bequaert (1948) quien consideró que la conquiliología era insuficiente para permitir este diagnóstico. Desde entonces se busca incluir las descripciones de la anatomía interna, con énfasis en el sistema reproductor (Leme 1973, 1989, 1993, Leme & Indrusiak 1990). Sin embargo, en Colombia no ha sido estudiada la morfología de las partes blandas de los Megalobulimus mencionados.
Si bien los caracteres morfológicos dan una buena resolución para la identificación taxonómica específica, se ha demostrado que algunos marcadores moleculares ofrecen de manera rápida y confiable la información necesaria para identificar las especies, por ello en la última década se ha propuesto un segmento del gen mitocondrial COI (Citocromo C Oxidasa Subunidad I) como marcador estándar global, ya que amplifica para una gran diversidad de especies, a través de un par de secuencias universales de iniciadores (Hebert et al. 2003).
No obstante, en algunos grupos de animales como los bivalvos, el marcador COI no amplifica, problema que se ha resuelto usando el gen mitocondrial 16S rRNA (Feng et al. 2011), el cual también provee suficiente variabilidad como para formar grupos monofiléticos con distancias intraespecíficas menores a las interespecíficas (Vences et al. 2005). En el caso particular de los Megalobulimus, se ha encontrado que este marcador amplifica mejor que el COI, con una eficacia del 100% (Congrains 2010, Ramírez et al. 2012).
Por lo tanto nuestra investigación se propuso establecer el estatus taxonómico de los caracoles terrestres, identificados a priori como Megalobulimus oblongus (Müller 1774) y depositados en la colección de Moluscos Vectores de la Universidad de Antioquia, utilizando la caracterización morfológica de las partes blandas del caracol, además de la identificación molecular con base en los marcadores mitocondriales 16S rRNA y COI.
Material y métodos
Material biológico.- Para esta investigación se analizaron 28 ejemplares de Megalobulimus oblongus adultos, ya que las conchas de todos tenían una altura total mayor de 90 mm y el labio externo doblado hacia afuera y rosado. Los especímenes forman parte de la colección de Moluscos Vectores de la Universidad de Antioquia VHET N°37 y provienen de seis localidades de Colombia (Tabla 1). Las partes blandas de los caracoles estaban conservadas en Railliet-Henry, las conchas almacenadas en seco y un trozo de tejido del pie conservado en alcohol al 96%.
Descripción morfológica del aparato reproductor y la concha de Megalobulimus.- Para la descripción del sistema reproductor se disectaron los 28 ejemplares de M. oblongus bajo estéreo microscopio (Nikon SMZ 745T); a cada una de las estructuras que lo componen se le midió el largo y el ancho, con la reglilla ocular. A las conchas se les midió el largo y ancho total y de la apertura, usando un calibrador digital (Uiustools® MT-0085cun, precisión de ±0.02 mm). Las dimensiones se dan en milímetros. Se realizaron registros fotográficos (Cámara Sony de 7.2 MP y ZOOM 2,8-5,2/ 6,3- 18,9 mm) y el aparato reproductor se ilustró en el software Corel Draw X3.
Extracción y amplificación de las secuencias
Extracción de ADN total.- El ADN se extrajo de un centímetro de tejido muscular del pie de cada uno de los 28 ejemplares de M. oblongus, usando el kit Qiagen DNeasy Blood & Tissue siguiendo las especificaciones del fabricante. La concentración de ADN extraído se comprobó por espectrofotometría en un nanodrop 1000 y se almacenó a -20 °C hasta la amplificación.
Amplificación de marcadores mitocondriales.- La amplificación se realizó por la técnica de reacción en cadena de la polimerasa (PCR) con los iniciadores del gen mitocondrial 16S rRNA: 16SFL104 (GACTGTGCTAAGGTAGCATAAT) y 16SRL472 (TCGTAGTCCAACATCGAGGTCA), diseñados especialmente para moluscos, que amplifican aproximadamente 330 pb (Ramírez 2004). Además, se usaron los iniciadores universales LCO 1490 (5'L GGTCAACAAATCATAAAGATATTGGL3') y HCO 2198 (5'L TAAACTTCAGGGTGACCAAAAAATCAL3') publicados por Folmer et al. (1994), que amplifican 700 pb, correspondientes a la Citocromo Oxidasa C subunidad I (COI).
La amplificación se llevó a cabo en un termociclador C1000 de Biorad. El volumen final fue de 30 µL, los cuales contenían: 3 µL de buffer de reacción de PCR a 10X, 2 µL de MgCl2 a 25 mM, 1 µL de cada dNTP a 5 mM, 1.8 µL de cada iniciador a 5 µM, 0.2 µL de Taq polimerasa a 0.5 unidades por reacción y 3 µL de ADN.
La PCR para el marcador 16S rRNA se llevó a cabo con una desnaturalización inicial de 96 ºC por 1', seguida de 35 ciclos de: desnaturalización a 94 ºC por 1', hibridación de los iniciadores a 45 ºC por 1' y extensión a 72 ºC durante 1', terminando con una extensión final a 72 ºC por 5'. Para el marcador COI, se realizó una desnaturalización inicial de 94 ºC por 4', seguida de 35 ciclos de: desnaturalización a 92 ºC por 2', hibridación de los iniciadores a 45 ºC por 1' y extensión a 72 ºC durante 1', terminando con una extensión final a 72 ºC por 10'.
Visualización del ADN por electroforesis.- Para comprobar la amplificación de los marcadores mitocondriales, se hizo una electroforesis en gel de agarosa al 1% con bromuro de etidio; se utilizó un marcador de tamaño molecular Hyperladder® de 100 a 1000pb. La electroforesis se corrió durante 45 minutos a 100 voltios. Las bandas se visualizaron y fotografiaron en un transiluminador.
Purificación y secuenciación del ADN.- Los productos de amplificación se purificaron usando el sistema Wizard® PCR Preps DNA Purification System Kit (Promega); 25 µL de los productos se enviaron a la compañía MACROGEN Inc. Korea (www.macrogen.com) para su secuenciación. Las secuencias están depositadas en el GenBank bajo los números de accesión KJ546457 (16S rRNA) y KJ546458 (COI).
Edición y análisis de las secuencias.- Los cromatogramas producto del secuenciamiento, se editaron manualmente usando el programa BioEdit v7.0.9 (Hall 1999), y se formó una secuencia consenso de cada individuo. Para garantizar que los resultados fueran verídicos y no producto de amplificación de contaminantes con ADN de otros organismos, se realizó una comparación de cada una de las secuencias contra la base de datos del GenBank (Benson et al. 2013) con la herramienta BLASTn. Para los alineamientos múltiples se incluyeron las secuencias registradas y disponibles en el GenBank (Tabla 2) y se utilizó el programa ClustalX 2.0 (Larkin et al. 2007). Como grupo externo se utilizaron las secuencias de Natalina kraussi (16S rRNA FJ262234, COI FJ262300) y N. beyrichi (16S rRNA FJ262179, COI FJ262245). Finalmente, con el programa MEGA v4.02 (Kumar et al. 2008) se construyeron árboles filogenéticos por el método de Neighbour-Joining (NJ) con corrección de distancias genéticas bajo el modelo de substitución nucleotídica Kimura 2-parámetros y un bootstrap de 1000 réplicas.
Resultados
Descripción de la concha de M. oblongus de Colombia
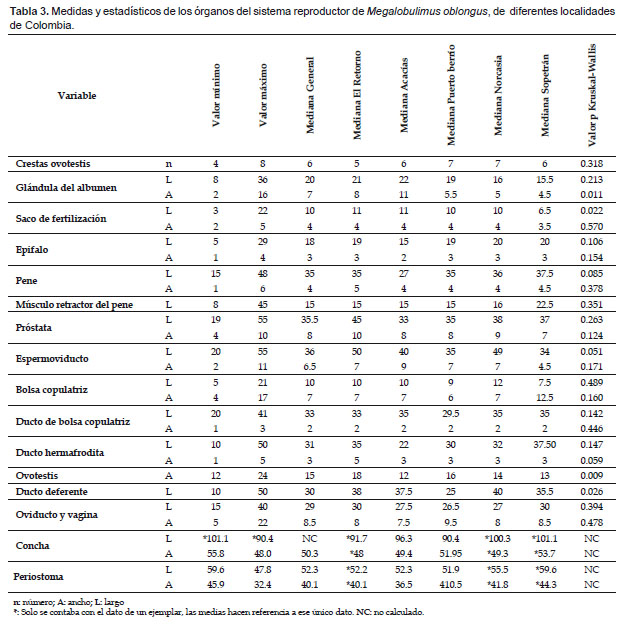
La concha de los 28 M. oblongus analizados es dextrógira, oblonga y más larga que ancha (Tabla 3). De color beige a marrón claro. Tiene de 5 a 6 vueltas que aumentan de tamaño del ápice a la apertura. Estos anfractos se encuentran delimitados por suturas bien definidas y forman ángulos de 145º a 180º. Entre sutura y sutura se observan líneas tenues de crecimiento de tono más claro que el resto de la concha. La apertura de la concha es oval-semilunar, con un labio externo, doblado hacia afuera, de color rosa brillante, muy grueso en el adulto. La pared parietal es rosa, presenta un ligero pliegue y se encuentra revestida por un callo transparente y brillante. El ombligo está cubierto casi en su totalidad por el borde inferior de la apertura (Fig. 1).
Descripción del sistema reproductor
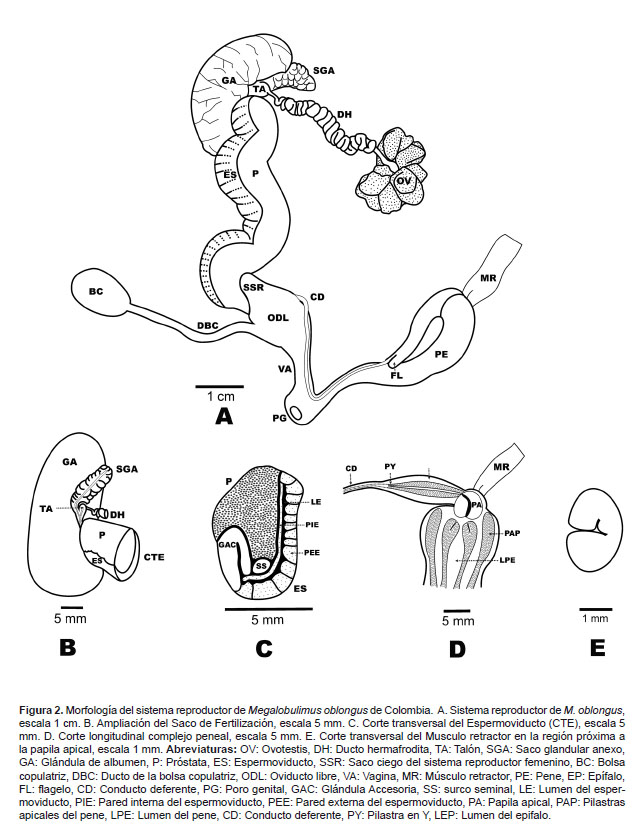
La ovotestis está localizada en la cara interna de la glándula digestiva entre el segundo y tercer anfracto, es relativamente pequeña y está conformada por 5 a 7 acinos de color marrón; de la parte inferior de la ovotestis sale el ducto hermafrodita, delgado, liso y de aspecto translúcido, en cuanto evoluciona a la región distal toma apariencia tortuosa de aspecto sinusoide. El complejo de fertilización está caracterizado por un pequeño talón adosado a la parte basal de un saco prominente, ovalado, de superficie granular y blancuzco, denominado "saco glandular anexo" (Baker 1926, Borda & Ramírez 2013), está embebido parcialmente en la glándula del albumen.
La glándula del albumen es una estructura verde oliva, de aspecto sólido y homogéneo, con surcos transversales en la superficie y un ducto interno muy delgado que la atraviesa. Adosada al costado se encuentra la región posterior de una gran estructura plegada en forma de "S", conformada por la próstata y el espermoviducto, que se extiende hasta el oviducto libre. En un corte transversal de esta estructura se evidencia: (1) El espermoviducto compuesto por dos paredes, la externa gruesa y rugosa, y la interna delgada y lisa, con un pequeño lumen entre ambas; además una glándula accesoria con forma de gancho. (2) La próstata como una masa sólida y lisa. (3) El surco seminal, entre el espermoviducto y la próstata, conformado por dos ductos estrechamente unidos. El surco seminal viaja longitudinalmente hasta el extremo posterior del oviducto libre, en donde se divide en un ducto delgado que se une a la pared de este y en un ducto grueso que da origen al conducto deferente.
El oviducto libre se inicia en la región anterior del espermoviducto, éste es un ducto robusto de paredes gruesas que internamente se encuentra totalmente plegado, en el lado derecho posee una dilatación ovoide llamada saco ciego. Al lado opuesto se desprende la porción más amplia del ducto de la bolsa copulatrix; ésta es una estructura periforme u oval. La vagina es una porción delgada contigua al oviducto libre que converge en el atrio genital, donde también desemboca el pene; el atrio culmina en el poro genital, ubicado en la región cefálica del caracol, detrás del tentáculo superior derecho.
La primera porción del conducto deferente se encuentra inmersa en la superficie del oviducto libre, y emerge al lado derecho de éste, para recorrer la superficie del oviducto libre, la vagina y el pene, hasta unirse al epifalo. Durante su recorrido disminuye su diámetro, siendo más delgado en el extremo que se une al epifalo.
El complejo peneal está compuesto por el pene y el epifalo plegado sobre el pene, con paredes sólidas de color crema; estos aumentan su diámetro en el extremo donde se encuentra la papila apical. Respecto a su tamaño, el pene es 1.94 veces más largo y 1.33 veces más ancho que el epifalo. En la región externa del epifalo cerca de donde se inserta el ducto deferente, se observa una pequeña extensión denominada flagelo. En la región interna se observa que el conducto deferente se fusiona con una pilastra que lo continúa hasta su extremo terminal para unirse a la papila apical del pene; dicha pilastra tiene forma de "Y" y pliegues sinuosos.
Al interior del pene se observan cuatro pilastras definidas, que disminuyen su diámetro en la región basal donde se conecta con el atrio genital; las pilastras tienen pliegues sinuosos y presentan un ensanchamiento en la región apical. La papila apical del pene es una estructura sólida de paredes lisas, localizada en la región proximal del pene, dicha estructura establece un puente entre el pene y el epifalo. El músculo retractor del pene viene del diafragma y se adhiere al pene con dos cortos haces, mide 15 mm de longitud (Fig. 2, Tabla 3).
Todos los especímenes estudiados correspondieron a Megalobulimus oblongus (Müller 1774), debido a que sus características morfológicas y conquiliológicas coincidieron con la descripción realizada para esta especie por Baker (1926).
En las conchas de los especímenes estudiados no se hallaron mayores diferencias. En cuanto a las estructuras que componen el sistema reproductor, las características típicas descritas por Baker (1926) se encontraron mejor expresadas en el ejemplar 366 proveniente de Puerto Berrío-Antioquia, a su vez el ejemplar más divergente fue el 114, proveniente de Sopetrán-Antioquia.
Análisis genético
En el 100% (28) de los caracoles estudiados, los iniciadores utilizados amplificaron un segmento de 328 pb del gen mitocondrial 16S rRNA. En tanto que solo en el 35% (10/28) de los mismos ejemplares los iniciadores "Folmer" amplificaron un fragmento de 672 pb, del gen mitocondrial Citocromo Oxidasa subunidad I (COI). De cada gen se obtuvo un único haplotipo.
En cuanto al contenido nucleotídico, las secuencias del marcador mitocondrial 16S rRNA presentan una relación de T+A de 66.4% y C+G de 33.5% y las del marcador COI una relación de T+A de 64.4% y C+G de 35.5%.
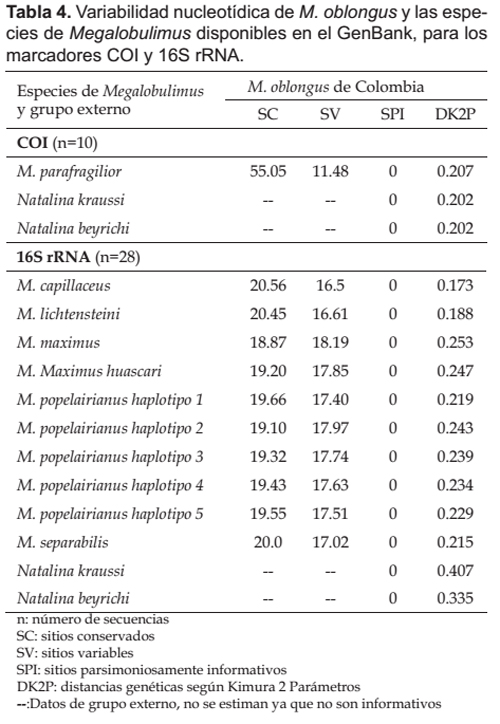
El alineamiento de las secuencias del marcador 16S rRNA, con las 30 secuencias de Megalobulimus del GenBank, mostró que existen 182 sitios conservados (20.56%) y 161 sitios variables (18.19%), de los cuales ninguno corresponde a sitios parsimoniosamente informativos. Para las secuencias obtenidas del COI también se realizó el alineamiento con la única secuencia de Megalobulimus encontrada en el GenBank mostrando que existen 556 sitios conservados (55.05%) y 116 sitios variables (11.48%) y tampoco presentó sitios parsimoniosamente informativos.
En relación a la distancia genética interespecífica mostrada por el marcador 16S rRNA, la menor divergencia se presentó con M. capillaceus (0.173), y la más alta divergencia se presentó con M. maximus (0.253). Con las demás especies los valores de Kimura 2 parámetros varían de 0.188 (M. lichtensteini) a 0.252 (M. huascari). Las secuencias del gen COI de M. oblongus y M. parafragilior presentan una distancia genética de 0.207 (Tabla 4).
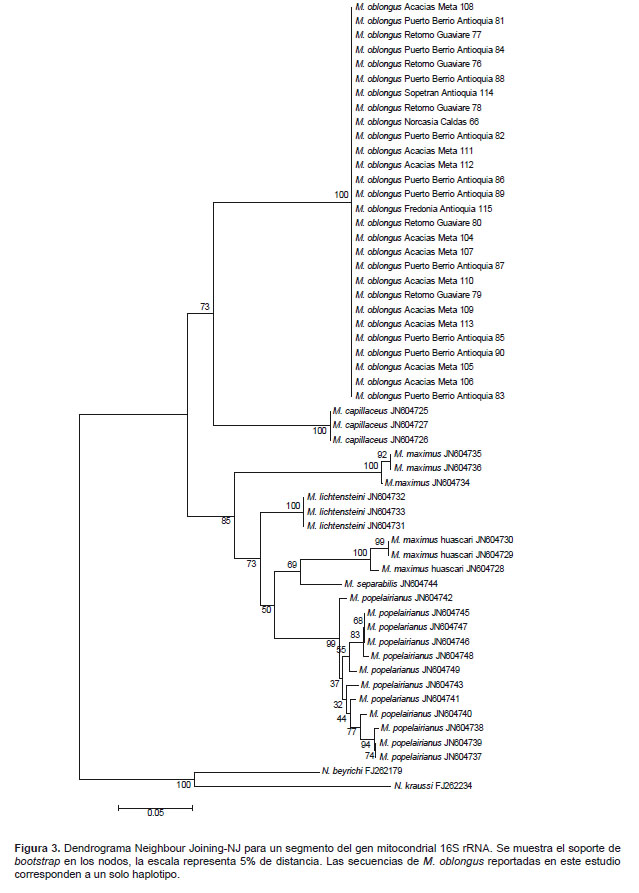
El dendrograma Neighbour Joining-NJ del marcador mitocondrial 16S rRNA, construido con las secuencias de M. oblongus de Colombia, las secuencias de seis especies de Megalobulimus disponible en el Genbank y las dos secuencias del grupo externo, se muestra en la Figura 3. Éste evidenció la formación de dos grupos monofiléticos; las secuencias del haplotipo de M. oblongus de Colombia presente en los 28 ejemplares estudiados, tienen una clara relación con M. capillaceus al compartir un clado con un valor de bootstrap de 73%. El otro clado, con un bootstrap del 85%, contiene las demás especies (M. popelairianus, M. huascari, M. lichtensteini, M. maximus y M. separabilis).
El dendrograma NJ del marcador mitocondrial COI construido con las secuencias de M. oblongus de Colombia, junto con la única secuencia de Megalobulimus disponible en el Genbank para este marcador y las dos secuencias del grupo externo, se muestra en la Figura 4. El haplotipo de M. oblongus de Colombia presente en los 10 individuos en los que se pudo amplificar el segmento "Folmer" del gen mitocondrial COI se agrupó con la única secuencia del género presente en el GenBank, correspondiente a la especie brasilera M. parafragilior.
Discusión
Esta es la primera publicación sobre una especie de Megalobulimus de Colombia, que establece el estatus taxonómico específico a partir de las características morfológicas del sistema reproductor de los especímenes objeto de esta investigación, los cuales fueron designados como Megalobulimus oblongus (Müller 1774), debido a que sus características morfológicas y conquiliológicas coinciden con las mencionadas para esta especie por Baker (1926), Bequaert (1948) y Leme (1973). También es la primera inclusión en el genbank de secuencias de M. oblongus de los marcadores mitocondriales 16S rRNA y COI, con los cuales se confirma que todos los especímenes de esta investigación corresponden a un mismo haplotipo.
De acuerdo con ambos marcadores, M. oblongus es más cercano a M. capillaceus que a otras especies con distribución en las vertientes orientales de los Andes. Este mismo resultado fue encontrado usando un marcador nuclear (Ramírez et al. 2012).
El único haplotipo encontrado en cada uno de los dos marcadores mitocondriales utilizados, contrasta con la variación estadísticamente significativa entre las dimensiones de la glándula del albumen, el saco glandular anexo del complejo de fertilización, la ovotestis y el ducto deferente, de M. oblongus procedentes de diferentes localidades. Ello puede estar relacionado con el estado reproductivo de los especímenes y la expresión de la variabilidad genética.
Por ello, cuando los caracteres morfológicos son insuficientes para clasificar individuos a nivel de especie, se recurre a la taxonomía molecular, siendo el COI el marcador estándar universal. Sin embargo, los resultados de esta investigación mostraron que el marcador mitocondrial 16S rRNA es más exitoso en la amplificación de secuencias de Megalobulimus que el COI, lo que concuerda con los hallazgos de Congrains (2010).
Al considerar los resultados de los análisis moleculares realizados a nuestros especímenes clasificados como M. oblongus, encontramos nula diversidad genética, evidenciada en la existencia de un solo haplotipo para ambos loci mitocondriales, aunque los animales hayan habitado lugares geográficamente distantes y en ecosistemas muy diferentes. Esto se debe quizás a la sinantropía que tiene esta especie y al beneficio económico que las personas obtienen de ella; como consecuencia especímenes son transportados para su comercio ilegal contribuyendo de esta manera a su dispersión por el territorio colombiano.
De otro lado, M. oblongus no es criado ni reproducido en cautiverio en Colombia, por lo que todos los ejemplares comercializados son extraídos de las poblaciones silvestres, hecho que merma las densidades naturales potenciando la endogamia, lo que reduce la variabilidad genética y conlleva a la disminución considerable de su capacidad de adaptación a los cambios del ambiente y conduce a un "cuello de botella" genético; como el que Bonnel y Selander (1974) observaron en una población del elefante marino Mirounga angustirostris, que mostraba baja diversidad genética debido a la caza desmedida (Hoelzel et al. 1993). Otro ejemplo de cuello de botella natural es el que sufrió el guepardo Acinonyx jubatus durante las glaciaciones del Pleistoceno; esta especie de guepardo se originó a partir de la especie Acinonyx pardinensis la cual fue sometida a condiciones medio ambientales complejas. La nueva especie resultante, Acinonyx jubatus, es un mamífero con muy baja diversidad génica, viéndose reflejado esto en la alta tasa de contagio de enfermedades, comparándolo con otros felinos (Ruiz et al. 2007).
Este estudio sugiere que M. oblongus se encuentra en peligro, por lo que urgen estudios sobre reproducción, genética poblacional y biogeografía, para impedir su extinción en Colombia. Demuestra además que las colecciones de referencia y los bancos de tejidos son fuentes de información de gran valor, ya que posibilitan la obtención de información relacionada con el riesgo en que se encuentran las especies y que sirven de insumo para el diseño de acciones de conservación.
Finalmente, disponer de información taxonómica es fundamental para la conservación biológica, ya que proporciona la base para el reconocimiento y la protección de las especies amenazadas. Da una percepción de la organización de los seres vivos (Avise 2004) y es la base para el desarrollo de programas de conservación y manejo de los recursos biológicos. Una clasificación errónea puede conducir a una gestión inadecuada y provocar la pérdida de especies (Avise 1989, May 1990, O´Brien & Mayr 1991).
Agradecimientos
Al CODI por la financiación (acta de Inicio IMB-046-2012). Al Dr. Iván Darío Vélez director del PECET-Programa de Estudio y Control de Enfermedades Tropicales, por su apoyo permanente a las investigaciones en malacología. A los integrantes de la unidad de Biología Molecular del PECET, por su colaboración en la estandarización de las PCR. A las personas que donaron los ejemplares de Megalobulimus a la colección de Moluscos Vectores de la Universidad de Antioquia VHET N°37.
Literatura citada Avise J.C. 1989. A role of molecular genetics in the recognition and conservation of endangered species. Trends in Ecology & Evolution 4: 279-281. doi: 10.1016/0169-5347(89)90203-6
Avise J.C. 2004. Molecular Markers, Natural History, and Evolution (Second Edition). Sinauer, Sunderland, MA. 684pp.
Baker H.B. 1926. The Mollusca collected by the University of Michigan _ Williamson Expedition in Venezuela. Part IV. Occasional Papers of the Museum of Zoology, University of Michigan 167: 1-49.
Benson D.A., I. Karsch-Mizrachi, K. Clark, et al. 2013. GenBank. Nucleic Acids Research 41(D1): D36-42. doi: 10.1093/ nar/gkq1079
Bequaert J. 1948. Monograph of the Strophocheilidae, a Neotropical family of terrestrial mollusks. Bulletin of the Museum of Comparative Zoology 1: 1 _ 210.
Bonnell M.L. & R.K. Selander. 1974. Elephant seals: genetic variation and near extinction. Science 184: 908-909. doi:10.1126/ science.184.4139.908
Borda V. & R. Ramírez. 2013. Re-characterization of the Red-lip Megalobulimus (Gastropoda: Strophocheilidae) from Peru with description of a new species. Zoologia 30(6): 675_691. http://dx.doi.org/10.1590/S1984-46702013005000008
Breure A.S.H. & P. Romero. 2012. Support and surprises: molecular phylogeny of the land snail superfamily Orthalicoidea using a three-locus gene analysis with a divergence time analysis and ancestral area reconstruction (Gastropoda: Stylom-matophora). Archiv für Molluskenkunde: International Journal of Malacology, 141(1): 1-20. doi: 10.1127/arch. moll/1869-0963/141/001-020
Congrains C. 2010. Ayudando a descifrar el enigma taxonómico, el código de barras de ADN de Megalobulimus spp. (Mollusca, Gastropoda) del Departamento de San Martín - Perú. Tesis, Título Profesional de Biólogo Genetista y Biotecnólogo. Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos.
Daniel H. 1942. Apuntes sobre algunos moluscos colombianos. Revista de la Academia Colombiana de Ciencias Exactas, Físicas y Naturales 4 (15-16): 372 _ 379.
Feng Y., Li Q., L. Kong & X. Zheng. 2011. DNA barcoding and phylogenetic analysis of Pectinidae (Mollusca: Bivalvia) based on mitochondrial COI and 16S rRNA genes. Molecular Biology Reports 38: 291 - 299. doi: 10.1007/s11033-010-0107-1
Folmer O., M. Black, W. Hoeh, et al. 1994. DNA primers for ampliûcation of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology 3 (5): 294-299.
Hall T. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series 41: 95-98.
Hausdorf B. & P. Bouchet. 2005. Working classiûcation of the Gastropoda. Pulmonata. In: P. Bouchet and J.P. Rocroi, eds. Classiûcation and nomenclator of gastropod families. Malacologia 47 (1/2): 263-283.
Hebert P., A. Cywinska, S. Ball & J. Waard. 2003. Biological identiûcations through DNA barcodes. Proceedings B of the Royal Society, London. 270: 313 - 321. doi: 10.1098/ rspb.2002.2218
Hoelzel A.R., J. Halley, S.J. O'Brien, et al. 1993. Elephant seal genetic variation and the use of simulation models to investigate historical population bottlenecks. Journal of Heredity 84: 443-449.
Jousseaume F. 1887. Mollusques nouveaux de La Republiquedel Equa-teur. Bulletin de la Société Zoologique de France 12:165-186
Kumar S., J. Dudley, M. Nei & K. Tamura 2008. MEGA: A biologist-centric software for evolutionary analysis of DNA and protein sequences. Brieûngs in Bioinformatics 9: 299-306. doi: 10.1093/bib/bbn017
Larkin M., G. Blackshields, N. Brown, et al. 2007. Clustal W and Clustal X version 2.0. Bioinformatics 23: 2947-2948. doi: 10.1093/bioinformatics/btm404
Leme J. 1973. Anatomy and systematics of the neotropical Strophocheiloidea (Gastropoda, Pulmonata) with the description of a new family. Arquivos de Zoologia 23(5): 295 _ 337.
Leme J. 1975. Ensaios ûlogenéticos em Pulmonata e sua importância na nova conceituação da Superfamília Strophocheiloidea (Gastropoda, Stylommatophpra). Archivos do Museu Nacional do Rio de Janeiro 55: 79 _ 84.
Leme J. 1989. Megalobulimus lopesi sp. n., uma nova espécie de Pulmonata terrestre da Mata Atlântica brasileira (Mollusca, Gastropoda, Megalobulimidae). Memórias do Instituto Oswaldo Cruz 84(4): 313 _ 318. http://dx.doi.org/10.1590/S0074-02761989000800055
Leme J. 1993. Estúdio anatômico sobre Megalobulimus auritus (Sowerby, 1838) (Gastropoda, Megalobulimidae). Papéis Avulsos de Zoologia 38(7): 95 _ 105.
Leme J. & L. Indrusiak 1990. Megalobulimus parafragilior sp.n., uma nova espécie de Pulmonata terrestre da Serra do Mar (Gastropoda, Megalobulimidae). Papéis Avulsos de Zoologia 37(5): 97 _ 105.
Linares E.L. & M. Vera. 2012. Catálogo de moluscos continentales de Colombia. Bogotá: Instituto de Ciencias Naturales, Facultad de Ciencias Universidad Nacional de Colombia. 360 pp.
Martens E. 1876. Vorläuûge Mittheilungen über die Molluskenfauna von Süd-Georgien. Sitzungs-Bericht der Gesellschaft Natur-forschender Freunde 3: 89 - 94.
May R.M. 1990. Taxonomy as destiny. Nature 347: 129-130. doi:10.1038/347129a0
Miller K. 1878. Die Binnenmollusquen von Ecuador. Malakozoologische Blätter 25: 153 _ 159.
Moussalli A., D.G. Herbert & D. Stuart-Fox. 2009. A phylogeny of the cannibal snails of southern Africa, genus Natalina sensu lato (Pulmonata: Rhytididae): assessing concordance between morphology and molecular data. Molecular Phylogenetics and Evolution 52 (1): 167-182. http://dx.doi.org/10.1016/j.ympev.2009.02.018
Müller O. 1774. Vermiunm terrestium et ûuviatilium, seu animalium infosurior Helmithicorum et Testaceorum, non marinorun succincta historia. Havniae et Lipsiae 14: 214.
Nyst H. 1845. Notice sur quelques Bulimus nouveaux ou peu connus. Bulletin de l'Academie Royale de Belgique 12: 146 _ 153.
O'Brien, S.J. & Mayr, E. 1991. Bureaucratic mischief: recognizing endangered species and subspecies. Science. 251: 1187-1188. DOI:10.1126/science.251.4998.1187
Pfeiûer L. 1842. Symbolae ad historiam heliceorum: Sectio Altera. Cassellis: Fischeri. 147pp.
Ramírez R. 2004. Sistemática e Filogeografía dos Moluscos do Ecos-sistema de "Lomas" do Deserto da Costa Central do Peru. Tese, Doutorado. Faculdade de Biociências, Pontiûcia Uni-versidade Católica do Rio Grande do Sul, Brasil.
Ramírez R., V. Borda, P. Romero, et al. 2012. Biodiversidad y ende-mismo de los caracoles terrestres Megalobulimus y Systrophia en la Amazonia occidental. Revista peruana de biología 19(1): 059 _ 074.
Restrepo M. 2009. Descripción morfológica de Megalobulimus oblongus (Müller, 1774) para Norcasia, Caldas. Tesis. Facultad de Ciencias Exactas y Naturales, Universidad de Antioquia, Colombia.
Ruiz García M., A. Murillo, C. Corrales, et al. 2007. Genética de poblaciones amazónicas: la historia evolutiva del jaguar, ocelote, delfín rosado, mono lanudo y piurí, reconstruida a partir de sus genes. Animal Biodiversity and Conservation 30.2: 115_130.
Vences M., M. Thomas, A. van der Meijden, et al. 2005. Comparative performance of the 16S rRNA in DNA barcoding of amphibians. Frontiers in Zoology 2: 5. doi:10.1186/1742-9994-2-5
Vera M. 2008. Lista de los géneros de moluscos terrestres de Colombia (Mollusca: Gastropoda: Prosobranchia: Mesogastropoda y Pulmonata: Stylommatophora). Biota Colombiana 9(1): 39 _ 62
Presentado: 13/12/2013
Aceptado: 16/03/2014
Publicado online: 26/05/2014
Journal home page: http://revistasinvestigacion.unmsm.edu.pe/index.php/rpb/index