Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.22 no.1 Lima 2015
TRABAJOS ORIGINALES
Resistencia a Phytophthora infestans linaje clonal EC-1 en Solanum tuberosum mediante la introducción del gen RB
Resistance to Phytophthora infestans EC-1 clonal lineage in Solanum tuberosum by introducing the RB gene
María Lupe Román1; Cristina Rivera1; Jeanette Orbegozo1; Fernando Serna1; Soledad Gamboa1; Willmer Perez1; Victor Suarez1; Greg Forbes1; Jan Kreuze1; Marc Ghislain1
1 Laboratorio de Biotecnología Aplicada, Centro Internacional de la Papa (CIP), Apartado 1558, Lima 12, Perú.
Resumen
Una de las opciones para el control de la enfermedad del tizón tardío de la papa es el desarrollo de variedades resistentes a Phytophthora infestans mediante la transferencia directa de genes de resistencia (R) por ingeniería genética. En el siguiente trabajo de investigación, se usó el gen RB de Solanum bulbocastanum, el cual otorga un amplio espectro de resistencia a razas de P. infestans. Para dicho fin, se transformó genéticamente vía Agrobacterium la variedad susceptible de papa Desiree (Solanum tuberosum) con el vector binario pCIP68 que contiene el gen RB. Como resultado, se obtuvieron 19 plantas transformadas con el gen RB, confirmadas por la prueba de resistencia a kanamicina y por la prueba de reacción en cadena de la polimerasa (PCR). Las 19 plantas transgénicas fueron sometidas a infección en invernadero bajo condiciones de bioseguridad con el aislamiento POX067 de P. infestans perteneciente al linaje clonal EC-1 que es dominante en el Perú. Tres de las 19 plantas ([RB]54, [RB]56 y [RB]70) presentaron un alto nivel de resistencia al aislamiento POX067 de P. infestans.
Palabras clave: Phytophthora infestans; gen RB; papa; transgénico; gen R.
Abstract
One of the most efficient options for the control of late blight disease in potato (Solanum tuberosum) is the development of resistant varieties to Phytophthora infestans mediated by the direct transfer of resistance (R) genes through genetic engineering. In the present work, we used Solanum bulbocastanum RB gene to confers broad spectrum resistance to P. infestans races. To that end, Agrobacterium - mediated genetic transformation was used to transform a susceptible potato variety, Desiree, with the binary vector pCIP68 harboring the RB gene. As a result, 19 transformed plants containing the RB gene were obtained. kanamycin resistance test and polymerase chain reaction (PCR) assays confirmed the integration of the T-DNA in the potato genome. The 19 transformed plants, also called transgenic events were subjected to infection under biosafety greenhouse conditions. Phytophthora infestans isolate POX067 of the EC-1 clonal lineage, commonly find in Peru, was used for the infection. Three of the 19 plants ([RB]54, [RB]56 and [RB]70) show high resistance levels to isolate POX067 of P. infestans.
Keywords: Phytophthora infestans; RB gene; potato; transgenic; R gene.
Introducción
La papa, Solanum tuberosum L., originaria de la zona sur del Perú (norte del Lago Titicaca), es cultivada en los Andes peruanos hace más de 7000 años (Spooner & Hetterscheid 2006). A nivel mundial este cultivo está considerado dentro de los cuatro alimentos básicos junto con el trigo, el maíz y el arroz. Esto se debe al importante valor nutritivo que posee: alto contenido de vitamina C, B1, B3, B6 y de aminoácidos adecuados para las necesidades humanas (FAO 2010, MINAGRI 2012). En Latinoamérica, Perú es uno de los principales productores de papa, con una producción promedio de 3.58 millones de toneladas y con un rendimiento promedio de 13.3 t/ha (MINAGRI 2012).
La principal amenaza biológica que ataca a este cultivo es el oomycete Phytophthora infestans (Mont.) de Bary causante de la enfermedad del tizón tardío de la papa, responsable de la pérdida del 10 - 15% de la producción global por año (Gebhardt & Valkonen, 2001). El valor económico de esta pérdida y el costo de protección del cultivo se estima en 6 mil millones de dólares anuales (Haas et al. 2009). La medida más usada para contrarrestar estas pérdidas es la aplicación de fungicidas. Sin embargo este método de control presenta un sinnúmero de desventajas como el difícil acceso a este producto, el costo elevado para los agricultores de escasos recursos económicos, los efectos negativos en la salud humana debido al uso inadecuado de estos químicos como es el caso del metalaxyl, y la aparición de nuevas razas resistentes a este fungicida (Fry 2008, Barona 2009). Este patógeno posee un alto potencial evolutivo debido a la plasticidad de su genoma (McDonald & Linde 2002) promoviendo la variación genética y aparición de poblaciones nuevas y agresivas (Raffaele et al. 2010). Otra fuente importante de variación en P. infestans es la reproducción sexual o recombinación entre dos tipos de apareamientos A1 y A2, complicando aún más el control del causante del tizón tardío.
Por otro lado, se ha reportado resistencia natural y duradera a P. infestans en la especie silvestre diploide Solamun bulbocastanum originaria de México. Esta resistencia está asociada con el gen Rpi- blb1 también denominado gen RB presente en el cromosoma VIII de S. bulbocastanum (Song et al. 2003, Colton et al. 2006). El gen RB codifica una proteína de 970 aminoácidos, perteneciente a la clase de proteínas R que exhiben dominios CC-NBS-LRR. Estos dominios están implicados en las funciones de interacción con los efectores del patógeno desencadenando la respuesta hipersensible (Vleeshouwers et al. 2008, Champouret et al. 2009). Debido a la incompatibilidad sexual entre S. bulbocastanum y S. tuberosum, se desarrollaron híbridos somáticos por fusión de protoplastos entre esta especie silvestre y variedades de papa comerciales logrando obtener resistencia similar al clon PT29 de S. bulbocastanum (Helgenson et al. 1998). Sin embargo, en ensayos de campo llevados a cabo en los Estados Unidos de América (USA) y México entre 1995 y 2002 se observó que P. infestans logró esporular en cultivares de papa derivadas del clon PT29. Esto significa que este fenotipo estaría reprimiendo pero no eliminaría los síntomas de la enfermedad, debido a esto la resistencia asociada a este material se denominó resistencia parcial, siendo efectiva para el uso en programas de mejoramiento para resistencia a este oomyceto (Dorrance et al. 2001).
Como alternativa al largo tiempo que se necesita para conferir resistencia al tizón tardío mediante los métodos convencionales del mejoramiento genético de papa, la efectividad y rapidez de la transferencia directa de genes mediante A. tumefaciens resultó una herramienta atractiva, y que conllevó a la obtención de plantas de papa transformadas con el gen RB a partir de las variedades Katahdin, Superior, Russet Burbank, entre otras. Estas plantas muestran altos niveles de resistencia a diversos linajes clonales de P. infestans: US-1 (Kuhl et al. 2007, Song et al. 2003), US-8 (Song et al. 2003, Kramer et al. 2009), US-10 (Halterman et al. 2008), US-14 (Bradeen et al. 2009), predominantes en USA, tanto en pruebas de invernadero como en campo.
En la actualidad, han aparecido nuevos linajes clonales en América del Norte como: US-22 (Kawchuk et al. 2011), US-23 (Hu et al. 2012) y US-24 (Kalischuk et al. 2012). El linaje clonal antiguo US-1 aun predomina en África subsahariana, así también como el nuevo linaje clonal KE-1 (Pule et al. 2013). En el Perú, el linaje clonal EC-1 demostró ser agresivo y resistente a metalaxil (Perez et al. 2009).
El presente trabajo tiene como objetivo obtener plantas transgénicas de papa de la variedad Desiree por la incorporación del gen RB vía A. tumefaciens resistentes al aislamiento POX067 de P. infestans pertenecientes al linaje clonal EC-1, predominante en Sud América (Ecuador, Perú).
Materiales y métodos
Material bacteriano.- Se utilizó Agrobacterium tumefaciens cepa EHA105 que contiene el vector binario pCIP68. El ADN de transferencia de este vector posee la secuencia codificante, promotora y terminadora del gen RB (accesión GenBank AY336128.1) y la secuencia codificante del gen neomicina fosfotransferasa II (nptII) como marcador de selección bajo la regulación de la secuencia promotora y de poli-adenilación del gen nopalina sintasa de A. tumefaciens (P Nos y pA Nos) (Fig. 1).
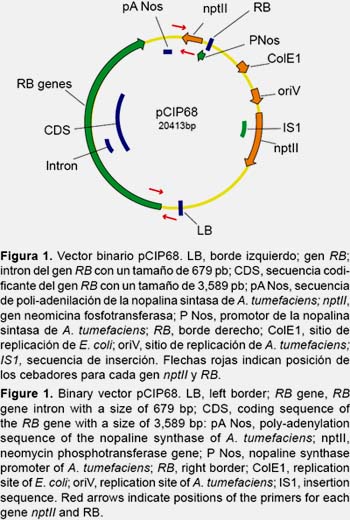
Material vegetal.- Plántulas in vitro de Solanum tuberosum var. Desiree (CIP800048) fueron obtenidas del Banco In Vitro del Centro Internacional de la Papa.
Aislamiento del patógeno.- El aislamiento POX067 de Phytophthora infestans, perteneciente al linaje clonal EC-1 con tipo de apareamiento A1 fue aislado de la variedad susceptible Amarilis en el área de Oxapampa, departamento de Pasco, Perú en el año 2000. (Villamón et al. 2005). La concentración utilizada en cada inoculación fue de 3000 esporangios/mL.
Propagación in vitro.- Se cortaron los segmentos vegetales que se encuentran entre los peciolos y tallos (nudos) del tercio superior de cada plántula. Estos fueron colocadas en magentas conteniendo 70 mL de medio semisólido de propagación (4.3 g/L de Murashige & Skoog, 25 g/L de sucrosa, 0.12 g/L de tiamina, 0.6 g/L de glicina, 0.15 g/L de ácido nicotínico, 0.15 g/L de piridoxina, 0.02 g/L de ácido giberélico, 2 g/L de gelrite y pH 5.6) y fueron incubadas por 3 semanas en cámaras de crecimiento a las siguientes condiciones ambientales: 20 – 22 °C, 16 horas de luz y 8 horas de oscuridad de fotoperiodo, 3000 lux de intensidad lumínica y a un rango de 60 - 70% de humedad relativa.
Transformación genética de papa.- La transformación fue vía organogénesis directa, siguiendo el protocolo de Medina-Bolívar y Cramer C. (2004) con modificaciones para Solanum tuberosum var. Desiree. Se tomaron los segmentos internodales de tallos de plantas de 4 semanas de edad propagadas in vitro en magentas, los cuales fueron cortados con una cuchilla estéril que fue puesta en contacto con A. tumefaciens (vector pCIP68). El crecimiento de A. tumefaciens fue a 28 °C por 48 horas en el medio semisólido Luria-Bertani (LB) suplementado con 100 mg/L de kanamicina. Los entrenudos infectados fueron colocados en el medio de co-cultivo semisólido (4.3 g/L de sales Murashige & Skoog, 25 g/L de sucrosa, 30 mg/L de acetosiringona, 2 g/L de gelrite, y pH 5.6) por 24 horas en oscuridad a 18 – 22 °C. Luego fueron transferidos al medio de regeneración (4.3 g/L de sales Murashige & Skoog, 20 g/L de sucrosa, 0.02 mg/L de ácido giberélico, 0.02 mg/L de ácido acético naftaleno, 2 mg/L de ribosido de zeatina, 100 mg/L de kanamicina, 250 mg/L de carbenicilina, 2 g/L de gelrite, y pH 5.6). Los explantes fueron cambiados a medio fresco cada 15 días y colocadas en las mismas condiciones de temperatura, humedad relativa y fotoperiodo que las usadas en la propagación in vitro. Después de 6 semanas, los regenerantes de 1 cm de altura fueron cortados y colocados en tubos conteniendo medio de propagación. Para evitar clones se cortó 1 regenerante por explante transformado.
Prueba de Resistencia a kanamicina.- Se cortaron segmentos de hojas de los posibles regenerantes transformados y fueron colocados en el medio de formación de callos (4.3 g/L de sales Murashige & Skoog, 20 g/L de sucrosa, 20 g/L de manitol, 0.5 g/L de MES, 0.5 g/L de PVP, 0.2 g/L de glutamina, 0.04 g/L de adenina-sulfato, 1 ml/L de vitaminas GAP, 1 mg/L de ácido acético naftaleno, 0.1 mg/L de 6-Bencyl-aminopurina, 200 mg/L de kanamicina, 2 g/L de gelrite, y pH 5.6). Se usaron segmentos de hojas de planta no transformada como control negativo y segmentos de hojas de planta transformada que contiene el gen neomicina fosfotransferasa (nptII) como control positivo (WIK). Este medio tiene la habilidad de inducir el desarrollo de callos en los explantes. La evaluación se realizó a las 4 semanas, durante este tiempo las placas que contienen el medio de formación de callos con las muestras de hojas fueron sometidas a las mismas condiciones ambientales de propagación in vitro.
Caracterización molecular por Reacción en cadena de la polimerasa (PCR).- Se extrajo ADN genómico a partir de 50 mg de hojas de plantas de papa resistentes a kanamicina y del control no transformado siguiendo el protocolo de Doyle y Doyle (1990). Para la prueba de PCR se usaron cebadores que amplifiquen a ambos genes: nptII y RB. Cebadores para el gen nptII: Km-1 S: 5’-ACG ATT CCG AAG CCC AAC C-3’ y Km-1 A: 5’-TCC TGT CAT CTC ACC TTG CTC C-3’, con un tamaño de amplicón de 597 pb y temperatura de hibridación de 55 °C. Cebadores que amplifiquen para el gen RB: RGA S: 5’-TCC TGT TTG ATG GTG GTT CCG A-3’y RGA A: 5’-CTT CCC TTC CTT TCT CGC CA-3, con un tamaño de amplicón de 299 pb y temperatura de hibridación de 54 °C. Los componentes por reacción del PCR se mezclaron en un volumen final de 15 µL donde se colocó 10.6 µL de agua libre de nucleasas, 1.5 µL de tampón de PCR 10X (New England Biolabs®), 0.3 µL dNTPs 10 mM (Promega®), 0.75 µL de cebadores sentido y antisentido (5 µM), 0.0375 µL de taq 5 U/ µL (New England Biolabs®) y 1 µL de ADN molde (100 ng). Las pruebas de PCR se llevaron a cabo en el termociclador de Applied Biosystem. El protocolo de PCR consistió de los siguientes 3 pasos: (1) denaturación inicial: 94 °C por 1 minuto, (2) denaturación: 94 °C por 1 minuto, hibridación (según el cebador) 54/55 °C por 1 minuto, extensión: 72 °C por 1 minuto por 35 ciclos y (3) extensión final: 72 °C por 10 minutos. Se corrieron los productos de PCR en un gel de agarosa al 1% teñido con bromuro de etidio y luego se tomaron imágenes de los geles haciendo uso del transiluminador de UV EpiChemi3 Darkroom. Para comparar el tamaño de los amplicones se usó como patrón de peso molecular al marcador Lambda digerido con PstI.
Las plantas transformadas confirmadas por las pruebas de resistencia a kanamicina y por la prueba de PCR se denominaron eventos transgénicos con su código correspondiente.
Trasplante del material bajo condiciones de invernadero.- Los eventos transgénicos y testigos o plantas controles fueron aclimatados a condiciones de invernadero en el Laboratorio de Bioseguridad del Centro Internacional de la Papa. Se trasplantaron 12 plántulas propagadas in vitro por evento y se colocaron en pequeños discos de sustrato conocido como Jiffy-7® (Jiffy Products International BV) por 15 días o hasta que enraizaron. Luego, los Jiffy-7® fueron trasplantados a macetas de plástico con el sustrato de cultivo PRO-MIX® (Premier Tech Horticulture), obteniendo 4 macetas por evento transgénico. El riego fue semanal directo al sustrato. Pasados 4 meses se cosecharon los tuberculillos de estas plantas pertenecientes a la primera generación vegetativa. Del mismo modo descrito anteriormente, se sembraron 12 tuberculillos uniformes repartidos en 4 macetas por cada evento, generando plantas de segunda generación vegetativa. Pasados los 45 días estas plantas fueron infectadas con el aislamiento POX067 de P. infestans como se detalla a continuación.
Ensayos de infección en planta completa.- Las pruebas de infección fueron realizadas bajo condiciones de invernadero (17 – 18 °C de temperatura y 80 – 100% de humedad relativa) óptimas para el desarrollo de la enfermedad. Se inoculó mediante aspersión, 3000 esporangios/mL del aislamiento seleccionado en el envés de los foliolos de cada planta (eventos transgénicos y testigos). Se utilizaron los siguientes testigos de Solanum tuberosum; como control positivo, el clon resistente a tizón tardío LBr40 (CIP387164.4); como controles negativos, la variedad de Desiree no transformada y la variedad Yungay (CIP002236). Cada planta fue cubierta con una bolsa plástica para crear un microclima extremo con mayor humedad. Las lecturas se realizaron al quinto día después de la infección mediante inspección visual, evaluando el porcentaje de severidad del área del tejido dañado o necrosado en la planta. Los valores del porcentaje de daño se encuentran en el rango de 0 – 100%.
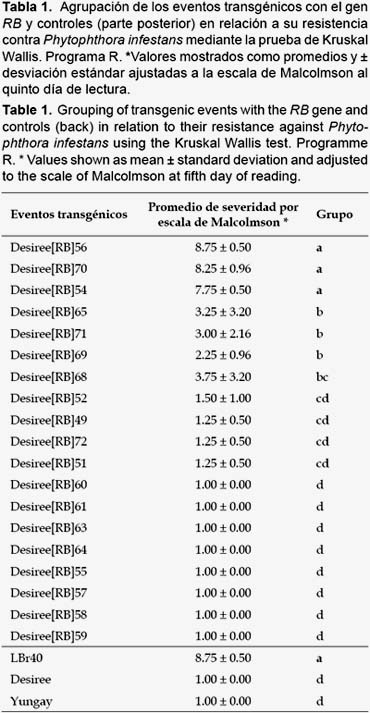
Análisis de datos.- Los datos de porcentaje de daño de los eventos fueron ajustados a la escala de Malcolmson (Cruickshank et al. 1982) donde el valor de 1 sería una planta altamente susceptible y el valor de 9 sería una planta altamente resistente. La descripción de la escala de Malcolmsom es la siguiente: 1= > 90%, 2= 81 – 90%, 3= 71 – 80%, 4= 61 – 70%, 5= 41 – 60%, 6= 26 – 40%, 7= 11 – 25%, 8= 1 – 10% y 9= 0%. Plantas que obtengan valores mayores a 7, son consideradas resistentes.
Análisis estadístico.- Para el análisis estadístico se usó la prueba no paramétrica de Kruskal Wallis (Corder et al. 2009) debido a que los datos no presentaron una distribución normal. Esta prueba es equivalente al ANOVA, se utiliza para comparar más de dos grupos de rangos y determinar que la diferencia no se deba al azar sino que la diferencia sea estadísticamente significativa. Los datos fueron procesados con el paquete Agricolae (De Mendiburu 2009) del Programa estadístico R (R Core Team, 2014).
Cuantificación del gen RB mediante qRT-PCR.- Se utilizó dos repeticiones biológicas por evento transgénico resistente (A y B). Se procedió a extraer el ARN de hojas frescas de cada repetición por evento, colectadas en tiempos diferentes: tiempo 0 o pre infección (prei), primer día pos infección (1 dpi), tercer día pos infección (3 dpi) y quinto día pos infección (5 dpi) usando TRIzol® (Invitrogen). Luego se procedió a digerir las muestras extraídas de ARN con ADNsa: RQ1 Rnase-free ADNse (Promega), para ambos pasos se siguieron los protocolos descritos por cada fabricante.
Cada muestra de ARN fue cuantificada mediante Nanodrop para sintetizar ADNc a partir de 2 µg utilizando el kit SuperscriptTM III First-Strand Synthesis System for RT-PCR (Invitrogen). Se usó hexámeros aleatorios siguiendo el protocolo del fabricante. Para las amplificaciones del ADNc molde, se usó el kit Power SYBRN Green Master Mix de Applied Biosystems y el termociclador Applied Biosystems StepOneTM. Para lo cual, se usó dos pares de cebadores, uno específico para el gen RB (Colton et al. 2006) y otro específico para el gen de referencia, factor de elongación ef1α, previamente identificado como el mejor gen de referencia usado en qRT-PCR para papa inoculada con P. infestans (Nicot et al. 2005, Saraiva et al. 2014). Para hallar la eficiencia de cada cebador se preparó una muestra representativa, es decir se mezcló 1 µL de cada muestra de ADNc y luego se procedió a hacer diluciones: 100 ng, 50 ng, 25 ng, 12.5 ng y 6.25 ng. Se corrieron 3 repeticiones técnicas por dilución. Para el cálculo de las eficiencias se siguió el modelo de regresión lineal a partir de la pendiente de la curva estándar la cual es determinada por el promedio de los valores Cq y los valores logarítmicos de cada concentración de ADNc. Se usó la siguiente fórmula para hallar la eficiencia de la PCR (Pfaffl et al. 2002): E=10[-1/pendiente] – 1.
Determinada las eficiencias de PCR se procedió a amplificar por triplicado cada muestra biológica por evento transgénico con ambos cebadores.
La reacción de qRT-PCR fue la siguiente: 3 µL de agua libre de nucleasas, 5 µL de SYBR Green, 0.5 µL de cebador sentido (5 µM), 0.5 µL de cebador antisentido (5 µM), 1 µL de ADNc molde (50 ng). El protocolo de qRT-PCR se basó en: activación de la enzima a 95ºC por 10 minutos. Denaturación: 94 °C por 10 segundos. 40 ciclos de hibridación: 55 °C por 30 segundos, extensión: 60 °C por 1 minuto y temperatura de fusión: 95 °C por 15 segundos, 60 °C por 1 minuto y 95 °C por 15 segundos. Los datos de Cq arrojados por el termociclador fueron procesados por el programa bioinformático: REST 2009 (Relative Expression Software Tool) de Qiagen. Este programa se basa en el método de cuantificación relativa de Pfaffl (Pfaffl et al. 2002), el cual normaliza los datos con el gen de referencia utilizando el valor de las eficiencias de cada gen.
Análisis estadísticos de los resultados de qRT-PCR.- Se utilizó la prueba de Dunnett para comparaciones múltiples como parte de la prueba de ANOVA de dos vías.
Para confirmar la existencia de diferencias significativas se compara las medias de todos los tratamientos contra un control, el cual a la vez, se considera como un tratamiento. El programa estadístico usado fue el GraphPad Prism 6 (GraphPad Software, Inc.).
Resultados
Prueba de resistencia a kanamicina.- Se obtuvieron 104 regenerantes putativamente transgénicos de 511 explantes transformados con una eficiencia de regeneración del 20.3%.
En la prueba de resistencia a kanamicina, de los 104 regenerantes solo 44 desarrollaron callos en presencia del antibiótico en altas concentraciones (200 mg/L). Los segmentos de hojas de los regenerantes susceptibles no formaron callos al igual que el control negativo (Fig. 2).

Análisis de los eventos transgénicos por PCR.- Los 44 regenerantes positivos para la prueba de resistencia a kanamicina fueron también positivos para la amplificación del gen nptII (Fig. 3). De estos 44 regenerantes, solo 19 eventos fueron positivos para los cebadores que amplifican parte del gen RB (Fig. 4).
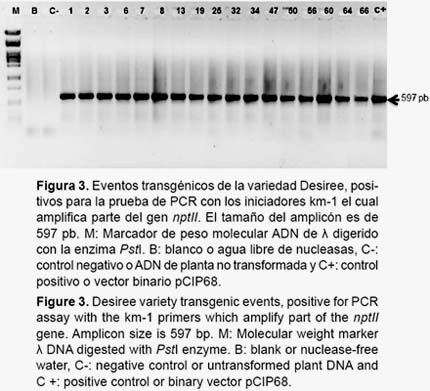
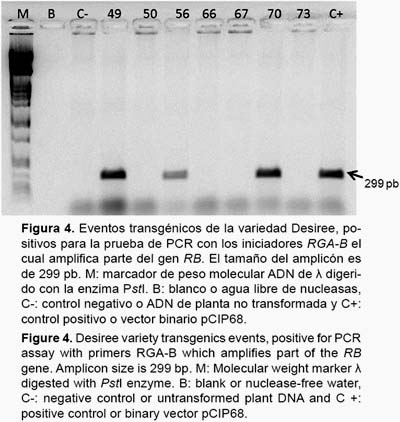
Por lo tanto mediante la caracterización molecular, se obtuvo 3.7% como eficiencia de transformación, identificándose 19 eventos positivos los cuales contenían el gen RB y el gen nptII, a partir de 511 explantes transformados.
Ensayos de infección con P. infestans en planta completa.- De los 19 eventos transgénicos con el gen RB, 3 mostraron alta resistencia a P. infestans (POX067), Desiree [RB]54, Desiree [RB]56, y Desiree [RB]70, al presentar mínimo daño en el tejido de la planta (Fig. 5).
La prueba de Kruskall Wallis agrupó los eventos transgénicos con el gen RB en 5 grupos, considerándose como eventos resistentes a Desiree [RB]56, Desiree [RB]70, y Desiree [RB]54 pertenecientes al grupo "a" (Tabla 1).
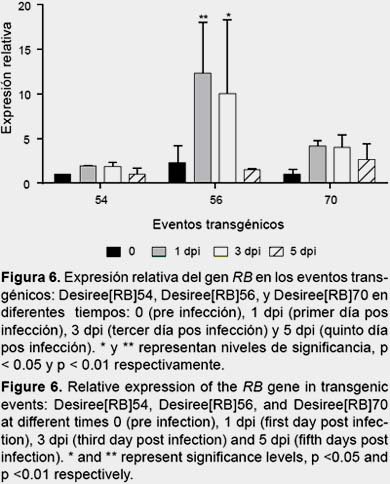
Cuantificación del gen RB mediante qRT-PCR.- El valor de las eficiencias para el gen RB y ef1α fueron de 99.0% y de 103.1%. La expresión relativa del gen RB en los eventos transgénicos fue normalizado con el gen ef1α y comparada con el valor de expresión basal o de pre infección (prei) del evento Desiree [RB]54 para todas las comparaciones por ser el menos resistente de los tres eventos seleccionados. El evento Desiree [RB]56 mostró tener mayor nivel de expresión o números de transcriptos del gen RB, seguida por los eventos Desiree [RB]70 y Desiree [RB]54. La expresión basal pre infección fue similar para los tres eventos. El evento [RB]56 mostró mayor nivel de expresión del gen RB con diferencias significativas al primer día pos infección (p < 0.01) y al tercer día pos infección (p < 0.05) con respecto al evento [RB]54. El evento [RB]70 no presentó diferencia significativa en el tiempo con respecto al evento [RB]54. Durante el quinto día pos infección, el nivel de número de transcriptos se reduce para todos los eventos, llegando a ser similar a nivel de expresión basal o del día pre infección (Fig. 6).
Discusión
La incorporación directa de genes de resistencia a P. infestans provenientes de especies silvestres de papa como Solanum bublbocastanum, es una vía muy prometedora para la obtención de variedades de papa resistentes al tizón tardío. Esta obtención de plantas de papa transgénicas es relativamente sencilla, rápida y su principal ventaja es que la nueva planta mantiene las características de la variedad original así como la adición de la resistencia a la enfermedad.
Se cosecharon 104 regenerantes siendo sometidos a la prueba de resistencia a kanamicina con la finalidad de verificar si todos fueron transformados. Esta prueba nos permitió evaluar de manera indirecta la expresión del gen nptII (Hellens et al. 2000). Sin embargo no se esperaba que más del 50% de regenerantes fueran escapes, es decir que no hayan sido transformados por A. tumefaciens o que el ADN-T se hubiese insertado en regiones de baja expresión. El alto número de escapes se puede deber a la gran capacidad de regeneración in vitro que caracteriza a la variedad Desiree (Cañedo & Cisneros 2004), permitiendo la proliferación y desarrollo de ciertos regenerantes no transformadas en presencia del agente selector. Como resultado, se obtiene una planta con resistencia o tolerancia natural. (López & Chaparro 2007). También es posible que varios de estos regenerantes no estuvieran en contacto permanente en el medio que contiene el agente selector, permitiendo su desarrollo (Monserrat et al. 2001). El agente selector utilizado fue el antibiótico kanamicina, el cual nos permitió seleccionar desde un inicio a los regenerantes que han incorporado adecuadamente el ADN-T (Estopá et al. 2001).
Los eventos que fueron positivos para la prueba de resistencia a kanamicina, también fueron positivos para prueba de PCR específico para el gen nptII como se esperaba. Sin embargo, para la misma prueba de PCR con los cebadores del gen RB, solo 19 eventos dieron positivos, correspondiendo a una eficiencia de transformación de 3.72%, la cual fue relativamente baja a pesar de que esta variedad tiene un alto índice de regeneración y responde bien a la transformación genética (Cañedo & Cisneros 2004, Beaujean et al. 1998). Un resultado similar donde se observó eventos transgénicos positivos para el gen nptII y negativos para el gen RB, ha sido reportado anteriormente (Kuhl et al. 2007). Esto se puede deber a que solo una porción del ADN-T se incorporó en el genoma de la planta debido a una deleción parcial o rearreglos del gen RB con uno o varios genes por su alta homología de secuencia mediante translocaciones e inversiones (Gheysen et al. 1990). El ADN-T ingresa por el borde derecho hasta el borde izquierdo. El vector pCIP68 proviene del vector pBIN19, el cual contiene el gen nptII cerca al borde derecho y el gen RB cerca al borde izquierdo. Esto significaría que primero ingresa el gen nptII y luego el gen de interés siendo esta una posible causa por la cual se hayan obtenido solo 19 eventos con el gen RB y nptII por PCR.
Para determinar la resistencia de los eventos transgénicos, estos se inocularon con P. infestans. En nuestro caso se realizó las pruebas de infección con el patógeno en planta completa en condiciones controladas anteriormente descritas (Dorrance & Inglis 1997, Pérez et al. 2009). Esta prueba tiene un valor cualitativo, basándose en la inspección visual al quinto día pos infección, del área afectada del total de la planta por el patógeno. Se obtuvieron con los porcentajes más bajo de daño a los eventos Desiree [RB]56, Desiree [RB]70 y [RB]54, y fueron agrupados según la prueba estadística de Kruskal Wallis en el grupo "a" como resistentes al aislamiento POX067, dando consistencia a los resultados arrojados por inspección visual. Ciertas diferencias en los porcentajes de daño entre repeticiones biológicas se podrían deber a que la aspersión del inóculo no fue homogénea entre los clones o a la diferencia morfológica de cada clon, lo cual influye en el desarrollo del patógeno. De otro lado, la prueba de infección en planta completa nos da una idea del comportamiento de la resistencia en campo o en el ambiente natural (Dorrance & Inglis 1997). La expresión del gen RB dirigida por su promotor endógeno, durante el tiempo previo a la infección con P. infestans es constitutivo en todos los eventos transgénicos. Sin embargo, se observa un aumento de la expresión del gen RB en presencia de P. infestans. Esta regulación positiva por el patógeno sobre los genes R ha sido observada en otros patosistemas (Gu et al. 2005, Halterman et al. 2003). El nivel de expresión del gen RB durante el tiempo, fue mayor para el evento [RB]56, seguido del evento [RB]70 y [RB]54 coincidiendo con los resultados anteriores. Estos resultados sugieren que el mayor nivel de expresión o mayor número de transcripto estaría relacionada a la resistencia mediada por este gen
Así mismo, el aumento de los transcriptos del gen RB durante las 24 horas pos infección jugaría un rol muy importante en los niveles de resistencia al retardar o evitar el desarrollo del patógeno en la planta. Esta característica se observa también en Solanum bulboscatanum cuando es infectada con P. infestans (Kramer et al 2009).
El aislamiento POX067 perteneciente al linaje clonal EC-1 de P. infestans que predomina en los Andes del Ecuador y Perú, es considerado uno de los más agresivos en nuestro país, sin embargo, tres eventos transgénicos mostraron ser resistentes. Esta observación nos permite proponer que los eventos transgénicos seleccionados podrían mostrar resistencia frente al linaje clonal US-1 prevalente en la mayoría de países de África subsahariana (Pule et al. 2013). Estudios previos, reportaron eventos transgénicos de papa con el gen RB resistentes a este linaje clonal(Kuhl et al. 2007, Song et al. 2003). De esta manera, este trabajo proveería de indicadores de resistencia a otro linaje clonal, reafirmando que el gen RB, puede generar altos niveles de resistencia a este patógeno y tener un amplio espectro de resistencia a diversos aislamientos provenientes de diferentes regiones del mundo. Esta adición de resistencia a la variedad Desiree podría permitir una significativa reducción en la cantidad de fungicidas utilizados en el control de la enfermedad, lo cual lograría incrementar la producción de papa y disminuir el daño causado por los aplicadores en el cultivo y el medio ambiente.
Agradecimientos
Se agradece al doctor Jiang de la universidad de Wisconsin (USA), por proveernos el fragmento de ADN que contiene la secuencia del gen RB y a los técnicos de investigación, Martin Ramos y Freddy Ventura.
Literatura citada
Barona P.D. 2009. Evaluación del impacto ambiental de tecnologías para producción de papa (Solanum tuberosum) con alternativas al uso de plaguicidas peligrosos. Tesis, Título en Ingeniaría Agrónoma. Facultad de Ciencias Agrícolas Universidad Central del Ecuador .http://cipotato.org/wp-content/uploads/Documentacion%20PDF/Tesis%20Dario%20Barona%20pdf.pdf
Beaujean A., R.S. Sangwan, A. Lecardonnel, et al. 1998. Agrobacterium-mediated transformation of three economically important potato cultivars using sliced internodal explants: an efficient protocol of transformation. Journal of Experimental Botany. 49(326):1589-1595. DOI: http://dx.doi.org/10.1093/jxb/49.326.1589.
Bradeen J., M. Iorizzo, D.S. Mollov, et al. 2009. Higher copy numbers of the potato RB transgene correspond to enhanced transcript and late blight resistance levels. Molecular Plant-Microbe Interactions 22(4):437-446. DOI: http://dx.doi.org/10.1094/MPMI-22-4-0437.
Cañedo V. & F. Cisneros. 2004. Clones de papa transformadas con la toxina de Bacillus thurigensis (Berliner) contra la polilla de la papa, Phthorimea operculella (Zeller). I. transformación de clones de papa y verificación de la presencia del gen cry1A (b). Revista Peruana de Entomología 44:89-93.
Champouret N., K. Bouwmeester, H. Rietman, et al. 2009. Phytophthora infestans isolates lacking class I ipiO variants are virulent on Rpi-blb1 potato. Molecular Plant-Microbe Interactions 22(12):1535-1545. DOI: http://dx.doi.org/10.1094/MPMI-22-12-1535.
Colton L.M., H.I. Groza, S.M. Wielgus, et al. 2006. Marker-assisted selection for the broad-spectrum potato late blight resistance conferred by gene RB derived from wild potato species. Crop Science 46(2):589-594. DOI: http://dx.doi.org/10.2135/cropsci2005.0112.
Corder G., W. Foreman & I. Dale. 2009. Nonparametric Statistics for Non-Statisticians. Hoboken: John Wiley & Sons. pp. 99-105.
Cruickshank G., H.E. Stewart & R.L. Wastie. 1982. An illustrated assessment key for foliage blight of potatoes. Potato Research 25(2):213-214. DOI: http://dx.doi.org/10.1007/BF02359807.
De Mendiburu F. 2009. Una herramienta de analisis estadistico para la investigacion agricola. Tesis. Universidad Nacional de Ingenieria (UNI-PERU).
Dorrance A.E. & D.A. Inglis. 1997. Assessment of greenhouse and laboratory screening methods for evaluating potato foliage for resistance to late blight. Plant Disease 81(10):1206-1213. DOI: http://dx.doi.org/10.1094/PDIS.1997.81.10.1206.
Dorrance A.E., D.A. Inglis, J.P. Helgeson, et al. 2001. Partial resistance to Phytophthora infestans in four Solanum crosses American Journal of Potato Research 78(1):9-17. DOI: http://dx.doi.org/10.1007/BF02874820.
Doyle J.J & J.L. Doyle. 1990. Isolation of plant DNA from fresh tissue. Focus 12:13-15. DOI: http://dx.doi.org/10.1313/1-92559-287-7:141.
Estopá M., V. Marfá, E. Melé, et al. 2001. Study of different antibiotic combinations for use in the elimination of Agrobacterium with kanamycin selection in carnation. Plant Cell, Tissue and Organ Culture. 65(3):211-220. DOI: http://dx.doi.org/10.1023/A:1010630726444.
FAO (Food and Agriculture organization of the United Nations). 2010. (en línea). FAOSTAT. Production quantity. <http://www.rlc.fao.or/es/agricultura/produ/papa.htm.>.Acceso 15/12/2013.
Fry, W. 2008. Phytophthora infestans: The plant (and R gene) destroyer. Molecular Plant Pathology 9(3):385-402. DOI: http://dx.doi.org/10.1111/j.1364-3703.2007.00465.x.
Gebhardt G. & J.P.T. Valkonen. 2001. Organization of genes controlling disease resistance in the potato genome. Annual Review of Phytopatology 39(1):79-102. DOI: http://dx.doi.org/10.1146/annurev.phyto.39.1.79
Gheysen G., R. Villarroel & M. Van Montagu. 1990. Illegitimate recombination in plants: a model for T-ADN integration. Genes & development 5(2):287-297. DOI: http://dx.doi.org/10.1101/gad.5.2.287.
Gu, K., B. Yang, D. Tian, et al. 2005. R gene expression induced by a type-III effector triggers disease resistance in rice. Nature 435:1122-1125. DOI: http://dx.doi.org/10.1038/nature03630.
Haas B.J., S. Kamoun, M.C. Zody, et al. 2009. Genome sequence and analysis of the Irish potato famine pathogen Phytophthora infestans. Nature. 461(7262):393-398. DOI: http://dx.doi.org/10.1038/nature08358.
Halterman, D., F. Wei & R. P Wise. 2003. Powdery mildew-induced Mla mRNAs are alternatively spliced and contain multiple upstream open reading frames. Plant Physiol. 131:558-567. DOI: http://dx.doi.org/10.1104/pp.014407.
Halterman D.A., L.C. Kramer, S.M. Wielgus, et al. 2008. Performance of transgenic potato containing the late blight resistance gene RB. Plant disease 92(3):339-343. DOI: http://dx.doi.org/10.1094/PDIS-92-3-0339.
Helgenson J.P., J.D. Pohlman, S. Austin, et al. 1998. Somatic hybrids between Solanum bulbocastanum and potato: a new source of resistance to late blight. Theoretical and Applied Genetics 96(6-7):738-742. DOI: http://dx.doi.org/10.1007/s001220050796.
Hellens R.P., E.A. Edwards, N.R. Leyland, et al. 2000. pGreen: a versatile and flexible binary Ti vector for Agrobacterium-mediated plant transformation. Plant Molecular Biology 42(6):819-832. DOI: http://dx.doi.org/10.1023/A:1006496308160.
Hu C.H., F.G Perez, R. Donahoo, et al. 2012. Recent genotypes of Phytophthora infestans in the Eastern United States reveal clonalpPopulations and reappearance of mefenoxam sensitivity. Plant Disease 96(9):1323-1330. DOI: http://dx.doi.org/10.1094/PDIS-03-11-0156-RE.
Kalischuk M.L., K.I. Al-Mughrabi, R.D. Peters, et al. 2012. Genetic composition of Phytophthora infestans in Canada reveals migration and increased diversity. Plant Disease 96(12):1729-1735. DOI: http://dx.doi.org/10.1094/PDIS-10-11-0859-RE.
Kawchuk L.M., R.J. Howard, R.D. Peters, et al. 2011. First report of Phytophthora infestans genotype US23 causing late blight in Canada. Plant Disease 95(7):873. DOI: http://dx.doi.org/10.1094/PDIS-01-11-0054.
Kramer L.C., M.J. Choudoir, S.M. Wielgus, et al. 2009. Correlation between transcript abundance of the RB gene and the level of the RB- mediated late blight resistance in potato. Molecular Plant-Microbe Interactions 22(4):447-455. DOI: http://dx.doi.org/10.1094/MPMI-22-4-0447.
Kuhl J.C., K. Zarka, J. Coombs, et al. 2007. Late Blight resistance of RB transgenic potato lines. Journal of the American Society for Horticultural Science 132(6):783-789.
López A. & A. Chaparro. 2007. A system for transformation potato plants (Solanum tuberosum sp. andigena var. Pastusa suprema) mediated through Agrobacterium tumefaciens. Agronomía Colombiana 25(1):16-25. ISSN 0120-9965.
Monserrat E., V. Marfa, E. Melé & J. Messenguer. 2001. Study of different antibiotic combinations for use in the elimination of Agrobacterium with kanamycin selection in cRNAation. Plant Cell Tissue and Organ Culture 65: 211-220. DOI: http://dx.doi.org/10.1023/A:1010630726444.
McDonald B.A. & C. Linde. 2002. Pathogen population genetics, evolutionary potential and durable resistance. Annual Review of Phytopathology 40(1):349-79. DOI: http://dx.doi.org/10.1146/annurev.phyto.40.120501.101443.
Medina-Bolívar F. & C. Cramer. 2004. Production of recombinant proteins by hairy roots cultured in plastic sleeve bioreactors. Methods in Molecular Biology. Vol. 267: Recombinant Gene Expression: Reviews and Protocols, Second Edition.
MINAGRI (Ministerio de Agricultura del Perú). 2012. (en línea). La Papa. <http://www.minag.gob.pe/portal/sector-agrario/agricola/cultivos-de-importancia-nacional/papa>. Acceso 16/03/2014.
Nicot N., J.F. Hausman, L. Hoffman, et al. 2005. Housekeeping gene selection for real-time RT-PCR normalization in potato during biotic and abiotic stress. Journal of Experimental Botany 56(421):2907-2914. DOI: http://dx.doi.org/10.1093/jxb/eri285
R Core Team 2014. R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL http://www.R-project.org/.
Pérez W., J. Lara & G.A Forbes, et al. 2009. Resistance to metalaxyl-M and cymoxanil in a dominant clonal lineage of Phytophthora infestans in Huánuco, Peru, an area of continuous potato production. European Journal of Plant Pathology 125:87-95. DOI: http://dx.doi.org/10.1007/s10658-009-9461-z
Pfaffl M., G.W. Horgan & L. Dempfle. 2002. Relative expression software tool (REST©) for group-wise comparison and statistical analysis of relative expression results in real-time PCR. Nucleic Acids Research 30(9):e36. DOI: http://dx.doi.org/10.1093/nar/30.9.e36.
Pule B.B., J.C. Meitz, A.H. Thompson, et al. 2013. Phytophthora infestans populations in central, eastern and southern African countries consist of two major clonal lineages. Plant Pathology 62(1):154-165. DOI: http://dx.doi.org/10.1111/j.1365-3059.2012.02608.x.
Raffaele S., R.A. Farrer, L.M. Cano, et al. 2010. Genome evolution following host jumps in the Irish potato famine pathogen lineage. Science 330(6010):1540-43. DOI: http://dx.doi.org/10.1126/science.1193070.
Saraiva K., D. Fernandes de Melo, V.D. Morais, et al. 2014. Selection of suitable soybean EF1α genes as internal controls for real-time PCR analyses of tissues during plant development and under stress conditions. Plant Cell Reports 33(9):1453-1465. DOI: http://dx.doi.org/10.1007/s00299-014-1628-1.
Song J., J.M. Bradeen, S.K. Naess, et al. 2003. Gene RB cloned from Solanum bulbocastanum confers broad spectrum resistance to potato late blight. Proceedings of the National of Science of the United States of America 100(16):9128-9133. DOI: http://dx.doi.org/10.1073/pnas.1533501100.
Spooner D. & W.L.A. Hetterscheid. 2006. Origins, evolution, and group classification of cultivated potatoes. In T. J. Motley, N. Zerega, and H. Cross [eds.], Darwin’s harvest: New approaches to the origins, evolution, and conservation of crops, 285-307. Columbia University Press, New York, New York, USA.
Villamón F.G., D.M. Spooner, M. Orrillo, et al. 2005. Late blight resistance linkages in a novel cross of the wild potato species Solanum paucissectum (series Piurana). Theoretical Applied Genetics 111(6):1201-1214. DOI: http://dx.doi.org/10.1007/s00122-005-0053-9.
Vleeshouwers V.G., H. Rietman, P. Krenek, et al. 2008. Effector genomics accelerates discovery and functional profiling of potato disease resistance and Phytophthora infestans avirulence genes. PlosOne 3(8):e2875. DOI: http://dx.doi.org/10.1371/journal.pone.0002875.
Fuentes de financiamiento:
El presente trabajo se realizó gracias al financiamiento de la Agencia de los Estados Unidos para el Desarrollo Internacional (USAID) Nº MTO 069018.
Información sobre los autores:
MG, JK, CR, MLR: realizaron el diseño experimental; MLR, JO, FS, CR, SG, WP: realizaron los experimentos; VS, MLR: analizaron los datos; MLR: redactó el manuscrito; MG, JK, WP, GF, CR, JO, FS, SG: revisaron y aprobaron el manuscrito.
Los autores no incurren en conflictos de intereses.
Autor para correspondencia
E-mail María Lupe Román: m.roman@cgiar.org
E-mail Cristina Rivera: c.rivera@cgiar.org
E-mail Jeanette Orbegozo: jorbegozoramirez@gmail.com
E-mail Fernando Serna: fserna@inia.gob.pe
E-mail Soledad Gamboa: s.gamboa@cgiar.org
E-mail Willmer Pérez: w.perez@cgiar.org
E-mail Victor Suarez: v.suarez@cgiar.org
E-mail Greg Forbes: g.forbes@cgiar.org
E-mail Jan Kreuze: j.kreuze@cgiar.org
E-mail Marc Ghislain: m.ghislain@cgiar.org
Presentado: 16/01/2015
Aceptado: 14/03/2015
Publicado online: 24/04/2015














