Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.23 no.3 Lima set./dic. 2016
http://dx.doi.org/10.15381/rpb.v23i3.12864
10.15381/rpb.v23i3.12864
TRABAJOS ORIGINALES
Expresión de efectores RXLR en el linaje clonal EC-1 de Phytophthora infestans en Perú
Expression of RXLR effectors in the EC-1 clonal lineage of Phytophthora infestans in Peru
Myriam Izarra y Hannele Lindqvist-Kreuze*
International Potato Center, P.O. Box 1558, Lima 12, Perú
*Autor para correspondencia Email Myriam Izarra: m.izarra@cgiar.org
ORCID Myriam Izarra: http://orcid.org/0000-0001-6272-6366
Email Hannele Lindqvist-Kreuze: h.lindqvist-kreuze@cgiar.org
Resumen
La papa (Solanum tuberosum L.) es en nuestros días uno de los cultivos alimenticios más importantes. Esta es propensa a la enfermedad de Tizón Tardío, causada por el oomiceto patógeno Phytophthora infestans, el cual secreta cientos de efectores que actúan como factores de virulencia. Poco se conoce sobre la diversidad de genes de virulencia de las cepas pertenecientes al linaje de reproducción clonal EC-1. En el presente estudio, mediante el secuenciamiento del transcriptoma de la interacción de la papa y P. infestans durante los primeros días después de la infección en las hojas de papa, se identificó la expresión diferencial de genes efectores tipo RXLR en dos cepas de P. infestans EC-1, siendo confirmados por qRT-PCR. Estas cepas, aisladas de papas cultivadas en el centro de los Andes peruanos, tienen diferentes patrones de virulencia. Los genes efectores, fueron silenciados en una cepa, para Avr-vnt1 en POX109 y para el homólogo Avh9.1 en POX067, pero expresados en la otra. Los resultados de transcriptoma fueron comparados con tres cepas adicionales del linaje EC-1. La información del repertorio de los efectores del patógeno y su expresión podrían ser informativos para el mejoramiento genético de la resistencia. El descubrimiento de efectores silenciados en las poblaciones del patógeno pueden guiar al uso de genes R específicos en los programas de mejoramiento genético. Por ejemplo, en el contexto de los Andes, donde el linaje clonal EC-1 predomina, el gen Rpi-vnt1 podría no ser recomendado.
Palabras claves: Phytophthora infestans; secuenciamiento del transcriptoma; efectores; resistencia al tizón tardío; virulencia.
Abstract
Potato (Solanum tuberosum L.) is nowadays one of the most important food crops. It is prone to the disease known Late blight caused by the oomycete pathogen Phytophthora infestans, which secrete a hundreds of effectors that act as virulence factors. Little is known of the diversity of virulence genes of the strains that belong to a clonally reproducing lineage EC-1. In this paper, through the transcriptome sequencing of potato and P. infestans interaction, we identified differentially expressed RXLR type effector genes in two P. infestans isolates EC-1, being confirmed by qRT-PCR. The isolates, originate from cultivated potato in the central Peruvian Andes, have different virulence patterns. Effector genes, were silenced in one isolate, such as Avr-vnt1 in POX 109 and for Avh9.1 homolog in POX 067, but expressed in the other. The transcriptomics results were compared with three additional isolates from the EC-1 lineage. The information on the pathogen effector repertoire and its expression should be informative for resistance breeding. Discovery of silenced effectors in the pathogen populations can guide the use of specific R genes in the breeding programs. For example in the Andean setting where EC-1 lineage dominates the Rpi-vnt1 would not be recommended.
Keywords: Phytophthora infestans; transcriptome sequencing; effectors; late blight resistance; virulence.
Introducción
Phytophthora infestans es un oomiceto patógeno de plantas. Los oomicetos son clasificados como Stramenopiles junto con las diatomeas y algas marrones, son formadores de hifas parecidos superficialmente a los hongos. El origen de P. infestans según recientes estudios sugiere que fue en México, y subsecuentemente migró a los Andes (Goss et al. 2014), teniendo en este centro de origen altos niveles de diversidad genética y fenotípica (Yoshida et al. 2013). Así mismo, la diversidad de P. infestans en el mundo es largamente conformada por migraciones (Fry et al. 2009). El estilo de vida hemibiotrópico de P. infestans comprende un ciclo de reproducción sexual y asexual produciendo diferentes formas de esporas: Oosporas y zoosporas respectivamente. La reproducción sexual ocurre entre tipos de apareamiento A1 y A2 (Fry et al. 1992). Phytophthora infestans es un patógeno extraordinariamente virulento y adaptable, causante de la enfermedad del Tizón tardío (Yoshida et al. 2013); este secreta varias proteínas efectoras que modulan la inmunidad innata del hospedero (Vleeshouwers et al. 2008), que resulta del reconocimiento de patrones moleculares asociados a patógenos (PAMPs) (Hogenhout et al. 2009).
La mayoría de sus hospederos pertenecen a la familia de Solanácea. En la interacción P. infestans / S. tuberosum, el tipo de resistencia más comúnmente estudiada, es la resistencia raza específica, la cual está gobernada por un sólo gen de resistencia-dominante (gen R), y por lo tanto, efectiva contra una determinada raza del patógeno (Estrada 2000). La resistencia a P. infestans es manifestada en una respuesta de hipersensibilidad, que es caracterizada por una rápida necrosis localizada en el sitio de la infección (Cornelissen & Melchers 1993). Todos los genes de avirulencia conocidos (Avr1, Avr2, Avr3a, Avr4, Avr-blb1, Avr-blb2 y Avr-vnt1) han sido identificados como pertenecientes a las clases de efector RXLR, estos genes codifican proteínas secretoras con el motivo RXLR para la translocación en las células (Vleeshouwers et al. 2011).
Phytophthora infestans tiene múltiples mecanismos para evadir el reconocimiento del gen R, así por ejemplo presenta cambios en el locus de avirulencia como un resultado de segregación somática por un mecanismo de mutación, la inserción de un transposón, la no disjunción/monosonía, la recombinación mitótica y por genes extra-cromosomales (Abu-El Samen et al. 2003). En el genoma de P. infestans, los genes codificantes de los efectores RXLR están presentes en las regiones dispersas que contienen los elementos transposables (TEs), que evolucionan rápidamente y sobrepasan la resistencia (Raffaele et al. 2010). El silenciamiento transcripcional es otro mecanismo asociado con la ganancia de virulencia (Qutob et al. 2009). Uno de los roles celulares clave es de restringir la actividad de los TEs, manteniendo la estabilidad del genoma (Castel & Martienssen 2013, Malone & Hannon 2009).
Phytophthora infestans presenta gran variabilidad en la región de los Andes peruanos y bolivianos, la misma que es considerada el centro de la diversidad de la papa, en esta región las cepas analizadas indicaron que la colección tenía cinco linajes clonales EC-1, US-1, PE-3, PE-5 y PE-6 (Perez et al. 2001). La población de P. infestans que infecta la papa cultivada en el Perú consiste mayormente en cepas que pertenecen al mismo linaje clonal EC-1. Este linaje apareció en los años 90 remplazando al linaje anterior US-1 y aunque es un linaje clonal, las cepas demuestran una gran variación en virulencia (Delgado et al. 2013), caracterizándose además por ser resistente a metalaxyl y por ser ampliamente virulento (Perez et al. 2001).
El CIP está trabajando en el acercamiento transgénico de papas resistentes al Tizón Tardío, utilizando dos genes provenientes de S. bulbocastanum (RB, Rpi-blb2) y un gen de S. venturii (Rpi-vnt1). Cada uno de estos reconoce un efector específico, Avr-blb1, Avr-blb2 y Avr-vnt1 respectivamente. Avr-blb1, está caracterizado por tener tres clases de variantes. Las clases I y II son requeridas para que la resistencia sea funcional, mientras que la clase III puede vencer la resistencia (Chen et al. 2012). Avr-blb2, miembros de esta familia existen en diferentes variantes alélicas, un residuo clave está en la posición 69 de la proteína efectora que determina si el gen Rpi-blb2 reconoce al patógeno, cuando es mutado desde Alanina (A), o Valina (V) hacia Fenilalanina (F), la activación del gen Rpi-blb2 se pierde (Oh et al. 2009). Con respecto a Avr-vnt1, P. infestans puede evadir el reconocimiento del gen Rpi-vnt1, por la reducción de su expresión, y por la variación epialélica de secuencias conservadas entre los alelos puntuales avirulentos y virulentos (Pel 2010).
Es de nuestro interés estudiar la población patogénica peruana evaluando la expresión de los genes de avirulencia debido al poco conocimiento de estos. El objetivo del presente trabajo fue identificar genes de virulencia diferencialmente expresados entre dos cepas del linaje clonal EC-1 aislados en Perú.
Materiales y métodos
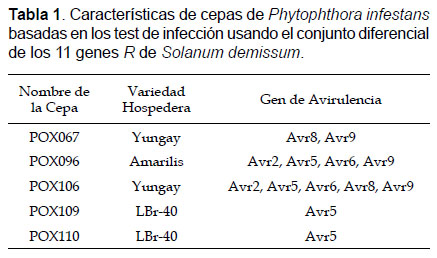
Cepas de P. infestans.- En total cinco cepas peruanas fueron caracterizadas en este estudio. Estas fueron colectadas desde variedades de papa cultivada en la región central de los Andes de Oxapampa y tienen diferentes patrones de virulencia determinados por el set diferencial de papa (Tabla 1). Estas fueron inoculadas en los tubérculos de la variedad de papa susceptible "Yungay" (CIP720064), y en un clon resistente, "LBr-40" (CIP387164.4).
Las plantas tuvieron de 45 días y la concentración del inóculo fue aproximadamente de 3000 esporangios/mL. Cada planta (4 plantas/genotipo/cepa) fue inoculada por rociamiento hasta el goteo con un rociador de mano. Luego de la inoculación, las condiciones de humedad fueron mantenidas, para el desarrollo del Tizón Tardío, con el reparto de vapor frío a través de un sistema de aspersión que opera en un termostato y por la cobertura de las plantas con bolsas plásticas aumenta la transpiración y retiene la temperatura, eso ayuda a la infección. La temperatura estuvo en el rango de 15 ‒ 20 °C y el régimen de luz fue de 12h. La evaluación del Tizón Tardío comenzó 3 días posteriores a la inoculación (dpi). Cada planta fue evaluada por el estado de la enfermedad (porcentaje de hoja afectada). La evaluación fue hecha con la escala de 0 ‒ 100% con 5-unidades de intervalos, donde el valor 0 = reacción no visible, 5 = Respuesta de hipersensibilidad, 10 ‒ 100 = Infección exitosa.
Muestreo y procesamiento.- Para el análisis de la expresión de genes el muestreo fue hecho al 1, 2 y 3 dpi. Los foliolos fueron colectados de la parte media de las plantas. Dos muestras que consistieron de 4 foliolos cada uno, fueron colectadas de cada genotipo. Estas dos muestras fueron consideradas como replicas biológicas, en total fueron seis muestras colectadas de cada interacción entre el genotipo de papa y la cepa de P. infestans. Los foliolos fueron inmediatamente congelados en nitrógeno líquido y almacenados −70 ºC hasta que fueron procesados. Las muestras de foliolos fueron triturados con mortero y pilón en nitrógeno líquido, y 50 ‒ 100 mg del polvo fue utilizado para la extracción total de ARN usando el reactivo de Trizol (Invitrogen®) siguiendo el protocolo del fabricante. El ARN de cada muestra fue extraído separadamente y cantidades equivalentes de muestras seleccionadas fueron juntadas para el secuenciamiento del transcriptoma. Las muestras secuenciadas fueron POX067 y POX109. El secuenciamiento del transcriptoma fue realizado por Fasteris Genomics®, y corrido con Illumina HiSeq2000, generando lecturas pareadas de 2x100 pb. Los datos del secuenciamiento fueron registrados en el archivo de lecturas de secuencias del NCBI [número de accesión SRP065209].
Análisis de datos.-El análisis del secuenciamiento del transcriptoma fue hecho siguiendo el protocolo de Trapnell et al. (2012). Las lecturas de dos bibliotecas fueron mapeadas en contra del genoma de referencia de P. infestans versión ASM14294v1.25 usando TopHat v2.1.0 y Bowtie2 v2.1.0 (Langmead & Salzberg 2012). Los genes y transcritos expresados fueron ensamblados, y los genes expresados diferencialmente fueron identificados usando Cufflinks v2.1.1.
La cinética de los genes expresados fue estudiada por PCR cuantitativo en Tiempo Real (qRT-PCR). La mezcla de la reacción fue preparada en un volumen total de 10 µL conteniendo: 4 µL de molde (50 ng), 0.4 µM de cada cebador, y PCR Master Mix Power SYBR® Green (Applied Biosystems® Stepone Real-Time PCR System). Se usó el siguiente programa de PCR: 95 °C por 3 minutos, 95 °C por 15 segundos y (Ta) °C por 45 segundos durante 40 ciclos, 95 °C por 1 minuto, 65 °C por 1 minuto y 65°C por 10 segundos (análisis de curva de desnaturalización).
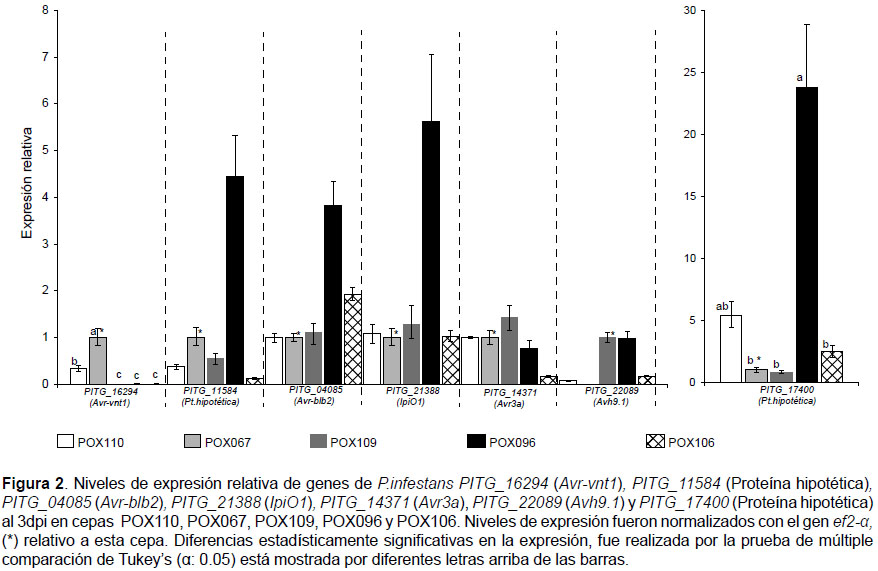
Los cebadores para qRT-PCR fueron diseñados en las secuencias de PITG de los genes flanqueados (Tabla 2) con la aplicación "Primerquest" de Integrated DNA Technologies® (http://www. idtdna.com/). Las eficiencias para los cebadores fueron obtenidas usando curvas de diluciones y un modelo de regresión lineal (Llorente et al. 2010, Pfaffl 2001). El gen de referencia ef2-α fue usado para normalizar los niveles de expresión, y la expresión diferencial fue determinada usando el programa REST® (http:// rest.gene-quantification.info/). Las diferencias estadísticamente significativas entre los resultados de qRT-PCR fueron determinadas mediante el ANOVA y la prueba de comparación múltiple de Tuckey usando el paquete estadístico SAS 9.1.3.
Resultados
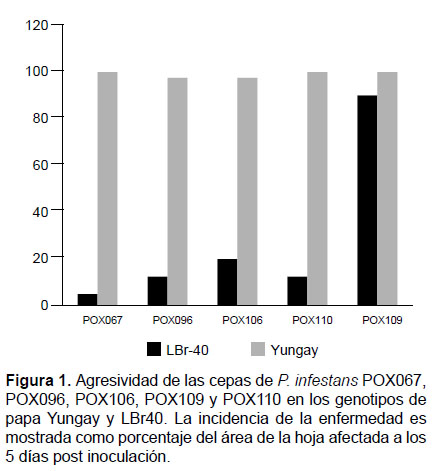
Virulencia de las cepas.- La evaluación de la agresividad de la enfermedad en las cinco cepas fue como sigue: La primera evaluación fue hecha cuando la reacción de hipersensibilidad fue visible en el genotipo de resistencia LBr40 al 3 dpi. La evaluación final (5 dpi) de la variedad susceptible Yungay tuvo un área de la hoja de 90 – 100 % de infección. De las cinco cepas probadas sólo POX109 fue capaz de infectar el genotipo de resistencia (Fig. 1).
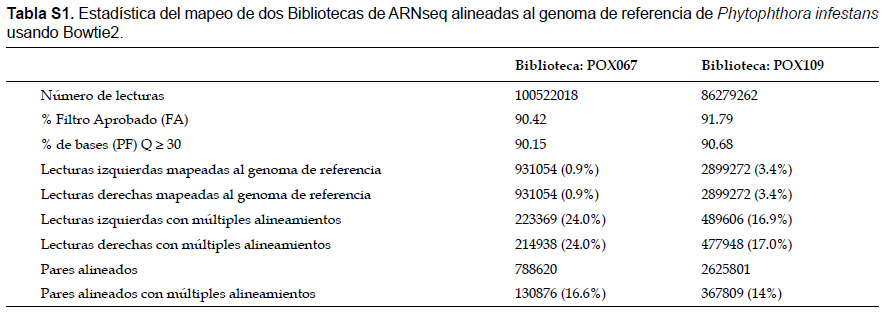
Expresión diferencial.- La expresión diferencial del gen de las cepas POX109 y POX067 fue analizada usando el secuenciamiento del transcriptoma (ARNseq). Como fue esperado, mayoría de los transcritos fueron de origen vegetal y sólo una fracción correspondió al genoma de P. infestans (Tabla S1).
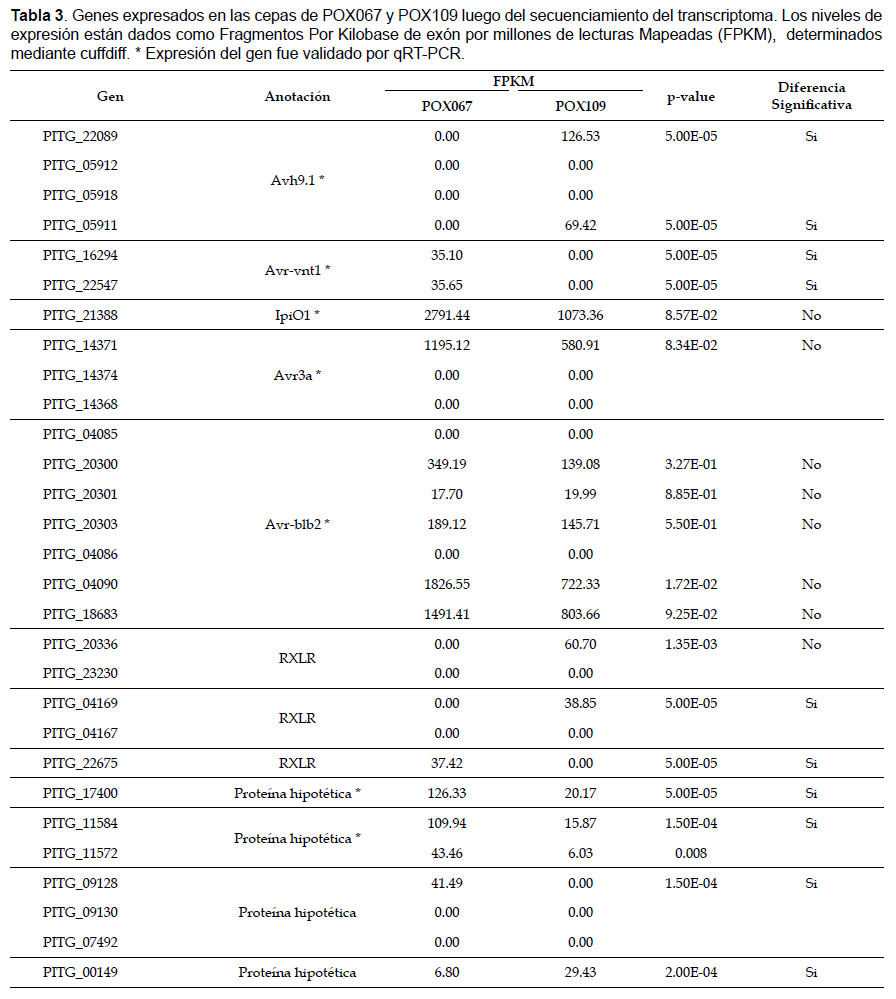
Los clústers que cumplen el criterio de calidad Q30 de Illumina, fueron sobre el 90% para ambas muestras. Los niveles de expresión de genes efectores RXLR y otros genes que aparecieron diferencialmente regulados entre las dos cepas determinados por Cuffdiff (Trapnell et al. 2009) están listados en la Tabla 3. La más notable diferencia entre las dos cepas es el silenciamiento de un número de genes efectores, incluyendo Avr-vnt1 y Avh9.1, y desconocidos tipos de genes RXLR en una de las cepas. Además, ambas cepas expresan Avr-blb1/ipiO, Avr-blb2 y Avr3a sin diferencias significativas.
Validación de la Expresión diferencial.- Para el qRT-PCR, muestras de ARN de plantas inoculadas con cepas de P. infestans colectadas al 1, 2 y 3 dpi, fueron analizadas por los genes efectores Avr-blb1/ipiO clase I (PITG_21388), Avr-vnt1 (PITG_16294), Avr-blb2 (PITG_04085), Avr3a (PITG_14371) y Avh9.1 (PITG_22089), dos genes de proteínas hipotéticas (PITG_17400, PITG_11584), así como por el gen de referencia ef2-α. En todas las cepas, el gen de referencia sólo fue detectado al 3 dpi, no obstante claros picos de desnaturalización fueron vistos en la mayoría de los genes efectores y en todas las cepas comenzando desde el día 1. Como el gen de referencia fue expresado sólo al 3 dpi nosotros sólo fuimos capaces de analizar la expresión diferencial en este día. En acuerdo con el secuenciamiento del transcriptoma, el análisis de qRT-PCR confirmó que POX109 no expresó Avr-vnt1, mientras todas las otras cepas si lo hicieron. La expresión de este gen en la cepa POX067 fue significativamente alta (p<0.05) cuando fue comparada con las otras cepas. Similarmente, Avh9.1 fue silenciado pero en la cepa POX067 y expresado en todas las otras cepas sin diferencias significativas (Fig. 2). Los otros tres efectores Avr (Avr-blb2, Avr-blb1/ipiO y Avr3a) fueron expresados en todas las cepas al mismo nivel.
Discusión
La era genómica, provee una idea de los mecanismos y procesos para el conocimiento de la patogenicidad de P. infestans (Fry et al. 2015). Los análisis de la expresión genética de alto procesamiento por el secuenciamiento de blibliotecas de ADNc, microarreglos y la tecnología de ARNseq, son herramientas valiosas para el estudio de las diferentes familias de genes presentes en la interacción planta-patógeno (Raffaele et al. 2010), por ello en el presente estudio se recurrió al uso de una de estas tecnologías. El reto de identificar genes Avr para la comprensión del patosistema papa-Phyophtora infestans en el contexto peruano es importante para el diseño de programas de diagnóstico, prevención y mejoramiento de la resistencia a P. infestans. Escogiendo cepas de P. infestans con diferentes patrones de virulencia, y con diferentes respuestas fenotípicas en una variedad de papa susceptible nos dió una mayor expresión de efectores para el presente estudio. Así, mediante el análisis del transcriptoma en condiciones de respuesta a la infección en la planta, fueron identificados genes RXLR que exhibieron su patrón de expresión en la fase biotrófica de infección, similar a lo estudiado previamente por Haas et al. (2009). Por otro lado, perfiles comparables de inducción durante la fase biotrófica han sido descritos para los genes efectores RXLR de P. sojae (Wang et al. 2011).
En el presente estudio las cepas de P. infestans en el linaje clonal EC-1 mostraron variación de expresión de efectores así como variación fenotípica de virulencia en papa. Algunos estudios en poblaciones de P. infestans indican una baja diversidad genética, representada por un linaje clonal dominante pero con alta diversidad de virulencia (Delgado et al. 2013). Aquí se demuestra que la variación es verdadera entre cepas a nivel fenotípico y de expresión del gen. Además, la misma cepa puede tener alterada la expresión del gen luego de su colecta en medio de cultivo. Cambios similares en estado de expresión del gen Avr de la misma cepa ha sido mostrado en P. sojae (Na et al. 2014). Esta es una explicación atractiva para el fenómeno observado comúnmente en el cambio de patrones de virulencia en la interacción papa-P. infestans. Algunas diferencias de expresión entre las dos cepas, pueden indicar que algunos efectores pueden ser esenciales para la infección de la papa, y no deben ser perdidos para su correcto reconocimiento (Fry et al. 2015). Así diferentes niveles de interacción entre genes Rpi-/Avr- pueden reflejar la activación de diferentes vías de defensa cuya activación está reflejada en diferentes mecanismos (Pel 2010).
El silenciamiento de genes efectores, especialmente los del tipo de efectores RXLR, es aparentemente un importante mecanismo de regulación de la virulencia/avirulencia en estas cepas. Esto ha sido mostrado en el caso del silenciamiento de Avr-vnt1 que lleva al escape del reconocimiento por el gen R Rpi-vnt1 en el linaje clonal EC-1 (Pel 2010). El gen de resistencia Rpi-vnt1 fue clonado de las especies de Sudamérica S. venturii (Foster et al. 2009) y luego fue mostrado que Rpi-phu1 originario de Solanum phureja (Zimnoch) es idéntico a Rpi-vnt1 (Pel 2010). El grupo de papa Phureja ha sido comúnmente cultivado en los Andes haciendo posible que el silenciamiento de Avr-vnt1 sea un resultado de la co-evolución. Sin embargo, se desconoce si este gen de resistencia es común en la totalidad de genes de Phureja. La regulación epigenética de la expresión del gen de Avr puede servir como un mecanismo reversible el cual permite la conservación de genes Avr para su uso futuro (Gijzen et al. 2014). Así mismo, copias pueden ser silenciadas sin comprometer la eficiencia de la infección del patógeno. Esto enfatiza porque el control de P. infestans permanece como un problema para la seguridad alimentaria (Fry et al. 2015). Así mismo, se puede atribuir que cambios entre la expresión de ciertos efectores son un mecanismo para la conservación de estos en el futuro (Gijzen et al. 2014).
Para Avr3a, en nuestros resultados no se evidencia silencia-miento ni diferencias significativas en la expresión entre las dos cepas. En las poblaciones de P. infestans, Avr3a tiene dos variantes alélicas mayores codificando las proteínas AVR3aKI y AVR3aEM, siendo el primero de estos el que induce la resistencia mediada por R3a (Armstrong et al. 2005).
Para Avr-blb1, sólo fue detectado IpiO1 perteneciente a la clase I, no fue detectada la variante del efector IpiO4 perteneciente a la clase III, aquella que es capaz de vencer la resistencia aún en presencia de IpiO1 (Halterman et al. 2010). Este resultado sugiere que estas cepas podrían ser avirulentes a las plantas de papa con el gen Rpi-blb1 / RB. Román et al. (2015) confirma que el efector Avr-blb1 en la cepa POX067 es reconocido por el transgén RB.
Para Avr-blb2, el aminoácido número 69 de la proteína AVRblb2 es crítico para el reconocimiento por Rpi-blb2, de modo que los homólogos AVRblb2 que son reconocidos por Rpi-blb2 tienen Val69, Ala69 o Ile69, y los que no son reconocidos tienen Phe69 (Oh et al. 2009). El estudio de Orbegozo et al. (2016) confirma, que el efector Avr-blb2 en la cepa POX067 está reconocido por el transgén. Además, POX067, que expresó variantes Ala69 y Phe69, fue probada para ver si su virulencia cambiaba por la sobre-inoculación con P. infestans. Sin embargo, no se registraron cambios en las respuestas sugiriendo que esta cepa no logró descartar o silenciar la variante avirulente de Avr-blb2. Se ha detectado anteriormente que la presencia de múltiples variantes de Avr-blb2 es común en las cepas de P. infestans a nivel mundial probablemente por tener alguna función importante en la patogenicidad.
En conclusión, el silenciamiento de genes de avirulencia, como Avr-vnt1 sugiere que el gen de resistencia Rpi-vnt1 no sería funcional en cepas presentes en el Perú. Este resultado es útil, para no considerar a este gen en futuros programas de mejoramiento de la resistencia en papa. En cambio los genes Avr-blb1 y Avr-blb2 sí podrían ser útiles en el desarrollo del mejoramiento genético para resistencia.
Agradecimientos
Agradecemos a Willmer Pérez en sugerir las cepas para el análisis y por realizar las inoculaciones, a Leticia Portal por realizar las extracciones de ARN y a Mirella Flores por la ayuda con los programas bioinformáticos y el manejo de la data.
Literatura citada
Abu-El Samen F., G. Secor & N. Gudmestad. 2003. Variability in virulence among asexual progenies of Phytophthora infestans. Phytopathology. 93(3): 293-304. doi: http://dx.doi.org/10.1094/PHYTO.2003.93.3.293 [ Links ]
Armstrong M.R., S.C. Whisson, L. Pritchard, et al. 2005. An ancestraloomycete locus contains late blight avirulence gene Avr3a, encoding a protein that is recognized in the host cytoplasm. Proceedings of the National Academy of Sciences of theUnited States of America. 102(21): 7766-7771. doi: http://dx.doi.org/10.1073/pnas.0500113102 [ Links ]
Castel S.E. & R.A. Martienssen. 2013. RNA interference in the nucleus: roles for small RNAs in transcription, epigenetics and beyond. Nature Reviews Genetics. 14(2): 100-112. doi:http://dx.doi.org/10.1038/nrg3355. [ Links ]
Cornelissen B.J. & L.S. Melchers. 1993. Strategies for Control ofFungal Diseases with Transgenic Plants. Plant physiology. 101(3): 709-712. doi: http://dx.doi.org/10.1104/pp.101.3.709 [ Links ]
Chen Y., Z. Liu & D.A. Halterman. 2012. Molecular determinants of resistance activation and suppression by Phytophthora infestans effector IPI-O. PLoS pathogens. 8(3): e1002595. doi: http://dx.doi.org/10.1371/journal.ppat.1002595 [ Links ]
Delgado R., A. Monteros-Altamirano, Y. Li, et al. 2013. Large subclonal variation in Phytophthora infestans populationsassociated with Ecuadorian potato landraces. Plant Pathology. 62(5): 1081-1088. doi: http://dx.doi.org/10.1111/ppa.12039 [ Links ]
Estrada N. 2000. La biodiversidad en el mejoramiento genético de la papa. PROINPA/CID/CIP. La Paz, 372 pp. [ Links ]
Foster S.J., T.-H. Park, M. Pel, et al. 2009. Rpi-vnt1. 1, a Tm-22 homolog from Solanum venturii, confers resistance to potato late blight. Molecular plant-microbe interactions. 22(5): 589-600. doi: http://dx.doi.org/10.1094/MPMI-22-5-0589. [ Links ]
Fry W., P. Birch, H. Judelson, et al. 2015. Five Reasons to consider Phytophthora infestans a re-emerging pathogen. Phytopathology. 105(7): 966-981. doi: http://dx.doi.org/10.1094/PHYTO-01-15-0005-FI10.1094/PHYTO-01-15-0005-FI [ Links ]
Fry W., S. Goodwin, J. Matuszak, et al. 1992. Population genetics and intercontinental migrations of Phytophthora infestans. Annual review of phytopathology. 30(1): 107-129. doi:http://dx.doi.org/10.1146/annurev.py.30.090192.000543 [ Links ]
Fry W.E., N.J. Grünwald, D.E.L. Cooke, et al. 2009. PopulationGenetics and Population Diversity of Phytophthorainfestans. In: Oomycete Genetics and Genomics. JohnWiley & Sons, Inc. pp. 139-164. doi: http://dx.doi.org/10.1002/9780470475898.ch7 [ Links ]
Gijzen M., C. Ishmael & S.D. Shrestha. 2014. Epigenetic control of effectors in plant pathogens. Frontiers in plant science. 5638.doi: http://dx.doi.org/10.3389/fpls.2014.00638. [ Links ]
Goss E.M., J.F. Tabima, D.E. Cooke, et al. 2014. The irish potato famine pathogen phytophthora infestans originated incentral mexico rather than the andes. Proceedings of the National Academy of Sciences. 111(24): 8791-8796. doi: http://dx.doi.org/10.1073/pnas.1401884111 [ Links ]
Haas B.J., S. Kamoun, M.C. Zody, et al. 2009. Genome sequence andanalysis of the Irish potato famine pathogen Phytophthora infestans. Nature. 461(7262): 393-398. doi: http://dx.doi.org/10.1038/nature08358. [ Links ]
Halterman D.A., Y. Chen, J. Sopee, et al. 2010. Competition betweenPhytophthora infestans effectors leads to increased aggressiveness on plants containing broad-spectrum late blightresistance. PLoS One. 5(5): e10536. doi: http://dx.doi.org/10.1371/journal.pone.0010536 [ Links ]
Hogenhout S.A., R.A. Van der Hoorn, R. Terauchi, et al. 2009. Emerging concepts in effector biology of plant-associated organisms. Molecular plant-microbe interactions. 22(2): 115-122. doi: http://dx.doi.org/10.1094/MPMI-22-2-0115. [ Links ]
Langmead B. & S.L. Salzberg. 2012. Fast gapped-read alignmentwith Bowtie 2. Nature methods. 9(4): 357-359. doi: http://dx.doi.org/10.1038/nmeth.1923 [ Links ]
Llorente B., F. Bravo-Almonacid, C. Cvitanich, et al. 2010. A quantitative real-time PCR method for in planta monitoring of Phytophthora infestans growth. Letters in applied microbiology. 51(6): 603-610. doi: http://dx.doi.org/10.1111/j.1472-765X.2010.02942.x [ Links ]
Malone C.D. & G.J. Hannon. 2009. Small RNAs as guardians of the genome. Cell. 136(4): 656-668. doi: http://dx.doi.org/10.1016/j.cell.2009.01.045 [ Links ]
Na R., D. Yu, B.P. Chapman, et al. 2014. Genome Re-Sequencing and Functional Analysis Places the Phytophthora sojaeAvirulence Genes Avr1c and Avr1a in a Tandem Repeat at a Single Locus. PloS one. 9(2): e89738. doi: http://dx.doi.org/10.1371/journal.pone.0089738. [ Links ]
Oh S.-K., C. Young, M. Lee, et al. 2009. In planta expression screens of Phytophthora infestans RXLR effectors reveal diversephenotypes, including activation of the Solanum bulbocastanum disease resistance protein Rpi-blb2. The Plant Cell Online. 21(9): 2928-2947. doi: http://dx.doi.org/10.1105/tpc.109.068247 [ Links ]
Orbegozo J., M.L. Roman, C. Rivera, et al. 2016. Rpi-blb2 gene from Solanum bulbocastanum confers extreme resistance to late blight disease in potato. Plant Cell, Tissue and Organ Culture (PCTOC). (1): 1-13. doi: http://dx.doi.org/10.1007/s11240-016-0947-z [ Links ]
Pel M. 2010. Mapping, Isolation and characterization of genes responsible for Late Blight Resistance in Potato. Wageningen. [ Links ]
Perez W.G., J.S. Gamboa, Y.V. Falcon, et al. 2001. Genetic structure of Peruvian populations of Phytophthora infestans. Phytopathology. 91(10): 956-965. doi: http://dx.doi.org/10.1094/PHYTO.2001.91.10.956 [ Links ]
Pfaffl M.W. 2001. A new mathematical model for relative quantification in real-time RT–PCR. Nucleic acids research. 29(9): e45. doi: http://dx.doi.org/10.1093/nar/29.9.e45 [ Links ]
Qutob D., J. Tedman-Jones, S. Dong, et al. 2009. Copy numbervariation and transcriptional polymorphisms of Phytophthora sojae RXLR effector genes Avr1a and Avr3a. PloS one. 4(4): e5066. doi: http://dx.doi.org/10.1371/journal.pone.0005066. [ Links ]
Raffaele S., J. Win, L.M. Cano, et al. 2010. Analyses of genome architecture and gene expression reveal novel candidate virulencefactors in the secretome of Phytophthora infestans. BMC genomics. 11(1): 637. doi: http://dx.doi.org/10.1186/14712164-11-637 [ Links ]
Román M.L., C. Rivera, J. Orbegozo, et al. 2015. Resistencia a Phytophthora infestans linaje clonal EC-1 en Solanum tuberosummediante la introducción del gen RB. Revista peruana de biología. 22(1): 63-70. doi: http://dx.doi.org/10.15381/rpb.v22i1.11122 [ Links ]
Trapnell C., A. Roberts, L. Goff, et al. 2012. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nature protocols. 7(3): 562-578. doi:http://dx.doi.org/10.1038/nprot.2012.016. [ Links ]
Trapnell C., L. Pachter&S.L. Salzberg. 2009. TopHat: discoveringsplice junctions with RNA-Seq. Bioinformatics. 25(9):1105-1111. doi: http://dx.doi.org/10.1093/bioinformatics/btp120 [ Links ]
Vleeshouwers V.G., S. Raffaele, J.H. Vossen, et al. 2011. Understandingand exploiting late blight resistance in the age of effectors. Annual review of phytopathology. 49507-531. doi: http://dx.doi.org/10.1146/annurev-phyto-072910-095326 [ Links ]
Vleeshouwers V.G., H. Rietman, P. Krenek, et al. 2008. Effector genomics accelerates discovery and functional profiling of potato disease resistance and Phytophthora infestans avirulence genes. PLoS One. 3(8): e2875. doi: http://dx.doi.org/10.1371/journal.pone.0002875 [ Links ]
Wang Q., C. Han, A.O. Ferreira, et al. 2011. Transcriptional programming and functional interactions within the Phytophthora sojae RXLR effector repertoire. The Plant Cell. 23(6): 20642086. doi: http://dx.doi.org/10.1105/tpc.111.086082. [ Links ]
Yoshida K., V.J. Schuenemann, L.M. Cano, et al. 2013. The rise and fall of the Phytophthora infestans lineage that triggered the Irish potato famine. Elife. 2. doi: http://dx.doi.org/10.7554/eLife.00731 [ Links ]
Tabla S1
Información sobre los autores: HL-K, MI: realizaron los experimentos, HL-K, MI: analizaron, interpretaron la data y escribieron el manuscrito. HL-K: Conceptualizó y planeó los experimentos. Los autores declaran que no tienen competencias de intereses. Los autores no incurren en conflictos de intereses.
Fuentes de financiamiento: Los fondos para esta investigación fueron proveídos por el CGIAR Consortium Research Program Roots Tubers and Bananas (RTB).
Presentado: 24/02/2016
Aceptado: 07/10/2016
Publicado online: 20/12/2016