Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista Peruana de Biología
On-line version ISSN 1727-9933
Rev. peru biol. vol.24 no.3 Lima Sept./Dec. 2017
http://dx.doi.org/10.15381/rpb.v24i3.13900
TRABAJOS ORIGINALES
Diferenciación genética de tilapia roja y gris (Oreochromis niloticus) mediante microsatélites y marcadores SCAR como indicadores del sexo genético
Genetic differentiation of red and gray tilapia (Oreochromis niloticus) using microsatellites and SCAR markers as indicators of genetic sex
Monica Arqueros, Linda Sánchez-Tuesta y Zulita Prieto*
Centro Experimental de Genética, Facultad de Ciencias Biológicas, Universidad Nacional de Trujillo, Perú. Tel. 5144-474835.
*Autora para la correspondencia:
Email Monica Arqueros: arqueros.monica@gmail.com
Email Linda Sánchez-Tuesta: santues2686@hotmail.com
Email Zulita Prieto: zprieto@unitru.edu.pe
Resumen
El objetivo del trabajo fue realizar la estandarización de los protocolos moleculares, diferenciar los linajes de la tilapia roja y gris mediante microsatélites de ADN y evaluar los marcadores SCAR5F-X/5R y SCAR-5F/5R-Y como indicadores del sexo genético de O. niloticus. El microsatélite UNH106 permitió diferenciar genéticamente los linajes de la tilapia roja de la gris. Los microsatélites UNH136, UNH115 y UNH995 presentaron loci monomórficos tanto en la tilapia roja como en la gris en el lote poblacional del Centro Experimental de Genética de la Universidad Nacional de Trujillo. Se describen los tamaños de fragmentos para los microsatélites y del gen de referencia β actin. También se confirmó la efectividad de los marcadores SCAR como informativos en la determinación del sexo genético evaluados en hembras XX y YY y machos XY y YY de O. niloticus roja y hembras XX y machos XY de O. niloticus gris.
Palabras clave: Tilapia; Oreochromis; microsatélites; marcadores SCAR; determinación del sexo.
Abstract
The aims of this work was to develop a standardized molecular protocols, to differentiate red and gray tilapia lineages using DNA microsatellites and to evaluate SCAR-5F-X/5R and SCAR-5F/5RY markers associated with the phenotypic sex of O. niloticus. The UNH106 microsatellite allowed differentiating genetically the lineages of red tilapia from gray. The markers UNH136, UNH115 and UNH995 presented monomorphic loci in both the red and gray tilapia in the population stock of the Centro Experimental de Genética of the Universidad Nacional de Trujillo. Fragment sizes for microsatellites and the reference gene β actin are described. The effectiveness of SCAR markers as informative in the determination of the genetic sex in XX and YY females and XY y YY males of red tilapia and XX females and XY males of gray tilapia was also confirmed.
Keywords: Tilapia; Oreochromis; microsatellites; SCAR markers; sex determination.
Introducción
Las especies de tilapia se cultivan en países tropicales y subtropicales con éxito productivo (Cnaani et al. 2008). En el Perú, la acuicultura de la tilapia no está muy desarrollada comparada con otros países latinoamericanos (Baltazar & Palomino 2004), como Brasil, Chile y Ecuador, que se ubican entre los 18 principales productores mundiales (FAO 2016).
Para obtener un óptimo cultivo de tilapia es necesario considerar un adecuado manejo, alimentación, buena calidad genética y monosexo masculino (Ruiz 2012). Independientemente de la especie, raza o línea de la tilapia, los machos tienen la propiedad de crecer más rápido que las hembras e invierten menos energía en la reproducción, por lo que muchas investigaciones han enfatizado las técnicas para la obtención de poblaciones 100% machos genéticamente, pues tienen entre 20 y 30% más peso que una hembra (Castillo 2011). Sin embargo, la gran diversidad de especies, poblaciones y linajes de tilapia, sumado a la similitud fenotípica de la morfología corporal, dificulta su diferenciación, lo que es indispensable para estudios de poblaciones, parentesco, manejo genético y productivo. En este sentido, la contribución de herramientas para la identificación molecular ha significado un claro aporte en la descripción de secuencias del ADN de la tilapia.
Sin embargo, las variaciones en los valores de diversidad genética son propias de un lote poblacional particular. Por ejemplo, Guyon et al. (2010) reportaron alrededor de 1358 marcadores, de los cuales 154 fueron microsatélites de importancia para la evaluación de la radiación de híbridos y otras investigaciones. Cuevas (2010) estudiando Oreochromis aureus, O. niloticus y O. mossambicus estimó la variabilidad genética con ocho microsatélites: UNH145, UNH155, UNH160, UNH166, UNH190, UNH207, UNH208 y UNH211; encontrando que O. mossambicus registraba los menores valores de heterocigosidad al ser comparada con O. aureus y O. niloticus. En el sur de China, con el uso de los microsatélites UNH995, UNH954, GM024, GM119, UNH738, UNH971, GM677, UNH860 y UNH932 se reportaron loci polimórficos y heterocigosidad sobre 0.39 en seis poblaciones de Oreochromis spp. (Dang-en Gu et al. 2014).
A diferencia de los anteriores marcadores, se reportan los microsatélites UNH995 y UNH104 asociados al sexo fenotípico en algunas familias de O. niloticus (Lee et al. 2003) y marcadores SCAR (sequence characterized amplified regions) relacionados con los cromosomas sexuales Y y X de O. niloticus (Sunet al. 2014). Estos marcadores son importantes para la diferenciación del sexo en juveniles, que solo es posible cuando los peces superan los 100 g con la visualización de gonopodios.
Estas investigaciones son relevantes para O. niloticus, ya que la comprobación de su repetitividad en diferentes familias facilitaría el sexaje a temprana edad. Las especies de tilapia tienen 44 cromosomas sin diferencias en el tamaño ni forma de los cromosomas sexuales (McAndrew et al. 2016) y presentan un período de indiferenciación en la morfogénesis hasta los 15 días después de la eclosión, que a pesar del sexo genético queda definida en la singamia. La diferenciación sexual fenotípica puede ser modificada por efectos de la variación de la temperatura, salinidad u otros agentes químicos, como hormonas (Faulkner et al. 1981, Devlin & Nagahama 2002).
Tanto los microsatélites como los marcadores SCAR son herramientas valiosas que deben ser evaluadas en las poblaciones con un plantel productivo y con mayor utilidad en un plantel orientado a la mejora genética, como el de la Universidad Nacional de Trujillo, que cuenta con poblaciones de tilapia de linaje distinto, tilapia gris procedente de San Martín (Perú) y tilapia roja de TilAqua International (Países Bajos).
El objetivo del presente trabajo fue evaluar los microsatélites del ADN para diferenciar linajes de tilapia gris y roja de la especie O. niloticus y la evaluación de los marcadores SCAR en hembras y machos de tilapia roja y gris en dos generaciones Go y G1.
Material y métodos
Material biológico.- Las muestras fueron obtenidas del Centro Experimental de Genética de la Universidad Nacional de Trujillo, Perú. Hembras XX y YY O. niloticus roja. G1 YY del cruce de machos YY con hembras YY O. niloticus roja. Machos XY y YY O. niloticus roja. Hembras XX y machos XY O. niloticus gris generaciones Go y G1.
La determinación del sexo fenotípico se realizó por la observación directa de las gónadas del individuo y el sexaje visual de las papilas genitales (Hussain 2004).
Extracción de ADN total.- Se cortó aproximadamente 15 mg de la aleta caudal de cada individuo. Las muestras fueron guardadas a -20 °C por 24 horas. La extracción del ADN total se realizó mediante la combinación y modificaciones de los métodos descritos por Cawthom et al.(2011), Aljanabi & Martínez(1997) y Kunhareang et al. (2010) modificado, que consistió en colocar las muestras de la aleta caudal en un tubo de microcentrífuga de 1.5 mL, al cual se le agregó 800 µL de buffer de digestión (10 mM TRIS-HCL pH 8.0; 25 mM EDTA pH 8.0; 20 mM NaCl; 2% SDS; tritón 0.5%, PVP 2%, mercaptoetanol 1%, 7 µL de proteinasa K 20 mg/mL), se agitó en vortex por 30 segundos, se dejó reposar los tubos en forma horizontal por 15 minutos y se incubaron a 55 °C por 1 hora. Se centrifugó a 14000 r.p.m. por 5 minutos, se transfirió 400 µL del sobrenadante a un tubo nuevo Eppendorf de 1.5 mL, se adicionó 100 µL de cloruro de sodio 6 M y se agitó en vortex por 30 segundos.
De inmediato, se añadió 300 µL de cloroformo frío y se centrifugó a 14000 r.p.m. por 10 minutos a 4 °C, luego se transfirió 200 µL de la fase acuosa superior a un nuevo tubo. El ADN se precipitó agregando 200 µL de isopropanol, posteriormente se incubó a -20 °C por 30 minutos. Se centrifugó a 14000 r.p.m. por 7 minutos a 4 °C y se descartó el sobrenadante. Se lavó el pellet con 1ml de etanol al 70% y se secó durante 15 minutos a temperatura ambiente. El ADN fue resuspendido en 50 µL de buffer TE (10 mM TRIS – 1mM EDTA con pH 8) y se almacenó a -20 °C.
Electroforesis.- La visualización de los fragmentos de ADN se realizó en gel de agarosa al 1.5% teñido con SYBR Safe DNA. La electroforesis se llevó a cabo a 100 V, 100 mA por 30 minutos. Las bandas de ADN fueron fotografiadas en el fotodocumentador de imágenes BIO-RAD Molecular Imager ChemiDoc XR+ System. Se verificó la calidad del ADN extraído mediante la amplificación del gen endógeno β- actin.
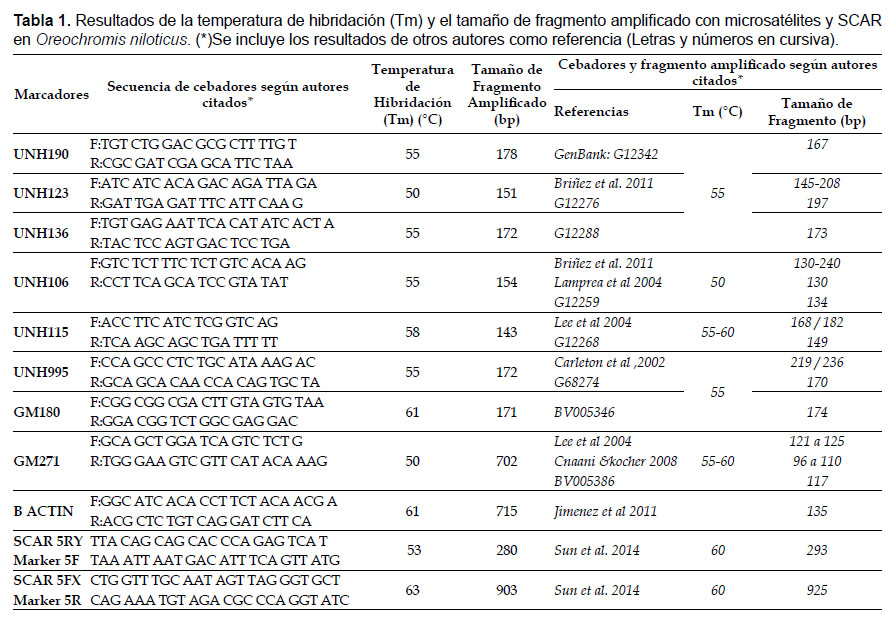
Amplificación de ADN con microsatélites.-Los cebadores usados fueron: UNH190 (5’-TGTCTGGACGCGCTTTTGT3’F y 5’-CGCGATCGAGCATTC-TAA-3’R), UNH136 (5-TGTGAGAATTCACATATCAC-TA-3’F y 5’-TACTCCAGTGACTCCTGA-3‘R), UNH123 (5’ATCATCACAGACAGATTAGA-3’F y 5´-GATTGA-GATTTCATTCAAG-3‘R) y UNH106 (5’-GTCTC- TTTCTCTGTCACAAG-3’F y 5’-CCTTCAGCATC-CGTATAT-3’R); UNH115 (5’-TCAAGCTAGCGATTTTT-3’R y 5’-ACCTTCATCTCGGTCAG-3’F), UNH995 (5’-G-CAGCACAACCACAGTGCTA-3’R y 5’-CCAGCCCTCTG-CATAAAGAC-3’F), GM271 (5’-TGGGAAGTCGTTCATACAAAG-3’R y 5’-GCAGCTGGATCAGTCTCTG3’F) y GM180 (5’-CGGCGGCGACTTGTAGTGTAA-3’F y 5’-GGACGGTCTGGCGAGGAC-3’R) (GenBank número de accesiones: G12342, G12288, G12276, G12259, G12268, G68274, BV005386, BV005346).
Se estandarizó la temperatura de hibridación de los microsatélites usando seis temperaturas distintas. Se usó el termociclador de Applied Biosystem Veriti de 96 pocillos de 0.2 mL.
La mezcla maestra se preparó de la siguiente manera: 2.5 µL de buffer 10X PCR-buffer MgCl2 Invitrogen, 0.75 µL MgCl2 50 mM Invitrogen, 0.5 µL dNTP 10 MmThermo Scientific, 1 µL primer, 0.1 µL Platinum® Taq DNA Polymerase Invitrogen, 18.15µl agua DEPC y 1 µL ADN. La PCR se realizó con las siguientes condiciones: desnaturalización inicial a 95 °C por 3 minutos, 35 ciclos de desnaturalización a 94 °C por 30 segundos; la Tm varió de acuerdo con el primer por 45 segundos, extensión 72 °C por 1 minuto; extensión final 72 °C por 7 minutos.
Con los microsatélites UNH106, UNH136 y UNH995, y β actin, se analizaron las poblaciones de tilapia roja y tilapia gris, en muestras de 25 individuos de cada linaje.
Amplificación con marcadores SCAR (Sun et al. 2014).- Los cebadores usados fueron SCAR-5F (TAAATTAATGACATTTCAGTTATG); SCAR-5R (CAGAAATGTAGACGCCAGGTATC); SCAR-5F-X (CTGGTTTGCAATAGTTAGGGTGCT); SCAR-5R-Y (TTA CAG CAC CCA GAG TCAT) (Sun et al. 2014).
Para la preparación de la mezcla maestra se usó 2.5 µL 10x Coral Load PCR-Buffer Qiagen, 0.5 µL dNTP 10 mM Thermo Scientific, 1 µL primer, 0.125 µL Hot Start Taq Plus, 15.875 µL agua DEPC, 4 µL ADN. La PCR se realizó con las siguientes condiciones: desnaturalización inicial a 94 °C por 5 minutos, 34 ciclos de desnaturalización a 94 °C por1 minutos; SCAR5F/5R-Y 53 °C por 1 minutos, SCAR-5F-X/5R 63 °C por un minuto, extensión 72 °C por un minuto; extensión final 72°C por 7 minutos.
Análisis de datos.-Se determinó el tamaño de cada producto amplificado mediante el software del fotodocumentador de geles ChemiDoc XR+ System. Se comparó el tamaño de bp en los genotipos de hembras y machos en Oreochromis niloticus "tilapia gris" y "tilapia roja" y se evaluó cualitativamente la presencia o la ausencia de bandas para cada marcador.
Resultados
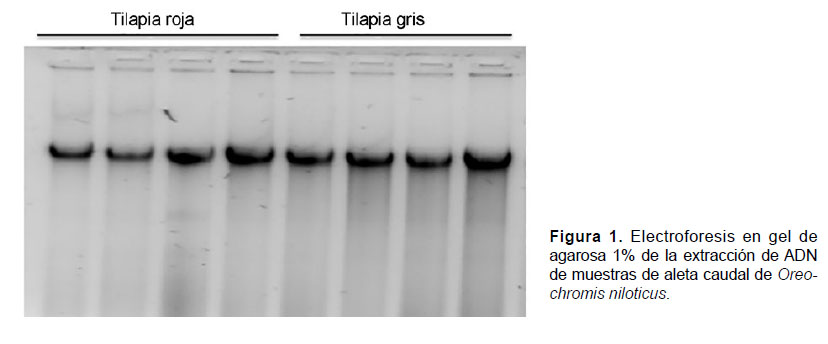
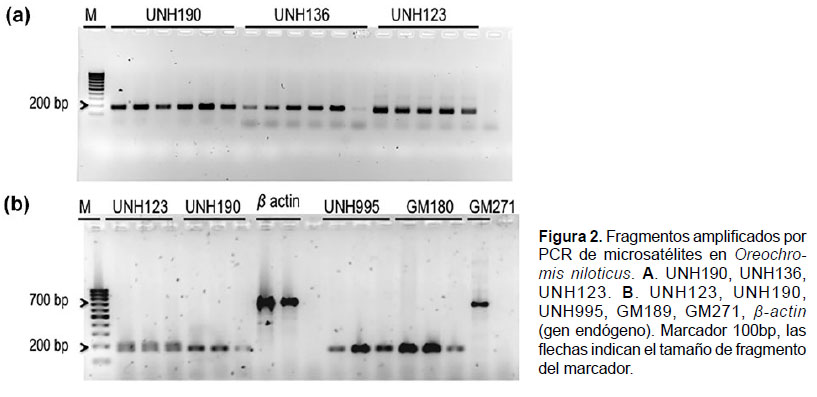
Se obtuvo ADN de tilapia de buena calidad con el protocolo modificado (Fig. 1). En la Tabla 1 y Figura 2 se presentan los resultados de estandarización de la temperatura de hibridación para los microsatélites y SCAR-5F-X/5R y SCAR-5F/5R-Y, y de β-actin, así como el tamaño de los productos amplificados por PCR. Los fragmentos de los microsatélites UNH190, UNH123, UNH136, UNH106, UNH115, UNH995, GM180 y GM271 fueron:178, 151, 172, 154, 143, 172, 171 y 702 bp, respectivamente. Para el SCAR-5F-X/5R fue de 903.30 ± 38.40 bp, para el SCAR-5F/5R-Y fue de 280 ± 17.29 bp y el tamaño del fragmento amplificado para β-actin fue de 715.0 ± 40.87 bp.
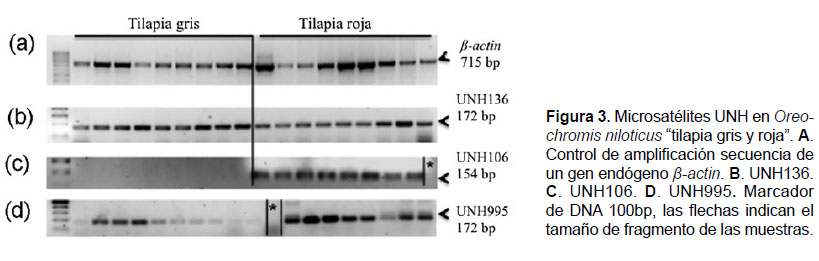
Los perfiles genéticos para los microsatélites UNH y GM fueron monomórficos. En la Figura 3, se observa muestras de individuos de tilapia gris y individuos de tilapia roja amplificados con UNH136 y UNH995 monomórficos con similar velocidad de migración para ambos linajes. No obstante, con el marcador UNH106 no hubo amplificación en ninguna muestra de los individuos de tilapia gris.
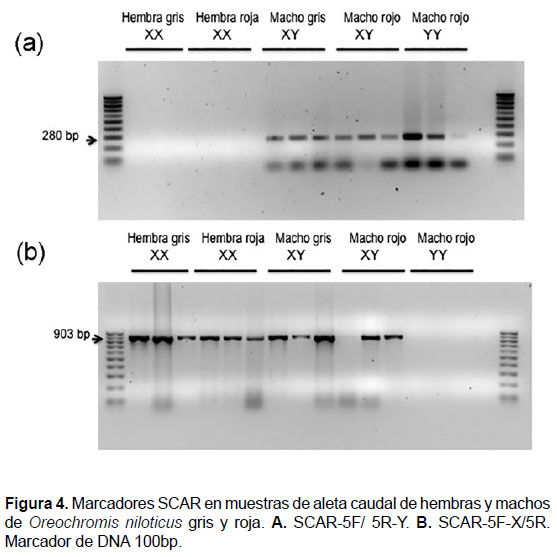
El marcador SCAR-5F-X/5R amplificó en todas las muestras de hembras grises y rojas, así como en los machos grises y rojos excepto en los machos YY; de manera opuesta, con el marcador SCAR-5F/5R-Y se obtuvo producto amplificado en todas las muestras de los machos grises y machos rojos XY y YY (Fig. 4 a y b).
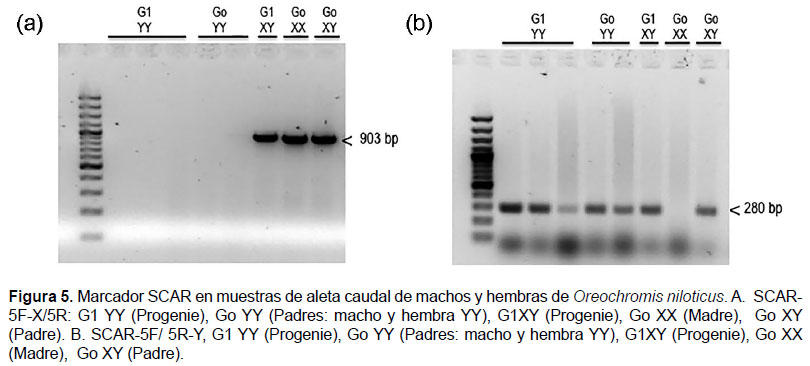
Así también, los marcadores SCAR-5F-X/5R y SCAR-5F/5RY correlacionaron con el sexo fenotípico de las progenies G1 del cruce de hembra YY O. niloticus roja con macho YY O. niloticus roja y G1 del cruce de hembra XX y macho XY tilapia gris (Fig. 5 a y b).
Discusión
Las modificaciones realizadas tomando como base los métodos de extracción de ADN de Aljanabi & Martínez (1997) y Kunhareang et al. (2010) permitieron mejorar la calidad del extracto de ADN de muestras de aleta caudal de tilapia. La adición de tritón al 0.5% en la mezcla del amortiguador de extracción fue lo más relevante de las modificaciones realizadas.
Los microsatélites, marcadores codominantes, se distinguen por su alto grado de polimorfismos que permiten detectar variaciones genéticas entre poblaciones y dentro de ellas (Romana-Eguia et al. 2004). Sin embargo, requieren de estandarización tanto en la calidad del ADN extraído como en la hibridación de los cebadores. Así también, el tamaño de fragmentos de los microsatélites puede diferir entre poblaciones, dependiendo del grado de diferenciación genética existente entre ellas.
En los resultados del trabajo, los tamaños de los fragmentos amplificados concordantes con los registrados en la base de datos del GenBank (htt//:www.ncbi.nlm.nih.gob) fueron: UNH136 (G12288),UNH190 (G12342), UNH123 (G12276), GM180 (BV005346), UNH106 (G12259)y UNH995 (G68274). Sin embargo, los fragmentos para UNH115 registraron 143 bp, lo que difiere de lo reportado por Lee et al. (2004) (G12268); para GM271 se registró 702 bp, distinto de los resultados de Lee et al. (2004) y Cnaani & Kocher 2008, quienes reportaron de 121 a 125 bp y de 96 a 110 bp, respectivamente. El fragmento amplificado del control interno PCR β actin fue 715 bp, distinto de lo registrado por Jiménez et al. (2011).
Las diferencias en los fragmentos de la PCR, particularmente con el marcador GM271, se explicaría debido a la amplificación intensificada en el proceso de replicación, que suele ocurrir por la presencia de secuencias repetidas como los microsatélites (Leffak et al. 2016). La secuencia del gen endógeno β-actin puede explicarse como consecuencia de una inserción en la secuencia. Sin embargo, se necesitan pruebas de secuenciación y realizar una alineación con la base de datos GenBank.
Del grupo de microsatélites evaluados en el Centro Experimental de Genética de la Universidad Nacional de Trujillo, el UNH106 fue el único microsatélite que permitió diferenciar genéticamente la tilapia roja de la tilapia gris y se obtuvo producto de la PCR en todas las muestras de tilapia roja; sin embargo, en las muestras de tilapia gris, la amplificación fue negativa, debido probablemente a los cambios en la secuencia de hibridación del genoma, lo que impidió la complementariedad de bases con uno o ambos cebadores utilizados.
Por otro lado, la presencia de los loci monomórficos analizados con los microsatélites UNH106, UNH136, UNH995 explica y confirma la variación genética reducida, propia de las líneas genéticas establecidas, con la evaluación con microsatélites adicionales podría evidenciar con mayor aproximación la variación genética existente. Estas difieren notoriamente de las poblaciones en las que se producen cruzamientos aleatorios y están sujetas a efectos evolutivos, como las mutaciones, selección natural, migraciones o deriva genética, como las reportadas por Hassanien & Gilbey (2005) en poblaciones de O. niloticus del río Nilo. Para el marcador UNH106 se observó polimorfismo con el valor de 6.01 para el número efectivo de alelos y heterocigosidad promedio de 0.76. Así también, Briñez et al. (2011) y otras investigaciones registraron polimorfismos con distintos microsatélites.
Con relación a la determinación del sexo en la tilapia, la plasticidad genética que presentan los genomas masculino y femenino, y la ocurrencia de cambios de expresión de los genes en las etapas iniciales de diferenciación gonadal dificultan el manejo de cruzamientos. Puede darse el caso de que fenotípicamente un individuo sea macho cuando genéticamente estaba programado para ser hembra y viceversa. Por ello, es necesaria la identificación del sexo genético de los individuos para programar cruzamientos con fines de mejora productiva.
En las especies de tilapia no se registran diferencias en los cromosomas sexuales, pues existe similitud en el tamaño y morfología de los pares de cromosomas (2n= 44) (Majumdar & McAndrew 1986) y hay secuencias de ADN repetido en todos los cromosomas (Martins et al. 2004). Según las evidencias cromosómicas en espermatocitos en paquiteno de O. niloticus, basado en la homología genética que ocurre entre cromosomas homólogos, se registró sinapsis parcial entre el par cromosómico de mayor tamaño (cromosoma 1); al respecto, Carrasco et al. (1999) propusieron que estos serían los cromosomas sexuales. Estudios sobre O. mossambicus mediante pruebas de hibridación in situ por fluorescencia FISH y la sinapsis de cromosomas homólogos en paquiteno de hembras y machos sostienen también que el par 1 correspondería a los cromosomas sexuales (Campos-Ramos et al. 2003). Ferreira & Martins (2008) mediante el análisis de secuencias repetidas por FISH reportaron diferencias entre los cromosomas del primer par de cromosomas y los propusieron como los sexuales.
Diversas investigaciones sobre microsatélites asociados a la segregación en función del sexo fenotípico de la tilapia han identificado grupos de ligamiento LG1 y LG23 relacionados con el sexo de O. aureus (Lee et al. 2004). En O.hornorum, el microsatélite UNH168 fue consistente en la amplificación y asociación al sexo fenotípico femenino, mientras que otros microsatélites estaban presentes en muestras de ambos sexos (Zhu et al. 2016). Los marcadores microsatélites UNH995 y UNH104 relacionados con la diferenciación del sexo en O. niloticus fueron determinados por Lee et al. (2003) y el microsatélite UNH898 fue propuesto como el mejor marcador asociado a los genes determinantes del sexo, por diferenciar machos de hembras con una sensibilidad de 96.71% (Naranjo et al. 2015). Además de los genes en los crosomomas sexuales, proponen la presencia de genes autosómicos en la determinación del sexo (Lee et al. 2003, Eshel et al. 2011).
Sun et al. (2014) diseñaron cebadores SCAR de muestras de O. niloticus y reportaron cuatro marcadores ligados al sexo asignados al grupo de ligamiento LG23 y que hibridan por FISH con los cromosomas más pequeños, el par 22. De estos marcadores, SCAR-5F-X/5R y SCAR-5F/5R-Y fueron probados en la presente investigación y se confirmó su asociación con la diferenciación del sexo fenotípico en hembras y machos de linajes distintos de tilapia roja y gris, producto de la PCR para SCAR-5F-X/5R de 903.3 ± 38.40 y para el SCAR-5F/5R-Y de 280.70 ± 17.29 bp. Esto concuerda con lo reportado por Sun et al. (2014), quienes proponen que SCAR-5F-X/5R y SCAR-5F/5R-Y se distinguen por la presencia de delecciones o inserciones de varios cientos de pares de nucleótidos que se diferencian en el tamaño de los fragmentos amplificados entre los marcadores y se asocian a los cromosomas sexuales YX.
Los resultados de varias investigaciones demuestran la posible existencia de genes determinantes del sexo en diferentes cromosomas del complemento, siendo los cromosomas candidatos a la presencia de genes mayores de determinación del sexo los pares cromosómicos 1 y 22 del complemento cromosómico.
Conclusiones
Los microsatélites UNH136, UNH115, UNH995 y los SCAR-5F/5R-Y, SCAR-F-X/5R presentaron loci monomórfico y no diferencian a O. niloticus "tilapia gris" de O. niloticus "tilapia roja". Mientras que con el microsatélite UNH106 sí se pudo diferenciar a O. niloticus "tilapia roja" de O. niloticus "tilapia gris". Para la β-actin, SCAR-5F/5R-Y y SCAR-5F-X/5R, el tamaño de fragmentos amplificados por la PCR es de 715, 280 y 903 pb, respectivamente. Mientras que en los microsatélites UNH190, UNH123, UNH136, UNH106, UNH115, UNH995, GM180 y GM271 los fragmentos fueron de 178, 151, 172, 154, 143, 172, 171 y 702 bp, respectivamente. La temperatura de hibridación óptima fue de 50 °C para UNH123 y GM271; para los marcadores SCAR-5F/5R-Y fue de 53 °C; para UNH190, UNH136, UNH106, UNH995 fue de 55 °C; para UNH115, 58 °C; para SCAR-5F-X/5R fue de 63 °C; y para β-actin y GM180 fue de 61°C.
Se logró determinar de manera inequívoca la asociación del sexo fenotípico con los marcadores SCAR (5F-X/5R y 5F/5R-Y) en hembras XX, machos XY y supermachos YY de O. niloticus "tilapia roja" y hembras XX y machos XY en O. niloticus "tilapia gris".
Agradecimientos. A los biólogos Julio León, Carlos Quijano y David Salirrosas por el apoyo en la investigación.
Literatura citada
Aljanabi S.M. & I. Martínez. 1997. Universal and rapid salt-extractionof high quality genomic DNA for PCR-based techniques.Nucleic Acids Research 25: 4692-4693. DOI https://doi.org/10.1093/nar/25.22.4692 [ Links ]
Baltazar P. & A. Palomino. 2004. Manual de cultivo de tilapia. Lima: Fondo Nacional de Desarrollo Pesquero. Fondo Nacional de Desarrollo Pesquero –FONDEPES, Agencia Española de Cooperación Internacional –AECI, Proyecto de Apoyo al Desarrollo del Sector Pesca y Acuícola del Perú –PADESPA. 115p [ Links ]
Briñez B., X Caraballo, M. Salazar. 2011. Genetic diversity of six populations of red hybrid tilapia, using microsatellites genetic markers. Revista MVZ Córdoba 16(2):2491-2498. [ Links ]
Campos-Ramos R., S. Harvey, J. Brendan, et al.2003. An investigation of sex determination in the Mozambique tilapia,Oreochromis mossambicus, using synaptonemal complex analysis, FISH, sex reversal and gynogenesis. Aquaculture 221:125-140. DOI https://doi.org/10.1016/S00448486(03)00072-3 [ Links ]
Carrasco, L.A., D.J. Penman & N. Bromage. 1999. Evidence for the presence of sex chromosomes in the Nile tilapia (Oreochromis niloticus) from synaptonemal complex analysis of XX, XY and YY genotypes. Aquaculture 173:207-218. DOIhttps://doi.org/10.1016/S0044-8486(98)00488-8 [ Links ]
Castillo C. L.F. 2011. Tilapia roja 2011. Una evolución de 29 años, de la incertidumbre al éxito. Colombia. Disponible en https://ag.arizona.edu/azaqua/ista/reports/TILAPIAROJA2010.doc [ Links ]
Cawthorn D.M., H.A. Steinman & R.C. Witthuhn. 2011. Comparative study of different methods for the extraction of DNA fromfish species commercially available in South Africa. Food Control 22(2):231-244. DOI https://doi.org/10.1016/j.foodcont.2010.07.003 [ Links ]
Cnaani A., B. Lee, N. Zilberman, C. Ozouf, et al. 2008. Genetics of sex determination in tilapiine species. Sexual Development 2(1): 43-54. DOI https://doi.org/10.1159/000117718 [ Links ]
Cuevas R., B.L. 2010. Evaluación productiva y variabilidad genética de especies de tilapia del género Oreochromis en el estado de Sinaloa. Tesis demaestría en recursos naturales y medio ambiente. Instituto Politecnico Nacional. Centro Interdisciplinario de Investigación para el Desarrollo IntegralRegional, UnidadSinaloa. http://itzamna.bnct.ipn.mx/bitstream/handle/123456789/9899/258.pdf?sequence=1 [ Links ]
Dang-en Gu, Xi-dong Mu, Hong-Mei Song, et al. 2014.Genetic diversity of invasive Oreochromis spp. (tilapia) populations inGuangdong province of China using microsatellite markers.Biochemical Systematics and Ecology, 55: 198-204. DOI https://doi.org/10.1016/j.bse.2014.03.035 [ Links ]
Devlin, R.H. & Y. Nagahama. 2002. Sex determination and sex differentiation in fish: an overview of genetic, physiological, and environmental influences. Aquaculture 208 (3-4): 191-364.DOI https://doi.org/10.1016/S0044-8486(02)00057-1 [ Links ]
Eshel O., A. Shirak, J.I. Weller, et al. 2011. Fine mapping of a locus on linkage group 23 for sex determination in Nile tilapia (Oreochromis niloticus). Anim. Genet. 42: 222-224. DOI https://doi.org/10.1111/j.1365-2052.2010.02128.x [ Links ]
FAO 2016. El estado mundial de la pesca y la acuicultura 2016.Contribución a la seguridad alimentaria y la nutrición para todos. Roma. 224 pp <http://www.fao.org/3/a-i5555s.pdf> [ Links ]
Faulkner, L.C., M.H. Pinedo, L.E. McDonald. 1981. Reproducción yendocrinología. 2ª.ed. México: Interamericana. pp. 180-182. [ Links ]
Feng Liu, Jian Li, Fei Sun, et al. 2013. A microsatellite-based linkage map ofsalt tolerant tilapia (Oreochromis mossambicus x Oreochromisspp) and mapping of sex-determining loci. BMC Genomics 14:58. DOI https://doi.org/10.1186/1471-2164-14-58 [ Links ]
Ferreira, I.A. &C. Martins. 2008. Physical chromosome mapping of repetitive DNA sequence in Nile tilapia Oreochromis niloticus: Evidences for a differential distribution of repetitive elements in the sex chromosome. Micron 39(4): 411-418. DOI https://doi.org/10.1016/j.micron.2007.02.010 [ Links ]
Guyon R., F. Senger, M. Rakotomanga, et al. 2010. A radiation hybridmap of the European sea bass (Dicentrarchus labrax) bases on 1581markers: Synteny analysis with model fish genomes.Genomics 96(4):228-238. DOI https://doi.org/10.1016/j.ygeno.2010.07.007 [ Links ]
Hassanien A.H. & J. Gilbey. 2005.Genetic diversity and differentiationof Nile tilapia (Oreochromis niloticus) revealed by DNA microsatellites. Aquaculture Research 36:1450-1457. DOI https://doi.org/10.1111/j.1365-2109.2005.01368.x [ Links ]
Hussain, M.G. (2004). Farming of tilapia: breeding plans, mass seed production and aquaculture techniques. Bangladesh Fisheries Research Institute. [ Links ]
Jiménez A., V. Tibatá, H. Junca, et al. 2011. Evaluating a nested-PCR assay for detecting Streptococcus agalactiae in red tilapia (Oreochromis sp.) tissue. Aquaculture 321(3-4):203-206. DOI https://doi.org/10.1016/j.aquaculture.2011.09.011 [ Links ]
Kunhareang S.,H. Zhou & J.G.H. Hickford.2010. Rapid DNA extraction of pig ear tissues. Meat Science 85(3):589-590. DOIhttps://doi.org/10.1016/j.meatsci.2010.02.028 [ Links ]
Lee, D., J.Penman & T.D. Kocher. 2003. Identification of a sex-determining region in Nile tilapia (Oreochromis niloticus) using bulked segregant analysis. International Society for Animal Genetics, Animal Genetics, 34: 379-38. DOI https://doi.org/10.1046/j.1365-2052.2003.01035.x [ Links ]
Lee B.Y.,G. Hulata & T.D. Kocher.2004. Two unlinked loci controllingthe sex of blue tilapia (Oreochromis aureus) Heredity, 59(2):543-549. DOI https://doi.org/10.1038/sj.hdy.6800453 [ Links ]
Leffak M., R. Gadgil, J. Barthelemy & T. Lewis. 2016. Replication stalling and DNA microsatellite instability. Biophysical Chemistry. DOI https://doi.org/10.1016/j.bpc.2016.11.007 [ Links ]
McAndrew B., D.J. Penman, M. Bekaert & S. Wehner. 2016. Tilapiagenomics studies. Genome in Aquaculture.Pp. 105-129.DOI https://doi.org/10.1016/B978-0-12-801418-9.00005-6 [ Links ]
Majumdaa, K.C. & B.J. McAndrew. 1986. Relative DNA content of somatic nuclei and chromosomal studies in three genera, Tilapia, Sarotherodon, and Oreochromis of the tribe Tilapiini (Pisces, Cichlidae). Genetica 68:175-188. DOI https://doi.org/10.1007/BF02424441 [ Links ]
Martins C., C. Oliveira, A. Wasko & J.M.Wright.2004. Physicalmapping of the Nile tilapia (Oreochromis niloticus) genomeby fluorescent in situ hybridization of repetitive DNAs to metaphase chromosomes – a review. Aquaculture 231:37-49.DOI https://doi.org/10.1016/j.aquaculture.2003.08.017 [ Links ]
Naranjo G., Ortiz D.R., Chávez M.A., Manjunatha B., Tirado J.O., Mu-oz D., Kundapur R.R. & Ravi M. 2015. Optimization of detection of sexual genotype through microsatellites in populations of Nile Tilapia (Oreochromis niloticus) from the breeding pools. Der Pharma Chemica 7(10):536-542. [ Links ]
Romana-Eguia M.R.R., M. Ikeda, Z.U. Basiao & N. Taniguchi. 2004.Genetic diversity in farmed Asian Nile and red hybrid tilapia stocks evaluated from microsatellite and mitochondrial DNA analysis. Aquaculture 236(1-4):131-150. DOI https://doi.org/10.1016/j.aquaculture.2004.01.026 [ Links ]
Ruiz, R.L. 2012. Estado de la acuicultura en el Perú. Revista AquaTIC. 37: 99-106. [ Links ]
Shirak A, E. Seroussi, N. Zilberman, et al. 2007. Mapping of candidate genes for sex determination in tilapias. Aquaculture. 272(1):S271. DOI https://doi.org/10.1016/j.aquaculture.2007.07.093 [ Links ]
Sun Y.L., D.N. Jiang, S. Zeng,et al.2014. Screening and characterization of sex-linked DNA markers and marker-assisted selection in the Nile Tilapia (Oreochromis niloticus).Aquaculture 433:19-27. DOI https://doi.org/10.1016/j.aquaculture.2014.05.035 [ Links ]
Zhu H., Liu Z., Lu M., et al. 2016. Screening and identification of a microsatellite marker associated with sex in Wami tilapia, Oreochromis urolepis hornorum. J. Genet 95(2):283-289. DOI https://doi.org/10.1007/s12041-016-0653-y. [ Links ]
Presentado: 04/01/2017
Aceptado: 22/09/2017
Publicado online: 28/10/2017
Información sobre los autores:
MA que realizó la parte experimental. LS-T realizó la parte experimental.ZP realizo el diseño experimental y planificación; MA, LS-T, ZP revisaron y aprobaron el manuscrito.
Los autores no incurren en conflictos de intereses.
Fuentes de financiamiento: El presente trabajo se realizó gracias al financiamiento del canon minero otorgado a la Universidad Nacional de Trujillo.
e