Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.25 no.3 Lima jul./set. 2018
http://dx.doi.org/10.15381/rpb.v25i3.15213
NOTA CIENTÍFICA
Hymenolepis microstoma (Cestoda: Hymenolepididae) en ratones caseros (Mus musculus ) de Lima, Perú
Hymenolepis microstoma (Cestoda: Hymenolepididae) in house mice (Mus musculus) from Lima, Peru
Luis A. Gomez-Puerta*, Cesar A. Valdivia-Carrera
Universidad Nacional Mayor de San Marcos. Facultad de Medicina Veterinaria. Laboratorio de Epidemiología y Economía Veterinaria. Av. Circunvalación 2800, San Borja. Lima, Perú.
Resumen
Un total de 12 cestodos adultos se colectaron de los conductos biliares de ratones domésticos (Mus musculus) provenientes de Lima, Perú. Diversas características del escólex y proglotis maduros del cestodo fueron observadas para la identificación morfológica. Así mismo, se realizó un diagnóstico molecular mediante un PCR y secuenciación parcial del gen mitocondrial citocromo c oxidasa subunidad 1 (cox1). Todos los cestodos fueron identificados como Hymenolepis microstoma por morfología y métodos moleculares. El aislado de H. microstoma de Perú mostró una similitud de secuencia significativa (> 99%) con los aislados de H. microstoma previamente reportados. Nuestro informe confirma la presencia del parásito en ratones de Lima.
Palabras claves: Hymenolepididae; cestodo; Hymenolepis; ratón domésticos; Mus musculus; citocromo c oxidasa subunidad I; cox1.
Abstract
A total of 12 adult cestodes were collected from the bile ducts of domestic mice (Mus musculus) from Lima, Peru. Various features of the scolex and mature proglottids of the tapeworms were observed for morphological identification. A molecular diagnosis was performed by PCR-based partial sequencing of mitochondrial gene of cytochrome c oxidase subunit 1 (cox1). All cestodes were identified as Hymenolepis microstoma by morphology and molecular methods. The H. microstoma isolate from Peru showed significant sequence similarity with previously reported isolates of H. microstoma (>99%). Our report confirms the presence of the parasite in mice from Lima.
Keywords: Hymenolepididae; cestode; Hymenolepis; domestic mouse; Mus musculus; cytochrome c oxidase subunit I; cox1.
Introducción
Los cestodos de la familia Hymenolepididae (Cyclophyllidea) son parásitos de diversas especies de aves y pequeños mamíferos, principalmente roedores, insectívoros y quirópteros (Czaplinski & Vaucher 1994). Algunas especies de esta familia tienen importancia para la salud pública y pueden causar enfermedades graves en personas inmunocomprometidas, principalmente las especies que parasitan a roedores (Macnish et al. 2003, Marangi et al. 2003, Olson et al. 2003).
Los principales reservorios de estos cestodos zoonóticos son los roedores comensales Rattus rattus, Rattus norvegicus y Mus musculus (Foronda et al. 2011). Estos roedores actúan como hospederos definitivos para Hymenolepis diminuta, Hymenolepis nana (=Rodentolepis nana) e Hymenolepis microstoma (Nkouawa et al. 2016, Foronda et al. 2011, Macnish et al. 2003). Estos cestodos tienen una distribución cosmopolita y son capaces de infectar a humanos (Nkouawa et al. 2016). El presente estudio utiliza el diagnostico morfológico y molecular, analizando parcialmente el gen mitocondrial citocromo c oxidasa 1 (cox1), para demostrar la presencia H. microstoma en el ratón casero (M. musculus), provenientes de Lima, Perú, agregando así un nuevo registro geográfico para el parásito.
Material y métodos
En Julio del 2017, haciendo un monitoreo parasitológico en roedores plaga, 12 cestodos fueron colectados del conducto biliar del ratón (M. musculus) provenientes del distrito de Santiago de Surco en Lima, Perú. Los cestodos fueron fijados en formol al 4% caliente y luego preservados en etanol al 70%. Algunos proglotis grávidos fueron preservados en etanol absoluto para estudios moleculares.
Estudio morfológico. Los proglotidos fueron teñidos con tricrómico de Gomori, deshidratados en series sucesivas de etanol hasta etanol absoluto, clarificados en eugenol y montados en bálsamo de Canadá. Los escólex fueron montados temporalmente en medio Berlese para facilitar la observación y medición de los ganchos rostelares. Las microfotografías se hicieron usando un microscopio Carl Zeiss (Axioskop 40) y las medidas se obtuvieron con el programa de análisis de imágenes Leica IM50 (Version, 4.0 R117). Las medidas se expresan en milímetros (mm) y micrómetros (µm). Las características métricas se mencionan en rango con el promedio y el error estándar (ES) en paréntesis.
Diagnóstico molecular. El ADN fue extraído de proglotis grávidos de algunos especímenes utilizando la metodología descrita por Gomez-Puerta et al. (2016). Para ellos, algunos segmentos de proglotis grávidos fueron colocados y deshidratados en viales de plástico de 0,2 ml. Luego se añadieron a los viales 100 µL de solución de Chelex al 5% y 10 µL de proteinasa K (20 mg/mL). Los viales se incubaron en un termociclador utilizando el siguiente programa: 1 hora a 57 °C, 10 minutos a 95 °C, 1 minuto a 37 °C, 10 minutos a 95 °C y finalmente 15 minutos a 15 °C. Las muestras de ADN se almacenaron a -70 °C hasta su uso. Se utilizó la reacción en cadena de la polimerasa (PCR) para amplificar un fragmento de aproximadamente 400 pares de bases (pb) del gen mitocondrial citocromo c oxidasa 1 (cox1) utilizando los protocolos y cebadores JB3 y JB4.5 descritos por Bowles y McManus, 1994. Los productos de PCR se secuenciaron usando un secuenciador automatizado ABI 3100 (Applied Biosystems). Las secuencias se ensamblaron utilizando el programa ChromasPro 1.7.6 (http://www.technelysium.com.au/ChromasPro.html). Todas las secuencias se compararon con secuencias de referencia obtenida del GenBank mediante el BLASTn (https://blast.ncbi.nlm.nih.gov). Las secuencias se alinearon utilizando el programa BioEdit (http://www.mbio.ncsu.edu/bioedit/bioedit.html). El Programa MEGA7 (http://www.megasoftware.net/) fue utilizado para generar un árbol filogenético a través del método "neighbor-joining" con la distancia de 2-parámetros de Kimura, este método es útil para comparar grandes juegos de datos de secuencias con bajos niveles de divergencia (Tamura et al. 2004). La secuencia obtenida en este estudio se depositó en el GenBank con acceso No MG570384 . Los especímenes estudiados fueron depositados en la Colección de Parásitos del Laboratorio de Epidemiologia y Economía Veterinaria de la Facultad de Medicina Veterinaria, UNMSM (FMV-599), Lima, Perú.
Resultados
Cestodos delgados y largos con una longitud total del estróbilo de 4.2 a 8.5 (5.5; ES= 2) cm y un ancho máximo de 832 – 1,384 (986; ES=12) µm. El escólex tiene 212 – 260 (228; ES=2) µm de ancho, con 4 ventosas de 80 – 111 (99; ES=4) µm de diámetro.
El róstelo mide 55 – 72 (66; ES=3) µm de largo y 32 – 50 (40; ES=3) µm de ancho, es armado con 24 – 26 ganchos de forma cricetoide. La longitud de los ganchos fue 14 – 16 (15; ES=0.1) µm de largo. El saco rostelar tiene 73 – 89 (79; ES=3) µm de profundidad y 48 – 66 (56; ES=3) µm de ancho máximo.
La disposición de los proglotis son de forma craspedote. Los proglotis inmaduros miden 92 – 164 (124; ES=4) µm de largo y 371 – 445 (409; ES=3) µm de ancho. Los proglotis maduros miden 180 – 271 (221; ES=3) µm de largo y 595 – 896 (752; ES=17) µm de ancho. Los proglotis grávidos tienen una longitud de 230 – 372 (297; ES=8) µm y un ancho de 1001 – 1384 (1159; ES=32) µm.
El sistema masculino está formado por tres testículos, uno poral y dos aporal, de forma oval o esféricos de 120 – 181 (145; ES=2) µm de largo y 115 – 166 (143; ES=2) µm de ancho. Presenta una vesícula seminal externa de 109 – 225 (162; ES=9) µm de longitud y 69 – 119 (91; ES=3) µm de ancho. El saco del cirro mide 143 – 207 (172; ES=3) µm de longitud y 37 – 57 (45; ES=2) µm de ancho máximo. La vesícula seminal interna tiene una longitud de 68 – 197 (101; ES=3) µm y un ancho máximo de 40 – 89 (55; ES=2) µm.
El sistema femenino consiste en un ovario lobulado , situado en la zona medial del proglotis, con una longitud de 77 – 121 (103; ES=6) µm y un ancho de 153 – 388 (261; ES=31) µm. La vagina es delgada y se localiza debajo del saco del cirro. El poro genital es unilateral y dextral, se abre en el tercio superior del lado del proglotis. Los proglotis grávidos contienen numerosos huevos, los cuales miden 83 – 94 (88; ES = 1) µm de largo y 69 – 87 (80; ES = 2) µm de ancho. Los huevos presentan 3 – 5 (4; ES = 0.2) filamentos polares. Las oncósferas tienen un diámetro de 38 – 44 (42, ES = 0.5) µm y los ganchos de las oncósferas miden 16 – 19 (17; ES = 0.2) µm de largo.
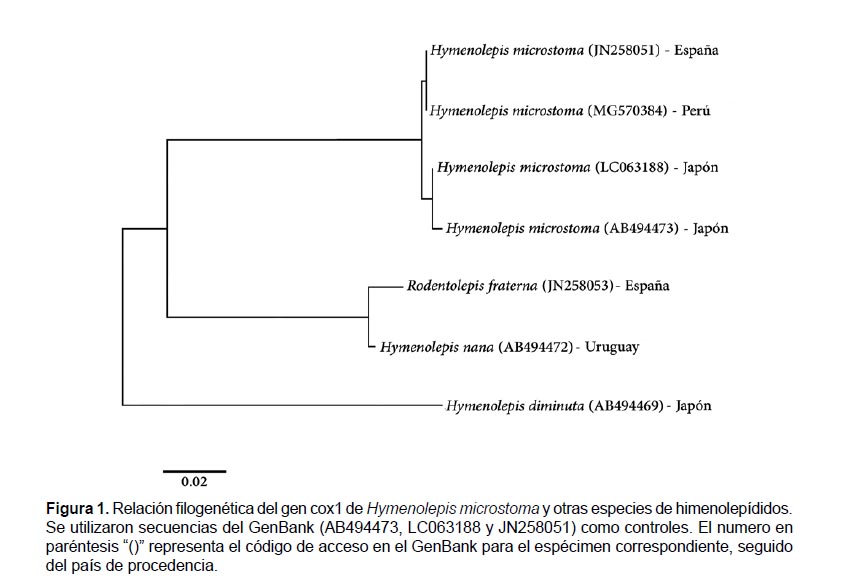
La identidad genética de H. microstoma se determinó por la alineación de la secuencia parcial del gen cox1. La secuencia obtenida en este estudio tuvo 418pb y fue alienada con secuencias de H. microstoma obtenidas del GenBank (JN258051, LC063188 y AB494473) (Nkouawa et al. 2016, Foronda et al. 2011, Okamoto et al. 1997). Las secuencias de H. microstoma obtenidas del GenBank fueron colectados de ratones (M. musculus) de España y Japón. Nuestro resultado mostro que las secuencias del presente estudio y las del GenBank tenían más del 99% de identidad entre ellas, con 2 y 3 nucleótidos de diferencia con las muestras de España y Japón, respectivamente. El análisis filogenético (Fig. 1) mostro dos ramas (clusters) para H. microstoma, una para los especímenes de Perú y España, y otra para los especímenes de Japón. Debido a esta baja variabilidad genética, esto podría indicar que H. microstoma se introdujo en Perú desde un país Europeo.
Discusión
Los parámetros morfológicos y la secuencia parcial del gen cox1 coincidieron con estudios previos de H. microstoma (Nkouawa et al. 2016, Cunningham & Olso 2010, Okamoto et al. 1997).
Esta especie de cestodo ha tenido mucha discrepancia respecto a su nomenclatura. Hymenolepis microstoma fue descrita por primera vez como Taenia microstoma, posteriormente fue incluida dentro del género Hymenolepis (Blanchard, 1891). Sin embargo, Spasskii (1954), realizó una revisión de la familia Hymenolepididae de mamíferos y trasladó a H. microstoma dentro del género Rodentolepis. Schmidt (1986) realizó una revisión de las especies de cestodos y consideró a Rodentolepis sinónimo del género Vampirolepis, quedando así el nombre de Vampirolepis microstoma. En la última revisión taxonómica de la familia Hymenolepididae, Czaplinski y Vaucher (1994) consideran valido al género Rodentolepis. Sin embargo, estudios actuales siguen considerando la nomenclatura del parásito como H. microstoma (Nkouawa et al. 2016).
Hymenolepis microstoma se caracteriza por parasitar el conducto biliar del hospedero mamífero. Los huevos que contienen oncósferas son expulsados con las heces al medio ambiente y pueden ser ingeridos por la etapa adulta o larval de un escarabajo apropiado (Tribolium confusum, T. castaneum, Tenebrio molitor y Oryzaephilus surinamensis), el cual actúa como hospedero intermediario (Dvorak et al. 1961). Los hospederos definitivos naturales para H. microstoma incluyen una amplia lista de roedores de los géneros Apodemus, Dendromus, Leggada, Mastomys y Mus), jerbos (Meriones Illiger) y ratones de campo (Microtus Schrank) (Schmidt 1986, Litchford 1963). Sin embargo, es más frecuente hallarlo en M. musculus tal como lo demuestra este estudio.
En un estudio se evaluó experimentalmente el poder infectivo de H. microstoma en Mus musculus, Rattus norvegicus y Mesocricetus auratus, y concluyeron que M. musculus es la especie más adecuada como hospedero definitivo. Esto debido a que el porcentaje de infección fue hasta 80% en M. musculus comparado con el hámster (M. auratus), el cual fue 30% y no logrando la infección en R. norvegicus (Dvorak et al. 1961).
Los estadios adultos de H. microstoma e H. nana son distinguibles morfológicamente (Baer & Tenora 1970, Czaplinski & Vaucher 1994). Algo peculiar es la forma morfológica de los huevos de H. microstoma, los cuales son muy similares con los huevos de H. nana, las medidas de los huevos y oncósferas, así como la posición de los filamentos polares son muy similares entre ellos (Macnish et al. 2003, Baer & Tenora 1970).
Los hallazgos de H. microstoma en humanos (Macnish et al. 2003) sugiere la importancia del uso de herramientas moleculares para la detección e identificación de cestodos hymenolepididos colectados de humanos, especialmente en especias morfológicamente similares. Debido a la confirmación de H. microstoma en ratones M. musculus, un roedor que tiene estrecha relación con los humanos, se requerirá realizar futuros estudios epidemiológicos para saber el estado actual de infección en humanos y la comprensión de la dinámica de transmisión del parásito.
Información sobre los autores:
LAGP y CAVC: realizaron los muestreos; analizaron los datos; redactaron, revisaron y aprobaron el manuscrito.
Los autores no incurren en conflicto de intereses.
Literatura citada
Baer J.G. & F. Tenora. 1970. Some species of Hymenolepis (Cestoidea) from rodents and from primates. Acta Scientiarum Naturalium Academiae Scientiarum Bohemoslovacae Brno 4:1–32. [ Links ]
Blanchard R. 1891. Histoire Zoologique et Médicale des Téniadés du genre Hymenolepis Weinland. Paris.112p [ Links ]
Bowles J. & D.P. McManus. 1994. Genetic characterization of the Asian Taenia, a newly described taeniid cestode of humans. The American Journal of Tropical Medicine and Hygiene 50(1): 33-44. PMid:7905720. https://doi.org/10.4269/ajtmh.1994.50.1.TM0500010033 [ Links ]
Cunningham L.J. & P.D. Olson. 2010. Description of Hymenolepis microstoma (Nottingham strain): a classical tapeworm model for research in the genomic era. Parasites & Vectors 31(3):123. doi: https://doi.org/10.1186/1756-3305-3-123 [ Links ]
Czaplinski B. & C. Vaucher. 1994. Family Hymenolepididae Ariola, 1899. In Keys to the Cestode Parasites of Vertebrates. Edited by: Khalil LF, Jones A, Bray RA. Wallingford, U.K.: CAB International.595-663. [ Links ]
Dvorak J.A., A.W. Jones, H.H. Kuhlman. 1961. Studies on the biology of Hymenolepis microstoma (Dujardin, 1845). The Journal of parasitology 39: 128 – 132. doi: https://doi.org/10.2307/3275481 [ Links ]
Foronda P., M. López-González, M. Hernández, V. Haukisalmi, C. Feliu. 2011. Distribution and genetic variation of hymenolepidid cestodes in murid rodents on the Canary Islands (Spain). Parasites & Vectors 26(4):185. doi: https://doi.org/10.1186/1756-3305-4-185 [ Links ]
Gomez-Puerta L.A., V. Alarcon, J. Pacheco, F. Franco, M.T. LopezUrbina, A.E. Gonzalez. 2016. Molecular and morphological evidence of Taenia omissa in pumas (Puma concolor) in the Peruvian Highlands. Revista Brasileira de Parasitologia Veterinaria 25(3):368-73. doi: https://doi.org/10.1590/S1984-29612016046. [ Links ]
Litchford R.G. 1963. Observations on Hymenolepis microstoma in three laboratory hosts: Mesocricetus auratus, Mus musculus, and Rattus novegicus. Journal of Parasitology 49(3):403410. https://doi.org/10.2307/3275808 [ Links ]
Macnish M.G., U.M. Ryan, J.M. Behnke, R.C.A. Thompson. 2003. Detection of the rodent tapeworm Rodentolepis (= Hymenolepis) microstoma in humans. A new zoonosis? International journal for parasitology 33:1079-1085. https://doi.org/10.1016/S0020-7519(03)00137-1 [ Links ]
Marangi M., B. Zechini, A. Fileti, G. Quaranta, A. Aceti. 2003. Hymenolepis diminuta infection in a child living in the urban area of Rome, Italy. Journal of clinical microbiology 41:39943995. https://doi.org/10.1128/JCM.41.8.3994-3995.2003 [ Links ]
Nkouawa A., V. Haukisalmi, T. Li, M. Nakao, A. Lavikainen, X. Chen, H. Henttonen, A. Ito. 2016. Cryptic diversity in hymenolepidid tapeworms infecting humans. Parasitology International 65(2):83-6. doi: https://doi.org/10.1016/j.parint.2015.10.009 [ Links ]
Okamoto M., T. Agatsuma, T. Kurosawa, A. Ito. 1997. Phylogenetic relationships of three hymenolepidid species inferred from nuclear ribosomal and mitochondrial DNA sequences. Parasitology 115(6):661-6. [ Links ]
Olson P.D., K. Yoder, L.F. Fajardo L-G, A.M. Marty, S. van de Pas, C. Olivier, D.A. Relman. 2003. Lethal invasive cestodiasis in immunosuppressed patients. J Infect Dis, 187:1962-1966. [ Links ]
Schmidt G.D. 1986. Handbook of Tapeworm Identification. Boca Raton, Florida: CRC Press; [ Links ].
Spasskii A.A. 1954. Classification of Hymenolepididae from mammals. Trudy Gel'mintologicheskoj Laboratorii Akademii Nauk 7:120-167. [ Links ]
Tamura K., M. Nei, S. Kumar. 2004. Prospects for inferring very large phylogenies by using the neighbor-joining method. Proceedings of the National Academy of Sciences of the United States of America 101(30): 11030-11035. http://dx.doi.org/10.1073/pnas.0404206101. [ Links ]
* Autor para correspondencia
Email Luis A. Gomez-Puerta: lgomezp@unmsm.edu.pe ; lucho92@yahoo.com
ORCID Luis A. Gomez-Puerta: http://orcid.org/0000-0002-7909-979X
Email Cesar A. Valdivia-Carrera: cesararturovc@gmail.com
ORCID Cesar A. Valdivia-Carrera: http://orcid.org/0000-0002-8597-0676
Presentado: 01/12/2017
Aceptado: 02/09/2018
Publicado online: 25/09/2018