Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.25 no.3 Lima jul./set. 2018
http://dx.doi.org/10.15381/rpb.v25i3.15216
NOTA CIENTÍFICA
Expresión diferencial de genes involucrados en la respuesta al estrés por helada en Solanum tuberosum subsp. andigena
Differential expression of genes involved in response to cold stress in Solanum tuberosum subsp. andigena
Diana Martinez *1, Demetrio López 2 y Edgar Neyra 1,3
1 Unidad de Genómica, Laboratorios de Investigación y Desarrollo (LID), Universidad Peruana Cayetano Heredia. Av. Honorio Delgado 430, Urb. Ingeniería, S.M.P. Lima - Perú.
2 Facultad de Ciencias e Ingeniería, Universidad para el Desarrollo Andino
3 Facultad de Medicina, Universidad Peruana Cayetano Heredia
Resumen
La helada es un fenómeno meteorológico caracterizado por la disminución de la temperatura del aire por debajo de cero grados centígrados que provocan estrés térmico en las plantas, es uno de los problemas de mayor incidencia e impacto en la agricultura andina. Por otro lado, estudios morfológicos y fisiológicos señalan a las papas nativas como fuentes de tolerancia genética a las bajas temperaturas. Sin embargo, las bases moleculares de los mecanismos de tolerancia en papa, aún son desconocidas. La tecnología del secuenciamiento de ARN (RNA-seq) es una excelente herramienta que permite identificar cambios en los perfiles de expresión génica tras el estrés por helada. En este trabajo, el transcriptoma de una variedad de papa tolerante y otra variedad susceptible a helada fueron secuenciados y comparados después de la exposición al estrés por helada (-8 °C) durante una hora, utilizando la técnica del RNA-seq para identificar diferencias en la expresión de genes entre ambas variedades. Se observó que más de 199 millones de lecturas de RNA-seq fueron alineadas al genoma de referencia ( Solanum tuberosum Grupo Phureja), lo que corresponde al 82.1% del total de las lecturas obtenidas tras el secuenciamiento. En cuanto a la modulación en la expresión de genes, la variedad tolerante presentó el mayor número de genes expresados diferencialmente ( Differentially Expressed Genes, DEGS) sobreexpresados (262 DEGs) y la variedad susceptible presentó el mayor número de DEGs reprimidos (37 DEGs) tras el estrés por helada.
Palabras claves : secuenciamiento ARN; transcriptómica; papa; helada; expresión.
Abstract
Freezing is abiotic stress characterized by a decrease air temperature below zero degrees Celsius. It is one of the highest incidence and impact problems in Andean´s agriculture. Morphological and physiological studies indicate that native potatoes are sources of genes related to plant frost tolerance. However, the molecular bases of tolerance mechanisms in potato are still unknown. RNA sequencing technology (RNA-seq) is an excellent tool to identify genes expression profile changes after freezing stress. In this work, the transcriptome of a freezing tolerant and another susceptible variety of potato were sequenced and compared after exposition to freezing stress (-8 °C) for an hour, using the RNA-seq technique to identify differences in the genes expression between both varieties. It was observed that more than 199 million reads were aligned against the reference genome (Solanum tuberosum Group Phureja), which correspond to 82.1% of the total reads obtained after sequencing. The tolerant variety showed the highest number of Differentially Expressed Genes (DEGs) upexpressed (262 DEGs), while the susceptible variety showed the highest number of down-expressed DEGs (37 DEGs).
Keywords : RNA sequencing; transcriptomics; potato; freezing; expression.
Introducción
La papa es una planta herbácea y anual perteneciente a la familia de las Solanáceas. Es el cuarto cultivo alimenticio de mayor importancia a nivel mundial, debido a su valor nutricional, adaptabilidad a diferentes climas y sistemas de cultivo (Devaux et al. 2010). En el Perú existen más de 2500 variedades de papas nativas, muchas de las cuales se caracterizan por tolerar diferentes tipos de estrés abiótico como sequía, salinidad o helada, constituyendo un valioso reservorio genético para futuros programas de mejoramiento vegetal (Ochoa 2001).
En los últimos años, el desarrollo de nuevas tecnologías de secuenciamiento de alto rendimiento, como el secuenciamiento de ARN (RNA-seq) ha permitido la detección y cuantificación de transcritos con mayor sensibilidad (Lister et al. 2009).
En la actualidad, se cuentan con estudios transcriptómicos (RNA-seq) de la respuesta al estrés por helada en cultivos comoArabidopsis thaliana, Oryza sativa,Triticum aestivum, Brassica juncea y Lilium lancifolium (Ren et al. 2015, Nakaminami et al. 2014, Yang et al. 2015, Gulick et al. 2005, Sinha et al. 2015, Wang et al. 2014). Sin embargo, hasta la fecha son limitados los estudios transcriptómicos de la respuesta a helada (a -8 °C) en papas nativas (Espinoza 2017 & Murata 2017).
Nuestro estudio permitió reportar y confirmar la diferencia en la expresión de genes tras el estrés por helada entre dos variedades de papa nativa (una tolerante y otra susceptible).
Materiales y métodos
Material vegetal.- Se realizaron evaluaciones fisiológicas de tolerancia al estrés por helada en cuatro variedades de papas nativas en invernaderos de la Universidad para el Desarrollo Andino (UDEA), localizados en el distrito de Lircay, provincia de Angaraes, región de Huancavelica (12°59'22"S, 74°43'14"W) a una altitud de 3278 m, con temperaturas que oscilan entre 2 y 18°C. En base a los resultados obtenidos en las evaluaciones se seleccionaron dos variedades tetraploides, nativas de Huancavelica (CIP y FEDECH, 2006), para el presente trabajo: Yana Manwa (Solanum tuberosum subsp.andigena), como variedad tolerante a helada y; Yuraq Gaspar ( Solanum tuberosum subsp. andigena), como variedad susceptible a helada (Morales, 2017).
Para el experimento, las variedades de papa seleccionadas, fueron propagadas a partir de esquejes de tallos laterales y regadas hasta la aparición de raíces. Posteriormente, las plántulas fueron trasplantadas a 12 macetas por variedad, en tierra vegetal con un perfil nutritivo (Musgo Sphagnum + Vermiculita + Perlita + Nutrientes), con riegos periódicos de solución nutritiva hasta alcanzar una altura de 30 - 40 cm., con temperaturas 18/2°C día/noche y 18 horas de fotoperiodicidad durante 12 semanas (Seppänen & Coleman 2003).
Diseño del experimento.- Transcurridos los tres meses de desarrollo, ambas variedades fueron sometidas a una simulación de helada artificial (estrés térmico - bajas temperaturas) mediante congelación real (-8 °C por una hora) en una cámara de temperatura controlada (INDUMELAB, Modelo OPCa-D-120) ubicada en UDEA.
El diseño experimental para ambas variedades (YM, variedad tolerante y YG, variedad susceptible) consistió en la colecta de hojas en dos tiempos: Tiempo Control (antes del estrés, T0) y Tiempo Estrés (después del estrés, T1), considerando 3 repeticiones (R1, R2 y R3) por cada tiempo. Cada planta fue considerada como una unidad experimental. Por lo tanto, se obtuvieron 12 muestras: 2 variedades, en 2 tiempos de muestreo con 3 repeticiones cada una. Tras la colecta de hojas, las muestras fueron conservadas a -80 °C hasta su uso.
Extracción de ARN.- Se extrajo el ARN a partir de 1 g de muestra de hojas, a través del reactivo Trizol (InvitrogenTM, Life Technologies) según las indicaciones del fabricante.
Para estimar la pureza del ARN extraído se calcularon las relaciones de absorbancia A260/A280 de cada una de las muestras. La cuantificación del material extraído se realizó en un espectro-fotómetro Eppendorf® y la integridad de las muestras de ARN fueron evaluadas en geles de agarosa al 2% (90V por 45 min).
Con el fin de eliminar restos de ADN genómico, el RNA extraído fue tratado con el kit DNase Treatment and Removal Reagents (Ambion ®, DNA-free).
Secuenciamiento de ARN.- La construcción y el secuencia-miento de las librerías se realizaron en el laboratorio Microarray Clinical Core de la Universidad de California, Los Angeles (UCLA). El secuenciamiento se realizó en un secuenciador de nueva generación (NGS) usando la plataforma Illumina HiSeqTM 3000 generando lecturas del tipo single-end de 50 bp.
Análisis bioinformático.- El procesamiento, montaje y análisis de datos del transcriptoma fueron realizados en un servidor instalado en el área de Hardware, Redes y Sistemas de la Universidad Peruana Cayetano Heredia, en el cual se encuentra instalado el sistema operativo LINUX (UBUNTU versión 14.04) en base al pipeline bioinformático desarrollado por Trapnell et al. (2012) para datos secuenciados en Illumina que poseen un genoma de referencia.
El control de calidad de las lecturas (reads) generadas del secuenciamiento, se realizó a través del programa FastQC (http:// www.bioinformatics.babraham.ac.uk/projects/fastqc/). Las lecturas de buena calidad se mapearon contra el genoma de referencia de la papa (DM1-3516R44) obtenido en http://solanaceae. plantbiology.msu.edu/pgsc_download.shtml, utilizando los programas Bowtie versión 2.2.3 (http://bowtie-bio.sourceforge. net/index.shtml) y TopHat versión 2.0.12 (http://ccb.jhu.edu/ software/tophat/index.shtml. Luego, para el montaje y cuantificación de los transcritos se utilizó el programa Cufflinks versión 2.2.1 (http://cole-trapnell-lab.github.io/cufflinks/). Finalmente, para la detección de los genes expresados diferencialmente (Dife rentially Expressed Genes, DEGs) se utilizó el programa Cuffdiff versión 2.2.1, incluido en el paquete Cufflinks.
Resultados
Extracción de ARN.- Las concentraciones del ARN extraído oscilaron entre 342.7 ng/µL y 980.2 ng/µL. En cuanto a la relación de la absorbancia A260/280, el valor osciló entre 1.8 y 2.0, indicando que todas las muestras se encontraron puras y sin contaminantes. La integridad del material extraído se determinó a partir de un gel de agarosa al 2%, visualizándose las bandas correspondientes a los ARN ribosomales (28S, 18S y 5S). Por lo tanto, la extracción de ARN con Trizol permitió obtener una adecuada cantidad y calidad de RNA para el secuenciamiento.
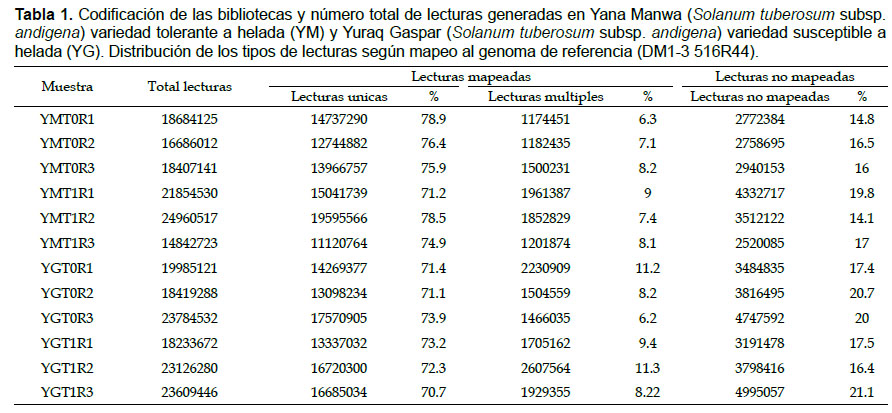
Control de Calidad.- La construcción y secuenciamiento de las bibliotecas fue realizada a partir del ARN extraído generándose un total de 12 librerías con lecturas de tamaño de 50 pb.
El secuenciamiento de las bibliotecas generó más de 242 millones de lecturas y en promedio más de 20 millones de lecturas por biblioteca. El mayor número de lecturas se generó en la biblioteca YMT1R2.fastq (24960517 lecturas) y el menor número de lecturas se generó en la biblioteca YMT1R3.fastq (14842723 lecturas) (Tabla 1).
En el programa FastQC, se evaluaron los valores de calidad de cada base de todas nuestras bibliotecas a través del valor del Phred Score. En todas nuestras bibliotecas, se observó, que el Phred score fue mayor a 30 (indicando una probabilidad de ocurrencia de 1 error en cada 1000 pb) (Ewing & Green 1998). Por lo tanto, la calidad por base generada en todas nuestras librerías fue óptima y confiable, no siendo necesario el descarte de ninguna lectura por baja calidad.
Luego, las lecturas fueron mapeadas al genoma de la papa (doubled monoploid S. tuberosum Group Phureja DM1-3). Se reporta un promedio de 72.3% de lecturas únicas mapeadas (aquellas que mapearon en una única posición del genoma), 9% fueron lecturas múltiples mapeadas (aquellas que mapearon en más de una posición en el genoma) y 17.7% que no mapearon al genoma, estas últimas fueron excluidas del análisis. Estos valores indicaron un rendimiento similar durante la construcción y el secuenciamiento entre librerías (Tabla 1).
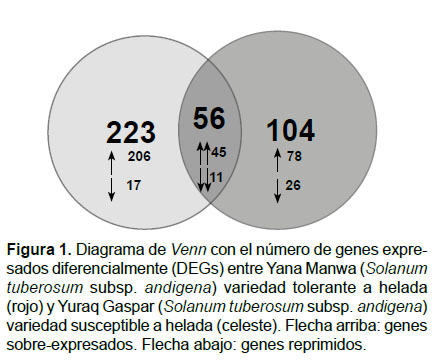
Genes expresados diferencialmente.- Los niveles de expresión diferencial entre las condiciones evaluadas fueron obtenidos con el programa CuffDiff. La unidad de medida de expresión que se utilizó fue el de lecturas por kilobase por millón de lecturas mapeadas (Read per Kilobase per million mapped reads, RPKM), la cual fue utilizada para normalizar los niveles de expresión de los genes (Garber et al. 2011). Tras el estrés se identificó un total de 383 DEGs para las variedades evaluadas, 279 DEGs de la variedad tolerante y 160 DEGs de la variedad susceptible, siendo 56 DEGs comunes para ambas variedades (Fig. 1).
En cuanto a la modulación en la expresión de genes, la variedad tolerante presentó un mayor número de DEGs cuya expresión fue modulada positivamente (genes sobreexpresados), mientras que, la variedad susceptible presentó un mayor número de DEGs cuya expresión fue modulada negativamente (genes reprimidos).
Dentro de los 56 DEGs comunes, se encontraron 45 DEGs sobreexpresados en ambas variedades y, 11 DEGs sobreexpresados en variedad tolerante y reprimidos en la variedad susceptible. No se observó represión y sobreexpresión de DEGs en la variedad tolerante y susceptible, respectivamente; ni represión de DEGs en ambas variedades.
Discusión
Chhangawala et al. (2015) demostraron que no existe diferencia significativa en la detección de expresión diferencial, al usar lecturas de 50pb single-end vs lecturas de 100bp paired-end. Por lo tanto, al no registrarse diferencias significativas, el secuenciamiento single-end resulta ser una alternativa igual de informativa y hasta económica, pues el secuenciamiento paired-end puede llegar a costar el doble de precio.
La precisión del análisis diferencial depende del alineamiento de las lecturas. Trapnell et al. (2012), indicaron que al menos 70% de las lecturas de RNA-seq deben ser alineadas al genoma de referencia, tasas de mapeo inferiores pueden indicar lecturas de baja calidad o presencia de contaminantes. En la Tabla 1, se observa que más de 199 millones de lecturas de RNA-seq fueron alineadas en el genoma de referencia (S. tuberosum), lo que corresponde al 82.1% del total de las lecturas obtenidas tras el secuenciamiento. Estos resultados indicaron que los datos del secuenciamiento fueron consistentes y adecuados para un análisis de transcriptoma de alta calidad.
En general, se encontró una mayor cantidad de DEGs en la variedad tolerante que en la variedad susceptible. Sin embargo, en Solanum goniocalix y Solanum stenotomum, Espinoza (2017) y Murata (2017) reportaron resultados opuestos, es decir un mayor número de DEGs en la variedad susceptible. Cabe mencionar que, en estos trabajos, las temperaturas de estrés fueron de 0 °C y -4 °C, posiblemente temperaturas que no representaron estrés significativo para la variedad tolerante, no siendo necesaria la activación de toda la maquinaria de respuesta al estrés.
Nuestra variedad tolerante presentó un mayor número de DEGs sobreexpresados comparado con la variedad susceptible, lo cual coincide con lo reportado por Chaudhary y Sharma (2015), quienes encontraron mayor sobre-expresión de DEGs en pepino amarillo (Hippophae rhamnoides) tolerante al estrés por helada, sugiriendo que la respuesta al estrés por helada está correlacionada directamente con la regulación positiva de genes.
Winfield et al. (2010) mencionaron que los transcritos de respuesta común en 3 variedades de maíz (dos variedades de invierno: Harnesk y Solstice y, una variedad de primavera: Paragon) tras la exposición a 4 °C por 2 días, estarían involucrados en la respuesta basal al estrés por frío. Mientras que los transcritos comunes únicamente en las variedades de invierno, estarían involucrados en la resistencia o capacidad de tolerancia al estrés por frío.
Los resultados muestran en general, que existe una expresión diferencial de genes relacionados a la respuesta al estrés por helada entre la variedad tolerante y susceptible. La expresión de los genes comunes brindará importante información sobre las rutas metabólicas basales que se activan en respuesta al estrés por helada. Mientras que, los genes sobreexpresados únicamente en la variedad tolerante, podrían estar relacionados con la capacidad de la planta para activar múltiples rutas que permitan la adaptación a las nuevas condiciones del medio.
Agradecimiento
Al Programa Nacional de Innovación para la Competitividad y Productividad -Innóvate Perú de acuerdo al Contrato 120-FINCyT-IA-2013, por haber financiado parte de la realización del presente trabajo.
Información sobre los autores:
DM: Diseño y realización del experimento. DL: obtención, siembra y monitoreo del desarrollo de las plantas. DM y EN: Análisis de datos y redacción del artículo. EN: revisión del artículo.
Los autores han declarado no incurrir en conflicto de intereses.
Fuentes de financiamiento: Programa Nacional de Innovación para la Competitividad y Productividad - Contrato 120-FINCyT-IA-2013.
Literatura citada
CIP & FEDECH. 2006. Catálogo de Variedades de papa nativa de Huancavelica. Centro Internacional de la Papa (CIP) yFederación Departamental de comunidades campesinas de Huancavelica. [ Links ]
Chaudhary S., & P.C. Sharma. 2015. DeepSAGE Based Differential Gene Expression Analysis under Cold and Freeze Stressin Seabuckthorn ( Hippophae rhamnoides L.). Plos One10(3): e0121982. doi: https://doi.org/10.1371/journal.pone.0121982 [ Links ]
Chhangawala S., G. Rudy, C.E. Mason, et al. 2015. The impact of read length on quantification of differentially expressed genesand splice junction detection. Genome Biology 16:131. doi: https://doi.org/10.1186/s13059-015-0697-y [ Links ]
Devaux A., M. Ordinola, A. Hibon, et al. 2010. El sector papa en la región andina. Diagnóstico y elementos para una visión estratégica (Bolivia, Ecuador y Perú). Perú: CIP (CentroInternacional de la Papa). [ Links ]
Espinoza M. R. 2017. Análisis transcriptómico de la respuesta a heladas en papas nativas. Tesis, Magíster en Bioquímica y Biología Molecular. Facultad de Ciencias y Filosofía. Universidad Peruana Cayetano Heredia. Acceso: 24/11/2018 [ Links ]
Ewing B., & P. Green. 1998. Base-calling of automated sequencer traces using phred. II. Error probabilities. Genome Research8(3): 175-185. [ Links ]
Garber M., M.G. Grabherr, M. Guttman, et al. 2011. Computational methods for transcriptome annotation and quantification using RNA-seq. Nature Methods 8(6):469-77. doi: https://doi.org/10.1038/nmeth.1613 [ Links ]
Gulick P., S. Drouin, Z. Yu, et al. 2005. Transcriptome comparison of winter and spring wheat responding to low temperature. Genome 48(5): 913–923. doi: https://doi.org/10.1139/g05-039 [ Links ]
Head S., H. Komori & S. LaMere. 2014. Library construction for nextgeneration sequencing: Overviews and challenges. BioTech 56 (2), 61-64. doi: https://doi.org/10.2144/000114133.eCollection2014 [ Links ]
Lister R., B. Gregory & J.R. Ecker. 2009. Next is now: new technologies for sequencing of genomes, transcriptomes, and beyond.Current Opinion Plant Biology 12: 107–118. doi: https://doi.org/10.1016/j.pbi.2008.11.004 [ Links ]
Morales S. 2017. Respuesta fisiológica de dos variedades de papasnativas ( Solanum tuberosum sp. andigena) de Huancavelica Yana Manwa y Yuraq Gaspar frente a estrés térmico. Tesis para optar el grado de Ingeniero Agrónomo. Facultad de Ciencias e Ingeniería. Universidad para el DesarrolloAndino. Acceso: 10/08/2018 [ Links ]
Murata E. J. 2017. Caracterización de la expresión genética en respuesta a temperaturas de congelamiento en papas nativas tolerantesy susceptibles. Tesis, Licenciada en Biología. Facultad de Ciencias y Filosofía. Universidad Peruana Cayetano Heredia. Acceso: 14/10/2018 [ Links ]
Nakaminami K, A. Matsui, H. Nakagami, et al. 2014. Analysis of differential expression patterns of mRNA and protein during cold-acclimation and de-acclimation in Arabidopsis. Molecular & Cell Proteomics 13: 3602–3611. doi: https://doi.org/10.1074/mcp.M114.039081 [ Links ]
Ochoa C. 2001. Las Papas de Sudamérica: Bolivia. Centro Internacional de la Papa (CIP), Instituto Francés de Estudios Andinos (IFEA), Lima, Perú [ Links ]
Ren L., D. Zhang, G. Chen G, et al. 2015. Transcriptomic profiling revealed the regulatory mechanism of Arabidopsis seedlings response to oxidative stress from cryopreservation. Plant CellReports 34 (12): 2161-2178. doi: https://doi.org/10.1007/s00299-015-1859-9 [ Links ]
Sinha S., V. Raxwal, B. Joshi, et al. 2015. De novo transcriptomeprofiling of cold-stressed siliques during pod filling stages in Indian mustard (Brassica juncea L.). Frontiers Plant Science 6: 932. doi: https://doi.org/10.3389/fpls.2015.00932 [ Links ]
Trapnell C., A. Roberts, L. Goff, et al. 2012. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nature Protocols 7(3): 562-578. doi: https://doi.org/10.1038/nprot.2012.016 [ Links ]
Trapnell C., B. Williams, G. Pertea, et al. 2010. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation.Nature Biotechnology 28(5):511-515. doi: https://doi.org/10.1038/nbt.1621 [ Links ]
Wang J., Y. Yang, X. Liu X, et al. 2014. Transcriptome profilingof the cold response and signaling pathways in Lilium lancifolium. BMC Genomics 15: 203. doi: https://doi.org/10.1186/1471-2164-15-203 [ Links ]
Winfield M., Ch. Lu, I. Wilson, et al. 2010. Plant responses to cold: transcriptome analysis of wheat. Plant BiotechnologyJournal 8(7): 749-771. doi: https://doi.org/10.1111/j.14677652.2010.00536.x [ Links ]
Yang Y., H. Chen, W. Jen, et al. 2015. Comparative transcriptome analysis of shoots and roots of TNG67 and TCN1 riceseedlings under cold stress and following subsequent recovery: Insights into metabolic pathways, phytohormones, and transcription Factors. PLoS One 10(7): e0131391. doi: https://doi.org/10.1371/journal.pone.0131391 [ Links ]
*Autor para Correspondencia
Email Diana Martinez: diana.martinez.cor@gmail.com
Email Demetrio López: dlopez@udea.edu.pe
Email Edgar Neyra: edgar.neyra@upch.pe
Presentado: 18/05/2017
Aceptado: 26/07/2018
Publicado online: 25/09/2018