Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.25 no.4 Lima oct. 2018
http://dx.doi.org/10.15381/rpb.v25i4.14312
TRABAJOS ORIGINALES
Asociación de la resistencia al mercurio con la resistencia a antibióticos en Escherichia coli aislados del litoral de Lima, Perú
Association of mercury resistance with resistance to antibiotics in Escherichia coli isolated from the coast of Lima, Peru
Marcos Alejandro Sulca López*, Débora Elizabeth Alvarado Iparraguirre
Universidad Nacional Mayor de San Marcos, Facultad de Ciencias Biológicas. Av. Venezuela s/n cuadra 34. Ciudad Universitaria. Lima 1-Perú.
*Autor para correspondencia
Email Marcos Sulca: ubiol67@gmail.com
Email Débora Alvarado: dalvaradoi@unmsm.edu.pe
Resumen
El objetivo de este estudio fue investigar la resistencia al mercurio y los antibióticos, y la transferencia de resistencia al mercurio por plásmidos conjugativos en 55 cepas de Escherichia coli aisladas de aguas superficiales del litoral de Lima, Perú. Se determinó la Concentración Mínima Inhibitoria (CMI) a diversos antibióticos y al mercurio en las cepas aisladas. Para confirmar la resistencia plasmídica al mercurio, se realizó la curación de estos con dodecil sulfato de sodio (SDS) 10%. El ensayo de transferencia de plásmidos por conjugación se realizó usando la cepa receptora E. coli DH5α sólo con las cepas que mostraron sensibilidad al mercurio después de la curación. La extracción de los plásmidos de resistencia fue realizada sólo en las cepas transconjugantes resistentes al mercurio. 41 (74.5%) cepas fueron resistentes al mercurio (HgR), presentando CMIs entre 30 μM (8.25 ppm) y 300 μM (82.5 ppm), de estás, 33 fueron HgR mediante plásmidos y de este último grupo, 14 fueron también resistentes a antibióticos. Sólo 6 cepas poseían plásmidos conjugativos con resistencia al mercurio, mostrando una frecuencia de transconjugación entre 9.41x10-4 y 4.76x10-2%. La alta prevalencia de cepas de E. coli HgR aisladas de la costa limeña podría ser un problema de salud pública y ambiental. En este sentido, los plásmidos congugativos pueden contribuir con la diseminación de mercurio y/o resistencia a antibióticos entre comunidades bacterianas en ambientes marinos.
Palabras clave: Escherichia coli; resistencia al mercurio; resistencia a los antibióticos; conjugación de plásmidos; Corriente de Humboldt.
Abstract
The Lima coast is highly affected by anthropogenic effluents from wastewater from contaminated urban rivers that flow into the coast. The objective of this study was to investigate the resistance to mercury and antibiotics, and the transfer of resistance to mercury by conjugative plasmids in 55 strains of Escherichia coli isolated of surface seawater from coastal Lima, Peru. The Minimum Inhibitory Concentration (MIC) was determined for various antibiotics and for mercury in the isolated strains. To confirm the plasmid resistance to mercury, the curing was carried out with 10% sodium dodecyl sulfate (SDS). The plasmid transfer assay by conjugation was performed using the E. coli DH5α as recipient strain only with the strains that showed sensitivity to mercury after curing. The extraction of the resistance plasmids was carried out only in the transconjugant strains resistant to mercury. 41 (74.5%) strains were resistant to mercury (HgR), with MICs between 30 μM (8.25 ppm) and 300 μM (82.5 ppm), of these, 33 were HgR by plasmids and of this last group, 14 were also resistant to antibiotics. Only 6 strains had conjugative plasmids with mercury resistance, showing a transconjugation frequency between 9.41x10-4 and 4.76x10-2%. The high prevalence of HgR in E. coli strains isolated from the coast of Lima could be a public and environmental health problem. In this sense, congugative plasmids can contribute to the spread of mercury and/or resistance to antibiotics among bacterial communities in marine environments.
Key words: Escherichia coli; resistance to mercury; resistance to antibiotics; conjugation of plasmids; Humboldt Current System.
Introducción
Entre los metales pesados, el mercurio no tiene ninguna función biológica beneficiosa y es extremadamente tóxico para todas las formas de vida (UNEP 2013, Govind & Madhuri 2014). Sin embargo, debido a las actividades antropogénicas como la extracción artesanal del mercurio, movilización y liberación de residuos conteniendo mercurio, se reporta la alta concentración de este metal en el medio ambiente y en la biota (plantas, animales y otros organismos vivos) (UNEP 2002).
El mercurio afecta a todos los seres vivos, incluido también a los microorganismos que generan resistencia para tolerar el efecto tóxico de este metal. En las bacterias, la resistencia al mercurio asociada con la resistencia a antibióticos está ampliamente distribuida, tanto en aislados clínicos y ambientales (Hobman et al. 2002, Cardonha et al. 2005), siendo los transposones y plásmidos conjugativos los elementos genéticos móviles que frecuentemente confieren resistencias a diversos antimicrobianos (Gonzáles et al. 2004, Moraga et al. 2003). Por tanto, el medio ambiente contaminado con antimicrobianos y metales pesados podría condicionar para la aparición de nuevas formas de resistencias en los procariotas. El litoral de Lima recibe efluentes sin tratamiento del río Rímac y Chillón, contaminados por aguas residuales de origen doméstico e industrial, (Bedregal et al. 2010, Reyes 2012) lo cual podría propiciar la aparición de resistencias a estos contaminantes.
Actualmente, en el Perú existen pocos estudios sobre el impacto de la contaminación del mercurio en ambientes marinos, así como el factor de riesgo para el desarrollo de resistencia a metales pesados y antibióticos en la comunidad bacteriana. Por tanto, el objetivo de este estudio fue investigar la resistencia al mercurio iónico y a antibióticos en cepas de Escherichia coli aisladas de ambientes marinos en la costa central del Perú y la asociación de la resistencia al mercurio con plásmidos conjugativos.
Material y métodos
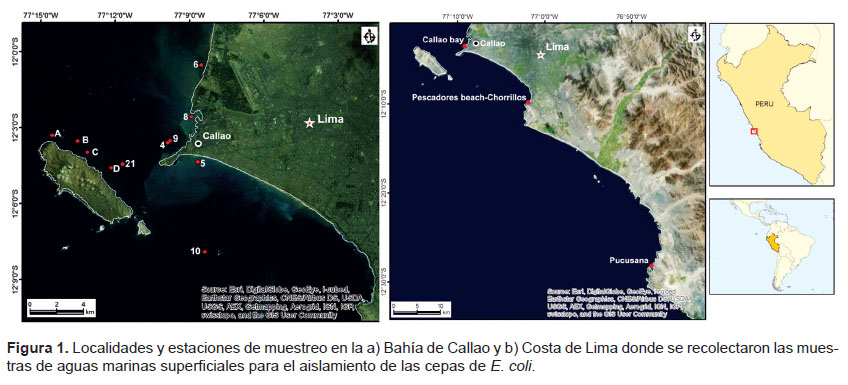
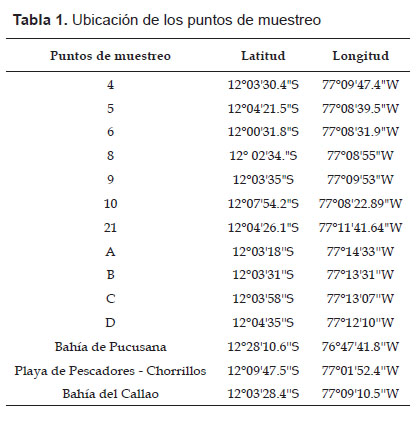
Cepas bacterianas.- Las cepas de E. coli (n=55) fueron aisladas de muestras de agua superficiales colectadas en los años 1999 y 2000 provenientes de las bahías de Pucusana, Miraflores y El Callao (Lima, Perú). Los puntos de muestreo y la ubicación de estos son mostrados en la Figura 1 y Tabla 1, respectivamente. Estas muestras fueron procesadas siguiendo los métodos estándar para el examen de aguas y aguas residuales (http://www.standardmethods.org), y los aislados almacenados en el Laboratorio de Microbiología Molecular y Biotecnología de la Facultad de Ciencias Biológicas de la Universidad Nacional Mayor de San Marcos. Para comenzar el estudio, las cepas fueron reactivadas en tubos con 10 mL de caldo Mueller-Hinton estéril, se verificó la pureza de las mismas sembrando en placas con Agar Mac Conkey, las condiciones de cultivo para ambos procedimientos fueron, incubación por 18-24 horas a temperatura ambiente. Las colonias lactosa positivas, característica de E. coli, fueron confirmadas mediante el uso de pruebas bioquímicas estándar (Farmer et al. 1985).
Determinación de la resistencia al mercurio y pruebas de susceptibilidad a los antimicrobianos.- La concentración mínima inhibitoria (CMI) al mercurio iónico (Hg+2) fue determinado por el método de macrodilución en caldo LB suplementado con cloruro de mercurio (HgCl2), cuya concentración varió entre 30 µM (≈ 8,25 ppm) a 400 µM (≈110 ppm). La incubación fue a temperatura ambiente por 24-48 horas, considerando cepas resistentes al mercurio (HgR) cuando estas mostraron crecimiento en caldo LB a partir de 30µM HgCl2, esto debido a que ensayos previos realizados con las cepas de E. coli ATCC 25922 y E. coli DH5, usadas como controles negativos sensibles al HgCl2, estas nos mostraron crecimiento cuando el caldo LB fue suplementado con 25µM HgCl2.
Adicionalmente, fueron realizados los perfiles de susceptibilidad a los antibióticos: cloranfenicol (30 µg), norfloxacina (10 µg), amikacina (30 µg), kanamicina (30 µg), ampicilina (10 µg); sulperazona (30 µg), tetraciclina (30 µg), aztreonam (30 µg), ceftazidima (30 µg), gentamicina (10 µg); amoxicilina (25 µg), sulfametoxazol-trimetoprima (25 µg), ácido nalidíxico (30 µg) y ciprofloxacina (5 µg) (Oxoid, Britain Greath). Estos perfiles se determinaron mediante el método de difusión en disco en Agar Mueller-Hinton, método que está basado por la presencia o ausencia de una zona de inhibición de crecimiento microbiano que es medido en milímetros. Los resultados de sensibilidad, sensibilidad intermedia y resistencia de los microorganismos a algún antimicrobiano está basado en la correlación entre el diámetro de la zona de inhibición con la concentración mínima inhibitoria de cada antibiótico para cada antimicrobiano. (NCCLS, 2000). La cepa E. coli ATCC 25922 se usó como control negativo.
Curación de los plásmidos.- Se utilizó el surfactante dodecil sulfato de sodio (SDS) 10% p/v en Caldo LB (Gerhardt et al. 1994). Se tomaron alícuotas de 20 µL de las cepas HgR cultivadas en caldo LB con 30 µM HgCl2 por 24 horas, cultivándolos en tubos con 2 mL del caldo LB libre de mercurio pero suministrado con SDS por 24-48 horas a temperatura ambiente; después se tomó 20 µL del cultivo curado y se sembró en caldos suministrado con 30 µM HgCl2, la inhibición de crecimiento confirma el origen de la resistencia al mercurio por plásmidos; paralelamente se sembró también en caldo LB sin mercurio, para comprobar la viabilidad del cultivo en SDS.
Ensayos de conjugación.- Los ensayos de conjugación se realizaron con la cepa E. coli DH5α como cepa receptora de plásmidos con las cepas de E. coli HgR que mostraron susceptibilidad al mercurio después del ensayo de curación del plásmido (Gerhardt et al. 1994). La frecuencia de transferencia del plásmido se calculó tomando el número de transconjugantes y cepas donadoras, siguiendo la fórmula:
Frecuencia de transconjugantes = # transconjugantes/mL / # donadoras/mL x100
Los plásmidos se extrajeron de las cepas transconjugantes de E. coli DH5α HgR mediante el método de lisis alcalina (Sambrook & Russell 2001) y sus pesos moleculares se determinaron comparando la movilidad relativa de los plásmidos con el marcador Lambda DNA/HindIII (Promega, Madison, WI).
Resultados
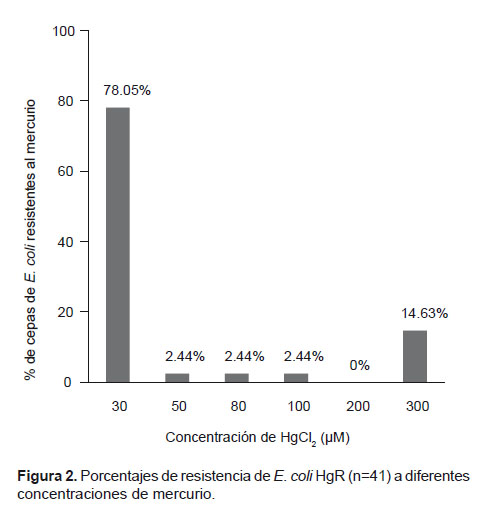
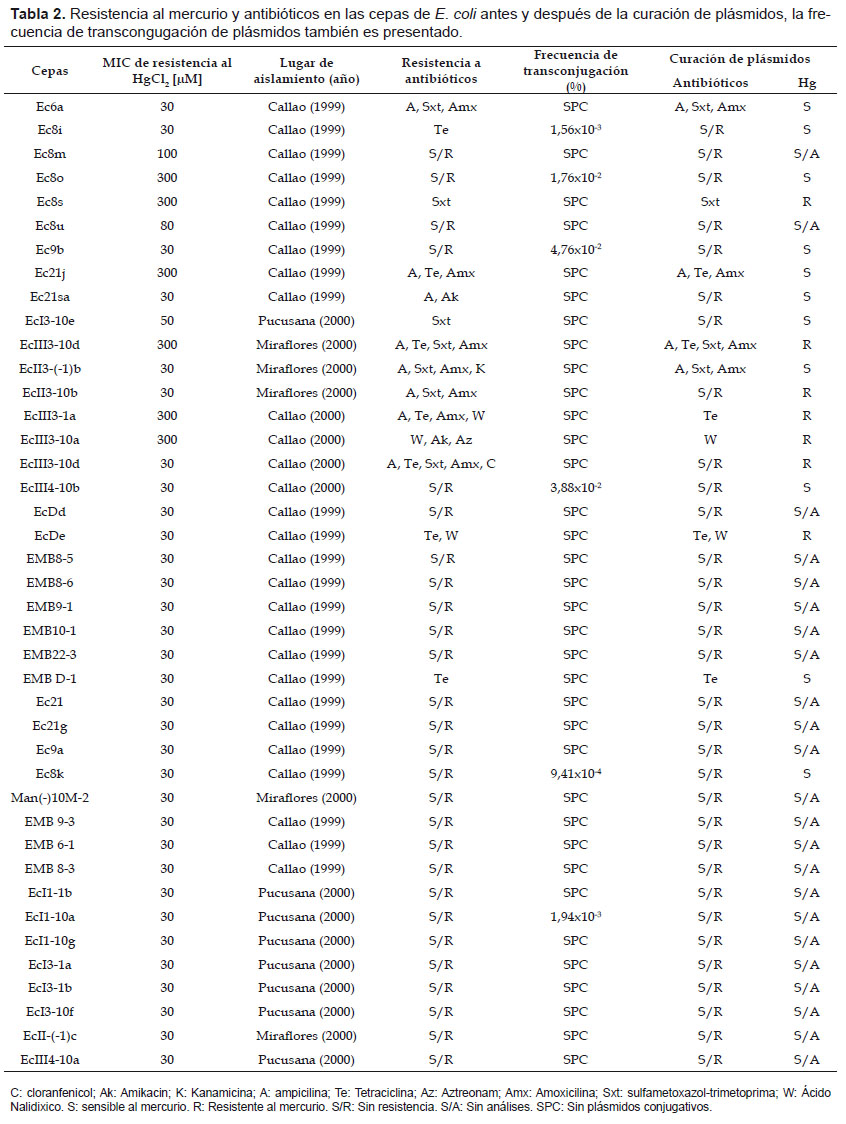
Los perfiles de resistencia al mercurio y a los antibióticos en las cepas de E. coli son mostrados en la Tabla 2, de las 55 cepas, 41 (74.54%) exhibieron resistencia al cloruro de mercurio (MIC90 >300 µM). En la Fig. 2 es mostrado los diversos porcentajes de resistencia de las cepas de E. coli al cloruro de mercurio.
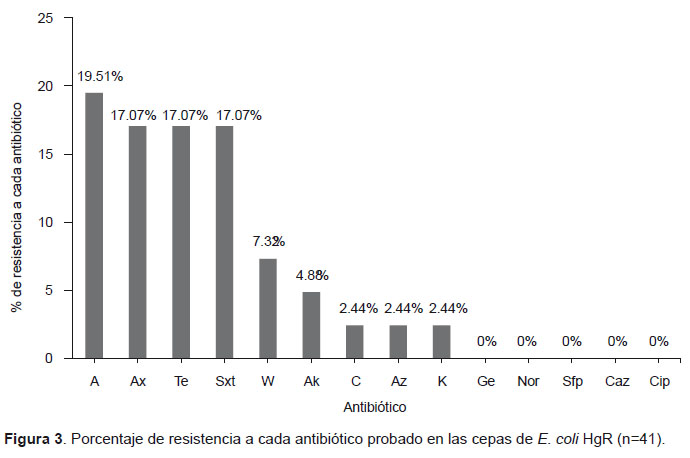
De todas las cepas resistentes al mercurio, 14 (34.2%) mostraron resistencia adicional a diversos antibióticos. Se observó diferentes fenotipos de resistencia a múltiples antibióticos (≥ 3 antibióticos diferentes) en ocho aislados (Tabla 2). Los patrones de resistencia a los antibióticos en las cepas de E. coli HgR fueron: ampicilina (19.5%), amoxicilina (17.1%), tetraciclina (17.1%), sulfametoxazol-trimetoprima (17.1%), siendo estos los más prevalentes. Se observó baja resistencia al ácido nalidíxico (7,32%), amikacina (4,88%), cloranfenicol (2,44%), aztreonam (2,44%) y kanamicina (2,44%). Estos resultados se muestran en la Fig. 3.
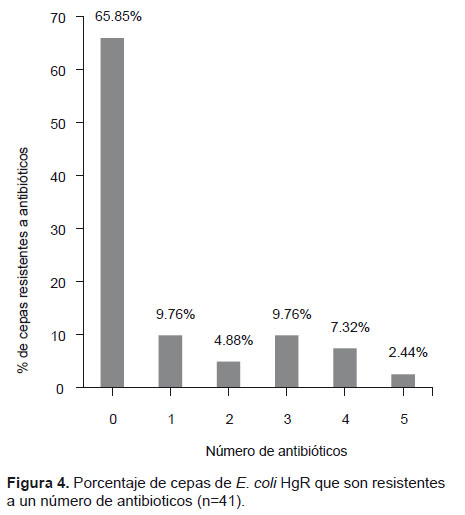
En relación al número de antibióticos que cada cepa de E. coli HgR es resistente, 2.44% fueron resistentes a cinco antibióticos, 7.32% a cuatro antibióticos, 9.76% a tres antibióticos, 4.88% a dos antibióticos, 9.76% a un antibiótico y 65.85% fueron sensibles a todos los antibióticos probados (Fig. 4).
De todas las cepas resistentes al mercurio (n=41), 34 (83%) fueron sensibles al mercurio después del ensayo de curación de los plásmidos, de estas, cinco cepas mostraron una ausencia total de resistencia a los antibióticos (Ver Tabla 2).
El resultado de los ensayos de conjugación mostró que seis cepas transfirieron algún plásmido de resistencia al mercurio, siendo que la frecuencia de transconjugación varió entre 4.76x10-2 y 9.41x10-4% (Tabla 2). Cabe resaltar que la conjugación se logró en cepas de E. coli HgR que tenían resistencia sólo al mercurio, con la excepción de una cepa de E. coli resistente a tetraciclina llamada Ec8i. Las cepas transconjugantes mostraron plásmidos con un peso molecular entre 1.3 y 24 Kpb.
Discusión
Los microorganismos presentes en aguas marinas impactadas por la actividad humana, están expuestas a diversas presiones selectivas de origen ambiental (temperatura del mar, pH, salinidad, oxígeno disuelto y otros parámetros variables) y compuestos tóxicos; desarrollando varios tipos de resistencias. Este es el primer estudio de E. coli aislado de ambientes marinos peruanos que tienen resistencia a diferentes antibióticos y mercurio, y además estas resistencias pueden ser transmitidas a cepas sensibles.
Varias cepas de E. coli fueron resistentes al mercurio, mostrando también resistencia a los antibióticos. Los mayores niveles de resistencia al mercurio se encontraron en aislamientos de la bahía de Callao, esto debido probablemente a la proximidad a la desembocadura del río Rímac y Chillón que transportan aguas residuales de origen urbano e industrial, principalmente de las mineras. Las cepas resistentes al mercurio mostraron resistencia a ampicilina, tetraciclina, amoxicilina y sulfametoxazol-trimetoprim; Cardonha et al. (2005) reportaron en cepas de E. coli aisladas de muestras de agua de alcantarilla y de mar, resistencia al mercurio asociada con la resistencia a la ampicilina y a sulfatomexazol-trimetoprina; Ferreira et al. (2007) aislaron cepas de Escherichia spp. Provenientes de aguas residuales no tratadas y tratadas, encontrando mayor prevalencia de resistencia a antibióticos como amoxicilina y tetraciclina, y también resistencia entre el mercurio y los antibióticos sulfametoxazol/trimetoprima y tetraciclina. Poiată et al. (2000), describieron resistencias tanto al mercurio y antibióticos como ampicilina y tetraciclina en aislados de origen clínico, concluyendo también que la resistencia al mercurio se relaciona con la resistencia a antibióticos de uso clínico. En relación con las bacterias de ambientes acuáticos, el uso generalizado de antibióticos en todo el mundo ha aumentado, aumentando también la resistencia a los antibióticos en estos microorganismos (Miller et al. 2009).
De todas las cepas resistentes al mercurio, sólo seis cepas presentaban plásmidos conjugativos, lo que demostraría que la resistencia al mercurio está dada principalmente por plásmidos no conjugativos; en las cepas Ec8i y EcI3-10e se observó que la resistencia a ambos inhibidores microbianos no está presente en el mismo plásmido conjugativo. Huddleston et al. (2006), también reportaron, en Aeromonas spp. resistentes a antibióticos y a mercurio, plásmidos conjugativos que codifican resistencia sólo al mercurio pero no a los antibióticos. Previamente, Seol et al. (2002), encontraron un plásmido de resistencia no conjugativo de 18.3 Kpb, con genes de resistencia para tetraciclina, estreptomicina y sulfisomidina en aislados de Shigella sonnei de origen clínico.
En cepas de origen ambiental, muchas especies bacterianas pertenecientes a los géneros Bacillus, Pseudomonas, Staphylococcus, Brevibacterium provenientes de diversos ambientes contaminados desarrollan resistencia al mercurio (≥ 10 ppm) y a otros metales pesados (De & Ramaiah 2007; Dash et al. 2014). En este estudio, 65.85% de las cepas resistentes al mercurio no han mostrado resistencia a algunos antibióticos pero mostraron resistencia a la concentración de mercurio más baja utilizada en este estudio de 30µM HgCl2, valor similar a lo reportado por Pike et al. (2002) y Ready et al. (2007) que fue de 32µM.
Seis cepas mostraron un nivel de resistencia diez veces mayor, de 300 µM HgCl2. Ball et al. (2007) reportaron resistencia al mercurio entre 25 y 300 µM en bacterias heterotróficas aisladas de estanques de relaves contaminados con mercurio. Ready et al. (2007) encontraron resistencia al mercurio entre 32 y 128 µM en bacterias expuestas a la amalgama. Zhang et al. (2012) describieron en Pseudomonas putida cepa SP1 aislado de muestras de agua de mar, capaz de tolerar hasta 280 µM, exhibiendo resistencia a otros metales pesados y diversos antibióticos. Paul (1991) reportó la presencia de plásmidos no conjugativos en bacterias de origen marino; en nuestro estudio pocas cepas tienen la capacidad de transferir resistencia al mercurio por conjugación. Los resultados mostraron que el porcentaje de transconjugación es alta comparado con los resultados obtenidos por Gupta & Ali (2004), quienes reportaron valores de 2x10-8 % y 4.6x10-8 %. La transferencia de plásmidos no fue afectada por el tamaño de estos, ya que los pesos moleculares varían entre 1.3 y 24 Kpb en las cepas transconjugantes; Gupta & Ali (2004) reportaron también plásmidos de 24 Kpb aproximadamente, Shoeb & Badar (2012) encontraron plásmidos entre 1-10 Kpb en Enterobacter cloacae aislados de agua de mar, con capacidad de conjugar estos plásmidos con genes de resistencia a metales pesados y antibióticos en cepas de E. coli receptoras.
Bacterias de estuarios y ambientes marinos continúan adquiriendo la resistencia al mercurio por transferencia horizontal de genes de resistencias con microorganismos de la misma especie como también de otras especies microbianas; siendo de interés la transferencia de diferentes tipos de resistencia con especies de bacterias marinas potencialmente patógenas para humanos (Dahlberg et al. 1998, Moraga 2003, Ramaiah & De 2003). Paul et al. (1992) demostraron en E. coli que la transferencia de plásmidos no conjugativos es dependiente del contacto celular, y en especies de Vibrio marinas la transformación natural por plásmidos no conjugativos pueden diseminarse a través de comunidades microbianas heterogéneas. Nuestros resultados mostraron la presencia de resistencia a antibióticos o metales pesados, asociados principalmente por plásmidos.
Sin embargo, debemos tener en cuenta que las cepas de E. coli usadas en el estudio no son bacterias nativas de ambientes marinos. El origen de estas cepas resistentes proviene de la alta concentración de coliformes fecales y metales pesados, que se encuentran en la desembocadura de los ríos Rímac y Chillón donde llegan efluentes de la ciudad de Lima. Estos efluentes de aguas residuales urbanas no son tratadas por lo que esto propiciaría el aumento de las cepas resistentes de E. coli en el litoral de Lima. Este escenario ya ha sido reportado por Kümmerer (2004), quien informó la presencia de antibióticos como penicilinas, tetraciclina, macrólidos, quinolonas, sulfonamidas y bacterias resistentes a estos antibióticos en efluentes hospitalarios, aguas residuales municipales, efluentes de plantas de tratamiento de aguas residuales y aguas superficiales. También, Reinthaler et al. (2003) aislaron cepas de E. coli de plantas de tratamiento de aguas residuales resistentes a varios antibióticos como penicilinas, cefalosporinas, quinolonas, tetraciclina y trimetoprima/sulfametoxazol; en comparación, en nuestro estudio las cepas fueron resistentes a la ampicilina, tetraciclina, trimetoprima/sulfametoxazol y amoxicilina principalmente.
Se ha encontrado que, cuando estas aguas no tratadas, con altas concentraciones de coliformes fecales resistentes al mercurio y antibióticos llegan a diversas playas y mares, la condición de resistentes continuaría permaneciendo en la bacteria (Nascimento et al. 1999). Eso podría ser porque los plásmidos de resistencia presentes en bacterias de origen ambiental muestran estabilidad incluso en ausencia de un agente selectivo, refutando el argumento de que los plásmidos se pierden durante generaciones sucesivas en ausencia de este agente, aumentando la probabilidad de producir una mayor propagación de genes de resistencia y/o determinantes de virulencia en bacterias nativas en un ecosistema dado (De Souza et al. 2006).
Conclusiones
Estas cepas resistentes presentes en aguas marinas de uso público representarían un riesgo para la salud pública, tanto cuando las personas entran en contacto con estas aguas contaminadas, como por el consumo de productos hidrobiológicos que contengan estos microorganismos. Además, nuestro estudio señala la importancia de procesos biológicos que conviertan al litoral de Lima en un reservorio de genes de resistencia al mercurio y a antibióticos, y que deben ser tomados en cuenta para el manejo de las aguas residuales de la ciudad.
Agradecimientos
A la Bióloga Mary Zuleyka Vicente Ruiz, a la Dra. Juana María Coha y al equipo de docentes y colaboradores del Laboratorio de Microbiología Molecular y Biotecnología de la Universidad Nacional Mayor de San Marcos. Esta investigación fue financiada en parte con el Proyecto CON CON N° 071001191 CSI-VRI-UNMSM.
Literatura citada
Ball M.M., P. Carrero, D. Castro, et al. 2007. Mercury resistance in bacterial strains isolated from tailing ponds in a gold mining area near El Callao (Bolívar State, Venezuela). Current Microbiol. 54(2): 149–154. doi: http://dx.doi.org/10.1007/s00284-006-0347-4 [ Links ]
Bedregal P., P. Mendoza, M. Ubillús et al. 2010. Evaluación de las aguas del río Rímac en Lima, Perú, utilizando el Índice de Calidad de Agua (ICA). Informe Científico Tecnológico 10:13-19. http://dspace.ipen.gob.pe/handle/ipen/623 [ Links ]
Cardonha A.M., R.H. Vieira, G. Peirano, et al. 2005. Resistance to antibiotics and heavy metals from Escherichia coli isolated from sea water and pluvial galleries. Acta Cir Bras. 20(Suppl. 1): 253-256. [ Links ]
Dahlberg C., M. Bergström, M. Andreasen, et al. 1998. Interspecies bacterial conjugation by plasmids from marine environments visualized by gfp expression. Molecular Biology and Evolution 15(4):385–390. https://doi.org/10.1093/oxfordjournals.molbev.a025935 [ Links ]
Dash H., N. Mangwani, S. Das. 2014. Characterization and potential application in mercury bioremediation of highly mercury-resistant marine bacterium Bacillus thuringiensis PW-05. Environ Sci Pollut Res. 21(4): 2642–2653. https://doi.org/10.1007/s11356-013-2206-8 [ Links ]
De J., & N. Ramaiah. 2007. Characterization of marine bacteria highly resistant to mercury exhibiting multiple resistances to toxic chemicals. Ecol Indicators. 7(3): 511–520. https://doi.org/10.1016/j.ecolind.2006.05.002 [ Links ]
De Souza M.J., S. Nair, P.A. Loka Bharathi, et al. 2006. Metal and antibiotic-resistance in psychrotrophic bacteria from Antarctic marine waters. Ecotoxicology 15(4): 379-384. https://doi.org/10.1007/s10646-006-0068-2 [ Links ]
Farmer J.J., B.R. Davis, F.W. Hickman-Brenner, et al. 1985. Biochemical identification of new species and biogroups of Enterobacteriaceae isolated from clinical specimens. Journal of Clinical Microbiology 21(1): 46–76. [ Links ]
Ferreira da Silva M., I. Vaz-Moreira, M. Gonzalez-Pajuelo, et al. 2007. Antimicrobial resistance patterns in Enterobacteriaceae isolated from an urban wastewater treatment plant. FEMS Microbiology Ecology 60(1): 166-176. https://doi.org/10.1111/j.1574-6941.2006.00268.x [ Links ]
Gerhardt P., R.G.E. Murray, W.A. Wood et al. 1994. Methods for General and Molecular Bacteriology. 2nd Edn., American Society for Microbiology, Washington, DC., ISBN: 9781555810481, pp: 343. [ Links ]
González G., S. Mella, R. Zemelman, et al. 2004. Integrones y cassettes genéticos de resistencia: estructura y rol frente a los antibacterianos. Revista médica de Chile 132(5): 619-626. https://doi.org/10.4067/S0034-98872004000500013 [ Links ]
Govind, P., S. Madhuri. 2014. Heavy Metals Causing Toxicity in Animals and Fishes. Research Journal of Animal, Veterinary and Fishery Sciences 2(2): 17-23. [ Links ]
Gupta N., & A. Ali. 2004. Mercury volatilization by R Factor Systems in Escherichia coli isolated from aquatic environments of India. Current Microbiology 48(2): 88–96. https://doi.org/10.1007/s00284-003-4054-0 [ Links ]
Hobman J.L., A.M. Essa, N.L. Brown. 2002. Mercury resistance (mer) operons in enterobacteria. Biochemical Society Transactions 30(4): 719-722. https://doi.org/10.1042/bst0300719 [ Links ]
Huddleston J.R., J.C. Zak, R.M. Jeter. 2006. Antimicrobial susceptibilities of Aeromonas spp. isolated from environmental sources. Applied and Environmental Microbiology 72(11): 7036-7042. https://doi.org/10.1128/AEM.00774-06 [ Links ]
Kümmerer, K. 2004. Resistance in the environment. Journal of Antimicrobial Chemotherapy 54(2): 311–320. https://doi.org/10.1093/jac/dkh325 [ Links ]
Miller R.V., K. Gammon, M.J. Day. 2009. Antibiotic resistance among bacteria isolated from seawater and penguin fecal samples collected near Palmer Station, Antarctica. Canadian Journal of Microbiology 55(1): 37-45. https://doi.org/10.1139/W08-119 [ Links ]
Moraga R., C. Merino, M. Mondaca. 2003. Resistencia a metales pesados en bacterias aisladas de la bahía de Iquique. Investigaciones Marinas 31(1): 91-95. https://doi.org/10.4067/S0717-71782003000100010 [ Links ]
Nascimento A.M., C.E. Campos, E.P. Campos, et al. 1999. Re-evaluation of antibiotic and mercury resistance in Escherichia coli populations isolated in 1978 from Amazonian rubber tree tappers and Indians. Research in Microbiology 150(6): 407-411. https://doi.org/10.1016/S0923-2508(99)80076-X [ Links ]
National Committee for Clinical Laboratory Standards (NCCLS). 2000. Performance standards for antimicrobial disk Susceptibility Test: Approved Standard. Seventh Edition M2-A7. [ Links ]
Paul J., M. Frischer, J. Thurmond. 1991. Gene transfer in marine water column and sediment microcosms by natural plasmid transformation. Applied and Environmental Microbiology 57(5): 1509-1515. [ Links ]
Paul J.H., J.M. Thurmond, M.E. Frischer, et al. 1992. Intergeneric natural plasmid transformation between E. coli and a marine Vibrio species. Molecular Ecology 1(1): 37-46. https://doi.org/10.1111/j.1365-294X.1992.tb00153.x [ Links ]
Pike R., V. Lucas, P. Stapleton, et al. 2002. Prevalence and antibiotic resistance profile of mercury-resistant oral bacteria from children with and without mercury amalgam fillings. Journal of Antimicrobial Chemotherapy 49(5): 777–783. https://doi.org/10.1093/jac/dkf019 [ Links ]
Poiată A., I. Bădicuţ, M. Indreş, et al. 2000. Mercury resistance among clinical isolates of Escherichia coli. Roumanian archives of microbiology and immunology 59(1-2): 71-79. [ Links ]
Ramaiah N., & J. De. 2003. Inusual rise in mercury-resistant bacteria in coastal environs. Microbial Ecology 45(4): 444-454. https://doi.org/10.1007/s00248-001-1068-7 [ Links ]
Ready D., J. Pratten, N. Mordan, et al. 2007. The effect of amalgam exposure on mercury- and antibiotic-resistant bacteria. International Journal of Antimicrobial Agents 30(1): 34-39. https://doi.org/10.1016/j.ijantimicag.2007.02.009 [ Links ]
Reinthaler F.F., J. Posch, G. Feierl, et al. 2003. Antibiotic resistance of E. coli in sewage and sludge. Water Research 37(8): 1685–1690. https://doi.org/10.1016/S0043-1354(02)00569-9 [ Links ]
Reyes C.M. 2012. Estudio de la contaminación de las aguas del río Chillón. Facultad de Ingeniería Geológica, Minera y Metalúrgica. Tesis. Magíster en Ciencias, mención en minería y medio ambiente. Universidad Nacional de Ingeniería, Lima Perú. http://cybertesis.uni.edu.pe/handle/uni/1082 [ Links ]
Sambrook J. & Russell D.W., 2001. Molecular cloning - a laboratory manual, 3rd ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor. [ Links ]
Seol S., Y. Jeong, H. Kang, et al. 2002. Epidemiologic analysis of Shigella sonnei isolates by using antimicrobial resistance gene probes. Journal of Bacteriology and Virology 32(4): 347-354. [ Links ]
Shoeb E., U. Badar, J. 2012. Horizontal gene transfer of stress resistance genes through plasmid transport. World Journal of Microbiology and Biotechnology 28(3): 1021-1025. https://doi.org/10.1007/s11274-011-0900-6 [ Links ]
United Nations Environment Programme-Chemicals (UNEP). Global Mercury Assessment. Geneva: 2002. [ Links ]
United Nations Environment Programme (UNEP). Global Mercury Assessment 2013: Sources, Emissions, Releases and Environmental Transport, UNEP Chemicals Branch, Geneva, Switzerland. 2013. [ Links ]
Zhang W., L. Chen, D. Liu. 2012. Characterization of a marine-isolated mercury-resistant Pseudomonas putida strain SP1 and its potential application in marine mercury reduction. Applied Microbiology and Biotechnology 93(3): 1305-1314. https://doi.org/10.1007/s00253-011-3454-5 [ Links ]
Información sobre los autores:
MASL, DEAI: realizaron el diseño experimental; los experimentos; analizaron los datos; redactaron el manuscrito; revisaron y aprobaron el manuscrito.
Los autores no incurren en conflictos de intereses.
Fuentes de financiamiento: El presente trabajo se realizó gracias al financiamiento del Proyecto N° 071001191 CSI-VRI-UNMSM.
Presentado: 15/02/2018
Aceptado: 29/07/2018
Publicado online: 07/12/2018