Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.26 no.1 Lima ene./mar. 2019
http://dx.doi.org/10.15381/rpb.v26i1.15911
TRABAJOS ORIGINALES
Identificación bioinformática de Polimorfismos de Nucleótido Simple (PNSs) en genes candidatos para las características de la fibra en alpacas (Vicugna pacos)
Bioinformatic identification of Single Nucleotide Polymorphisms (SNPs) in candidate genes for fiber characteristics in alpacas (Vicugna pacos)
Alvaro Gonzalo Fernández Suárez* 1, Gustavo Augusto Gutiérrez Reynoso1 ORCID: 0000-0002-1896-0048, Federico Abel Ponce de León Bravo2 ORCID: 0000-0001-8645-553X
1Universidad Nacional Agraria La Molina, Perú.
2University of Minnesota, United States.
Resumen
El objetivo fue identificar y predecir la ubicación de polimorfismos de nucleótido simple (PNSs) en genes relacionados al crecimiento de la fibra. Se realizó el estudio con un total de 31 genes de queratina (KRT9, KRT12, KRT13, KRT14, KRT16, KRT18, KRT20, KRT25, KRT1, KRT3, KRT5, KRT6a, KRT6b, KRT6c, KRT7, KRT8, KRT71, KRT80, KRT31, KRT32, KIRT40, KRT81, KRT82, KRT10, KRT15, KRT17, KRT19, KRT2, KRT4, KRT79 y KRT83) asociados con las características de lana, fibra y pelo en ovinos, cabras y humanos respectivamente, cuyas secuencias fueron encontradas en la base de datos del National Center for Biotechnology Information (NCBI). Mediante el uso de bases de datos y herramientas bioinformáticas como el Conserved Domains Database, Spling, y MegaBlast se logró ubicar secuencias únicas para cada gen. Estas secuencias fueron comparadas con los genomas de referencia Vicugna_pacos-2.0.2 y Vi_pacos_V1.0. Se identificaron 48 PNSs ubicados en las regiones intrónicas y exónicas de 22 genes. No se localizaron PNSs en o alrededor de los genes KRT10, KRT15, KRT17, KRT19, KRT2, KRT4, KRT6b, KRT6c y KRT79. El análisis comparativo entre las cuatro especies estudiadas permitió observar que los genes KRT81, KRT6b y KRT6c no están presentes en los genomas de referencia de alpaca, los genes KRT31, KRT14, KRT81, KRT83, KRT6b y KRT6c no están presentes en el genoma de referencia de ovino y los genes KRT31, KRT13, KRT81, KRT83, KRT6b y KRT6c no están presentes en el genoma de referencia de cabra.
Palabras clave: Alpaca; bioinformática; polimorfismos de nucleótido simple; queratina; haplotipo.
Abstract
The objective was to identify and predict the location of single nucleotide polymorphisms (SNPs) in genes related to fiber growth. The study was carried out with 31 keratin genes (KRT9, KRT12, KRT13, KRT14, KRT16, KRT18, KRT20, KRT25, KRT1, KRT3, KRT5, KRT6a, KRT6b, KRT6c, KRT7, KRT8, KRT71, KRT80, KRT31, KRT32, KIRT40, KRT81, KRT82, KRT10, KRT15, KRT17, KRT19, KRT2, KRT4, KRT79 y KRT83) associated with wool, fiber and hair characteristics in sheep, goat and human, respectively. These gene sequences were retrieved from the National Center for Biotechnology Information (NCBI) database. Using databases and bioinformatics tools such as the Conserved Domains database, Spling and Megablast, unique sequences for each gene were identified. These sequences were compared to the reference genomes: Vicugna_pacos-2.0.2 and Vi_pacos_V1.0 to identify single nucleotide polymorphisms (SNPs). In this manner, 48 SNPs were identified and localized in both intronic and exonic regions of 22 genes. We did not identify SNPs for KRT10, KRT15, KRT17, KRT19, KRT2, KRT4, KRT6b, KRT6c and KRT79. Comparative analysis among the four species studied allow to identify that sequences for KRT81, KRT6b and KRT6c genes are not present in the alpaca reference genomes. Similarly, genes KRT31, KRT14, KRT81, KRT83, KRT6b and KRT6c are not present in the ovine reference genome and, genes KRT31, KRT13, KRT81, KRT83, KRT6b and KRT6c are not present in the goat reference genome.
Keywords: Alpaca; bioinformatics; single nucleotide polymorphisms; keratin; haplotype.
Introducción
La alpaca es criada principalmente para la producción de fibra, siendo ésta apreciada por su finura, suavidad y la gran gama de tonalidades de colores naturales que se obtienen de ella. La industria textil considera que la fibra de alpaca es especial ya que los artículos que se confeccionan con ella son clasificados como artículos de lujo (Wang et al. 2003). Sin embargo, solo el 22% y 46% de la fibra ofertada en el mercado nacional son de calidad fina y semifina respectivamente (De los Ríos 2006), sugiriéndose que una de las vías para la mejora de su calidad seria a través del mejoramiento genético.
La principal proteína constituyente de las fibras de origen animal es la queratina, producida en los folículos pilosos de la piel (Zoccola 2014). Por tanto, sería importante identificar y conocer la función de los genes relacionados a la síntesis de queratina, así como la identificación de PNSs asociados a las características de calidad de fibra. Rogers (2004) menciona que el folículo piloso es una estructura muy conservada en los mamíferos. De la misma manera, Schweizer et al. (2006) indica que las secuencias de genes de queratina son conservadas en mamíferos. Por lo tanto, es de suponerse que el uso de las secuencias de genes ortólogos de ovinos, cabras y humanos facilitaran la identificación de secuencias similares en alpacas. De la misma manera la comparación de estas secuencias entre alpacas permitirá la identificación de marcadores moleculares genéticos.
El uso de marcadores moleculares como los PNSs puede ayudar a identificar regiones genómicas asociadas a las características de la fibra que pueden usarse para seleccionar animales superiores con los cuales se pueda lograr un mejoramiento genético acelerado y continuo.
Los PNSs son variaciones de un solo nucleótido en una secuencia, generalmente se originan de errores de replicación del ADN durante la división celular de las células germinales (Thavamanikumar et al. 2011). Según
Ramensky et al. (2002) los PNSs son abundantes y están distribuidos a lo largo del genoma. Sin embargo, para ser usados como marcadores genéticos se requiere que uno de sus alelos esté presente en al menos el 1% de la población (Brookes 1999). Además, Brookes (1999) señala que los PNSs se caracterizan por ser estables genéticamente y poseer una baja tasa de mutación.
Se han realizado estudios para la identificación de polimorfismos de nucleótido simple en alpacas. Así, Foppiano (2016) ubico 27 PNSs en genes de proteínas asociadas a la queratina (KRTAP1-2, KRTAP6-1, KRTAP9-2, KRTAP11-1 y KRTAP13-1). Por otro lado, Delgado de la Flor (2014) realizó la caracterización de marcadores PNSs en el gen de Tricohialina para estudiar su posible asociación con el fenotipo Suri y Huacaya, reportando la ubicación de 2 PNSs que se encuentran en desequilibrio de ligamiento con el gen Tricohialina, pero no encontró asociación de esos PNSs con el fenotipo Suri o con el Huacaya. Por otro lado, Guzmán (2011) reporto PNSs para el gen TLR4, sin embargo, no encontró asociación entre animales sanos y enfermos afectados por Pasteurella multocida. Diversos autores han reportado PNSs en la secuencia de genes
relacionados a la expresión del color del vellón en alpacas (Cransberg y Munyard, 2011; Chandramohan et al., 2013; Guridi et al., 2011; Chandramohan et al., 2015) También Mamani et al. (2017) con el objetivo de desarrollar una micromatriz semi-densa de PNSs identificó 50,686 PNSs en alpacas a partir del genotipado de un panel de radiación celular hibrido alpaca/hámster con una micromatriz de alta densidad de bovino.
A fin de realizar estudios de asociación entre marcadores moleculares y funciones biológicas específicas, el presente trabajo tuvo el objetivo de identificar y predecir la ubicación de PNSs en genes candidatos relacionados al crecimiento y características de la fibra de alpacas. Los PNSs identificados podrían ser incluidos en una futura micromatriz de PNSs de alpaca.
Material y métodos
Se estudiaron 31 genes de queratina reportados para ovinos, cabras y humanos entre los cuales tenemos 12 genes del epitelio de tipo I (KRT9, KRT10, KRT12, KRT13, KRT14, KRT15, KRT16, KRT17, KRT18, KRT19, KRT20 y KRT25), 13 genes del epitelio de tipo II (KRT1, KRT2, KRT3, KRT4, KRT5, KRT6a, KRT6b, KRT6c, KRT7, KRT8, KRT71, KRT79 y KRT80), 3 genes de la fibra de tipo I (KRT31, KRT32 y KRT40) y 3 genes de la fibra de tipo II (KRT81, KRT82 y KRT83) (Zhidong et al. 2006).
Se utilizó la base de datos del National Center for Biotechnology Information (NCBI, EEUU) para obtener las secuencias, número de exones y cromosoma al que pertenecen cada uno de estos genes. Las secuencias fueron obtenidas en formato FASTA. Una vez obtenida la secuencia para cada gen de interés se identificaron los exones que contenían secuencias que caracterizan los dominios de las superfamilias génicas (Conserved Domains Database, https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) para eliminar estas secuencias comunes de las comparaciones a seguir. El filtrado de estas secuencias comunes nos asegura el poder identificar PNSs en regiones específicas para cada gen.
Una vez eliminadas las regiones compartidas con otros genes de cada superfamilia, se delimitaron los intrones, exones y las regiones no traducidas mediante el uso del programa Splign (https://www.ncbi.nlm.nih.gov/sutils/splign/splign.cgi). Luego, utilizando el programa Mega Blast (https://blast.ncbi.nlm.nih.gov/Blast.cgi) se compararon e identificaron zonas compartidas de genes ortólogos de ovino, caprino y humano con el genoma de referencia VicPac 2.0.2. Para la identificación de PNSs se alinearon y compararon las secuencias de los genes de interés con el Vicpac 2.0.2 y el Vi.Pacos 1.0 que representan dos genomas distintos de referencia de alpaca.
Resultados
Se identificaron un total de 48 PNSs de los cuales 14 PNSs se encuentran distribuidos en 8 genes de queratina de epitelio de tipo I (KRT9, KRT12, KRT13, KRT14, KRT16, KRT18, KRT20, KRT25); 23 PNSs distribuidos en los 8 genes de queratina de epitelio de tipo II (KRT1, KRT3, KRT5, KRT6a, KRT7, KRT8, KRT71, KRT80); 4 PNSs en los genes de queratina de fibra de tipo I (KRT31, KRT32, KIRT40)y 7 PNSs en los genes de queratina de fibra de tipo II (KRT81, KRT82, KRT83). En el Anexo 1 se presentan las secuencias de los 48 PNSs identificados. La nomenclatura usada para designar cada PNS incluye el acrónimo (UNALM) de la institución que identifico el PNS seguido del acrónimo correspondiente al gene (KRT##) y la localización del PNS ya sea en un exón seguido del número del exón (E#) o un intrón seguido por número del intrón (I#) y por último siete dígitos numerales consecutivos que enumera cada PNS.
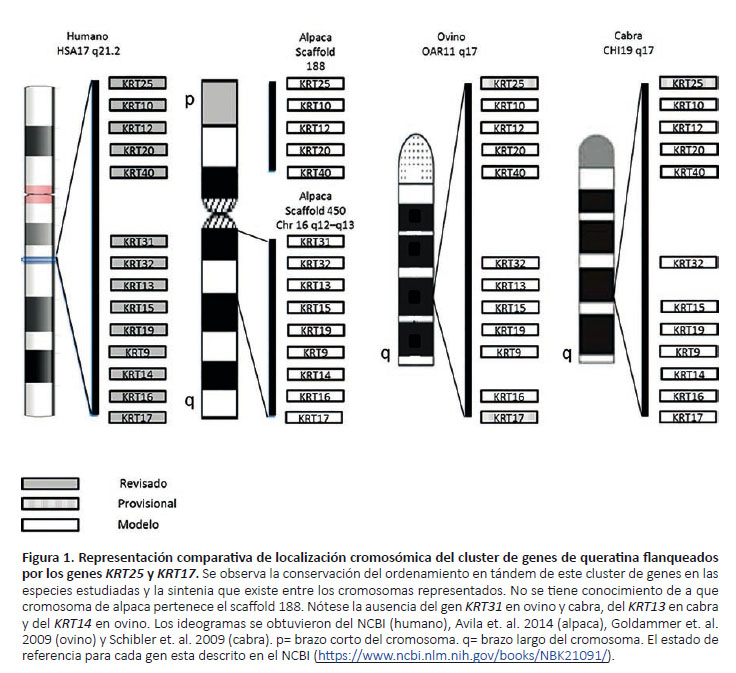
Los genes KRT6a, KRT6b y KRT6c se encuentran ubicados en tres loci en el genoma de referencia del humano,sin embargo, en el genoma de alpaca (VicPac 2.0.2) solose pudo ubicar una secuencia equivalente a la del gen KRT6a y la ausencia de secuencias equivalentes a los genes KRT6b y KRT6c. El análisis comparativo de secuenciasgénicas entre las cuatro especies estudiadas, indico quelos genes KRT25, KRT10, KRT12, KRT20, KRT40, KRT31, KRT32, KRT13, KRT15, KRT19, KRT9, KRT14, KRT16 y KRT17 están ubicados en el cromosoma 17, 11 y 19 de humano, ovino y cabra respectivamente; y en alpaca se encuentran en el scaffold 188 y 450 (Fig. 1). El scaffold 450ha sido asignado a VPA16 q12-q13 (Mendoza 2018). Los genes KRT31 y KRT14 no están anotados en ovinos y los genes KRT31 y KRT13 no están anotados en cabras. Los genes KRT80, KRT7, KRT81, KRT83, KRT82, KRT6a, KRT6b, KRT6c, KRT5, KRT71, KRT2, KRT1, KRT3, KRT4, KRT79, KRT8 y KRT18 están ubicados en el cromosoma 12 q13, 3q23, 5 q21 de humano, ovino y cabra respectivamente y elscaffold 36 en alpaca. Sin embargo, la orientación centró-mero – telómero de este cluster es inversa en ovinos y encaprinos (Fig. 2). Este estudio comparativo nos permitióobservar que los genes KRT81, KRT83, KRT6b y KRT6c no están anotados en ovinos y cabras y que el genoma referencial de alpaca (VicPac 2.0.2) no tiene secuencias y/oanotaciones para los genes KRT6b y KRT6c.
Los genes estudiados en los que se han identificado PNSs se encuentran en los scaffold 450, 188 y 36 del Vicpac 2.0.2 y las distancias entre ellos dentro de cada scaffold varían entre dieciséis mil a cien mil pares de bases. Se predicen posibles haplotipos que se encontrarían en la población de alpacas. En el scaffold 450, los posibles PNSs de los genes KRT31, KRT32, KRT13, KRT9, KRT14 y KRT16 formaron dos haplotipos, el 5´CTGAAG3´ y el 5´TCAGGA3´. De la misma manera, en el scaffold 188 los posibles PNSs de los genes KRT25, KRT12, KRT20 y KRT40 formaron dos haplotipos, 5´TAGT3´ y 5´CTAC3´. Así mismo, en el scaffold 36, los posibles PNSs de los genes KRT80, KRT7, KRT81, KRT83, KRT82 KRT6a, KRT5, KRT71, KRT1, KRT3, KRT8 y KRT18, formaron dos haplotipos, 5´CACTAAAGAGCC3´ y 5´TGTTAGGAGATA3´.
Discusión
Pariset et al. (2006) identificó un PNS en el exón 6 del gen KRT1 en ovinos. En el presente trabajo se identificaron en alpaca 3 PNSs en el gen KRT1, 2 en el intrón 2 y 1 en el exón 9. En ovinos Sulayman et al. (2018) identificó PNSs en los genes KRT27, KRT31, KRT36, KRT38, KRT81 y KRT85 y encontró que el gen KRT31 esta significativamente asociado al diámetro de lana, por ello, sería importante utilizar los dos PNSs reportados en este estudio para el gen KRT31 de la alpaca, porque también podrían estar asociados con el diámetro de fibra.
Schweizer et al. (2006) indica que la secuencia de los genes de queratina es conservada en los mamíferos. Pero, no todos los genes están presentes en las distintas especies. Los 31 genes de queratina utilizados para este trabajo fueron ubicados en su totalidad en humanos, los genes KRT6b, KRT6c, KRT31, KRT81 y KRT83 no están presentes y/o anotados para ovino y cabra en sus genomas referenciales en el NCBI y los genes KRT6a, KRT6b, KRT6c y KRT81 no están presentes y/o anotados en el Vicpac 2.0.2. Sin embargo, en el presente trabajo fueron ubicadas dos secuencias similares a los genes KRT6a y KRT81 en alpaca que aún no están anotadas en el VicPac 2.0.2. El gen KRT6a está ubicado entre el nucleótido 10536432 y el 10539642 del scaffold 36 y el gen KRT81 está ubicado entre el nucleótido 10411693 y el 10419541 del scaffold 36, por ello podemos concluir que estos genes existen en alpaca.
Hesse et al. (2004) desarrolló un análisis de cluster de genes de queratina en humanos y ratas identificando clusters de queratinas de tipo I en el cromosoma 17 y 10 respectivamente, con un total de 27 genes y 4 pseudogenes en humanos. También ubico un cluster de queratinas de tipo II en el cromosoma 12 y otro en el cromosoma 7 de humanos y ratas respectivamente, con un total de 26 genes y 5 pseudogenes en humanos. En el presente trabajo se identificó un cluster de 12 genes de queratinas en el scaffold 36, de 4 genes en el scaffold 188 y 6 genes en el scaffold 450. Mendoza (2018) ubicó mediante hibridación fluorescente in situ (FISH) que el gen KRT15 está ubicado en el cromosoma 16 q12-q13 en alpaca al igual queel scaffold 450, en consecuencia, los PNSs y genes ubicados en este scaffold se encuentran en el cromosoma 16 de alpaca (Fig. 1). Además, cabe la posibilidad de que el número de clusters y/o genes dentro de cluster sea mayor alo reportado en el presente estudio, debido a que solo se consideraron 31 genes de queratina en este análisis.
Desde que los PNSs predichos conforman haplotipos de PNSs, estos serán segregados conjuntamente, dada que la posibilidad de que exista una recombinación haplotipica es baja, la probabilidad de que dos individuos no emparentados posean el mismo haplotipo también es baja. Por lo cual el uso de estos haplotipos para estudios de asociación a características fenotípicas de la fibra es recomendable. En especial el haplotipo encontrado en el scaffold 450que incluye el gen KRT31 relacionado con finura de lana (Sulayman et al. 2018), en alpaca podría ser suficiente para identificar asociación entre estos genes y caracteres de la fibra desde que se analice una muestra adecuada de animales, facilitando de esta manera la implementación de selección asistida por marcadores moleculares.
De los 31 genes, en 9 genes (KRT10, KRT15, KRT17, KRT19, KRT2, KRT4, KRT6b, KRT6c y KRT79) no se pudo identificar ningún PNS exónico o intrónico. El gen KRT10 se encuentra en el scaffold 188 alineado en tandem con los genes KRT25, KRT12, KRT20 y KRT40 (Fig. 1). Los genes KRT15, KRT19 y KRT17 se encuentran en el scaffold 450 alineados en tandem con los genes KRT31, KRT32, KRT13, KRT9, KRT14 y KRT16 (Fig. 1). Los genes KRT2, KRT4 y KRT79 se encuentran en el scaffold 36 y están alineados en tandem con los genes KRT80, KRT7, KRT81, KRT83, KRT82, KRT6a, KRT5, KRT71, KRT1, KRT3, KRT8 y KRT18 (Fig. 2).
En conclusión, se logró ubicar un total de 48 PNSs, los cuales después de su validación in vitro podrán ser usados para realizar pruebas de asociación entre las características de la fibra y PNSs. Además, se pudo ratificar que los genes de queratina son genes conservados entre especies y que su distribución y ordenamiento en tándem en el genoma es muy similar entre especies. También se identificó que el cluster flanqueado por los genes KRT80 y KRT18 es conservado en las cuatro especies estudiadas pero que se encuentra invertido, con referencia al humano, en ovinos y cabras. Como no se conoce la localización cromosómica de este cluster en alpacas no se puede indicar su orientación con respecto al humano.
Agradecimientos:
Los autores desean expresar su agradecimiento a: ͵ Mg. Sci. Mayra Mendoza, Mg. Sci. Marcos Calderón y Mg.Sci. Manuel More. ͵ Consejo Nacional de Ciencia y Tecnología-CONCYTEC. ͵ Programa Nacional de Innovación Agraria-PNIA. ͵ Programa de Mejoramiento Animal, Universidad Nacional Agraria La Molina-UNALM. ͵ Minnesota Supercomputing Institute (MSI), University of Minnesota.
Rol de los autores:
FAPLB, GAGR: realizaron el diseño experimental y orientaron el análisis bioinformático; AGFS: realizo el análisis bioinformático y redacto el manuscrito; FAPLB, GAGR, AGFS revisaron y aprobaron el manuscrito.
Conflicto de intereses:
Los autores no incurren en conflictos de intereses.
Fuentes de financiamiento:
El presente trabajo es parte de los proyectos 125-015 FONDECYT, proyecto 028-2016 PNIA, Hatch project MIN-16-103 y del VLIR-UOS a través del programa IUC-UNALM.
Aspectos éticos / legales:
Este trabajo no involucró colecta o preservación de especímenes, y no usó técnicas invasivas para su desarrollo. Este trabajo no incurrió en ningún problema legal.
Literatura citada
Avila F. 2014. Comparative mapping of the alpaca genome. Doctoral dissertation, Texas A & M University. Available electronically from http://hdl. handle. net/1969.1/153632. [ Links ]
Brookes A.J. 1999. The essence of SNPs. Gene 234:177-186. https://doi.org/10.1016/S0378-1119(99)00219-X. [ Links ]
Chandramohan B., C. Renieri, V. La Manna & A. La Terza. 2013. The alpaca agouti gene: genomic locus, transcripts and causative mutations of eumelanic and pheomelanic coat color. Gene 521(2):303-10. https://doi.org/10.1016/j.gene.2013.03.060 [ Links ]
Chandramohan B., C. Renieri, V. La Manna & A. La Terza. 2015. The alpaca melanocortin 1 receptor: gene mutations, transcripts, and relative levels of expression in ventral skin biopsies. The Scientific World Journal https://doi.org/10.1155/2015/265751 [ Links ]
Cransberg R. & K. Munyard. 2011. Polymorphisms detected in the tyrosinase and matp (slc45a2) genes did not explain coat colour dilution in a sample of Alpaca (Vicugna pacos). Small Ruminant Research 95(2-3):92-96. https://doi.org/10.1016/j.smallrumres.2010.10.004 [ Links ]
De Los Ríos E. 2006. Producción textil de fibras de camélidos sudamericanos en el área altoandina de Bolivia, Ecuador y Perú. Organización de las Naciones Unidas para el Desarrollo Industrial (UNIDO). (http://infoalpacas.com.pe/wp-content/uploads/2015/05/58563_camelidos_final.pdf) [ Links ]
Delgado De La Flor, I. 2014. Caracterización de nuevos marcadores genéticos microsatélites e identificación deSNP en el gen de Tricohialina en alpacas (Vicugna pacos). Tesis Magister en Bioquímica y Biología Molecular. Lima, Perú, Universidad Peruana Cayetano Heredia. [ Links ]
Foppiano F. 2016. Caracterización de marcadores genéticosen genes que codifican a proteínas asociadas a queratina y evaluación de la asociación del gen KRTAP111 al diámetro de fibra en alpaca (Vicugna pacos) siguiendo una aproximación de gen candidato. Tesis Magister en Bioquímica y Biología Molecular. Lima, Perú, Universidad Peruana Cayetano Heredia. [ Links ]
Goldammer T., GP. Di Meo, G. Luhken G., C. Drogemuller, CH. Wu, J. Kijas, BP. Dalrymple, FW. Nicholas, JF. Maddox, L. Iannuzzi & NE. Cockett. 2009. Molecular Cytogenetics and Gene Mapping in Sheep (ovis aries, 2n=54). Cytogenetic and Genome Research 126(1-2):63-76. https://doi.org/10.1159/000245907. [ Links ]
Guridi M., B. Soret, L. Alfonso & A. Arana. 2011. Single nucleotide polymorphisms in the Melanocortin 1 Receptor gene are linked with lightness of fibre colour in Peruvian Alpaca (Vicugna pacos). Animal Genetics. 42(6):679-82. https://doi.org/10.1111/j.13652052.2011.02205.x. [ Links ]
Guzmán K. 2011. Identificación de polimorfismos del gen tlr4 en crías de alpacas con cuadros de neumonías por Pasteurella multocida. Tesis EAP medicina veterinaria. Lima, Perú, Universidad Nacional Mayor de San Marcos. [ Links ]
Hesse M., A. Zimek, K. Weber, & T. Magin. 2004. Comprehensive analysis of keratin gene clusters in humans and rodents. European Journal of Cell Biolgy 83:19-26. https://doi.org/10.1078/0171-9335-00354. [ Links ]
Mamani C., G. Gutierrez & F.A. Ponce de León 2017. Identificación de polimorfismos de nucleótido simple en alpaca (Vicugna pacos) usando un panel de células híbridas irradiadas alpaca/hámster. Revista RICBA 1(2): 92-95. http://dx.doi.org/10.25127/ricba.20171.242. [ Links ]
Mendoza M. 2018. localización de genes y marcadores moleculares de alpaca (vicugna pacos) mediante la técnica de hibridación fluorescente in situ (fish). Tesis para optar el grado de magister en producción animal. Lima, Perú, Universidad Agraria la Molina. [ Links ]
Pariset L., I. Cappuccio, P. Ajmone-Marsan, M. Bruford, S. Dunner, O. Cortes, G. Erhardt, E. Prinzenberg, K. Gutscher, S. Joost, I. Pinto-Juma Nijman, J. Lenstra, T. Perez, A. Valentini & E. Consortium. 2006. Characterization of 37 Breed-Specific Single-Nucleotide Polymorphisms in Sheep. Journal of Heredity 2006:97(5):531–534. DOI: 10.1093/jhered/esl020 [ Links ]
Ramensky V., P. Bork & S. Sunyaev. 2002. Human non-synonymous SNPs: server and survey. Nucleic Acids Research 30:3894-3900. [ Links ]
Rogers G. 2004. Hair follicle differentiation and regulation. The International Journal of Developmental Biology.48:163-170. https://doi.org/10.1387/ijdb.021587gr. [ Links ]
Schibler L., Di Meo GP, Cribiu EP & Lannuzzi L. 2009. Molecular cytogenetics and comparative mapping in goats (Capra hircus, 2n=60). Cytogeneticand Genome Research. 126:77–85. https://doi. org/10.1159/000245908. [ Links ]
Schweizer J., P. Bowden, P. Coulombe, E. Lane, L. Magin, M. Omary, D. Parry, M. Rogers & M. Wright. 2006. New consensus nomenclature for mammalian keratins. Journal of Cell Biology. 174: 169–174. https://doi. org/10.1083/jcb.20060316. [ Links ]
Sulayman A., M. Tursun, Y. Sulaiman, X. Huang, K. Tian, Y. Tian, X. Xu, X. Fu, A. Mamat, & H. Tulafu. 2018. Association analysis of polymorphisms in six keratin genes with wool traits in sheep. Asian-Australasian Journal of Animal Sciences. 31:775-783. https://doi.org/10.5713/ajas.17.0349 [ Links ]
Thavamanikumar S., L.J. Mcmanus, J.F.G. Tibbits & G. Bossinger. 2011. The significance of single nucleotide polymorphisms (SNPs) in Eucalyptus globulus breedingprograms. Australian Forestry. 74: 23-29. https://doi.org/10.1080/00049158.2011.10676342 [ Links ]
Wang X., L. Wang & X. Liu. 2003. The Quality and Processing Performance of Alpaca Fibres: A report for the RuralIndustries Research and Development Corporation.RIRDC Publication Nº 03/128: 132 pág. (https://www.agrifutures.com.au/wp-content/uploads/publications/03-128.pdf) [ Links ]
Zoccola, M. 2014. Fibras proteicas: pelos finos (camélidos y cabra). Identificación, caracterización, clasificacióny acondicionamiento. INTI, UE. Cuaderno tecnológico N° 14. Proyecto mejora de las economías regionales y Desarrollo local. (http://infoalpacas.com.pe/wp-content/uploads/2017/02/cuadernillo14.pdf) [ Links ]
Zhidong, Y., J. Wildermoth, O. Wallace, S. Gordon, N. Maqbool, P. Maclean, A. Nixon, & A. Pearson. 2011. Annotationof sheep keratin intermediate filament genes and their patterns of expression. Experimental Dermatology, 20: 582-588. https://doi.org/10.1111/j.16000625.2011.01274.x [ Links ]
Correspondencia:
*Autor para correspondencia
1Universidad Nacional Agraria La Molina. Av. La Molina S/N, Lima 12.
2University of Minnesota, St. Paul, MN 55108, United States.
Email AGFS: 20110366@lamolina.edu.pe
Email GAGR: gustavogr@lamolina.edu.pe
Email FAPLB: apl@umn.com
Presentado: 31/07/2018
Aceptado: 03/02/2019
Publicado online: 30/03/2019