Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.26 no.1 Lima ene./mar. 2019
http://dx.doi.org/10.15381/rpb.v26i1.15912
TRABAJOS ORIGINALES
Microorganismos tolerantes a metales pesados del pasivo minero Santa Rosa, Jangas (Perú)
Heavy metals tolerant microorganisms from mine tailing wastelands Santa Rosa, Jangas (Peru)
Loyer Muñoz-Silva 1, Percy Olivera-Gonzales 2, Miguelina SantillánTorres 3, y Carmen Tamariz-Angeles* 2
1 Facultad de Ciencias Ambientales, Universidad Nacional Santiago Antúnez de Mayolo.
2 Laboratorio de Biología, Facultad de Ciencias, Universidad Nacional Santiago Antúnez de Mayolo.
3 Laboratorio de Química, Facultad de Ciencias, Universidad Nacional Santiago Antúnez de Mayolo.
Resumen
En el presente trabajo estudiamos el grado de tolerancia a metales pesados de hongos y bacterias aisladas de suelos con y sin rizósfera, con el propósito de conocer su potencial para aplicaciones en biorremediación. Las muestras procedían del pasivo minero de Santa Rosa de Jangas. Los hongos y bacterias aislados fueron identificados taxonómicamente mediante el análisis de la región ITS y 16S ADNr, respectivamente. El índice de tolerancia a metales pesados se calculó usando medio salino suplementado con 1 mM a plomo (II), cobre (II), niquel (II) o zinc (II), y 0.1 mM a plata (I), cromo (VI) o cadmio (II). En total se aislaron 23 hongos y 18 bacterias. Las cepas de hongos con mejores índices de tolerancia fueron: Fusarium temperatum CTLM05 (Pb+2), Fusarium temperatum CTLM08 (Zn+2), Fusarium oxysporum CTLM18 (Ni+2 y Cd+2), Fusarium oxysporum CTLM12 (Ag+1), Fusarium inflexum CTLM22 (Cu+2) y Penicillium vanluykii CTLM11 (Cr+6). Las cepas de bacterias con mayores índices de tolerancia fueron Bacillus licheniformis SSR18 (Cd+2, Ni+2 y Zn+2), Bacillus subtilis SSR3 (Pb+2), Serratia sp. SSR15 (Cu+2), Serratia sp. SSR13 (Ag+1) y Bacillus cereus SSR01 (Cr+6). También se encontró que los hongos mostraron mejores índices de tolerancia que las bacterias. Finalmente, los suelos del pasivo ambiental minero de Santa Rosa de Jangas poseen una microflora interesante, probablemente con mecanismos para su adaptación, crecimiento y desarrollo sobre metales pesados y pueden ser de utilidad para el desarrollo de procesos biotecnológicos y biorremediación.
Palabras clave: Contaminación de suelos; Fusarium; Penicillium; Bacillus; Serratia; rizosfera.
Abstract
In this work, we studied the degree of tolerance to heavy metals of fungi and bacteria isolated from soils with and without rhizosphere, in order to know its potential for applications in bioremediation. The samples came from Santa Rosa de Jangas mining liability. The fungi and bacterial strains were taxonomically identified by ITS region and 16S rDNA analysis, respectively. Heavy metal tolerance indices were calculate using salt medium supplemented with 1mM of lead (II), cupper (II), nickel (II) or zinc (II); and 0.1 mM of silver (I), chromium(VI) or cadmium (II). It was isolated 23 fungi and 18 bacteria strains. The fungi with better tolerance indices were Fusarium temperatum CTLM05 (Pb+2), Fusarium temperatum CTLM08 (Zn+2), Fusarium oxysporum CTLM18 (Ni+2 and Cd+2), Fusarium oxysporum CTLM12 (Ag+1), Fusarium inflexum CTLM22 (Cu+2), and Penicillium vanluykii CTLM11 (Cr+6). Likewise, the bacterial strains with better tolerance indices were Bacillus licheniformis SSR18 (Cd+2, Ni+2 and Zn+2), Bacillus subtilis SSR3 (Pb+2), Serratia sp. SSR15 (Cu+2), Serratia sp. SSR13 (Ag+1) and Bacillus cereus SSR01 (Cr+6). Too, it was found that fungi showed better tolerance indices than bacterial strains. Finally, the soil from Santa Rosa waste mine have an interesting microflora, probably with mechanisms for their adaptation, growth, and development were heavy metals are present and they could be useful to perform biotechnology and bioremediation processes.
Keywords: Polluted soils; Fusarium; Penicillium; Bacillus; Serratia.
Introducción
Una de las fuentes de contaminación con metales pesados es la minería, que es considerada como la segunda actividad humana más antigua, pero deja suelos y materiales indeseables en forma de vertederos recubiertos, relaves y pozas de cenizas reemplazando los ecosistemas naturales (Juwarkar et al., 2010). BecerraCastro et al. (2012) califica a la minería como la actividad más contaminante de suelos; por lo tanto para la adecuada protección y restauración de estos ecosistemas contaminados con metales pesados se requieren su caracterización y remediación (Wuana & Okieimen, 2011). En el Perú, la minería también es una actividad económica importante, pero debido a los inadecuados planes de cierre y biorremediación – especialmente de las minas antiguas – numerosos residuos (pasivos) mineros han sido dejados abandonados a lo largo de todo el país, constituyéndose un riesgo para la salud. Así mismo, en los países en desarrollo, la restauración de estos ecosistemas es escaza pero urgente porque reduciría los riesgos asociados y repondría suelos para la agricultura (Wuana & Okieimen, 2011). Por otro lado, estos ambientes contaminados poseen una biota de interés (Colpaert et al. 2011) porque los organismos resistentes a estos metales pueden ser utilizados para limpiar ambientes contaminados a través de aplicaciones biotecnológicas (Colin et al. 2012) gracias a que poseen mecanismos de sobrevivencia y desintoxicación (Muñoz et al. 2012) que les permite tolerar y acumular los metales pesados (Colin et al. 2012); es así que, los microorganismos tolerantes aislados de ambientes contaminados con metales pesados son una buena alternativa para limpiar y remediar estos ecosistemas (Vargas-García et al. 2012, Krishna et al. 2013). Así mismo como primer paso para el diseño de los procesos de biorremediación se sugiere determinar los microrganismos existentes en las áreas perturbadas (Guo et al., 2010), porque los microorganismos nativos además de tolerar los metales también están adaptadas a las condiciones ambientales de temperatura, humedad, pH, etc. del área interés. En este contexto, debido a que en la zona sierra peruana existen suelos perturbados por la industria minera cuya microflora no ha sido o está escazamente caracterizada y estudiada, el objetivo de esta investigación fue conocer la composición microbiana de los suelos del pasivo minero Santa Rosa de Jangas (Ancash, Perú) y su tolerancia a varios metales pesados con el fin de identificar microorganismos promisorios que permitan desarrollar técnicas biotecnológicas para la bioremediación de ambientes contaminados con metales pesados.
Material y métodos
Colecta y caracterización fisicoquímica del suelo.-
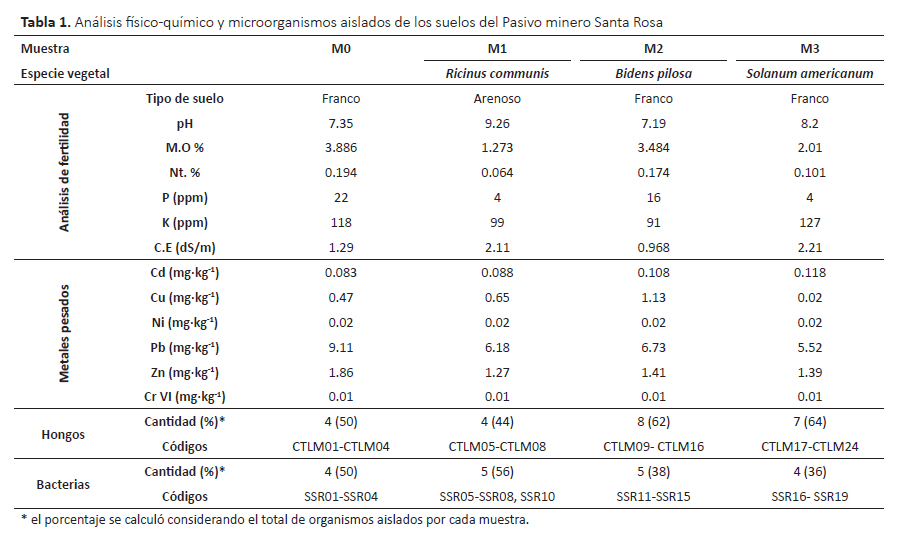
La colecta se realizó en el mes de marzo del 2014 y se tomaron cuatro muestras de suelo de aprox. 1 kg, a 25 cm de profundidad. El área de muestreo fue de aprox. 250 m2 correspondiente al pasivo minero de la planta concentradora Santa Rosa de Jangas de la Universidad Nacional Santiago Antúnez de Mayolo (Ancash-Perú) ubicado a 9°23’38.4"S, 77°34’55.2"W y 2893 m de altitud, a 1 km del pueblo de Jangas y 150 m del río Santa aprox. Las muestras fueron transportadas al laboratorio en recipientes estériles a 8 °C aproximadamente. También se colectaron plantas asociadas a los suelos, éstas fueron identificadas en el Herbario David Smith de la Universidad Nacional Santiago Antúnez de Mayolo (UNASAM).
Una parte de los suelos se usó para su caracterización físico química: composición de metales pesados de acuerdo al APHA (Standard methods for examination of water and wastewater) códigos MS08 (Cd), MS11(Cu), MS22(Ni), MS24(Pb), MS32(Zinc) y MS33 (Cr VI) en el Laboratorio de Calidad Ambiental de la Universidad Nacional Santiago Antúnez de Mayolo; y el análisis físicoquímico de fertilidad de suelos donde se evaluó el tipo de suelo, pH, conductividad eléctrica (CE), composición de materia orgánica (MO), nitrógeno total (Nt), fósforo (P) y potasio (K) en el Laboratorio de Suelos de la Universidad Nacional Santiago Antúnez de Mayolo. La otra parte del suelo fue utilizada para el aislamiento de los microorganismos.
Aislamiento de hongos y bacterias.- Previo al aislamiento, se enriqueció el cultivo inoculando 10 g de suelo en 90 mL del medio salino LPS (Jiang et al. 2008) con la diferencia que se agregó sólo 5 gL-1 de glucosa, y el LPS estéril fue suplementado con plomo (II) 0.1 mM estéril. Se incubó a 24 °C y 60 rpm en un baño maría con agitación horizontal por 7 días y se sub-cultivó tres veces transfiriendo 1 mL del cultivo a un medio fresco.
El aislamiento se realizó por el método de diluciones seriadas de 10-1 a 10-6, 100 µL de las diluciones fueron inoculadas en placas con LPSA (LPS más 15 g·L-1 de agaragar) suplementado con 1 mM Pb (II). Adicionalmente, para seleccionar hongos se agregó 0.01% de Tritón X–100, 50 µg·mL-1 de tetraciclina y 50 µg·mL-1 de estreptomicina; mientras que para bacterias se agregó 100 µg·mL-1 de cicloheximida. Todas las siembras se hicieron por duplicado e incubadas a 28 °C por 7 días.
Las colonias diferentes fueron aisladas y sembradas por estrías en placas con Agar Sabouraud (SA) en el caso de hongos y Agar Tripticasa de Soya (TSA) en el caso de bacterias; luego fueron cultivadas en tubos con medio de cultivo inclinado a 28 °C y almacenados a 4 °C.
Caracterización molecular de hongos y bacterias.Los hongos se cultivaron en caldo Saboraud a 28 °C con agitación orbital (180 rpm) por 72 h. Se colectó el micelio mediante centrifugación y se extrajo el ADN con el Kit AxyPrep Multisource Genomic Miniprep (Axygen, USA) usando el protocolo del fabricante. Se amplificó la región ITS del ADNr usando los primers ITS1-ITS4 y el secuenciamiento se realizó en Canadian Center for DNA Barcoding (CCBD) con los primers ITS5-ITS4.
El ADN de las bacterias se extrajo con el Kit AxyPrep Bacterial Genomic Miniprep (Axygen, USA) de acuerdo al protocolo del fabricante. La amplificación (PCR) del gen 16S del ADNr se realizó siguiendo la metodología descrita por Tamariz-Angeles et al. (2014). El secuenciamiento se realizó en la compañía Macrogen Korea Inc. (http:// www.macrogen.com) con los primers 518F y 800R.
La limpieza y el análisis de secuencias se realizaron con el software CodonCode v.7.2. Se obtuvieron secuencias similares a las secuencias consenso preferentemente de los registros tipo del GenBank mediante el BlastN del NCBI (http://www.ncbi.nlm.nih.gov/). Todas las secuencias se alinearon con ClustralX v.2.0 y se elaboraron los árboles filogenéticos con Mega v.6 usando la metodología Maximum likelihood y Neighbor-Joinning, Kimura 2-parámetros y 1000 bootstraps.
En el caso de los hongos los datos taxonómicos, fotografías y secuencias se ingresaron a la plataforma BOLDSYSTEMS -The Barcode of Life Data Systems (www. boldsystems.org/) para la elaboración de los códigos de barras de ADN.
Tolerancia a metales pesados.- Para el caso de los hongos se siguió la metodología descrita por Muñoz et al. (2012) con algunas modificaciones. La tolerancia a metales se realizó sobre placas Petri con LPSA suplementado con los metales pesados: 1.0 mM de Pb+2, Cu+2, Ni+2 ó Zn+2 preparados a partir de Pb(NO ) , Cu(NO ) .5H O, 0.08-0.1. Todas las soluciones stock de los metales pesados fueron esterilizados por filtración y se agregaron al medio de cultivo antes de la inoculación. Se inocularon discos de 5 mm de diámetro de los cultivos de 5 días en Agar Saboraud. Se incubaron por 7 días a 28 °C y se determinó el nivel de tolerancia como porcentaje de tolerancia (%T ) que se obtuvo multiplicando x 100 el índice de tolerancia (IT ); el mismo que fue calculado como IT = Diámetro del hongo en el medio con metal / Diámetro del hongo en medio sin metal (Muñoz et al. 2012).
Para las bacterias se utilizó 5mL de LPS suplementado con metales pesados a las mismas concentraciones que se indica para el caso de los hongos. Como inóculo se usó 20 µL de cultivo joven (16 horas) a OD entre 0.08-0.1. Luego se incubó a 24°C y 60 rpm de agitación horizontal por 24 horas. Se calculó el porcentaje de tolerancia de la siguiente manera (%T ) = [(Densidad óptica del cultivo con el metal – Densidad del medio con metal sin inóculo)/Densidad del cultivo sin metal] x 100.
Análisis estadísticos.- Para evaluar la tolerancia a los metales pesados se hicieron cuatro repeticiones en un diseño completamente al azar (DCA), donde se calculó promedios, desviaciones estándares y se compararon las medias mediante el análisis de variancia ANOVA y la comparación de las medias de Duncan (P < 0.05).
Resultados y discusión
Características físico-químicas del suelo y aislamiento de los hongos y bacterias. En el pasivo minero Santa Rosa se encontró escaza vegetación representada principalmente por Bidens pilosa; esta especie también fue reportada en pasivos mineros de cobre en China (He et al. 2010).
Respecto a las características de los suelos, las muestras M1 y M3 mostraron pH alcalino, salinidad ligera, materia orgánica, nitrógeno y fósforo fue pobre; mientras que los suelos MO y M2 mostraron pH neutro, suelo no salino, materia orgánica, nitrógeno y fósforo en cantidades medianas (Tabla 1). Entre los metales pesados, el plomo (Pb) fue el más abundante, por lo tanto este metal fue utilizado en el medio de cultivo durante el enriquecimiento y aislamiento de los microorganismos.
Se aislaron un total de 41 cepas incluyendo 22 hongos filamentosos, 1 levadura y 18 bacterias. Según Guo et al. (2010), los microorganismos juegan un rol importante en la biorremediación de metales pesados tanto como bioacumuladores (manera directa) o interactuando con las plantas para facilitar la bioacumulación de las plantas (manera indirecta); así mismo su diversidad depende de la etapa de restauración del ecosistema por lo cual también podrían ser usados como bioindicadores (Li et al. 2016).
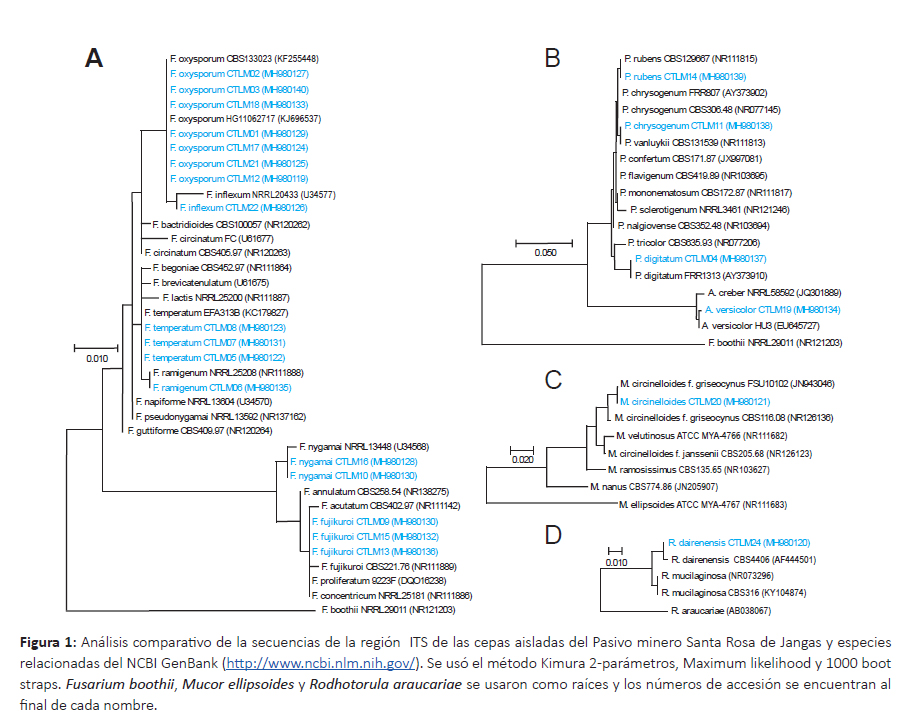
Caracterización molecular de hongos y bacterias.Las secuencias del ITS de los hongos aislados fueron editadas a un tamaño de 590 bp que corresponde a la región completa del ITS (ITS1-5.8S-ITS2). En el análisis BlastN se obtuvo 98 – 100% similitud de identidad con cepas tipo del GenBank. Las secuencias fueron depositadas al Genbank con los números de accesión MH980118MH980140 que se encuentran después del código de la cepa (Fig. 1). También se elaboraron los códigos de barra de ADN que se adjunta en el material suplementario DOI: dx.doi.org/10.5883/DS-CTLM.
De acuerdo al análisis filogenético de los hongos se identificaron 12 especies distribuidos en 5 géneros (Fig. 1A-D), donde el género Fusarium estuvo representada por 6 especies: F. fujikuroi, F. inflexum, F. nygamai, F. oxysporum, F. ramigenum y F. temperatum; el género Penicillium estuvo representada por 3 especies: P. digitatum, P. rubens y P. vanluykii; mientras que los géneros Aspergillus, Mucor y Rhodotorula sólo tuvieron una especie: A. versicolor, M. griseocynus y R. dairenensis, respectivamente. Estas especies están ampliamente distribuidos en los suelos; pero a su vez varias investigaciones señalan la potencialidad de especies del género Fusarium para la biorremediación de metales pesados (Chen et al. 2010, Parameswari et al. 2010, Muñoz et al. 2012, Iram et al. 2013, Oladipo et al. 2018a). Del mismo modo varias especies del género Penicillium, Aspergillus, Mucor y Rhodotorula aislados de ambientes contaminados han mostrado capacidades para la bioremediación de metales pesados (Iram et al. 2013, Mani & Kumar 2014, Muñoz et al. 2012, Oladipo et al. 2018a, Parameswari et al. 2010, Vargas-García et al. 2012).
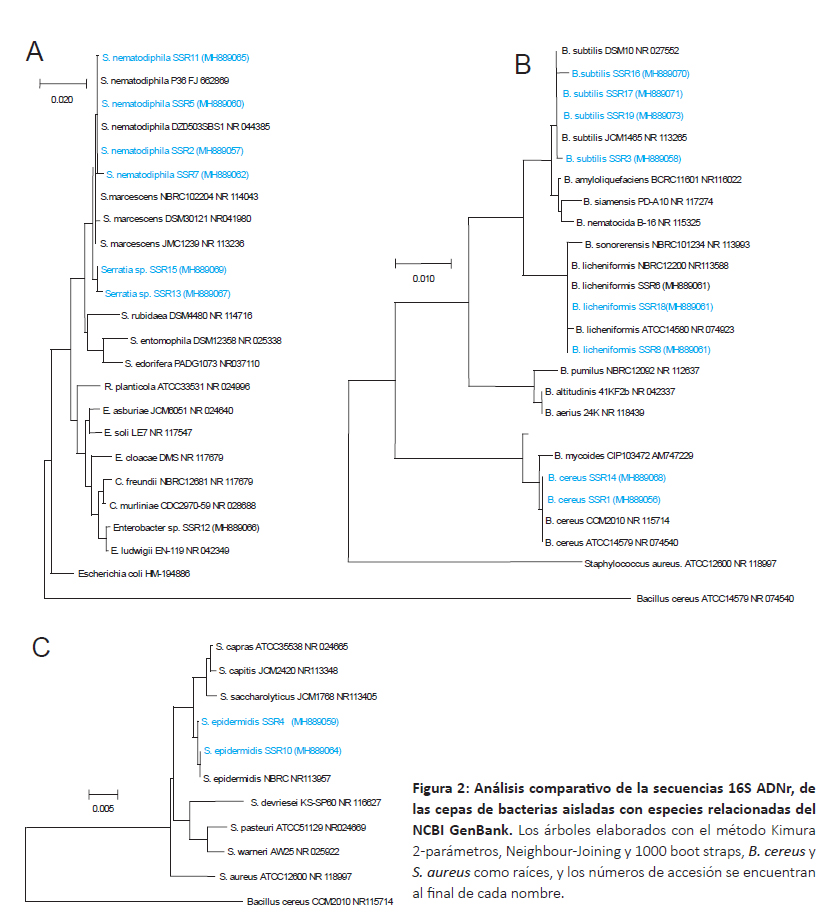
Respecto a las bacterias aisladas, todas las secuencias fueron editadas a un tamaño de 1200 – 1450bp correspondiente al gen 16S ADNr. En el análisis BlastN se obtuvo porcentajes de identidad de 97 – 100% con cepas tipo del GenBank. Las secuencia de las cepas aisladas fueron depositadas al Genbank y su número de accesión se encuentra posterior al código de la cepas en la Figura 2.
El análisis filogenético de las bacterias muestra la presencia de 7 especies agrupadas en 4 géneros (Fig. 2A-C), donde el género Bacillus estuvo representada por tres especies: B. licheniformis, B. subtilis y B. cereus; también se aislaron cepas de Staphylococcus epidermidis y especies de los géneros Serratia y Enterobacter. Numerosas investigaciones reportan la amplia utilidad biotecnológica de las especies del género Bacillus; entre las cuales, las cepas de Bacillus sp. y Bacillus cereus aisladas de ambientes contaminados han mostrado tolerancia a metales pesados así como capacidades para la bioremediación (Zahoor & Rehman 2009, He et al. 2010, Guo et al. 2010, Valverde et al. 2011). Ahemad (2014) reporta que varias especies del género Bacillus promotores de crecimiento vegetal vienen siendo aplicadas a procesos de desintoxicación de ambientes contaminados con metales pesados; Gupta y Diwan (2017) encontraron que algunos microorganismos -entre ellos especies de Bacillustienen potencialidades para contribuir con la desintoxicación de ambientes contaminados mediante la producción exopolisacáridos con capacidad de quelar metales pesados. Respecto a las especies de los géneros Serratia, Enterobacter y Staphylococcus, Chen et al. (2010) aislaron algunas cepas de Serratia nematodiphila y Enterobacter aerogenes endofíticas resistentes a metales pesados; así mismo Yu et al. (2014) encontraron una cepa de Staphylococcus epidermidis con capacidad de reducir Hg (II) debido a la presencia del operon mer, además una cepa de Staphylococcus capitis mostró tolerancia a cadmio, plomo, zinc, cobre, níquel y cromo (IV) reduciendo este último metal en porcentajes cercanos a 80% (Zahoor & Rehman 2009).
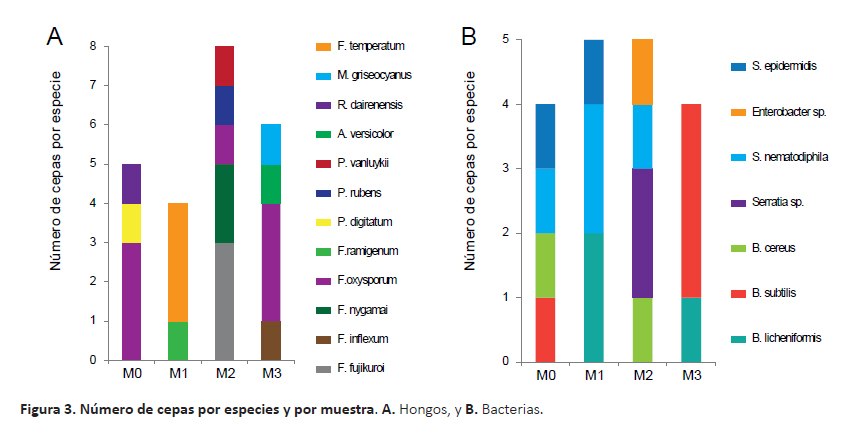
Respecto a las especies por muestra de suelo, en las muestras M2 (rizósfera de B. pilosa) y M3 (rizósfera de S. americanum) se aislaron los mayores números de géneros y especies de hongos; mientras que en M1 (rizósfera de R. communis) sólo se aislaron especies del género Fusarium (Fig. 3A). Así mismo, se aislaron hasta 4 especies de bacterias de las muestras M0 y M2; mientras que en M1 y M3 se encontraron 3 y 2 especies, respectivamente. También se observó que F. oxysporum estuvo presente en las muestras M0, M2 y M3 (Fig. 3A); mientras que por lo menos una cepa del género Bacillus estuvo presente en todas las muestras y cepas del género Serratia se encontraron en las muestras M0, M1 y M2 (Fig. 3B).
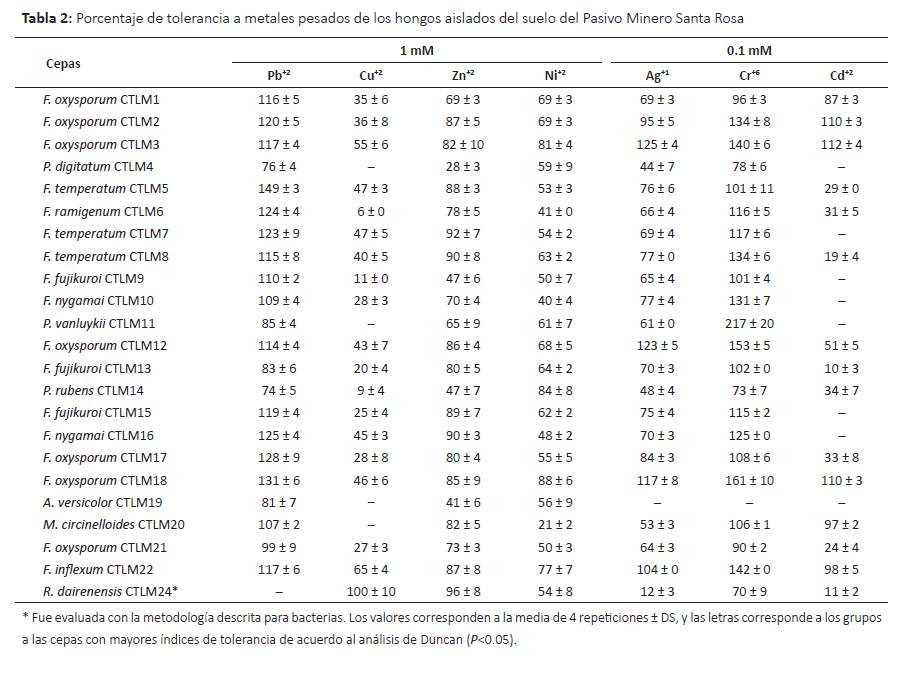
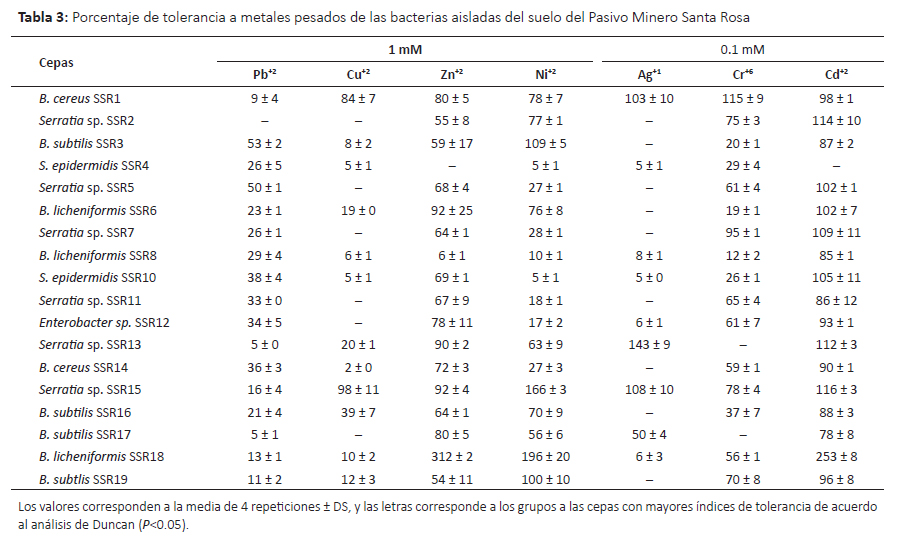
Tolerancia a metales pesados.Se evaluó la capacidad de tolerancia a siete metales pesados con diversos niveles de toxicidad y los resultados se muestran en las Tablas 2 y 3. Oladipo et al. (2018a) consideran tolerancia muy baja cuando el índice de tolerancia (IT) se encuentra entre 0.00–0.39, tolerancia baja cuando el IT es 0.40– 0.59, tolerancia moderada con IT 0.60–0.79, tolerancia alta con IT 0.80–0.99 y tolerancia muy alta con IT ≥ 1.00; en este sentido se encontró que varios microorganismos mostraron alta y muy alta tolerancia.
En el caso del plomo, 70% de los hongos mostraron alta tolerancia a 1mM de plomo. Este metal es uno de los cuatro metales más tóxicos de la tierra porque causa problemas en la salud humana y ecosistema (Rhee et al. 2014), es uno de los más persistentes en el ambiente (Mani & Kumar 2014) y respecto a los tres ciclos pasados su presencia como contaminante se ha incrementado hasta 1000 veces más (Naik & Dubey 2013). Por lo tanto, los microorganismos con habilidades de sobrevivir y tolerar concentraciones altas de Pb (II) son punto de partida para mayores estudios con fines de biorremediación. Los hongos con muy alta tolerancia y mejores resultados fueron Fusarium temperatum CTLM05 seguido de F. oxysporum CTLM17 y F. oxysporum CTLM18. En concordancia, especies del género Fusarium han sido reportadas como tolerantes a Pb(II), Cd (II), entre otros metales pesados (Vargas-García et al. 2012). En el caso de las bacterias sólo B. subtilis SSR3 y Serratia sp. SSR2 mostraron tolerancia moderada a 1mM de plomo (II), mientras que las demás cepas presentaron baja o muy baja tolerancia. Syed y Chinthala (2015) encontraron cepas del género Bacillus entre ellas B. subtilis NSPA13 con significativa capacidad de biosorción de plomo.
Respecto al cobre, este metal es un micronutriente esencial para los seres vivos pero puede ser tóxico en bajas concentraciones (Ahemad 2014, Cornu et al. 2017). Por lo tanto la célula posee mecanismos homeostáticos para regular su concentración y minimizar su toxicidad pero su exceso puede afectar al cerebro, riñón, hígado, intestino (Colin et al. 2012, Cornu et al. 2017). Su alta toxicidad está relacionada con su capacidad de formar radicales libres (Rohini & Jayalakshme 2015). La contaminación con este metal se debe a factores naturales y la actividad humana tales como la minería, industria, agricultura y combustión (Cornu et al. 2017, Hock et al. 2017). Frente a este problema, algunas bacterias (Cornu et al. 2017) y ciertos hongos (Hock et al. 2017) son promisorios para los procesos de biorremediación porque han mostrado capacidades para bioabsorber este metal. En este sentido, se evaluó el nivel de tolerancia a 1 mM de cobre (II), encontrándose que la levadura Rhodotorula dairenensis CTLM24, y las bacterias Serratia sp. SSR15 y Bacillus cereus SSR1 mostraron tolerancia muy alta a diferencia de la mayoría de microorganismos evaluados que mostraron tolerancia baja a este metal. En relación a R. dairenensis, la especie R. mucilaginosa UANL-001L ha sido reportada como resistente a varios metales pesados entre ellos cobre (Garza-Gonzalez et al. 2016). Así mismo, estudios sobre resistencia a cobre de otras cepas de R. mucilaginosa muestran que éstas poseen la capacidad de bioabsorver el cobre y mecanismos para tolerarlo (Colin et al. 2012, Irazusta et al. 2012). Este grupo de levaduras producen pigmentos de tipo carotenoides que probablemente minimizan la toxicidad de los metales pesados (Irazusta et al. 2012). Respecto a las bacterias, Cidre et al. (2017) han reportado algunas cepas del género Serratia y Bacillus con alto nivel de resistencia a cobre, habiendo encontrado la presencia de genes pertenecientes al cluster pco multicobre oxidasa y al cluster sil involucrado en la resistencia a Cu y Ag en especies del género Serratia y otras gram negativas. Del mismo modo, Behera et al. (2014) en el estudio de respuesta antioxidativa de B. cereus frente a cadmio y cobre, encontraron que ambos metales inducían diferentes respuestas, donde la respuesta frente al cobre estaría relacionada con la producción de catalasas. Así mismo, algunas cepas de B. cereus mostraron alta capacidad de biosorción de cobre (Rohini & Jayalakshme 2015, Oladipo et al. 2018b), encontrándose la presencia de genes de resistencia al cobre en el cromosoma y que el grado de biosorción era dependiente a las condiciones del medio cultivo y entorno (Rohini & Jayalakshme 2015).
El zinc como elemento traza es un metal importante para el desarrollo normal de diversas vías metabólicas celulares y en la salud del ser humano (Wuana & Okieimen 2011, Ahemad 2014). La contaminación de aguas y suelos con zinc proveniente de actividades humanas principalmente la minería, la combustión de carbón y desechos, y la industria del acero viene generando riesgos en la salud y ecosistema (Wuana & Okieimen 2011), por lo tanto es necesario los esfuerzos para la biorremediación de ecosistemas contaminados con este metal. Entre los microorganismos evaluados y cultivados con 1mM de Zn (II) se encontró que los hongos R. dairenensis CTLM24, F. temperatum CTLM7 y CTLM8, y F. nigamai CTLM16 mostraron alta tolerancia a zinc. Así mismo, las bacterias mostraron niveles de tolerancia variados, de los cuales la cepa B. licheniformis SSR18 alcanzó un índice de 3.00, es decir no solo fue altamente tolerante sino que fue estimulada por este metal; también B. licheniformi SSR06 y Serratia sp. SSR15 mostraron alta tolerancia. En concordancia, Muñoz et al. (2012) encontraron que las bacterias y hongos aisladas de aguas residuales toleraron altas cantidades de zinc, pero la cepa R. mucilaginosa mostró menor tolerancia. Sin embargo, Garza-Gonzalez et al. (2016) encontraron que R. mucilaginosa UANL-001L mostraba alta tolerancia a zinc y plomo, atribuyendo esta propiedad a la formación de exopolisacáridos. Contrariamente, R. dairenensis CTLM24 no toleró 1mM de plomo, pudiendo dar indicios de la presencia de otros mecanismos involucrados. Por otro lado, Krishna et al. (2013) encontraron una cepa de Bacillus sp. que bio-acumulaba altos niveles de zinc a pH 9.
El níquel es contaminante industrial que está presente en el ambiente sólo en muy bajas concentraciones (Wuana & Okieimen 2011), como otros metales es esencial para los seres vivos en muy bajas dosis pero a mayores concentraciones es causante de varios tipos de cáncer (Wuana & Okieimen 2011, Macomber & Hausinger 2011). Wuana y Okieimen (2011) indican que a pesar de su efecto negativo en el crecimiento de los microorganismo, algunos desarrollan resistencia. En concordancia, se encontró que todas las cepas de hongos y bacterias mostraron tolerancia a 1mM de niquel. Los hongos con mejor respuesta fueron F. oxysporium CTLM18, P. rubens CTLM14 y F. oxysporum CTLM3 que mostraron alta tolerancia. Entre las bacterias B. licheniformis SSR18 seguido de Serratia sp. SSR15 mostraron muy alta tolerancia con valores mayores a 1.96 ± 0.02 y 1.66 ± 0.3, respectivamente. Se han encontrado varios mecanismos de tolerancia en bacterias y eucariotas (Macomber and Hausinger, 2011), donde la tolerancia de Bacillus cereus está asociada a un gen cromosomal (Shoeb et al. 2010).
Respecto a la tolerancia a plata, todas las cepas de hongos excepto A. versicolor CTLM19 mostraron algún grado de tolerancia a 0.1mM de plata (I) donde las cepasF. oxysporium CTLM03, CTLM12, CTLM18 y F. inflexum CTLM22 fueron muy altamente tolerantes. Por lo contrario, la mayoría de bacterias no mostraron tolerancia a este metal. Sin embargo, las cepas Serratia sp. SSR13, Serratia sp. SSR15 y B. cereus SSR1 mostraron muy alta tolerancia. Este metal es tóxico para las bacterias porque forman complejos con el azufre (Ahemad 2014), esta actividad antibacteriana está siendo rescatada por la nanotecnología y se están estudiando algunos hongos tolerantes capaces de formar nanopartículas de plata (Devi & Joshi 2015, Majeed et al. 2017).
El cromo (VI) es altamente tóxico, mutagénico y carcinogénico, deriva principalmente de la actividad humana, por su alta solubilidad se moviliza a otros lugares diferentes de su origen (Ahemad 2014, Viti et al. 2014). Atraviesa la membrana celular por la vía de ingreso del sulfato y genera iones intermediario Cr(V) y/o Cr(IV), radicales libres y como producto final Cr(III) que afecta la replicación del ADN, causa mutagénesis, y afecta a las enzimas (Viti et al. 2014). En el caso de los hongos la mayoría mostraron alta y muy alta tolerancia, entre ellos destacó F. nygamai CTLM10 que alcanzó un índice de 3.17 seguido de F. oxysporum CTLM12 y CTLM18. Concordantemente, Iram et al. (2013) aislaron varias especies de hongos resistentes a Cr y Pb a partir de suelos, entre ellos a Fusarium solani. La mayoría de bacterias mostraron algún nivel de tolerancia a este metal, entre ellas B. cereus SSR1 mostró muy alta tolerancia. Concordantemente, Upadhyay et al. (2017) encontraron que B. subtilis MNU16 aislada de suelos de minas de carbón con un gran nivel de resistencia a cromo (VI), el mismo que es reducido a Cr (III).
Junto con el mercurio y el plomo, el cadmio es el tercer metal más contaminante sin función biológica conocida (Wuana & Okieimen 2011). La contaminación con este metal proviene de la minería, efluentes de las textileras, industrias de electroplatinados y galvanizados y las baterías de cadmio (Fazli et al. 2015). Catorce cepas de hongos mostraron algún nivel de tolerancia a 0.1 mM de cadmio (II) de los cuales F. oxysporum CTLM02, F. oxysporum CTLM03, y F. oxysporum CTLM18 mostraron muy alta tolerancia. Concordantemente, otras investigaciones señalan el alto potencial de los hongos para la descontaminación del cadmio (An et al. 2015, Fazli et al. 2015). Así mismo, la mayoría de bacterias mostraron tolerancia a este metal y el 44% mostró muy alta tolerancia. B. licheniformis SSR18 creció hasta 150% más que el control sin metal (IT 2.53). La biomasa viva o muerta o los EPS (polisacáridos extracelulares) de algunas especies del género Bacillus han mostrado resistencia y potencial de remover cadmio (Shameer 2016, Wu et al. 2016).
Finalmente, la aplicación de los sistemas microbianos para la biorecuperación y bioprocesos de los metales ha recibido gran importancia en los últimos años (Liang & Gadd 2017). En este sentido, en los suelos del pasivo minero Santa Rosa se ha encontrado una microflora de hongos y bacterias resistente a metales pesados, los cuales podrían jugar el rol de promotores de crecimiento vegetal protegiendo a las plantas de los efectos tóxicos de los metales (Jiang et al. 2008), pero también podrían tener el rol de bioacumulación y reducción de los metales pesados. Estos bioprocesos podrían ser aprovechados para procesos de bioremediación, por lo cual son necesarias mayores investigaciones.
Material suplementario
Códigos de barra de ADN de los hongos se encuentra en la siguiente dirección DOI dx.doi.org/10.5883/DS-CTLM
Agradecimientos:
-
Biodiversity Institute of Ontario Herbario (BIO) de la Universidad
de Guelph (Canadá)
-
La Dra. Nataly Ivanova y el Dr. Alex Borisenko del BIO
-
Canadian Center for DNA barcoding (CCDB)
Rol de los autores:
LMS, POG, MST y CTA diseñaron la investigación, LMS desarrolló la parte experimental, POG y MST apoyaron en la parte experimental, CTA realizó el análisis molecular y escribió el borrador del manuscrito, LMS, POG, MST y CTA aprobaron el manuscrito. Los autores manifiestan que no existen conflictos de intereses.
Conflicto de intereses:
Los autores no incurren en conflictos de intereses.
Aspectos éticos / legales:
Este trabajo no incurrio en ningun problema legal.
Fuentes de financiamiento:
El secuenciamiento de los hongos tuvo el apoyo del Canadian Center of DNA Barcoding (CCDB) y Biodiversity Institute of Ontario (BIO) de la Universidad de Guelph a través del proyecto "DNA barcoding to support the biodiversity conservation, sustainable harvesting and trade in Perú".
Citación:
Muñoz-Silva L., P. Olivera-Gonzales, M. Santillán- Torres & C. Tamariz-Angeles. 2019. Microorganismos tolerantes a metales pesados del pasivo minero Santa Rosa Jangas (Perú). Revista peruana de biología 26(1): 109 118 (Febrero 2019). doi: http://dx.doi.org/10.15381/rpb.v26i1.15914
Literatura citada
Ahemad M. 2014. Remediation of metalliferous soils through the heavy metal resistant plant growth promoting bacteria: Paradigms and prospects. Arabian Journal of Chemistry. doi: https://doi.org/10.1016/j.arabjc.2014.11.020 [ Links ]
An H., Y. Liu, , X. Zhao, et al. 2015. Characterization of cadmium-resistant endophytic fungi from Salix variegata Franch. in Three Gorges Reservoir Region, China. Microbiological Research 176: 29–37. doi:10.1016/j.micres.2015.03.013 [ Links ]
Becerra-Castro C., C. Monterroso, A. Prieto-Fernández, et al. 2012. Pseudometallophytes colonising Pb/Zn mine tailings: A description of the plant-microorganismrhizosphere soil system and isolation of metal-tolerant bacteria. Journal of Hazardous Materials 217– 218: 350-359. doi:10.1016/j.jhazmat.2012.03.039
Behera M., J. Dandapat & C.C. Rath. 2014. Effect of heavy metals on growth response and antioxidant defense protection in Bacillus cereus. Journal Basic Microbiology 54 (11): 1201–1209. doi:10.1002/jobm.201300805 [ Links ]
Chen L., S. Luo, X. Xiao, et al. 2010. Application of plant growthpromoting endophytes (PGPE) isolated from Solanum nigrum L. for phytoextraction of Cd-polluted soils. Applied Soil Ecology 46 (3): 383-389. doi:10.1016/j.apsoil.2010.10.003 [ Links ]
Cidre I., R.P. Pulido, J.G. Burgos, et al. 2017. Copper and Zinc Tolerance in Bacteria Isolated from Fresh Produce. Journal of Food Protection 80 (6): 969-975. doi: 10.4315/0362-028X.JFP-16-513. [ Links ]
Colin V.L., L.B. Villegas & C.M. Abate. 2012. Indigenous microorganisms as potential bioremediators for environments contaminated with heavy metals. International Biodeterioration & Biodegradation 69: 28-37. doi:10.1016/j.ibiod.2011.12.001 [ Links ]
Colpaert J.V., J.H.L.Wevers, E. Krznaric, et al. 2011. How metal-tolerant ecotypes of ectomycorrhizal fungi protect plants from heavy metal pollution. Annals of Forest Science 68 (1): 17-24. doi:10.1007/s13595-010-0003-9 [ Links ]
Cornu J.Y., D. Huguenot, K. Jézéquel, et al. 2017. Bioremediation of copper-contaminated soils by bacteria. World Journal Microbiology Biotechnology 33(26): 1-9. doi:10.1007/s11274-016-2191-4 [ Links ]
Devi L.S. & S.R. Joshi. 2015. Ultrastructures of silver nanoparticles biosynthesized using endophytic fungi. Journal of Microscopy and Ultrastructure. 3(1): 29-37. doi:10.1016/j.jmau.2014.10.004 [ Links ]
Fazli M.M., N. Soleimani, M. Mehrasbi, et al. 2015. Highly cadmium tolerant fungi: Their tolerance and removal potential. Journal of Environmental Health Science &. Engineering 13 (19): 1-9. doi:10.1186/s40201-015-0176-0 [ Links ]
Garza-Gonzalez M.T., D. Barboza, A. Vazquez, et al. 2016. Metal-induced production of a novel bioadsorbent exopolysaccharide in a native Rhodotorula mucilaginosa from the mexican northeastern region. PLoS One 11 (2): 1–18. doi:10.1371/journal.pone.0148430 [ Links ]
Guo H., S. Luo, L. Chen, et al. 2010. Bioremediation of heavy metals by growing hyperaccumulaor endophytic bacterium Bacillus sp. L14. Bioresource Technology 101(22): 8599–8605. doi:10.1016/j.biortech.2010.06.085 [ Links ]
Gupta P. & B. Diwan. 2017. Bacterial Exopolysaccharide mediated heavy metal removal: A Review on biosynthesis, mechanism and remediation strategies. Biotechnology Reports 13: 58–71. doi:10.1016/j.btre.2016.12.006 [ Links ]
He L.Y., Y.F. Zhang, H.Y. Ma, et al. 2010. Characterization of copper-resistant bacteria and assessment of bacterial communities in rhizosphere soils of coppertolerant plants. Applied Soil Ecology 44(1): 49–55. doi:10.1016/j.apsoil.2009.09.004 [ Links ]
Hock Ong G., X. Han Ho, S. Shamkeeva, et al. 2017. Biosorption study of potential fungi for copper remediation from Peninsular Malaysia. Remediation Journal 27 (4): 59– 63. doi:10.1002/rem.21531 [ Links ]
Iram S., A. Zaman, Z. Iqbal et al. 2013. Heavy Metal Tolerance of Fungus Isolated from Soil Contaminated with Sewage and Industrial Wastewater. Polish Journal of Environmental Studies 22 (3): 691–697. [ Links ]
Irazusta V., C. Estévez, M.J. Amoroso, et al. 2012. Proteomic study of the yeast Rhodotorula mucilaginosa RCL11 under copper stress. BioMetals 25 (3): 517-527. doi:10.1007/s10534-012-9531-0 [ Links ]
Jiang CY., X.F. Sheng, M. Qian, et al. 2008. Isolation and characterization of a heavy metal-resistant Burkholderia sp. from heavy metal-contaminated paddy field soil and its potential in promoting plant growth and heavy metal accumulation in metal-polluted soil. Chemosphere 72 (2): 157–164. doi:10.1016/j.chemosphere.2008.02.006 [ Links ]
Juwarkar A.A., S.K. Singh & A. Mudhoo. 2010. A comprehensive overview of elements in bioremediation. Reviews in Environmental Science and Bio/Technology 9(3): 215-288. doi:10.1007/s11157-010-9215-6 [ Links ]
Krishna M.P., R. Varghese, V.A. Babu, et al. 2013. Bioremedaition of zinc using Bacillus sp. isolated from metal-contaminated industrial zone. In A. and A. Augustine, eds. Prospects in Bioscience: Addressing the Issues. Springer, India. Pp. 11–18. doi:10.1007/978-81-322-0810-5 [ Links ]
Li Y., Z. Jia, Q. Sun, et al. 2016. Ecological restoration alters microbial communities in mine tailings profiles. Scientific Reports 6: 1–11. doi:10.1038/srep25193 [ Links ]
Liang X. & G.M. Gadd. 2017. Metal and metalloid biorecovery using fungi. Microbial Biotechnology. 10(5): 1199– 1205. doi:10.1111/1751-7915.12767 [ Links ]
Macomber L. & R.P. Hausinger. 2011. Mechanisms of nickel toxicity in microorganisms. Metallomics 3(11): 1153– 1162. doi:doi:10.1039/c1mt00063b
Majeed S., M. Danish, B. Zahrudin, A.H. & G.K. Dash. 2017. Biosynthesis and characterization of silver nanoparticles from fungal species and its antibacterial and anticancer effect. Karbala International Journal Modern Science 4(1): 86-92. doi:10.1016/j.kijoms.2017.11.002
Mani D. & C. Kumar. 2014. Biotechnological advances in bioremediation of heavy metals contaminated ecosystems: An overview with special reference to phytoremediation. International Journal of Environmental Science and Technology 11(3): 843-872. doi:10.1007/s13762-013-0299-8 [ Links ]
Muñoz A.J., E. Ruiz, H. Abriouel, et al. 2012. Heavy metal tolerance of microorganisms isolated from wastewaters: Identification and evaluation of its potential for biosorption. Chemical Engineering Journal. 210 (1): 325-332. [ Links ]
Naik M.M. & S.K. Dubey. 2013. Lead resistant bacteria: lead resistance mechanisms, their applications in lead bioremediation and biomonitoring. Ecotoxicology and Environmental Safety 98(1): 1-7. doi: 10.1016/j.ecoenv.2013.09.039 [ Links ]
Oladipo O.G., O.O. Awotoye, A. Olayinka, et al. 2018a. Heavy metal tolerance traits of filamentous fungi isolated from gold and gemstone mining sites. Brazilian Journal of Microbiology 49(1): 29-37. doi:10.1016/j.bjm.2017.06.003 [ Links ]
Oladipo O.G., O.T. Ezeokoli, M.S. Maboeta, et al. 2018b. Tolerance and growth kinetics of bacteria isolated from gold and gemstone mining sites in response to heavy metal concentrations. Journal of Environmental Management 212: 357-366. doi:10.1016/j.jenvman.2018.01.038 [ Links ]
Parameswari E., A. Lakshmanan & T. Thilagavathi. 2010. Biosorption and metal tolerance potential of filamentous fungi isolated from metal polluted ecosystem. Electronic Journal of Environmental, Agricultural and Food Chemistry 9(4): 664–671. [ Links ]
Rhee Y.J., S. Hillier, H. Pendlowski, et al. 2014. Fungal transformation of metallic lead to pyromorphite in liquid Medium. Chemosphere 113:17-21 doi:10.1016/j.chemosphere.2014.03.085 [ Links ]
Rohini B. & S. Jayalakshme. 2015. Bioremediation potential of Bacillus cereus against copper and other heavy metals. International Journal of Advanced Research in Biological Sciences 2(2): 200–209. [ Links ]
Shameer S. 2016. Biosorption of lead, copper and cadmium using the extracellular polysaccharides (EPS) of Bacillus sp., from solar salterns. 3 Biotech 6(194): 1-10. doi:10.1007/s13205-016-0498-3
Shoeb E., N. Ahmed, P. Warner, P. et al. 2010. Identification of a unique mechanism of tolerance against nickel in Bacillus Cereus isolated from heavy metal contaminated sites. The Internet Journal of Microbiology 9(1): 1-7.
Syed S. & P. Chinthala. 2015. Heavy Metal Detoxification by Different Bacillus Species Isolated from Solar Salterns. Scientifica 2015: 1–8. doi:10.1155/2015/319760 [ Links ]
Tamariz-Angeles C., P. Olivera-Gonzales, G.K. Villena et al. 2014. Isolation and Identification of Cellulolytic and Xylanolytic Bacteria from Huancarhuaz Hot Spring, Peru. Annual Research & Review in Biology 4(19): 2920–2930. [ Links ]
Upadhyay N., K. Vishwakarma, J. Singh, et al. 2017. Tolerance and Reduction of Chromium(VI) by Bacillus sp. MNU16 Isolated from Contaminated Coal Mining Soil. Frontiers In Plant Science 8, 1–13. doi:10.3389/fpls.2017.00778 [ Links ]
Valverde A., M. González-Tirante, M. Medina-Sierra, et al. 2011. Diversity and community structure of culturable arsenic-resistant bacteria across a soil arsenic gradient at an abandoned tungsten-tin mining area. Chemosphere 85(1): 129–134. doi:10.1016/j.chemosphere.2011.06.025 [ Links ]
Vargas-García M., M.J. López, F. Suárez-Estrella, et al. 2012. Compost as a source of microbial isolates for the bioremediation of heavy metals: In vitro selection. Science of The Total Environment 431: 62–67. doi:10.1016/j.scitotenv.2012.05.026 [ Links ]
Viti C., E. Marchi, F. Decorosi & L. Giovannetti. 2014. Molecular mechanisms of Cr(VI) resistance in bacteria and fungi. FEMS Microbiology Reviews 38(4): 633–659. doi:10.1111/1574-6976.12051 [ Links ]
Wu H., Q. Wu, G. Wu, et al. 2016. Cd-resistant strains of B. cereus S5 with endurance capacity and their capacities for cadmium removal from cadmium-polluted water. PLoS One 11(4), 1–25. doi:10.1371/journal.pone.0151479 [ Links ]
Wuana R.A. & F.E. Okieimen. 2011. Heavy Metals in Contaminated Soils: A Review of Sources, Chemistry, Risks and Best Available Strategies for Remediation. International Scholarly Research Network Ecology 2011: 1–20. doi:10.5402/2011/402647 [ Links ]
Yu Z., J. Li, Y. Li, et al. 2014. A mer operon confers mercury reduction in a Staphylococcus epidermidis strain isolated from Lanzhou reach of the Yellow River. International Biodeterioration & Biodegradation. 90: 57–63. https://doi.org/10.1016/j.ibiod.2014.02.002 [ Links ]
Zahoor A. & A. Rehman. 2009. Isolation of Cr(VI) reducing bacteria from industrial effluents and their potential use in bioremediation of chromium containing wastewater. Journal of Environmental Sciencees 21(6): 814– 820. doi:10.1016/S1001-0742(08)62346-3 [ Links ]
Correspondencia:
* Autor para correspondencia
Email LMS: loyermunoz@gmail.com
Email PO-G: poliverag@unasam.edu.pe
Email MS-T: msantillant@unasam.edu.pe
Email CT-A: ctamariz@unasam.edu.pe
Presentado: 10/10/2018
Aceptado: 13/01/2019
Publicado online: 18/03/2019




![[ARTÍCULORETRACTADO]Toxicidad y actividad molusccidal del aceite esencial Pimenta dioica contra el caracol Biomphalaria glabrata](/img/es/prev.gif)