Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.26 no.2 Lima abr./jun. 2019
http://dx.doi.org/10.15381/rpb.v26i2.16370
TRABAJOS ORIGINALES
Diversidad genética de tres poblaciones de Physalis peruviana a partir del fraccionamiento y patrón electroforético de proteínas de reserva seminal
Genetic diversity in three populations of Physalis peruvianausing fractionation and electrophoretic patterns of seed storage protein
Henry Bonilla, Yajahaira Carbajal, María Siles ORCID iD: 0000-0003-4956-8310 y Alberto López* ORCID iD: 0000-0001-6070-5836
Universidad Nacional Mayor de San Marcos, Facultad de Ciencias Biológicas, Grupo de Investigación en Recursos Genéticos (RecGen), Lima, Perú.
Resumen
En el presente trabajo, es estudiada la diversidad genética de tres poblaciones atribuidas a ecotipos de aguaymanto, Physalis peruaviana. Las tres poblaciones eran atribuidas a los ecotipos Agroandino (provincia de San Pablo), Celendino (provincia de Celendín) y Cajabamba (provincia de Cajabamba) del departamento de Cajamarca. Se realizó la cuantificación proteica y evaluó el polimorfismo de las proteínas de reserva seminal (SSPs) mediante electroforesis en gel de poliacrilamida denaturante (SDS-PAGE). Además, se identificaron características bioquímicas de las proteínas seminales en esta especie. No se hallaron diferencias entre las tres poblaciones basados en la cuantificación proteica. Las globulinas (82.4%) fueron la fracción mayoritaria seguida por las albuminas (13.9%), glutelinas (3.7%) y prolaminas (0.7%). Sólo las albuminas mostraron polimorfismo, hallándose 21 proteínas entre ~ 6.5 a ~45 kDa y tres perfiles electroforéticos diferentes, los cuales fueron compartidos entre las poblaciones. Se identificaron las leguminas y vicilinas en la fracción globulina. Las glutelinas mostraron proteínas de mismo peso molecular (PM) a las leguminas; y las prolaminas sólo una banda de bajo PM. La población de San Pablo fue completamente homogénea a diferencia de la población de Cajabamba que mostró la mayor diversidad genética seguida de Celendín. No fue posible diferenciar las poblaciones designadas como ecotipos Agroandino, Cajabamba y Celendino basados en el análisis de proteínas seminales.
Palabras clave: Aguaymanto; Solanaceae; diferenciacion genética; ecotipos; Perú; SDS-PAGE.
Abstract
The genetic diversity of three populations designated as ecotypes of golden berry (Physalis peruaviana) is studied using protein quantification and polymorphism of seed storage proteins (SSPs) by denaturating polyacrylamide gel electrophoresis (SDS-PAGE). As well, biochemical characteristics of seed proteins were identified. The populations were from San Pablo province (Agroandino ecotype), Celendín province (Celendino ecotype) and Cajabamba province (Cajabamba ecotype), all from Cajamarca Department. There was not difference among the three populations based on protein quantification. Globulins (82.4%) were the majority fraction followed for albumins (13.9%), glutelins (3.7%) and prolamins (0.7%). Only albumins showed polymorphism, showing 21 proteins between ~6.5 to ~45 kDa and three different electrophoretic profiles, which were share among the three populations. Legumins and vicilins were identified in globulin fraction. Glutelins showed proteins of same molecular weight (MW) to legumins; and prolamins only a band of low MW. San Pablo province population (Agroandino ecotype) was completely uniform, while Cajabamba population showed higher genetic diversity followed by Celendin population. Our results shows that, based on seed proteins analyses is not possible to distinguish the three populations designated as Agroandino, Cajabamba and Celendino ecotypes.
Keywords: golden berry; Solanaceae; genetic difference; ecotypes; Peru; SDS-PAGE.
Introducción
Physalis peruaviana L. conocida en el Perú como aguaymanto, uchuva en Colombia, uvilla en Ecuador y golden berry en inglés, es una planta herbácea nativa de los Andes, que ha ganado importancia económica en los últimos años debido a los componentes bioactivos presentes en su fruto, por lo cual ha sido considerada como un alimento funcional (Puente et al. 2011). El fruto es una baya esférica amarillo-naranja que contiene entre 150- 300 semillas pequeñas (Fischer et al. 2014). La demanda local, nacional y extranjera de este fruto se encuentra en estado creciente, tanto en su forma fresca como producto transformado (INDECOPI 2015), por lo que el conocimiento de su variabilidad genética permitiría adecuados programas de mejora genética y conservación de este cultivo. Es así que, Morillo et al (2011) señalan que, debido a que es un cultivo con gran demanda de exportación, es necesario el conocimiento de su variabilidad genética para las actividades de selección y premejoramiento; así mismo refieren que la utilización plena del potencial de cualquier cultivo depende de un conocimiento genético amplio, por lo que sería de suma importancia conocer, conservar y manejar la diversidad de fitorecursos con gran potencial como es el caso de P. peruviana.
Se entiende como ecotipo a aquella población que manifiesta una expresión fenotípica particular frente a un ambiente determinado; así, según la Biblioteca Nacional de Agricultura de los EE.UU. (NAL 2017) un ecotipo es un grupo subespecífico que se adapta genéticamente a un hábitat particular. En Physalis peruviana se ha determinado ecotipos diferenciados por caracteres morfológicos, tres ecotipos procedentes de Colombia, Kenia y Sudáfrica son lo que comúnmente se cultivan en todo el mundo; sin embargo, existen referenciados más de 80 ecotipos (Puente et al. 2011). En el Perú, el departamento de Cajamarca tiene la mayor área cultivada de aguaymanto. Los agricultores y las empresas agroindustriales, dedicadas a la producción de aguaymanto, hacen esfuerzos para tratar de identificar, seleccionar y cultivar distintos ecotipos, sin embargo, no se ha profundizado en conocimiento genético que sustente la selección de estos ecotipos.
Aunque, tradicionalmente la diversidad genética fue evaluada usando caracteres morfológicos, estos han sido superados por marcadores moleculares, como los bioquímicos y los basados en ADN (Schlötterer 2004). Uno de estos marcadores bioquímicos lo conforman las proteínas de reserva de semillas, que constituyen la fuente de aminoácidos para los procesos de síntesis que tienen lugar durante la germinación (Martin et al. 2010). Las proteínas de reserva de semillas (SSPs, por sus siglas en inglés) se clasifican en base a su solubilidad, así las albúminas son solubles en agua, las globulinas son solubles en soluciones salinas, las prolaminas son solubles en alcohol y las glutelinas son solubles en ácidos o alcalis (Osborne 1909). El análisis de SSPs ha sido utilizado para detectar diferencias entre especies y cultivares de Solanaceas (Vladova et al. 2000, Vladova et al. 2004), obteniendo perfiles electroforéticos distintos mediante electroforesis en gel de poliacrilamida desnaturante (SDS-PAGE). También, las SSPs han permitido la diferenciación entre ecotipos de tomates y frejoles (Mennella et al. 2001, Mennella et al. 2003). Además, se ha señalado que los perfiles electroforéticos de proteínas seminales tienen una alta estabilidad e independencia de las condiciones ecológicas (Florina 2012; Samah et al. 2015), por lo que también se le ha utilizado para diferenciar y caracterizar poblaciones (Ramirez et al 2016). Aunque los marcadores basados en ADN son reconocidos por proporcionar resultados más específicos, los marcadores bioquímicos que utilizan la SDS-PAGE tienen la ventaja de ser técnicas simples y de bajos requerimientos, adecuados para estudios iniciales a gran escala en los programas de mejoramiento genético de plantas (Gao et al. 2010). Al respecto, Galussi et al (2006) sostienen que el conocimiento del perfil de proteínas seminales de cada cultivar permitiría reconocer las posibles diferencias entre ellos; mientras que, Moscoso et al (2017) señalan que los estudios de fraccionamiento y caracterización de proteínas de semilla permiten identificar variedades con valor nutricional, lo que conlleva a estudios que permitirían desarrollar nutraceúticos ricos en péptidos bioactivos. Por otro lado, Gardiner y Forde (1992) demostraron que la SDS-PAGE de 200 semillas como mínimo produce un patrón electroforético representativo y reproducible para la población analizada.
En el presente trabajo se analiza la diversidad genética de tres poblaciones de aguaymanto, designados por sus productores como ecotipos Agroandino, Celendino y Cajabamba, procedentes del departamento de Cajamarca.
Material y métodos
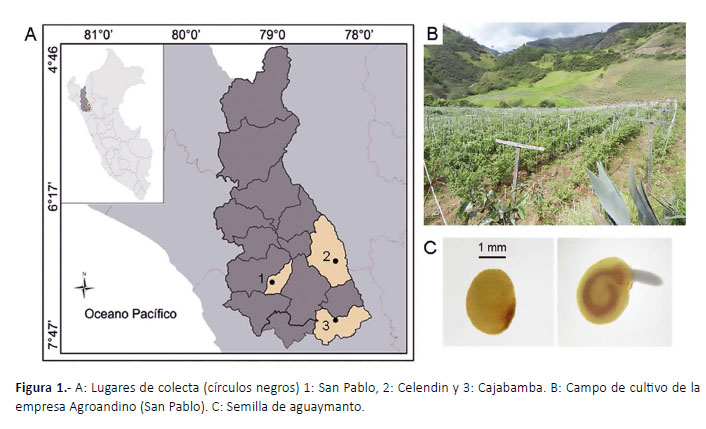
Material biológico.- Frutos maduros de treinta plantas de P. peruviana fueron colectados durante el año 2015, en tres poblaciones cultivadas de las provincias de San Pablo, Celendín y Cajabamba, en la región de Cajamarca (Fig. 1; Tabla 1). Las muestras fueron colectadas de los campos de cultivo de la empresa Agroandino-Peru (San Pablo), AZ Ingenieros EIRL (Celendin) y las proporcionadas por el Ing. Lenin Abanto (Cajabamba) quienes designan a sus poblaciones como ecotipos Agroandino, Celendino y Cajabamba respectivamente. Estos ecotipos son caracterizados por diferencias morfológicas, como la forma y color del fruto. Se colectaron diez frutos maduros de planta, cada fruto contiene aproximadamente 200 semillas, lo que hace un total de 100 frutos y 20000 semillas por población. El estado de madurez de los frutos fue catalogado de acuerdo a la norma técnica colombiana NTC 4580 para P. peruviana. Se enviaron muestras herborizadas al Museo de Historia Natural de la Universidad Nacional Mayor de San Marcos para su identificación taxonómica (Códigos: 222-USM-2015, 223-USM-2015, 224-USM-2015).
Extracción de las fracciones proteicas.- La extracción proteica se realizó por planta, así las semillas de los diez frutos de cada planta fueron mezcladas y se extrajo una muestra de 0.2 g, lo que equivale aproximadamente 250 semillas. Estas semillas fueron trituradas hasta obtener una harina fina con un mortero, a partir de la cual se obtuvo las cuatro fracciones de proteínas de almacenamiento seminal sucesivamente en base a la técnica de fraccionamiento por solubilidad según Osborne (1909).De esta manera, se extrajo primero la albúmina, para locual se mezcló la harina con agua destilada (1:5 p/v) durante 60 minutos y se centrifugó a 14500 rpm durante 20 minutos. El sobrenadante, fracción albúmina, se extrajoy almacenó a -20 ºC. El sedimento se lavó dos veces conagua destilada y se añadió la solución de extracción deglobulinas (NaCl 0.5 M, Tris HCl 50 mM pH 8), y se continuó con el mismo protocolo utilizado para la fracción anterior. De forma similar, se extrajeron la fracción glutelina (NaOH 0.1 M, SDS al 2%, 2-mercaptoetanol al 5%)y prolamina (etanol al 70%). Además, cada fracción (1:4v/v) se desgrasó con acetona /metanol (8:1 v/v) durante 2 horas a -20ºC, se centrifugó a 14500 rpm durante 15 minutos y se resuspendió el sedimento en SDS al 0.1%.La cuantificación de proteínas se realizó utilizando absorbancia a 280 nm en NanoDrop Lite (Thermo Scientific).La cuantificacion fue corregida de acuerdo con la fórmula de Waddell (Aitken & Learmonth 2002) y el rendimiento proteico (g proteína/100 g harina de semilla) se calculó utilizandolos datos de cuantificación y volumen de extracción.
Electroforesis de proteínas.- La electroforesis se realizó usando un equipo de electroforesis vertical (Mi-ni-PROTEAN, Bio-Rad Laboratories) utilizando un sistema discontinuo-denaturante (SDS-PAGE) según Laemmli (1970). Se prepararon geles de resolución (14%, pH 8.8) y empaquetamiento (4%, pH 8.6) utilizando una solución de acrilamida-bisacrilamida, tampón Tris-HCl, sodio dodecilsulfato (SDS), persulfato de amonio y tetrametil-etilendiamina (TEMED). Se analizaron condiciones reductoras y no reductoras utilizando tampón de carga 4X (Tris HCl 250 mM pH 6.8, glicerol al 25%, azul de bromofenol al 0.02%, SDS al 8% y mercaptoetanol al 8%) según Grabski et al (2001) con ligeras modificaciones. Para las condiciones no reductoras, sólo se evitó el uso de 2-mercaptoetanol. La mezcla fue calentada en baño maria (90°C) durante 5 minutos, se centrifugó y se usó inmediatamente para la separación electroforética. Los geles se tiñeron con Coomassie Blue R-250 de acuerdo con Lawrence et al. (2009).
Análisis de los datos.-Se evaluó la distribución normal (prueba de Shapiro-Wilk, P <0.05) y homogeneidad de varianzas (prueba de Levene, P <0.05) para la cuantificación de proteínas. La comparación de las medias entre los ecotipos se evaluó mediante ANOVA (paramétrica) o Kruskal-wallis (no paramétrica). Todas las pruebas estadísticas se realizaron utilizando el programa Rstudio v3.1.2 (R Core Team 2014).
Las comparaciones de patrones de proteínas fueroncualitativas, basadas en la movilidad de bandas en condiciones reductoras. Se elaboró una matriz binaria de presencia/ausencia de proteína. Las bandas débiles seasignaron como datos perdidos y se descartaron del análisis aquellas bandas con un porcentaje de datos perdidos superior al 10% de las treinta plantas analizadas. El índice de Shannon (H) se utilizó para analizar la diversidad genética (Lewontin 1972), el cual se calculó en dos niveles usando el programa Popgene v1.32 (Yeh et al. 1999), asi Hpop es la diversidad dentro de las poblaciones, y Hsp es la diversidad de la especie, con estos parámetros se determinó la proporción de diversidad dentro de las poblaciones (Hpop/Hsp), y la proporción de diversidad entre poblaciones (Hsp-Hpop/ Hsp) (King & Schaal 1989). Las masas moleculares de las proteínas se estimaron mediante comparación con el marcador de proteína de 6,5 a 200 kDa (SERVA Electrophoresis GmbH), y se realizó el análisis densitométrico usando el programa Gelanalyzer 2010a.
Resultados
Fraccionamiento proteico.- La extracción sucesiva de harina de semilla de P. peruviana con diferentes disolventes produjo albúmina, globulina, prolamina y glutelina en proporciones variables (Tabla 2). La globulina fue la mayor fracción proteica (82.4%), presentando el 1.08, 1.15, 1.12 g/100g en la población de San Pablo, Celendin y Cajabamba respectivamente. La albúmina aportó 13.9%, seguida de la glutelina (3.7%) y la prolamina (0.7%) que presentó niveles insignificantes. No hubo diferencias significativas en el contenido proteico de las fracciones entre las poblaciones (P < 0.05) (Tabla 2).
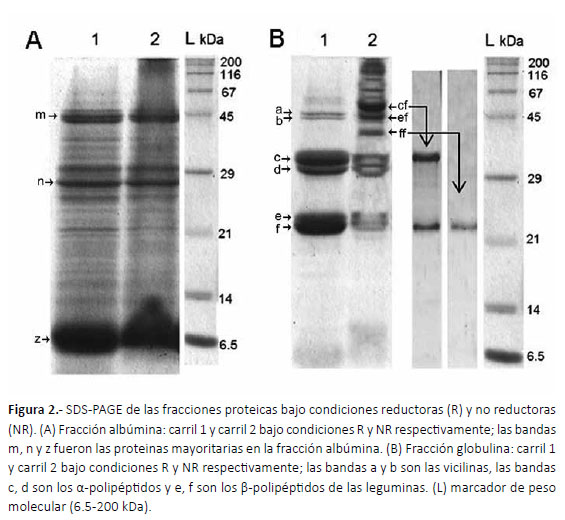
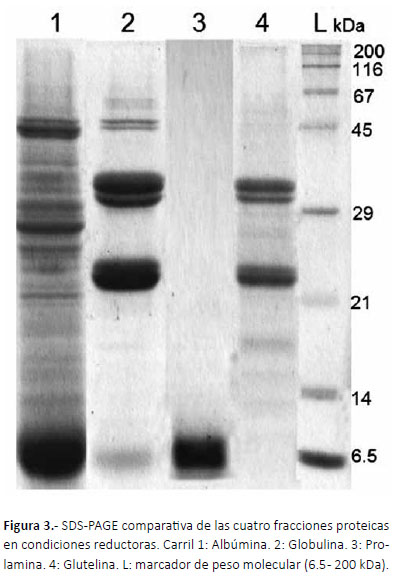
SDS-PAGE.- El SDS-PAGE de la fracción albúmina en condiciones reductoras mostró proteínas entre 6.5 a 45 kDa (Fig. 2 (a), carril 1). Bajo condiciones no reductoras se encontró un patrón similar (Fig. 2 (a), carril 2), pero variando sólo en la banda de bajo peso molecular 6.5 kD lo que indicaría la presencia de enlaces disulfuro. El análisis densitométrico reveló tres regiones principales, siendo la proteína de bajo peso molecular (banda z) la mayoritaria seguida por las bandas m y n. La fracción globulina en condiciones reductoras mostró 6 proteínas, que se visualizaron en tres grupos compuestos por dos proteínas cada uno (Fig. 2 (b), carril 1). Así, en el Grupo 1, las bandas a y b mostraron un peso molecular de aproxximadamente 45 kDa; las bandas c y d de ~34 y 32 kDa respectivamente en el grupo 2; y las bandas e y f presentaron ~24 y 22 kDa en el grupo 3, siendo estas últimas las bandas mayoritarias. Además, los grupos 2 y 3 en conjunto representaron el 83.4% de la fracción globulina. Por otro lado, en condiciones no reductoras, se encontraron aproximadamente 12 proteínas en esta fracción. Además, se identificaron subunidades de algunas bandas proteicas (cf, ef y ff en la Fig. 2 (b), carril 2) extrayéndolas del gel y resolviéndolas bajo condiciones reductoras. La fracción prolamina se resolvió en una banda de bajo peso molecular (6.5 kDa), con una ligera variación bajo condiciones no reductoras similares a la banda z en la fracción albúmina. Las glutelinas sólo se lograron extraer bajo condiciones reductoras, mostrando 4 bandas de PM similar a la fracción globulina (Fig. 3, carril 4).
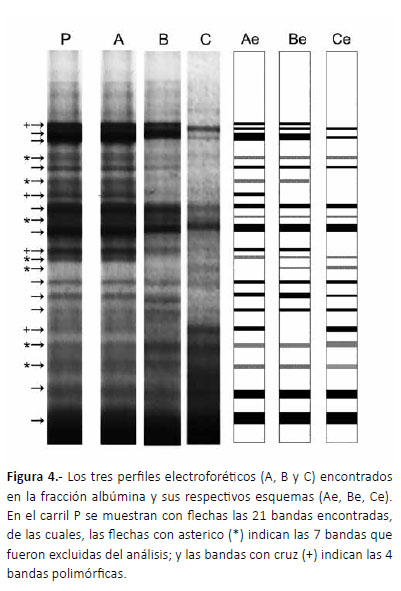
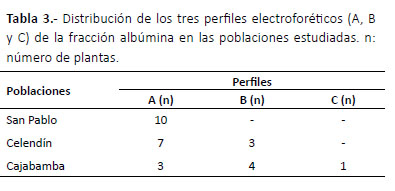
Diversidad genética.- Sólo la fracción albúmina presentó polimorfismo, observándose 21 bandas de las cuales, debido al porcentaje de datos perdidos, fueron analizadas 14; obteniéndose tres perfiles proteicos diferentes (perfil A, B y C en Fig. 4). Todas las plantas de San Pablo presentaron un solo perfil, el perfil A, el cual fue compartido con las otras poblaciones. La población de Celendín mostró dos perfiles (A y B), siendo el perfil A compartido con San Pablo y el perfil B con Cajabamba. La población de Cajabamba presentó los tres perfiles, siendo uno de ellos hallado solo en esta población (perfil C), además no se incluyó en el análisis el perfil electroforético de dos individuos pues presentaron un patrón atipico. Los perfiles A, B y C se hallaron en 20, 7 y 1 individuo respectivamente (Tabla 3). Las fracciones globulina y glutelina se resolvieron en un único perfil para todas las plantas analizadas. Debido a que solo la fracción albúmina presento polimorfismo, el índice de Shannon se calculó sobre la base de esta fracción. La población de San Pablo mostró diversidad nula (H = 0,00 ± 0,0), mientras Celendín y Cajabamba mostraron baja diversidad 0,1 ± 0,25 y 0.17 ± 0.29 respectivamente, siendo Cajabamba el más diverso. Respecto a la estructuración genética, la diversidad se halló distribuida principalmente dentro de las poblaciones (63%), mientras la diversidad entre estas fue 37%.
Discusión
Sogi et al. (2002) estudiando semillas de tomate (Solanaceae) encontraron una tendencia similar a nuestros resultados en aguaymanto, asi, la globulina fue la fracción mayoritaria (61%), seguida de albúmina (23.4%), glutelina (8.6%) y prolamina (7.1%). Además, Sheoram et al. (2005) utilizando electroforesis 2D y espectrometría de masas, también indican a las globulinas como las proteínas mayoritarias, pues identificaron una alta frecuencia de estas (46%), seguidas por las albúminas (2%) a partir de la identificación de 47 proteínas de una extracción total de proteínas. Sin embargo, en la sandía, una cucurbitácea, la composición proteica varía un poco, siendo la fracción mayoritaria la globulina seguida de glutelinas, albuminas y prolaminas (Wani et al., 2011). Estos resultados apoyan los hallazgos que especies de diferentes familias presentan distintas composiciones en sus fracciónes de proteína seminal.
En Perú no han sido reportados estudios de perfiles electroforéticos de proteinas seminales de aguaymanto, por lo que el presente trabajo sería un primer reporte. No hubo diferencias en la concentración de las fracciones proteicas entre las poblaciones, lo cual indica su alta similitud. Wani et al. (2011) estudiando fracciones proteícas en dos variedades certificadas de sandía, hallaron diferencias significativas en la concentración de las fracciones. Resultados similares fueron obtenidos por Tchiagam et al. (2011) en 10 variedades de frijoles. Teniendo en cuenta lo anterior, nuestros resultados indicarían que las tres poblaciones serian similares.
Varios autores mencionan a la albúmina 2S, como la proteína mayoritaria en la fracción albúmina (Salmanowicz & Przybylska 1994). La albúmina 2S es una proteína heterodimérica compuesta de una subunidad grande y una pequeña unidas por enlaces disulfuro (SS) (Shewry et al. 1995). Oguri et al. (2003) encontraron la subunidad grande y pequeña con pesos moleculares (PM) de alrededor de 8 y 4 kDa respectivamente en tomate, usando un sistema Tricina-SDS-PAGE. Por lo tanto, la banda de 6,5 kDa (Fig. 2 (a), banda z en el carril 1) correspondería a la albúmina 2S. Sin embargo, no fue posible distinguir las dos subunidades porque el sistema utilizado Glicine-SDS-PAGE no permite la resolución de proteínas de bajo peso molecular (Schägger 2006).
La fracción de globulina se compone de dos grupos en base a su coeficiente de sedimentación: 7s y 11s, llamados vicilinas y leguminas, respectivamente (Tai et al. 2001). Sheoram et al. (2005) encontró que las vicillinas poseen mayor PM que las leguminas en tomate. Además, Tai et al. (2001) identificaron una vicilina de 45 kDa en esta misma especie, por lo cual las bandas a y b (grupo 1) (Fig. 2 (b), carril 1) podrían considerarse vicilinas. Las leguminas están compuestas por dos polipéptidos llamados α y β que están unidos por enlaces S-S (Fukushima, 1991). Además, los α-polipéptidos usualmente tienen PM entre 30-40 kDa mientras que los β entre 20-25 kDa, asi los α-polipéptidos poseen mayor PM que los β (Alché et al. 2006). Por lo tanto, el grupo 2 (34 y 32 kDa) y 3 (24 y 22 kDa) serían los α y β-polipéptidos respectivamente. La separación de los dímeros ef, cf y ff (Figura 2 (b), carril 2) también confirmarían esta designación. Además, Chileh et al. (2010) indicaron a las leguminas como las globulinas mayoritarias, lo que estaría de acuerdo con nuestros resultados densitométricos.
Las prolaminas presentaron un PM similar a la banda z (~ 6,5 kDa) de la fracción albúmina, la cual ha sido designada como la albúmina 2s de aguaymanto. Actualmente, la albúmina 2S se ha clasificado como miembro de la superfamilia prolamina basándose en relaciones estructurales (Sherry et al. 2002). Por lo tanto, las prolaminas de P. peruviana podrían ser isoformas solubles en alcohol de la albúmina 2S. Esto podría deberse a que la albúmina 2S está codificada para familias multigénicas que generan varias isoformas (Oguri et al., 2003). La glutelina sólo se extrajo bajo condiciones reductoras como también mencionan Smith y Desborough (1987) en Solanaceaes. El perfil de esta fracción proteica fue similar al de las legumininas, lo que indicaria que las glutelinas de P. peruviana podría ser isoformas de este grupo de proteínas.
Las tres poblaciones mostraron baja diversidad genética, incluso San Pablo presentó nula. Los valores bajos de diversidad genética se generan por la presencia de una población estructurada, que puede ser generada en poblaciones cultivadas por dos razones principales: (1) el tipo de reproducción vegetal, principalmente en autógamas, lo cual no es el caso pues aguaymanto es principalmente alógama (Cely et al. 2015) y (2) la presencia de un programa de mejora genetica; esto último fue mencionado por los representantes de las empresas que cultivan los denominados ecotipos Agroandino y Celendino, quienes aplican el método de selección de masal. Por lo tanto, esta sería la razón de la baja diversidad genética encontrada. Aunque no se encontró variación en globulinas y glutelinas, su existencia no se descarta pues se ha reportado que la técnica SDS-PAGE oculta variabilidad que es posible revelar con otras técnicas como HPLC (Menella et al. 2001; Menella et al. 2003; Menella et al. 2005).
El análisis electroforético es una valiosa herramienta que permite separar y aislar proteínas, y los perfiles o patrones electroforéticos que se obtienen permiten detectar variabilidad en cuanto a tipos y concentraciones de proteínas en muestras de diferente origen (Abarca et al. 2002). Además, se ha demostrado que es posible distinguir variedades tradicionales, variedades certificadas y ecotipos basándose en un perfil electroforético específico (Driedger et al. 1993). Así, los tres perfiles encontrados en la fracción albúmina indicarían la existencia de tres posibles ecotipos, dos de ellos, perfil A y B, (Fig. 4) hallados en varios individuos (20 y 7 respectivamente) y por tanto se sugeriría su existencia, sin embargo, el tercero (perfil C, Fig. 4) solo fue hallado en un individuo de Cajabamba y, por tanto, no se podría confirmar su presencia. En trabajos previos se ha empleado a las glutelinas como un marcador proteico en la diferenciación de variedades en la familia solanácea (Smith & Desborough 1987; Menella et al. 2001), sin embargo, nuestros hallazgos indicarían que la fracción albúmina también debería ser considerada como un marcador a tener en cuenta para el análisis de variedades en aguaymanto. La presencia de variedades o ecotipos debe ser corroborada empleando técnicas de identificación morfológica, agronómica y ADN.
En resumen, se reporta por primera vez un análisis de los perfiles electroforéticos de proteínas seminales de tres poblaciones de aguaymanto provenientes del departamento de Cajamarca, los perfiles electroforéticos son compartidos y no muestran diferencia en la concentración proteica de semillas, lo que sería consecuencia de la baja diferenciación genética entre las poblaciones de San Pablo, Celendín y Cajabamba. Esto indicaría que aún no se han diferenciado como ecotipos. Por otro lado, la mayor homogeneidad de la población de San Pablo se debería a una mejor eficiencia en el programa de selección y mejora genética implementado por la empresa.
Agradecimientos:
Los autores agradecen a las empresas Agroandino y AZingenieros, y al Ing. Lenin Abanto por las facilidades durante la colecta y suministro de frutos de sus campos de cultivos de Physalis peruviana. Así también al Vice Rectorado de Investigación de la UNMSM por la subvención a los proyectos Nº 151001017 y 161001311.
Fuentes de financiamiento:
El presente trabajo se realizó gracias al financiamiento de la Universidad Nacional Mayor de San Marcos, Vicerrectorado de Investigación, Proyectos Nº 151001017 y 161001311.
Conflicto de intereses:
Los autores no incurren en conflictos de intereses.
Rol de los autores:
HB diseño el estudio; HB y YC recolección de las muestras; HB, YC y AL analizaron e interpretaron los datos; HB, YC, MS, y AL redactaron; HB, YC, MS, y AL intervinieron para la aprobación de la versión final; todos contribuyeron para la revisión crítica.
Aspectos éticos / legales:
Los materiales proceden de fuentes comerciales por lo que no se requieren permisos especificos.
Citación:
Bonilla H., Y. Carbajal, M. Siles y A. López. 2019. Diversidad genética de tres poblaciones de Physalis peruviana a partir del fraccionamiento y patrón electroforético de proteínas de reserva seminal. Revista peruana de biología 26(2): 243 - 250 (Julio 2019). doi: http://dx.doi.org/10.15381/rpb.v26i2.16370
Literatura citada
Abarca N., J.A. Avila, N. Naranjo, J. Herrera & M. Gonzales. 2002. Perfiles electroforeticos de las proteinas de semilla de pinos como caracteres taxonómicos. Revista Colombiana de Biotecnología 4 (2): 38-44. https://doi.org/10.15446/rev.colomb.biote [ Links ]
Aitken A. & Learmonth M.P. 2002. Protein Determination by UV Absorption, in: Walker J.M. (Ed) The Protein Protocols Handbook. Humana Press, pp. 3-6. https://doi.org/10.1385/1-59259-169-8:3 [ Links ]
Alché J., J. Jiménez-López, W. Wang, A. Castro-López & M. Rodríguez-García. 2006. Biochemical Characterization and Cellular Localization of 11 S Type Storage Proteins in Olive (Olea europaea L.) Seeds. Journal of agricultural and food chemistry 54: 5562–5570. https://doi.org/10.1021/jf060203s [ Links ]
Cely J., Rodríguez F., Almario C. & Meneses L. 2015. Variabilidad genética de parentales y poblaciones f1 inter e intraespecíficas de Physalis peruviana L. y P. floridana Rydb. Revista Brasileira de Fruticultura 37(1): 179-192. https://doi.org/10.1590/0100-2945-002/14 [ Links ]
Galussi A., Reinoso P., Zimmermann L., Soldá G. & Lui L. 2006. Identificación de cultivares de Lotus spp. por análisis de proteínas seminales. Revista de la Facultad de Agronomía, La Plata 106 (1): 21-26. [ Links ]
Chileh T., B. Esteban-García, D. Alonso & F. García-Maroto. 2010. Characterization of the 11S Globulin Gene Family in the Castor Plant Ricinus communis L. Journal of Agricultural and Food Chemistry 58: 272–281. https://doi.org/10.1021/jf902970p [ Links ]
Driedger D., B. Watts, A. Hussain & L. Elias. 1994. Isoenzyme and cotyledon protein variation for identification of black beans (Phaseolus vulgaris L.) with similar seed morphology. Euphytica 74: 27–34. https://doi.org/10.1007/BF00033763 [ Links ]
Fischer G., P. Almanza-Merchán & D. Miranda. 2014. Importancia y cultivo de la uchuva (Physalis peruviana L.). Revista Brasileira de Fruticultura 36: 01–15. https://doi.org/10.1590/0100-2945-441/13 [ Links ]
Florina F. 2012. Assessment of genetic diversity in a collection oflocal tomatoes by SDS-PAGE method. Journal of Horticulture, Forestry and Biotechnology 16: 133- 136. [ Links ]
Fukushima D. 1991. Structures of plant storage proteins and their functions. Food Reviews International 7:353–381. https://doi.org/10.1080/87559129109540916 [ Links ]
Galvez M.J., H.A. Castro & C.B. Villamil. 2009. Antigenic patterns of seed proteins in Opuntioideae (Cactaceae). Biochemical Systematics and Ecology 37: 91-97. https://doi.org/10.1016/j.bse.2008.12.004 [ Links ]
Gao L., W. Ma, J. Chen, K. Wang, J. Li, et al. 2010. Characterization and Comparative Analysis of Wheat High Molecular Weight Glutenin Subunits by SDS-PAGE, RP-HPLC, HPCE, and MALDI-TOF-MS. Journal of Agricultural and Food Chemistry 58: 2777–2786. https://doi.org/10.1021/jf903363z [ Links ]
Gardiner S.E., Forde M.B. 1992. Identification of Cultivars of Grasses and Forage Legumes by SDS-PAGE of Seed Proteins. In: Linskens H.F., Jackson J.F. (eds) Seed Analysis. Modern Methods of Plant Analysis, vol 14. Springer, Berlin, Heidelberg. https://doi.org/10.1007/978-3662-01639-8_3 [ Links ]
Grabski C. & R. Burgess. 2001. Preparation of Protein Samples for SDS-Polyacrylamide Gel Electrophoresis: Procedures and Tips. InNovations 13: 10-12. http://wolfson.huji.ac.il/purification/PDF/PAGE_SDS/NOVAGEN_Prepare_Sample_PAGE_SDS.pdf. (acceso 17.01.02). [ Links ]
INDECOPI (Instituto Nacional de Defensa de la Competencia y de la Protección de la Propiedad Intelectual) 2015. BIOPAT/PERU Tema: Aguaymanto. Año 1 Nº1. Enero 2015. (acceso 31.08.07) [ Links ]
Jean Baptiste N.T., M.B. Joseph, M. Antoine, Y. Nicolas & Y. Emmanuel. 2011. Genetic analysis of seed proteins contents in cowpea (Vigna unguiculata L. Walp.). African J. Biotechnol. 10: 3077–3086. https://doi.org/10.5897/AJB10.2469 [ Links ]
King, L.M. & B.A. Schaal. 1989. Ribosomal-DNA Variation and Distribution in Rudbeckia missouriensis. Evolution (N.Y). 43: 1117. https://doi.org/10.2307/2409592 [ Links ]
Laemmli U.K. 1970. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4. Nature 227: 680–685. https://doi.org/10.1038/227680a0 [ Links ]
Lawrence A. & H. Besir. 2009. (en linea). Staining of Proteins in Gels with Coomassie G-250 without Organic Solvent and Acetic Acid. J. Vis. Exp. https://doi.org/10.3791/1350 [ Links ]
Lewontin R.C. 1972. The Apportionment of Human Diversity, in: Evolutionary Biology. Springer US, Boston, MA. 381–398. https://doi.org/10.1007/978-1-46849063-3_14 [ Links ]
Martin M.A., S. Muñoz, F. Muñoz, M. Uribe, J. Molina, M. Herrera, L. Martín & J. Alvarez. 2010. Primeros resultados en el desarrollo de un marcador genético basado en las proteinnas de reserve en dos especies del género Nothofagus. Bosque (Valdivia), 31(3) 252-257. https://doi.org/10.4067/S07117-92002010000300010 [ Links ]
Mennella G., V. Sanaja & A. D’Alessandro. 2001. Tomato ecotype characterization by anionic exchange high performance liquid chromatography analysis of endosperm seed proteins. Acta Hortic. 453–457. https://doi.org/10.17660/ActaHortic.2001.546.61
Mennella G., V. Sanaja, A. D’Alessandro, M. Milone & D. Perrone. 2003. HPLC analyses of seed storage proteins reveal polymorphism in Italian common bean (Phaseolus vulgaris L.) ecotypes. Euphytica 134: 85–95. https://doi.org/10.1023/A:1026127004224
Mennella G., V. Sanaja, A. D’Alessandro & A. Desiderio. 2005. Biochemical characterization of white onion landraces (Allium cepa L.) through HPLC analysis of endosperm seed proteins. Euphytica 141: 169–180. https://doi.org/10.1007/s10681-005-6804-5
Morillo A.T., D.E. Villota, T.C. Lagos & H.R. Ordoñez. 2011. Caracterizaciòn morfológica y molecular de 18 introducciones de Uchuva Physalis peruviana L. de la colecciòn de la Universidad de Nariño. Ver.Fac.Nal.Agr.Medellìn 64(2): 6043-6053. https://doi.org/10.15446/rfnam [ Links ]
Moscoso G., Zavaleta A., Mujica A., Santos M. & Calixto R. 2017. Fraccionamiento y caracterización electroforética de las proteínas de la semilla de kañihua (Chenopodium pallidicaule Aellen). Revista chilena de nutrición 44 (2): 144-152 . https://doi.org/10.4067/S0717-75182017000200005 [ Links ]
NAL. 2017. (en línea). Tesauro y Glosario Agrícola. Acceso 31/08/2017 [ Links ]
NTC 4580. 1999. Norma Técnica Colombiana. Frutas frescas. Uchuva. Especificaciones. Instituto Colombiano de Normas Técnicas y Certificación (ICONTEC) [ Links ]
Oguri, S., M. Kamoshida, Y. Nagata, Y. Momonoki & H. Kamimura. 2003. Characterization and sequence of tomato 2S seed albumin: a storage protein with sequence similarities to the fruit lectin. Planta 216: 976-984. https://doi.org/10.1007/s00425-002-0950-y [ Links ]
Osborne T.B. 1909. The vegetable proteins, by Thomas B. Osborne. Longmans, Green, London. https://doi.org/10.5962/bhl.title.18912 [ Links ]
Puente L.A., C. Pinto-Muñoz, E. Castro, & M. Cortés. 2011. Physalis peruviana Linnaeus, the multiple properties of a highly functional fruit: A review. Food Research International 44: 1733–1740. https://doi.org/10.1016/j.foodres.2010.09.034 [ Links ]
Ramirez J., Herrera A., Aguirre C., Covarrubias J., Iturriaga G & Raya J. 2016. Caracterización de las proteínas de reserva y contenido mineral de semilla de melon (Cucumis melo L.) Revista Mexicana de Ciencias Agrícolas 7 (7): 1667- 1678. https://doi.org/10.29312/remexca.v7i7.158 [ Links ]
R Core Team. 2014. R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL http://www.R-project.org/. [ Links ]
Salmanowicz B.P. & J. Przybylska. 1994. Electrophoretic patterns of seed albumins in the Old-WorldLupinus species (Fabaceae): Variation in the 2S albumin class. Plant Systematics and Evolution 192: 67–78. https://doi.org/10.1007/BF00985909 [ Links ]
Samah S., E. Ventura-Zapata & E. Valadez-Moctezuma. 2015. Fractionation and electrophoretic patterns of seed protein of Opuntia genus. A preliminary survey as a tool for accession differentiation and taxonomy. Biochemical Systematics and Ecology 58: 187–194. https://doi.org/10.1016/j.bse.2014.12.005 [ Links ]
Schägger H. 2006. Tricine–SDS-PAGE. Nature Protocols 1: 16– 22. https://doi.org/10.1038/nprot.2006.4 [ Links ]
Schlötterer C. The evolution of molecular markers — just a matter of fashion? 2004. Nature Reviews Genetics. Vol. 5, p. 63-69. https://doi.org/10.1038/nrg1249 [ Links ]
Sheoran I.S., D. Olson, A. Ross, & V. Sawhney. 2005. Proteome analysis of embryo and endosperm from germinating tomato seeds. Proteomics 5: 3752–3764. https://doi.org/10.1002/pmic.200401209 [ Links ]
Shewry P.R. 1995. Seed Storage Proteins: Structures and Biosynthesis. Plant Cell Online 7: 945–956. https://doi.org/10.1105/tpc.7.7.945 [ Links ]
Shewry P.R. 2002. Cereal seed storage proteins: structures, properties and role in grain utilization. Journal of Experimental Botany 53: 947–958. https://doi.org/10.1093/jexbot/53.370.947 [ Links ]
Smith J.A. & S. Desborough. 1987. The endosperm seed protein Solin: biochemical characterization, induction by ABA and species-specific subunits. Theoretical and Applied Genetics 74. https://doi.org/10.1007/BF00247551 [ Links ]
Sogi D., M. Arora, S. Garg & A. Bawa. 2002. Fractionation and electrophoresis of tomato waste seed proteins. Food Chemistry 76: 449–454. https://doi.org/10.1016/S0308-8146(01)00304-1 [ Links ]
Tai S.S., T. Lee, C. Tsai, T.-J. Yiu & J. Tzen. 2001. Expression pattern and deposition of three storage proteins, 11S globulin, 2S albumin and 7S globulin in maturing sesame seeds. Plant Physiology and Biochemistry 39: 981–992. https://doi.org/10.1016/S0981-9428(01)01314-6 [ Links ]
Tchiagam J-BN, J.M. Bell, A.M. Nassourou, N.Y. Njintang, E. Youmbi. 2011. Genetic analysis of seed proteins contents in cowpea (Vigna unguiculata L. Walp.). African Journal of Biotechnology 10(16):3077–3086. https://doi.org/10.5897/AJB10.2469. [ Links ]
Vladova R., Pandeva R. & Petcolicheva K. 2000. Seed Storage Proteins in Capsicum Annuum Cultivars. Biologia Plantarum. 43: 291. https://doi.org/10.1023/A:1002776915689 [ Links ]
Vladova R., V. Tsanev & K. Petcolicheva. 2004. Seed Storage Proteins in Solanaceae and Cucurbitaceae Species. Biologia Plantarum 48: 601–603. https://doi.org/10.1023/B:BIOP.0000047159.91119.c5 [ Links ]
Wani A.A., D.S. Sogi, P. Singh, I.A. Wani, and U.S. Shivhare. 2011. Characterisation and functional properties of watermelon (Citrullus lanatus) seed proteins. Journal of the Science of Food and Agriculture 91:113-121. https://doi.org/10.1002/jsfa.4160 [ Links ]
Yeh C., R. Yang & T. Boyle. 1999. POPGENE v1.31. University of Alberta and Centre for International Forestry Research. Canada. [ Links ]
Presentado: 23/07/2018
Aceptado: 15/08/2018
Publicado online: 06/07/2019
Correspondencia:
*Autor para correspondencia
Henry Bonilla: hnrb109@gmail.com
Yajahaira Carbajal: nevenka.cg36@gmail.com
María Siles: msiles@unmsm.edu.pe
Alberto López: alopezs@unmsm.edu.pe
Otros datos de los autores / biografía:
ORCID Alberto López: 0000-0001-6070-5836
ORCID María Siles: 0000-0003-4956-8310