Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.26 no.2 Lima abr./jun. 2019
http://dx.doi.org/10.15381/rpb.v26i2.16383
NOTA CIENTÍFICA
Caracterización molecular de bacterias cultivables y no cultivables procedentes de pozas de lixiviación con cianuro
Molecular characterization of culturable and non-culturable bacteria from leaching pools with cyanide
Yacory Anahis Sernaque Aguilar 1 ORCID Id:0000-0002-8708-9487, Melitza Cornejo La Torre 2, Jerome Pierre Regard 4, Eric Louis Mialhe Matonnier 3
1. Universidad Nacional de Piura, Piura, Perú.
2. Cooperativa de trabajadores BIOTECOOP, Tumbes, Perú.
3. INCABIOTEC SAC, Tumbes, Perú.
4. WF Silva Ingenieros S.A.C., Lima, Perú.
Resumen
En el presente estudio, las comunidades bacterianas en muestras de suelo y agua, procedentes de pozas artesanales de lixiviación con cianuro, fueron caracterizadas por análisis dependientes e independientes de cultivo. Para la caracterización de la comunidad bacteriana cultivable, se emplearon técnicas clásicas de microbiología hasta la obtención de cepas puras, las cuales fueron identificadas a nivel molecular. Por otro lado, las comunidades bacterianas no cultivables fueron caracterizadas por secuenciación de próxima generación del gen ARNr 16S. La comunidad bacteriana cultivable estaba principalmente representada por los géneros Pseudomonas, Bacillus y Acinetobacter; mientras que las comunidades no cultivables, predominantes en muestras de suelo, fueron los filos Proteobacteria (12.91%), Firmicutes (11.32%), Actinobacteria (11.25%) y Bacteroidetes (10.16%). Por otro lado, en muestras de agua predominaron los filos Firmicutes (59.16%) y Actinobacteria (38.99%).
Palabras clave: Minería; pozas de lixiviación; cianuro; secuenciación de próxima generación; bacterias.
Abstract
In the present study, bacterial communities in soil and water samples, from artisanal leaching pools with cyanide, were characterized by dependent and independent culture analyzes. For the characterization of the culturable bacterial community, classical techniques of microbiology were used, until obtaining pure strains, which were identified at the molecular level. On the other hand, uncultured bacterial communities were characterized by next-generation sequencing of the 16S rRNA gene. The culturable bacterial community was mainly represented by the genera Pseudomonas, Bacillus and Acinetobacter; while the predominant uncultured communities, in soil samples, were the proteobacteria (12.91%), Firmicutes (11.32%), Actinobacteria (11.25%) and Bacteroidetes (10.16%). On the other hand, in water samples, the edges of Firmicutes (59.16%) and Actinobacteria (38.99%) predominated.
Keywords: Mining; leaching pools; cyanide; next-generation sequencing; bacteria.
Introducción
La minería artesanal se define como un sistema informal y no regulado de minería en pequeña escala. Generalmente, emplea tecnologías muy simples, y no hay planificación para la rehabilitación después del cierre de la operación minera. El resultado más visible, es la destrucción del medio ambiente a través de la contaminación por metales pesados de la tierra y el agua que rodea la operación de minería artesanal, debido a la eliminación de residuos de rocas y relaves (Krisnayanti et al.2012).
Existen sistemas de ingeniería convencionales para rehabilitar terrenos mineros degradados, pero son costosos y se basan en tecnología que puede no estar disponible en las peores áreas de minería artesanal (Krisnayanti et al.2012).
En los últimos años, la biotecnología ha experimentado grandes avances que se han visto reflejados en múltiples aplicaciones industriales. Los métodos independientes de cultivo, especialmente el análisis metagenómico de las comunidades microbianas, se han convertido en herramientas útiles en la detección e identificación de nuevas especies, genes o enzimas, importantes para la biotecnología industrial, además de proporcionar información valiosa sobre la función, estructura, interacción y dinámica de las comunidades microbianas en un ecosistema (Montaña 2015).
Estudios dependientes e independientes de cultivo sobre las minas de oro en Japón (Inagaki et al. 2003, Nunoura et al. 2005), Sudáfrica (Takai et al. 2001, Baker et al. 2003) y Estados Unidos (Nemergut et al. 2004) han mostrado poblaciones microbianas activas, compuestas de diversos grupos de microorganismos con propiedades fisiológicas inusuales, las cuales pueden ser aplicadas en procesos de biorremediación.
El presente estudio tuvo como objetivo identificar y analizar molecularmente, mediante análisis dependientes e independientes de cultivo, las comunidades bacterianas en muestras de suelo y agua de pozas artesanales de lixiviación con cianuro. Los resultados mostraron que la comunidad bacteriana cultivable estuvo representada por los géneros Pseudomonas, Bacillus, Acinetobacter, entre otros; mientras que los filos Proteobacteria y Firmicutes, fueron los más representativos de la comunidad no cultivable.
Material y métodos
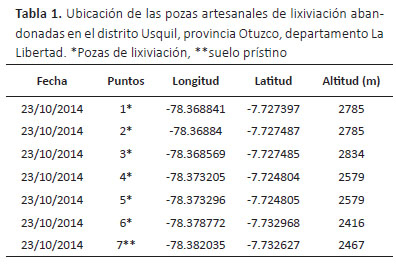
Fuente de los aislados de bacterias.- En el presente estudio las muestras de suelo y agua fueron colectadas de seis pozas artesanales de lixiviación abandonadas, en área próxima al proyecto minero "Yerba buena" de la empresa WF SILVA INGENIEROS SRL, ubicadas en distrito Usquil, provincia Otuzco, departamento La Libertad. Adicionalmente, se colectó una muestra de suelo y agua de un sitio de referencia que no había sido afectado por actividad minera y representaba la situación más próxima a un suelo y agua prístina, en el que se determinó la ausencia de cianuro. En cada punto de colecta se evaluaron diversos parámetros de campo como temperatura, pH y concentración de cianuro, empleando el medidor portátil HANNA HI 98185-01, pH/ORP/ISE y el fotómetro portátil HANNA HI 96714, respectivamente. Así mismo, se registraron las coordenadas de cada punto muestreado, de las seis pozas de lixiviación (1 - 6) y del suelo prístino (7) (Tabla 1).
Aislamiento y condiciones de cultivo.- Las bacterias fueron aisladas empleando el medio general Agar tripticasa de soya (TSA), y el medio específico Cetrimide fue usado para especies del género Pseudomonas. Para la siembra de las muestras de suelo y agua se realizaron diluciones sucesivas colocando 1mL de cada muestra de agua en tubos estériles (FALCON), que contenían 9 mL de agua destilada estéril. Luego, las diluciones 10-1, 10-2 y 10-3 fueron sembradas en placas e incubadas a 37 °C por 24 - 48 horas. Las cepas bacterianas fueron replicadas mediante siembras sucesivas hasta la obtención de cepas puras, luego fueron conservadas con glicerol al 15% y almacenadas a -20 °C.
Extracción de ADN genómico bacteriano.- El cultivo bacteriano (1.2 mL) fue sedimentado en un tubo de microcentrifuga a 10000 rpm por 2 min. Posteriormente, se eliminó el sobrenadante y resuspendió el sedimento en 500 µL de Buffer Fosfato Salino (PBS 1X) esterilizado. Luego, se centrifugó a 10000 rpm por 2 min., se eliminó el sobrenadante, y el pellet fue resuspendido en 200 µL de la solución TE (Tris 10 mM / 1 mM EDTA). Después, para lisar las células se llevó a ebullición por 10 min. e inmediatamente se colocó sobre hielo por 5min. y se centrifugó a 10000 rpm por 1min. Luego, se transfirió el sobrenadante a otro microtubo y se trató con 1 µL ARNasa agregando a cada muestra e incubó a 65 °C por 15 minutos. Finalmente, pasado el tiempo de incubación se conservó el ADN a -20 °C, para investigaciones posteriores.
Determinación de secuencias del gen ADNr 16S.- La amplificación del gen ARNr 16S, de las bacterias aisladas, se realizó mediante la técnica de reacción en cadena de la polimerasa (PCR), empleando los primers 27F (5´AGAGTTTGATCMTGGCTCAG 3´) y 1492R (5´ TACGGYTACCTTGTTACGACTT 3´). Las reacciones fueron realizadas en un volumen final de 25 µL, 2.5 µL de Buffer 10X, 2.5 µL de MgCl2 50 mM, 0.1 µL de Taq polimerasa (5U/ µL) (Thermo Fisher scientific), 0.5 µL de dNTPs a 10 mM, 0.6 µL de cada primer a 15 pmol, 2 µL de ADN extraído y agua ultra pura. La amplificación de los productos fue verificada por electroforesis utilizando un gel de agarosa de 1.5%.
Extracción de ADN metagenómico a partir de suelo y agua.- La caracterización de comunidades bacterianas de suelo y de agua prístinos y contaminadas por el uso de cianuro, fue realizada empleando ADN total extraído directamente de las muestras, usando protocolos previamente estandarizados (Tekere et al. 2011) y el kit ADN PowerSoil (Mo Bio Laboratories, USA) siguiendo las instrucciones del proveedor. Luego, los análisis moleculares dirigidos a la región variable V4 del gen 16S rRNA (Gong et al. 2013), se realizaron mediante "secuenciación de próxima generación" (Ion Torrent, MR DNA).
Identificación de especies.- Las secuencias obtenidas fueron ensambladas empleando el software MEGA v6.0 y luego comparadas con la base de datos del GenBank, del Centro Nacional de Información Biotecnológica (NCBI), usando la herramienta BLASTN. Se consideraron sólo aquellas secuencias que presentaban un porcentaje de identidad superior a 98% con la secuencia sometida. Así mismo, se empleó la herramienta bioinformática Microsoft Excel para el procesamiento de datos, elaboración de gráficos y tablas.
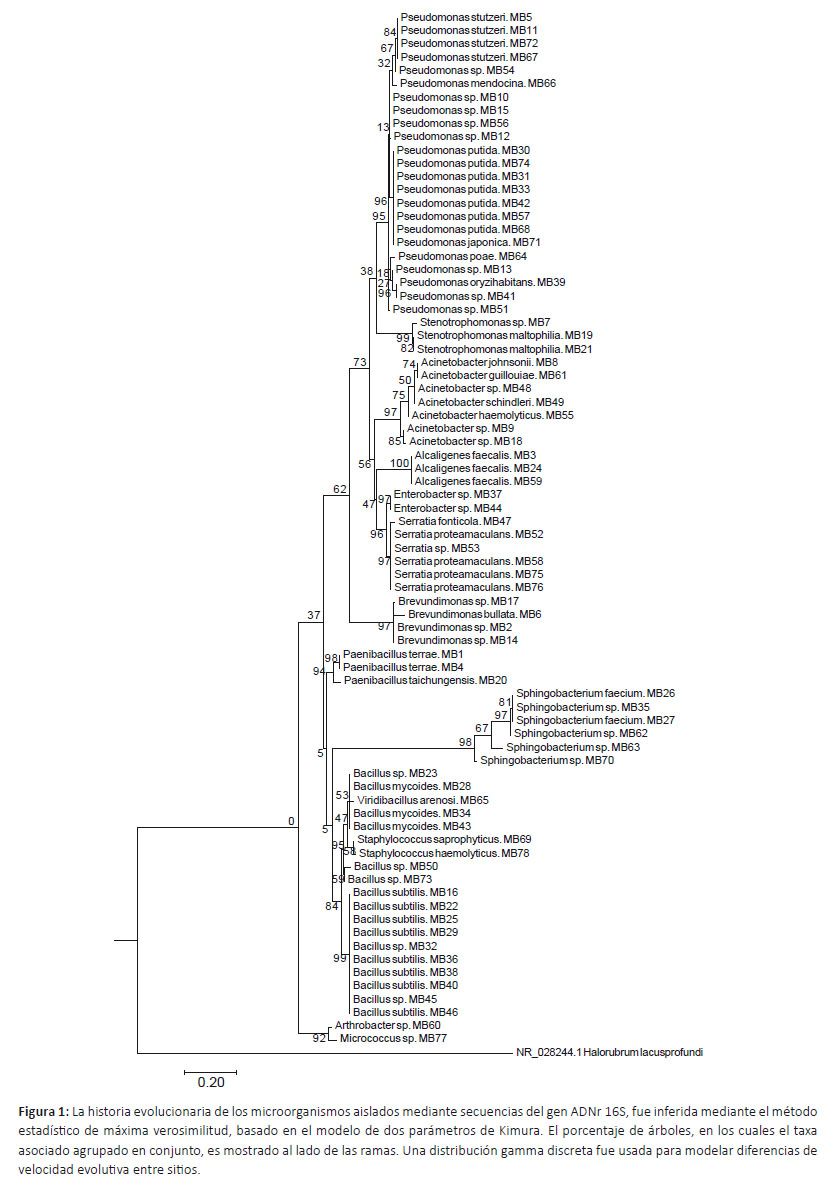
Análisis filogenético.- El análisis filogenético y el alineamiento fue realizado utilizando el software MEGA v6.0 y las secuencias del gen ADNr 16S. La historia evolucionaria fue inferida empleando el método estadístico máxima verosimilitud (máximum likelihood) basado en el modelo de dos parámetros de Kimura con 1000 réplicas de bootstrap. Halorubrum lacusprofundi (número de accesión NR_028244.1) fue empleada como grupo externo para la elaboración del árbol filogenético dirigido al gen ADNr 16S.
Resultados
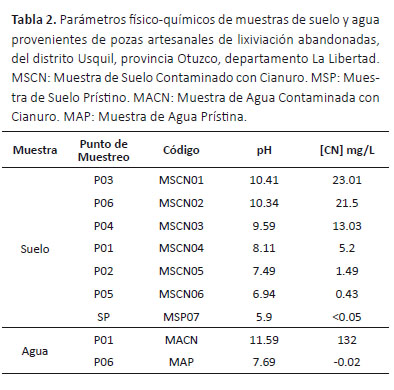
Parámetros físico-químicos de las muestras de suelo y agua.- En las muestras suelo evaluadas la concentración de cianuro fue de 23.01 mg/L y 21.5 mg/L en dos puntos muestreados, mientras que en el resto de muestras de suelo las concentraciones fueron de 13.03mg/L, 5.2mg/L y 1.49mg/L. Por otro lado, la muestra de agua presentó una concentración de cianuro de 132 mg/L y un pH de 11.59. Los valores de pH obtenidos de las muestras de suelo se encontraron en un rango de 6 - 10 (Tabla 2).
Identificación molecular de bacterias aisladas.- El estudio a nivel molecular de muestras de pozas de lixiviación ubicadas en La Libertad, permitió identificar un total de 78 bacterias, de las cuales sólo 54 corresponden a cepas con diferentes números de accesión (Tabla 3) como Pseudomonas, Bacillus, Acinetobacter, Sphingobacterium, Alcaligenes, Serratia, Enterobacter, entre otros. Así mismo, el 69% de la comunidad bacteriana identificada corresponde al grupo de bacterias Gram negativas, mientras que el 31% pertenece a bacterias Gram positivas. La identificación de las secuencias de nucleótidos de los aislados, proporcionó entre 90 ⎼ 100% de identidad para bacterias al compararse con la base de datos del GenBank. El árbol filogenético para bacterias es mostrado en la Figura 1.
Análisis metagenómicos de muestras de suelos contaminados con cianuro y derivados tóxicos.- Los análisis metagenómicos de las muestras de suelos contaminados con cianuro mostraron 22 filos bacterianos identificados, predominando los filos Proteobacteria (12.91%), Firmicutes (11.32%), Bacteroidetes (10.16%), Actinobacteria (11.25%), Verrucomicrobia (8.20%) y con abundancias menores se encuentraron Chloroflexi (0.73%), Synergistetes (0.59%), Thermodesulfobacteria (0.49%), Chlorobi (0.33%), Fibrobacteres (0.001%) y Gemmatimonadetes (0.001%); además, se reportaron también secuencias bacterianas no clasificadas, las cuales representan el 14.48% del total (Tabla 4).
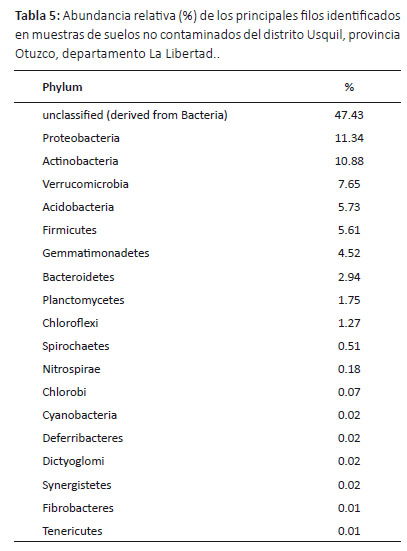
Análisis metagenómicos de suelos no contaminados con cianuro.- En las muestras de suelos no contaminados con cianuro 19 filos fueron identificados por metagenómica directa. Las secuencias no clasificadas derivadas de bacterias representan el 47.42%, Proteobacteria (11.33%), Actinobacteria (10.88%) y Verrucomicrobia (7.65%). Así mismo, se determinaron otros filos con menor abundancia desde 6% hasta menos del 1% (Tabla 5).
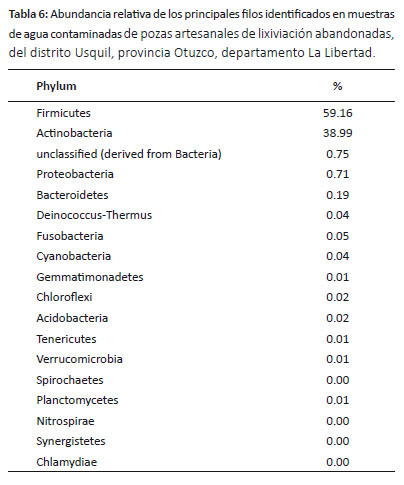
Análisis metagenómicos de agua contaminada con cianuro.- En las muestras de agua contaminados con cianuro 17 filos fueron identificados, siendo Firmicutes (59.16%), Actinobacteria (38.99%) los filos con mayor abundancia. Por otro lado, en los filos con menor abundancia se encuentran Proteobacteria (0.71%), Bacteroidetes (0.19%), Fusobacteria (0.05%), Deinococcus-Thermus (0.04%), Cyanobacteria (0.04%), Acidobacteria (0.02%), Chloroflexi (0.02%), Gemmatimonadetes (0.01%), Tenericutes (0.01%), Planctomycetes (0.01%), Verrucomicrobia (0.01%), Spirochaetes (0.003%), Nitrospirae (0.002%), Synergistetes (0.001%), Chlamydiae (0.0006%) (Tabla 6).
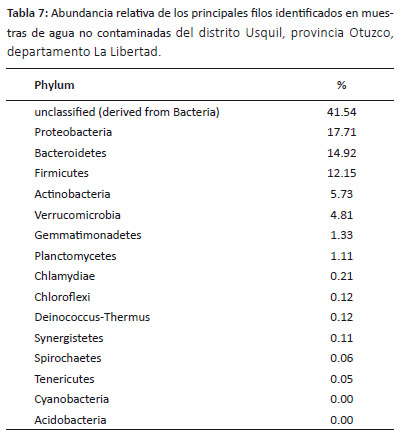
Análisis metagenómicos de agua no contaminada con cianuro.- La diversidad bacteriana en muestras de agua no contaminada con cianuro estuvo representada por los filos Proteobacteria y bacteroidetes, con una abundancia de 17.71% y 14.92%, respectivamente. Por otro lado, los filos presentes en menor abundancia fueron Actinobacteria (5.73%) y Verrucomicrobia (4.81%) (Tabla 7).
Discusión
Los ambientes contaminados con cianuro han sido ampliamente estudiados a fin de obtener la información necesaria para la implementación de estrategias de biorremediación (Adams et al. 2012, Akcil & Mudder 2003, Dasha et al. 2009, Deloya 2012, Luque et al. 2016). Varios microorganismos han sido evaluados y aislados de suelos procedentes de minería, siendo los géneros más representativos Exiguobacterium, Aeromonas, Bacillus, Pseudomonas, Escherichia, y Acinetobacter (Anderson & Cook 2004). Por otra parte, Alcaligenes sp. perteneciente a la familia Alcaligenaceae y clasificado como 𝛽-Proteobacteria, ha sido reportado como degradador y habitante nativo de lugares contaminados con cianuro (Baxter & Cummings 2006, Bhalla et al. 2012) y metales pesados (Abbas et al. 2015). En el presente trabajo, dentro de la comunidad bacteriana cultivable, se han identificado tanto los géneros más representativos de zonas contaminadas con cianuro (Pseudomonas, Alcaligenes, Bacillus y Acinetobacter), como géneros bacterianos poco reportados en otros estudios (Sphingobacterium, Paenibacillus y Stenotrophomonas), lo que permite conocer más acerca de las comunidades bacterianas cultivables presentes en estas zonas contaminadas, estudiar a mayor profundidad su función en estos nichos, y revelar sus posibles aplicaciones en procesos de tratamiento de zonas afectadas por la actividad minera.
Así mismo, las bacterias presentes en lodo activado son generalmente Gram-negativas e incluyen Pseudomonas, Achromobacter, Flavobacterium, Nocardia, Bdellovibrio y Mycobacterium (Sharifi et al. 2001). El género Pseudomonas es considerado entre los mejores para la degradación de cianuro debido a su versatilidad metabólica, amplia capacidad oxidativa y una alta adaptabilidad para utilizar una gran variedad de sustancias químicas como fuente de carbono y nitrógeno (Quispe et al. 2011). En el presente estudio, también se ha identificado un mayor porcentaje de bacterias Gram negativas (69%) principalmente del género Pseudomonas, cuya tolerancia a ambientes alcalinos con muy poca cantidad de nutrientes (Akcil & Mudder 2003, Chung 2008), así como su capacidad para degradar cianuro, explicaría su mayor abundancia en las pozas de lixiviación evaluadas. Solo el 31% de la comunidad identificada fueron bacterias Gram positivas pertenecientes a los géneros Staphylococcus, Micrococcus, Chryseomicrobium y Bacillus.
Por otro lado, la metagenómica se resalta como una herramienta importante para la biorremediación (Gabor et al. 2007, George et al. 2011), permitiendo entender la estructura, interacción y potencial metabólico de las comunidades microbianas no cultivables, capaces de degradar cianuros (Wang et al. 2015, Kantor et al. 2015, Luque et al. 2016). Los miembros del filo Proteobacteria son frecuentemente encontrados en suelos de diferentes tipos de minas (Moreels et al. 2008, Khan et al. 2013, Li et al. 2014). Estudios sobre sistemas de tratamientos de aguas residuales conteniendo cianuro han reportado que la biodegradación exitosa de este contaminante fue asociada con miembros de Proteobacteria, su importancia se debe a la vía más eficiente de degradación de cianuro, hidrolítica, la cual es comúnmente encontrada en este filo (Shin et al. 2019). Por otro lado, las bacterias responsables de la biolixiviación, mesófilas o termófilas moderadas, se distribuyen principalmente en los grupos filogenéticos: Proteobacteria, Firmicutes, y Actinobacteria (Clark & Norris 1996, Norris et al. 2000). El filo Firmicutes es uno de los grupos bacterianos más comunes en ambientes extremos, ya que muchos de sus miembros tienen la capacidad de tolerar una amplia gama de condiciones desfavorables (Khan et al. 2013).
Dentro de la gran diversidad, también se reportaron los filos Proteobacteria y Firmicutes como los más abundantes en las muestras de suelo y agua evaluadas. Su participación en procesos de biorremediación y biolixiviación, explica la importancia de identificar y estudiar estos filos. Por otro lado, el filo Actinobacteria fue reportado en menor abundancia, con 11.25% y 38.99% en muestras de suelo y agua, respectivamente. Considerando que este filo contiene pocos miembros cultivables (Janssen 2006), sus roles fisiológicos en ambientes naturales, incluso en ecología de mina, sigue siendo desconocido (Rastogi et al.2009).
El presente estudio muestra la diversidad bacteriana cultivable y no cultivable presente en zonas contaminadas con cianuro producto de la actividad minera en la región La Libertad, conocimiento que coadyuvara a la implementación de técnicas de tratamiento en zonas afectadas por la actividad minera en nuestro país. Sin embargo, aún se requieren estudios que permitan conocer a mayor profundidad la función y potencial metabólico de estos microorganismos para que puedan ser empleados en diferentes campos de investigación.
Agradecimientos:
Se agradece el apoyo brindado por la Concesionaria WF. Silva Ingenieros S.R.L durante la obtención de muestras y ejecución del proyecto; así mismo a la empresa INCABIOTEC SAC por poner a disposición las instalaciones del laboratorio de biología molecular.
Rol de los autores:
YASA contribuyó en el desarrollo conceptual y experimental, MCLT realizó la colección de muestras, JPR asesoró en la toma de muestras, ELMM participó en la concepción del proyecto, YASA, MCLT, JPR, ELMM revisaron, redactaron y aprobaron el manuscrito.
Conflicto de intereses:
Los autores no incurren en conflictos de intereses.
Fuentes de financiamiento:
Esta investigación fue financiada por el proyecto titulado "Bioremediación de suelos y aguas afectados por actividades mineras auríferas y argentíferas en la Región La Libertad mediante el uso de un consorcio de bacterias y/o hongos nativos productores de enzimas degradadoras del cianuro y de sus derivados tóxicos", con código PIPEI-7-P-286-073-13 (Fincyt e Innóvate Perú, la concesionaria WF. Silva Ingenieros S.R.L y la Universidad Nacional de Tumbes).
Aspectos éticos / legales:
El material biológico y el proceso de investigación no requirieron permisos específicos.
Literatura citada
Abbas S., I. Ahmed, I. Iida, et al. 2015. A heavy metal tolerant novel bacterium, Alcaligenes pakistanensis sp. nov., isolated from industrial effluent in Pakistan. Antonie van Leeuwenhoek. 108(4): 859–870. http://doi.org/10.1007/s10482-015-0540-1 [ Links ]
Anderson C. R. & G.M. Cook. 2004. Isolation and characterization of arsenate reducing bacteria from arsenic-contaminated sites in New Zealand.Current microbiology. 48 (5): 341-347. https://doi.org/10.1007/s00284-003-4205-3 [ Links ]
Adams D.J., J. Van Komen & T.M. Pickett. 2012. Biological cyanide degradation. In: C. Young, ed. Cyanide: Social, Industrial and Economic Aspects. The Metals Society, Warrendale, PA. Pp. 203–213. [ Links ]
Akcil A. & T. Mudder. 2003. Microbial destruction of cyanide wastes in gold mining: process Review. Biotechnology Letters. 25 (6): 445- 450. https://doi.org/10.1023/A:1022608213814 [ Links ]
Bhalla T. C., N. Sharma & R.K. Bhatia. 2012. Microbial Degradation of Cyanides and Nitriles. In: A. Prakash, T. Satyanarayana and B. N. Johri, eds. Microorganisms in Environmental Management. Springer, Dordrecht. Pp.569–587 [ Links ]
Baxter J. & S.P. Cummings. 2006. The current and future applications of microorganism in the bioremediation of cyanide contamination. Antonie van Leeuwenhoek. 90(1): 1–17. http://doi.org/10.1007/s10482-006-9057-y [ Links ]
Baker B.J., D.P. Moser, B.J. MacGregor, et al. 2003. Related assemblages of sulphate-reducing bacteria associated with ultradeep gold mines of South Africa and deep basalt aquifers of Washington State. Environ. Microbiol. 5 (4). 267-277. https://doi.org/10.1046/j.1462-2920.2003.00408.x [ Links ]
Clark D.A. & P.R. Norris. 1996. Acidimicrobium ferrooxidans gen. nov., sp. nov.: mixed-culture ferrous iron oxidation with Sulfobacillus species. Microbiology. 142 (4): 785-790. https://doi.org/10.1099/00221287-142-4-785 [ Links ]
Chung B. 2008. Control de los contaminantes químicos en el Perú. Revista Peruana de Medicina Experimental y Salud Publica 25(4): 413-418. http://dx.doi.org/10.17843/rpmesp.2008.254.1307 [ Links ]
Dasha R.,A.Gaur & C.Balomajumder.2009.Cyanide in industrial wastewaters and its removal: a Review on biotreatment. Journal of Hazardous Materials. 163(1):1–11. https://doi.org/10.1016/j.jhazmat.2008.06.051 [ Links ]
Deloya A. 2012. Tratamiento de desechos del cianuro por bioremediación. Tecnología en Marcha. 25(2): 61-72. doi:10.18845/tm.v25i2.317 [ Links ]
Gabor E., K. Liebeton, F. Niehaus, et al. 2007. Updating the metagenomics toolbox. Biotechnology Journal. 2(2):201–206. https://doi.org/10.1002/biot.200600250 [ Links ]
George I.F., E. Bouhajja & S.N. Agathos. 2011. Metagenomics for Bioremediation. En Comprehensive Biotechnology, de M Moo-Young. Elsevier, Amsterdam. Pp. 47-57. [ Links ]
Gong J. 2013. Metagenomic technology and genome mining. Microbiol Biotechnol. 97 (15): 6603–6611. https://doi.org/10.1007/s00253-013-4932-8 [ Links ]
Inagaki F., K. Takai, H. Hirayama, et al. 2003. Distribution and phylogenetic diversity of the subsurface microbial community in a Japanese epithermal gold mine. Extremophiles. 7(4):307-317. https://doi.org/10.1007/s00792-003-0324-9 [ Links ]
Janssen P.H. 2006. Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA genes. Appl. Environ. Microbiol. 72(3): 1719–1728. https://doi.org/10.1128/AEM.72.3.1719-1728.2006 [ Links ]
Kantor S., W. van Zyl, P. van Hill, et al. 2015. Bioreactor microbial ecosystems for thiocyanate and cyanide degradation unravelled with genome-resolved metagenomics.Environmental microbiology. 17(12):4929–4941. https://doi.org/10.1111/1462-2920.12936 [ Links ]
Khan N.H., F.V. Bondici, P.G. Medihala, et al. 2013. Bacterial Diversity and Composition of an Alkaline Uranium Mine Tailings-Water Interface. Journal of Microbiology. 51(5): 558–569. https://doi.org/10.1007/s12275-013-3075-z [ Links ]
Krisnayanti B.D.,C.W. Anderson, W.H. Utomo, et al.2012. Assessment of environmental mercury discharge at a four-year-old artisanal gold mining area on Lombok Island, Indonesia. Journal of Environmental Monitoring. 14(10):2598-607. https://doi.org/10.5539/jas.v2n2p202 [ Links ]
Li Y., H. Wen, L. Chen, et al. 2014. Succession of Bacterial Community Structure and Diversity in Soil along a Chronosequence of Reclamation and Re- Vegetation on Coal Mine Spoils in China. PLoS ONE. 9(12): e115024 https://doi.org/10.1371/journal.pone.0115024 [ Links ]
Luque V., C. Moreno & M. Roldán. 2016. Biodegradation of cyanide waste from mining and jewellery industries. Current Opinion in Biotechnology.38:9-13. https://doi.org/10.1016/j.copbio.2015.12.004 [ Links ]
Moreels D., G. Crosson, C. Garafola, et al. 2008. Microbial community dynamics in uranium contaminated subsurface sediments under biostimulated conditions with high nitrate and nickel pressure. Environ. Sci. Pollut. Res. 15(6):481–491. https://doi.org/10.1007/s11356-008-0034-z [ Links ]
Montaña J.S. 2015. Aproximación metagenómica para la identificación de enzimas lipolíticas en suelo de bosque alto andino del parque nacional natural los nevados. Tesis para optar el grado de doctor. Facultad de ciencias Pontificia Universidad Javeriana. http://hdl.handle.net/10554/17002 [ Links ]
Nemergut D.R., A.P. Martin & S.K. Schmidt. 2004. Integron diversity in heavy-metal-contaminated mine tailings and inferences about integron evolution. Appl. Environ. Microbiol. 70 (2). 1160-1168. https://doi.org/10.1128/AEM.70.2.1160-1168.2004 [ Links ]
Norris P.R., N.P. Burton & N.A. Foulis. 2000. Acidophiles in bioreactor mineral processing. Extremophiles. 4(2):71-6. https://doi.org/10.1007/s007920050139 [ Links ]
Nunoura T., H. Hirayama, H. Takami, H. Oida, et al. 2005. Genetic and functional properties of uncultivated thermophilic crenarchaeotes from a subsurface gold mine as revealed by analysis of genome fragments. Environ. Microbiol. 7(12). 1967-1984. https://doi.org/10.1111/j.1462-2920.2005.00881.x [ Links ]
Quispe L., M. Arteaga, E. Cardenas, et al. 2011. Eliminación de cianuro mediante sistema combinado UV/H2O2/Ti2. Revista Boliviana de química. 28 (2):113-118. [ Links ]
Rastogi G., L.D. Stetler, B.M. Peyton, et al. 2009. Molecular analysis of prokaryotic diversity in the deep subsurface of the former Homestake gold mine, South Dakota, USA. The Journal of Microbiology. 47(4): 371-384. https://doi.org/10.1007/s12275-008-0249-1 [ Links ]
Sharifi-Yazdi M. K., C. Azimi & M.B. Khalili. 2001. Isolation and identification of bacteria presents in the activated sludge unit, in the treatment of industrial waste water. Iranian Journal of Public Health. 30(3-4): 91-94. [ Links ]
Shin D., J.Park, H. Park, et al. 2019. Key Microbes and Metabolic Potentials Contributing to Cyanide Biodegradation in Stirred-Tank Bioreactors Treating Gold Mining Effluent. Mineral Processing and Extractive Metallurgy Review. 40(3): 1-11. https://doi.org/10.1080/08827508.2019.1575213 [ Links ]
Takai K., D.P. Moser, M. DeFlaun, et al. 2001. Archaeal diversity in waters from deep South African gold mines. Appl. Environ. Microbiol. 67 (12).5750-5760. https://doi.org/10.1128/AEM.67.21.5750-5760.2001 [ Links ]
Tekere M., A. Lötter, J. Olivier, et al. 2011. Metagenomic analysis of bacterial diversity of Siloam hot water spring, Limpopo, South Africa. African Journal of Biotechnology. 10(78):18005-18012. http://hdl.handle.net/2263/18099 [ Links ]
Wang Z., L. Liu, F. Guo, et al. 2015. Deciphering Cyanide-Degrading Potential of Bacterial Community Associated with the Coking Wastewater Treatment Plant with a Novel Draft Genome. Environmental Microbiology. 70(3):701-709. https://doi.org/10.1007/s00248-015-0611-x [ Links ]
Correspondencia:
*Autor para correspondencia
Yacory A. Sernaque A.: yacoryanahis@gmail.com
San Pedro Mz 8 lote 20. San Pedro-Piura.
Melitza Cornejo La Torre: mcornejo@biotecoop.org
Mz. L1 LT. 2 AAHH La Primavera. Castilla-Piura.
Jerome Pierre Regard: jerome.regard@gmail.com
2 bis avenue Laure Monsarrat 81220 saint Paul cap de joux France.
Eric Louis Mialhe Matonnier: ericmialhe@yahoo.fr
Jirón Filipinas N° 212.Tumbes.
Presentado: 14/10/2018
Aceptado: 02/05/2019
Publicado online: 06/07/2019